肠球菌CRISPR-Cas系统基因结构的分析及其与耐药基因的关系
兰 蕾, 郑 旭, 迟媛媛, 王雪婷, 王梦园, 朱元祺, 闫志勇*
(1.青岛大学 医学部,山东 青岛 266071;2.青岛大学 附属青岛市立医院,山东 青岛 266001;3.青岛大学 附属青岛妇女儿童医院,山东 青岛 266034;4.青岛大学 附属医院,山东 青岛 266071)
肠球菌属(Enterococcus)为一群兼性厌氧的革兰阳性球菌,包括屎肠球菌(Enterococcusfaecium)、粪肠球菌(Enterococcusfaecalis)、盲肠球菌(Enterococcuscecorum)、鸟肠球菌(Enterococcusavium)等多个种,正常栖居于哺乳动物肠道。作为动物和人类肠道中天然菌群的一部分,通常认为肠球菌的致病性低,只感染特殊人群,如肿瘤、血液病、肾病或器官移植等免疫功能低下患者,以往主要为内源性感染[1]。近年来,该菌已成为医院获得性感染的主要原因之一,是各种伤口及手术部位感染的常见病原体,其临床株分离率排在前列,其中以粪肠球菌(E.faecalis)和屎肠球菌(E.faecium)最为突出。肠球菌对许多一线抗菌药物具有天然抗性,如克林霉素、头孢菌素、复方磺胺和低浓度氨基糖苷类等[2]。此外,还具有获得诸如抗生素抗性基因、毒力基因等可移动遗传元件的能力,使其对大环内酯类、四环素类、喹诺酮类、糖肽类和链霉菌素等的耐药日渐普遍。其中,粪肠球菌属于所谓的“ESKAPE”病原体(粪肠球菌、金黄色葡萄球菌(Staphylococcusaureus)、肺炎克雷伯菌(KlebsiellaPneumoniae)、鲍曼不动杆菌(Acinetobacterbaumannii)、铜绿假单胞菌(Pseudomonasaeruginosa)和肠杆菌属(Enterobacter)),引起大部分医院感染,并能逃逸抗菌药物的作用[3]。因此,肠球菌感染的治疗非常困难,给临床带来严峻挑战,尤其是过去二十年中,高死亡率耐万古霉素肠球菌的出现更令人震惊[4]。规律成簇的间隔短回文重复序列(Clustered Regularly Interspaced Short Palindromic Repeats, CRISPR)是日本学者在大肠埃希菌中首次发现的一种细菌“适应性免疫”系统[5]。“适应性”是指针对特定外来入侵者的特异性防御。完整的CRISPR-Cas系统包括三部分(cas基因簇、前导序列和CRISPR阵列)。该系统发挥作用时,先将外源基因片段整合到CRISPR阵列中形成新的间隔序列,当相同外源核酸再次入侵时,系统可以识别并将其切割或沉默其表达,从而抵抗外来遗传物质的侵入、维持自身物种基因组的稳定。古生菌和细菌均面临着不断入侵的外来DNA的威胁,这些入侵者主要是可移动的遗传元件,如噬菌体、质粒、转座子等[6]。CRISPR-Cas系统可以保护原核生物免受外来基因组的侵害,因此,理论上讲可抑制耐药基因的水平转移。本研究对肠球菌的CRISPR系统基因结构及耐药基因进行了分析,以期能深入了解该系统及其与耐药性的关系。
1 材料与方法
1.1 材料
从NCBI数据库(https://www.ncbi.nlm.nih.gov/)下载235株肠球菌全基因组及基因注释等信息,利用CRISPRCasFinder在线软件查找其CRISPR-Cas系统。235株肠球菌中共包括10个种。
1.2 方法
1.2.1 肠球菌CRISPR-Cas系统的筛查 从NCBI数据库下载了235株肠球菌的信息,通过在线软件CRISPRCasFinder(https://crisprcas.i2bc.paris-saclay.fr/CrisprCasFinder/Index)筛查其CRISPR-Cas系统。
1.2.2cas基因的分析与CRISPR-Cas系统的分型 以CRISPRCasFinder等软件筛查肠球菌基因组中的cas基因,并根据cas的种类和组成对系统进行分型、绘制基因结构图。此外,截取cas基因序列用MEGA7.0软件以ClustalW算法进行比对和系统发育分析。
1.2.3 重复序列分析 将肠球菌“确定(confirmed)”CRISPR基因座中的重复序列筛重后,利用MEGA7.0软件中ClustalW算法比对并进行系统发展分析。最小自由能(MFE)和二级结构预测通过RNAfold在线软件完成。
1.2.4 间隔序列分析 肠球菌确定CRISPR基因座中间隔序列的靶向分析通过Nucleotide BLAST完成,以间隔序列与靶向序列的一致率(Identity)和覆盖率(Cover)均大于85%作为同源标准,分类整理拟定同源序列的来源信息。
1.2.5 耐药基因分析 通过CARD在线软件(https://card.mcmaster.ca/)查找肠球菌各菌株基因组中携带的耐药基因。
1.2.6 统计学分析 采用GraphPad Prism 5.0软件中卡方检验法进行数据的统计学处理,P<0.05为有统计学意义,分别对菌株中是否含有确定CRISPR阵列、cas基因簇和完整CRISPR-Cas系统与耐药基因携带率之间的关系进行分析。
2 结果与分析
2.1 肠球菌CRISPR-Cas系统分布情况
纳入研究的肠球菌有10种共235株,包括屎肠球菌155株、粪肠球菌50株,其中含完整CRISPR-Cas系统(同时含有阵列和cas基因簇)的肠球菌35株。共43株含有至少1个确定CRISPR阵列(18.3%)且仅分布于染色体上,其中包含1、2、3、4、5个确定CRISPR阵列的肠球菌分别为31、9、1、1、1株,且含有8个孤儿CRISPR阵列(无cas基因簇)。共检测出确定CRISPR基因座61个和可疑CRISPR基因座345个。cas簇阳性株为45株。如表1所示,除屎肠球菌以外,其他肠球菌cas基因簇和确定CRISPR阵列的携带率整体较高。

表1 235株肠球菌CRISPR-Cas系统分布情况Table 1 Distribution of CRISPR-Cas system in 235 strains ofEnterococcus
2.2 cas基因簇结构分析及cas1基因系统发育构建
纳入研究的肠球菌CRISPR-Cas系统分为4种类型,分别为II-C型、II-A型、I-C型和I-B型(图1)。其中粪肠球菌菌株CVM N60443F基因组中同时含有II-C型和II-A型cas基因簇,其余携带菌株仅含1套cas基因簇。在45株肠球菌中共发现46套cas基因,包括37套II-A型(80.4%),7套II-C型(15.2%),I-C型和I-B型各1套。10株仅存在可疑CRISPR基因座的菌株中也发现了cas基因的存在,且均为II-A型。在屎肠球菌质粒中发现了2个单独存在的cas2基因(屎肠球菌菌株ATCC 700221和FA3)。
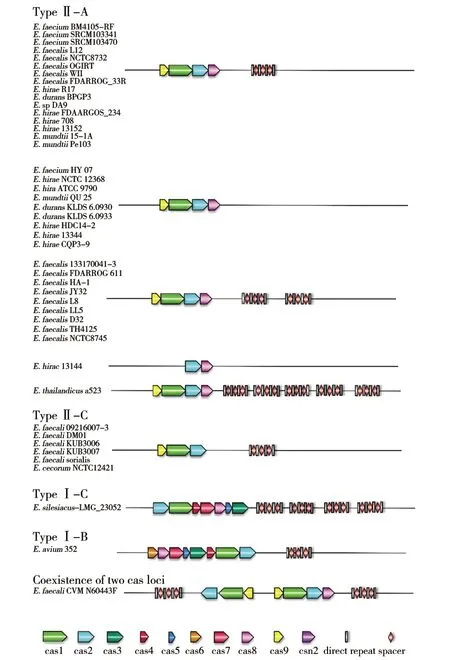
图1 肠球菌CRISPR-Cas系统基因结构图Fig.1 The genetic structure ofEnterococcusCRISPR-Cas system
肠球菌中II-C型、II-A型、I-C型和I-B型CRISPR-Cas系统的基因组成各不相同。cas1、cas2和cas9为II-C型、II-A型CRISPR-Cas系统所共有,cas1和cas2为4型共有,其中cas2基因仅存在于肠球菌II-A型CRISPR-Cas系统中。cas1基因长度较为一致,大多867 bp。对cas1基因进行比对分析发现,除I-C型和I-B型2株外,其余各菌株cas1基因较为保守。对45株存在cas1基因簇的肠球菌cas1基因进行系统进化树构建,结果如图2所示,多数细菌存在于2支上,基本与CRISPR-Cas系统的分型结果一致。
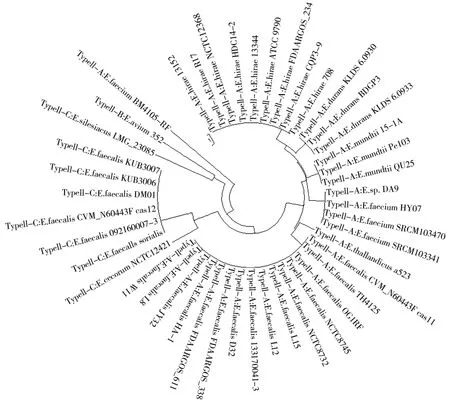
图2 基于肠球菌cas1基因构建的系统发育树Fig.2 The phylogenetic tree based onEnterococcuscas1gene
2.3 肠球菌CRISPR阵列重复序列分析
235株肠球菌中含有确定CRISPR阵列61个,共发现重复序列17种,长度为30~38 bp,重复数量5~50不等,最多的是盲肠球菌菌株NCTC12421,其余均为可疑CRISPR位点(图3),II-A型和II-C型CRISPR-Cas系统中的重复序列在5′端和3′端保守性较好。相同重复序列在不同菌株中CRISPR-Cas系统的型别较为一致。将17种重复序列输入到RNAfold在线软件中进行分析,预测其RNA二级结构及最小自由能,结果有12个能形成茎环结构(图4);其MFE最高-0.2 kcal/mol,最低-11.9 kcal/mol。

图3 确定CRISPR阵列17种重复序列比对结果Fig.3 The alignment results of 17 repeats in confirmed CRISPR array
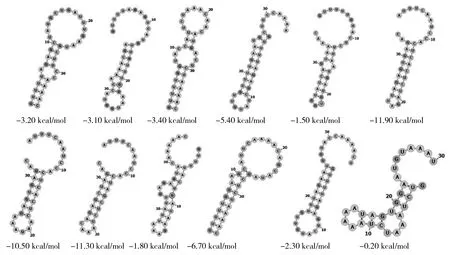
图4 部分重复序列的二级结构模型及最小自由能(MEF)Fig.4 Secondary structure and MEF of partial repeats
2.4 肠球菌CRISPR阵列间隔序列分析
肠球菌CRISPR阵列间隔序列数最高为49,最低为4,平均8个,共609条,剔除其中重复者后余426种,序列长度在29~38 bp之间,且多为30 bp。在GenBank核酸数据库中对426种序列进行Blastn检索,有69种(16.2%)靶向噬菌体,23种(5.4%)靶向质粒,5种(1.2%)靶向转座子,另外有5种(1.2%)靶向细菌自身基因组非CRISPR阵列(表2)。所有间隔序列均靶向非自身细菌基因组。其中部分间隔序列同时靶向多种质粒或噬菌体,如粪肠球菌中的序列AATAGATGGGTAAGAACTGTTTATGTCAAA靶向10种质粒,ATGG-GTGGACCAACGATTCACAGTTCCGCA靶向4种噬菌体。
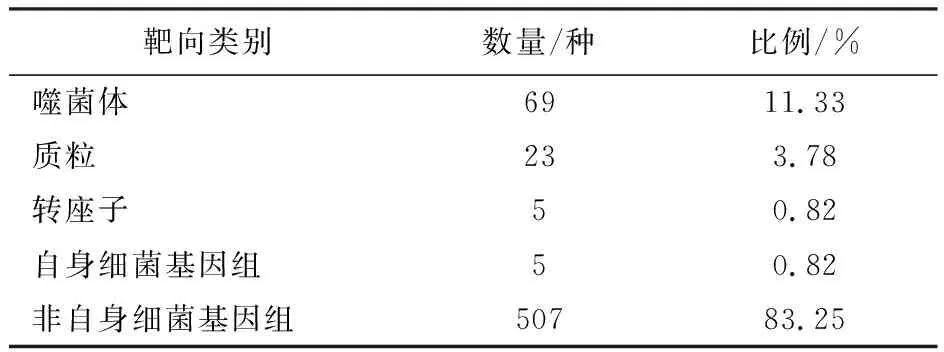
表2 肠球菌间隔序列靶向的同源序列分析Table 2 Homologous sequence analysis of spacer sequence targeting inEnterococcus
2.5 肠球菌CRISPR-Cas系统与耐药基因携带情况的分析
235株肠球菌基因组中筛查出的耐药基因见表3。多药耐药基因(eatAv)、二氨基嘧啶抗生素耐药基因(dfrF)、四环素类抗生素耐药基因[tet(45)]以及肽类抗生素耐药基因(LiaRmutant、LiaSmutant)携带率在不含确定CRISPR阵列组(23.44%、25.52%、16.67%、11.98%、13.02%)明显高于确定CRISPR阵列组(9.3%、0、0、0、0);二氨基嘧啶抗生素耐药基因(dfrF)、四环素类抗生素耐药基因[tet(45)]以及肽类抗生素耐药基因(LiaRmutant、LiaSmutant)携带率在不含cas基因簇组(25.79%、16.84%、12.11%、13.16%)明显高于cas基因簇组(0、0、0、0);二氨基嘧啶抗生素耐药基因(dfrF)以及四环素类抗生素耐药基因[tet(45)]携带率在不含完整CRISPR-Cas系统组(24.50%、16.00%)明显高于完整CRISPR-Cas系统组(0、0),分布差异有统计学意义。其余耐药基因分布差异均无统计学差异。

表3 肠球菌CRISPR-Cas系统与耐药基因的关系Table 3 The relationship between CRISPR-Cas system and drug resistance genes inEnterococcus
3 讨 论
CRISPR介导的完整适应性免疫依次包括适应、表达和干扰三个步骤[7]。在适应阶段中来自入侵元件的部分外源DNA片段(protospacers)被处理并作为新的间隔子整合到CRISPR阵列中。表达阶段涉及CRISPR阵列的转录,然后将前体转录物(pre-crRNA)加工成成熟的CRISPR RNA(crRNA),其与一种或多种cas蛋白组装成CRISPR核糖核蛋白(crRNP)复合物。干扰阶段涉及在crRNP复合物中通过cas核酸酶对入侵的同源病毒或质粒核酸进行crRNA定向切割[8]。
本研究通过对来自NCBI数据库的235株肠球菌全基因组等信息进行分析,共发现确定CRISPR基因座61个和可疑CRISPR基因座303个;其中确定CRISPR基因座中包括8个孤儿CRISPR阵列(orphan CRISPR array)。除屎肠球菌外,其余肠球菌确定CRISPR基因座携带率均较高。确定CRISPR基因座仅存在于染色体上,可疑CRISPR基因座还在质粒中发现。通过分析61个确定CRISPR阵列,共发现重复序列17种,长度为30~38 bp,重复数量在5~50个不等。间隔序列是存储细菌对外来核酸入侵的记忆库,通过对肠球菌间隔序列进行balstn分析,发现不同菌株间隔序列靶向的目的序列同源性较高,可靶向同种质粒、噬菌体或其他细菌,这表明不同菌株在遗传进化过程中可受到相同外来核酸的入侵。
CRISPR-Cas系统主要被分为Ⅰ、Ⅱ、Ⅲ三种常见类型,以及不常见的类型Ⅳ、Ⅴ、Ⅵ,均拥有各自特异性CAS蛋白[9]。所有系统都包含两个共有基因:cas1,一种金属依赖性DNA酶,无序列特异性,与外源DNA(间隔子)整合到CRISPR盒中有关;cas2,一种金属依赖性核糖核酸内切酶,可能参与间隔序列的获取。Ⅰ型系统目前分为Ⅰ-A~Ⅰ-F六个亚型,其特征基因为cas3基因[10]。Ⅱ型系统也称链球菌样或Nmeni亚型“HNH”系统,特异基因为cas9[11]。Ⅲ型中均具有特征基因cas10,分为Ⅲ-A和Ⅲ-B两种亚型[12]。本研究中45株肠球菌含有46套cas基因簇,其中Ⅱ-A型含量最高,其次是Ⅱ-C型。Cas1和Cas2是唯一存在于CRISPR-Cas系统所有亚型的CAS蛋白。对肠球菌cas1基因进行系统发育分析,除Ⅰ-C型、Ⅰ-B型以及屎肠球菌BM4105-RF以外,其余各菌株cas1基因分为两支,与CRISPR-Cas系统的分型结果相符。
CRISPR系统被称为原核生物适应性免疫系统,在原核生物中普遍存在。本研究结果显示,整体上肠球菌完整CRISPR-Cas系统总携带率为10.77%,低于铜绿假单胞菌(40.1%)和克雷伯菌(35.2%),高于葡萄球菌(7.7%)和沙雷菌(10.2%)[13]。但是屎肠球菌CRISPR-Cas系统携带率极低(2.6%),其他肠球菌如粪肠球菌、孟氏肠球菌及海氏肠球菌等携带率分别可达40%、33.3%、40%。粪肠球菌和屎肠球菌在肠球菌中检出率最高, 且与疾病发生最为紧密;二者CRISPR-Cas系统含量却存在较大差异,值得进一步研究。本研究还发现,粪肠球菌完整CRISPR-Cas系统携带率为40%,而屎肠球菌仅为1.9%。Castillo-Rojas等[14]通过对血液、脑脊液等临床样本中的肠球菌进行耐药检测发现,屎肠球菌菌株耐药性最高;Chou等[15]对12名患者进行回顾性研究发现屎肠球菌对氨苄青霉素和替考拉宁的耐药率均高于粪肠球菌;Palmer等[16]研究证实多重耐药的肠球菌缺乏CRISPR-Cas系统,揭示了原核生物中的CRISPR系统对阻碍耐药传播可能具有重要影响。该现象表明这些生物面临的选择性压力导致了权衡取舍,以失去对潜在有害入侵DNA的某些保护作用。
肠球菌是人类和动物肠道的正常栖居菌,但也被世界卫生组织列为全球耐多药病原体,其许多抗生素抗性特征都位于质粒上,并有可能通过水平基因转移而传播[17]。本研究发现四环素类耐药基因(tetM、tet(45))、大环内酯类抗生素耐药基因(ErmB、ErmA、Erm)、二氨基嘧啶抗生素耐药基因(dfrE、dfrF、dfrG)等均可通过可移动遗传元件在肠球菌等细菌间传播。尤为重要的是,本研究发现在肠球菌中ErmB、dfrE、dfrF以及tet(45)等耐药基因携带情况在不含有完整CRISPR-Cas系统组明显高于完整CRISPR-Cas系统组,提示CRISPR-Cas系统在细菌耐药基因的传播方面具有不可忽视的作用。作为医院获得性感染的主要原因,肠球菌已成为临床实践中的重要问题,它们在环境中生存和耐受消毒剂的能力,固有的抗微生物能力以及出色的基因组可塑性,已帮助该微生物成为医院感染的“常客”[18]。水平基因转移在肠球菌临床分离株的进化和发展中起关键作用。
本研究分析了肠球菌属CRISPR-Cas系统基因结构及其与耐药性的关系,发现除屎肠球菌外,肠球菌属CRISPR-Cas系统携带率较高,并且揭示CRISPR-Cas系统可能阻碍耐药基因的传播。尽管本研究纳入分析的菌株数量有限,但对于肠球菌院内感染的防控治疗可以提供参考。