大麦TIFY基因家族成员鉴定及表达分析
刘梦迪 倪守飞 王月雪 母景娇 蔡倩 赵彦宏 王艳芳

摘要:【目的】對大麦TIFY基因家族成员进行鉴定及表达分析,为进一步探究TIFY基因家族在大麦生长发育与胁迫响应中的作用机理打下基础。【方法】基于TIFY家族蛋白的保守域特征,利用HMMER从大麦中鉴定TIFY基因家族成员,利用采用生物信息学软件对其理化性质、保守基序、特征结构域、顺式作用元件、基因结构、系统进化及表达模式进行预测分析。【结果】从大麦中鉴定出15个HvTIFYs基因(HvTIFY1~HvTIFY15),分布于5条染色体上,且大多数基因在染色体上成簇分布。15个HvTIFYs蛋白均具有TIFY家族蛋白的特征结构域(TIFY),根据所含保守结构域的不同,可分为ZML(4个)和JAZ亚族(11个),且亲水性蛋白(14个)和偏碱性蛋白(11个)居多,但均定位于细胞核;二级结构相似度较高,均由α-螺旋、β-转角和无规则卷曲组成,除HvTIFY7蛋白外,其余蛋白二级结构所占比排序:无规则卷曲>α-螺旋>β-转角。HvTIFYs基因结构存在明显差异,其中,JAZ亚族11个基因的内含子数为0~6;ZML亚族4个基因的内含子数为6~7个,系统发育进化树上相邻分支的基因具有较相似的基因结构。HvTIFYs基因启动子区域富含光、激素和胁迫等顺式作用元件,种类及分布均呈多样性。5个物种的79条TIFY蛋白分为4个组,恰好与TIFY家族的4个亚族对应,其中,ZML、TIFY和JAZ亚族包含单、双子叶植物的TIFY蛋白,而PPD亚族仅含有双子叶植物的TIFY蛋白。15个HvTIFYs基因在不同组织器官中的表达量存在明显差异,其中HvTIFY1、HvTIFY2和HvTIFY8基因在8个组织中的表达量均较高,HvTIFY10和HvTIFY15基因表达量中等,HvTIFY6基因表达量较低;HvTIFY11基因不表达。15个基因在根的不同组织中对盐胁迫的敏感程度不同。【结论】从大麦中鉴定出的15个HvTIFYs基因存在一定的功能分化,具有明显的组织和时空特异性,推测其在大麦逆境响应和激素调节中具有重要调控作用。
关键词: 大麦;TIFY基因家族;鉴定;生物信息学;表达分析
中图分类号: S512.303.53 文献标志码: A 文章编号:2095-1191(2022)02-0417-13
Identification and expression analysis of TIFY gene family members in barley
LIU Meng-di NI Shou-fei WANG Yue-xue MU Jing-jiao CAI Qian ZHAO Yan-hong1 WANG Yan-fang
(1Agricultural College, Ludong University/Key Laboratory of Molecular Module Breeding for High Yield and Stress Resistance of Crops in Colleges and Universities of Shandong Province, Yantai, Shandong 264025, China;
2College of Life and Science,Ludong University, Yantai, Shandong 264025, China)
Abstract:【Objective】The identification and expression analysis of barley TIFY gene family members could lay a foundation for the further exploration and understanding of the mechanism of TIFY gene family in barley growth and development and response to stress. 【Method】TIFY gene family members were identified from barley by HMMER based on their conserved domain characteristics. Their physicochemical properties, conserved motifs,characteristic domains, cisac-ting elements,gene structure,phylogeny and expression patterns were predicted using bioinformatics software. 【Result】In this study,15 TIFY genes (HvTIFY1-HvTIFY15) were identified in barley,which were distributed on 5 chromosomes, some of them in clusters. The 15 HvTIFY proteins had the characteristic domain (TIFY) of the TIFY family. According to their different contents of conserved domains, they could be divided into ZML(4) and JAZ(11) subgroups. Most of them were hydrophilic (14) and lightly basic (11) proteins,but all of them were located in the nucleus. With the exception of HvTIFY7, the similarity of secondary structure was high, which was proportionally composed of random coil>α-helix>β-angle. There were, however, obvious differences in the structure of HvTIFYs genes. The number of introns ranged from 0 to 6 for the 11 genes in the Jaz subfamily and from 6 to 7 for the 4 genes in the ZML subfamily. The promoter regions of HvTIFY genes were rich in cis-acting elements such as light, hormone and stress, and its species and distribution were diverse. The 79 TIFY proteins of 5 species were divided into 4 subfamilies. The ZML,TIFY and JAZ subfamilies contain TIFY proteins of monocotyledons and dicotyledons, while the PPD subfamily only contains TIFY proteins of dicotyledons. There were significant differences in the expression of the 15 HvTIFY genes in different tissues and organs,in which HvTIFY1, HvTIFY2 and HvTIFY8 genes were highly expressed in 8 tissues,HvTIFY10 and HvTIFY15 genes were modera-tely expressed, HvTIFY6 expression was low and HvTIFY11 expression was not detected. Different HvTIFY genes displayed different sensitivities to salt stress in different root tissues. 【Conclusion】There is some functional differentiation in the 15 HvTIFY genes identified from barley with obvious tissue and spatiotemporal specificity, which are speculated to have important regulatory roles in hormone regulation and the barley response to adverse conditions.
Key words: barley; TIFY gene family; identification; bioinformatics; expression analysis
Foundation items: National Key Research and Development Program of China(2019YFD1005002-0304)
0 引言
【研究意义】大麦(Hordeum vulgare)是全球普遍栽培的第四大禾谷类作物,在食用、饲料、酿造业等多个方面均具有重要用途(龚谨,2020;郭铭,2021),但其在生长过程常因遭遇诸多逆境胁迫而导致减产,如何提高大麦的抗逆能力已成为研究热点。TIFY基因家族是一类植物特有的转录因子,因其编码的蛋白均具有高度保守的TIFY结构域而得名(Vanholme et al.,2007;Zhu et al.,2013)。该基因家族不仅与植物生长发育密切相关,而且在非生物胁迫(低温、干旱和高盐)及激素调节(茉莉酸信号转导)等多种生物学反应中发挥重要作用(Zhang et al.,2015a;刘俊等,2021)。对大麦TIFY基因家族进行系统鉴定及表达分析,并解析TIFY基因家族在大麦应对逆境胁迫中的作用机制,对培育高产抗逆的大麦品种具有重要意义。【前人研究进展】自Nishiiet等(2000)首次从拟南芥中发现了TIFY基因家族成员AtTIFY1(也称为AtZIM)以来,陆续从拟南芥(Vanholme et al.,2007)、水稻(Ye et al.,2009)、葡萄(Zhang et al.,2012)、野生大豆(Zhu et al.,2013)、二穗短柄草(Zhang et al.,2015)、玉米(Zhang et al.,2015b)、棉花(Zhao et al.,2016)、小麥(Xie et al.,2019)、大麻(温东等,2020)、柑桔(张沪等,2020)和杜仲(刘俊等,2021)中分别鉴定出18、20、19、34、21、30、30、63、3、14和14个TIFY基因。TIFY蛋白除了包含TIFY结构域外,通常还含有GATA、CCT、PPD和Jas结构域。根据蛋白包含保守结构域的不同,可将其分为4个亚族:TIFY、PPD、JAZ和ZML。其中,只含有TIFY结构域的蛋白归属于TIFY亚族;同时包含TIFY和Jas结构域的蛋白归属于JAZ亚族;同时含有TIFY、GATA和CCT结构域的蛋白归属于ZML亚族;除包含TIFY结构域外,N端还含有1个PPD结构域的蛋白归属于PPD亚族(White,2006;Chini et al.,2007;Yan et al.,2007;Cai et al.,2014;Tian et al.,2019)。前人研究发现,TIFY基因家族成员与植物的生长发育密切相关,而且在逆境胁迫中发挥重要作用。例如,拟南芥AtTIFY1基因对叶柄和下胚轴的伸长具有重要调控作用(Shikata et al.,2004),AtPPD1和AtPPD2基因对叶片生长具有重要调控作用(White,2006);香蕉MaTIFY1基因被丙烯诱导表达后可加快果实成熟(罗冬兰等,2017);水稻OsJAZ1基因通过与OsMYC2和OsCOI1b相互作用调控花的发育和根的伸长,其核心序列被替换或删除均会影响花和根发育过程中茉莉酸信号的特异性和敏感性(Chini et al.,2007;Yan et al.,2007;Cai et al.,2014;Tian et al.,2019);葡萄的19个VvTIFY基因对生物胁迫(如白粉病菌和葡萄卷叶病毒GLRaV-3)并不敏感,但环境(如低温、干旱和高盐等)胁迫和激素(如茉莉酸和脱落酸等)胁迫能诱导部分VvTIFY基因的差异表达(Zhang et al.,2012);大豆TIFY10基因在响应碱胁迫中发挥重要调控作用(Zhu et al.,2014);二穗短柄草中的部分TIFY基因参与茉莉酸信号通路,响应至少一种非生物胁迫(如干旱、高盐、低温、高温等)(Zhang et al.,2015a);核桃PnJAZ1基因表达可提高种子萌发率和早期发育中的盐胁迫耐受性(Liu et al.,2019)。【本研究切入点】目前,TIFY基因家族在很多物种中已被鉴定出来,但鲜见有关大麦TIFY基因家族成员鉴定及表达分析的研究报道。【拟解决的关键问题】利用生物信息学方法鉴定出大麦TIFY基因,分析其理化性质、保守基序、蛋白结构、顺式作用元件、基因结构、系统进化及基因表达模式,为解析该基因家族在大麦生长发育和逆境胁迫中的功能提供理论参考。
1 材料与方法
1. 1 TIFY基因家族的鉴定及定位
从EnsemblPlant数据库(http://plants.ensembl.org/index.html)和UniProt蛋白数据库(https://www.uniprot.org/)中下载大麦所有的蛋白序列,从Pfam数据库(http://pfam.xfam.org/)提取TIFY家族蛋白特征种子文件(PF06200)。基于种子文件PF06200,利用HMMER从已下载的大麦蛋白质序列中筛选出候选TIFY蛋白序列,然后利用SMART(http://smart.embl-heidelberg.de/)对候选大麦TIFY蛋白进一步鉴定及验证。
从Phytozome数据库(https://phytozome.jgi.doe.gov/pz/portal.html)获取大麦TIFY基因在染色体上的位置信息,然后用MapChart绘制大麦TIFY基因的染色体位置图。使用Cell-PLoc2.0(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)对大麦TIFY蛋白进行亚细胞定位预测。
1. 2 蛋白理化性质及结构预测
利用Protparam(https://web.expasy.org/protpa-ram/)预测大麦TIFY蛋白的基本理化性质。使用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)预测大麦TIFY蛋白的二级结构。利用SWISS-MODEL(https://swissmodel.expasy.org/interactive)预测大麦TIFY蛋白的三级结构。
1. 3 蛋白保守结构域和基因结构分析
使用MEME(http://memesuite.org/tools/meme)预测大麦TIFY蛋白的保守基序(motif),motif最大数值设定为8。利用GSDS(http://gsds.cbi.pkuedu.cn/)分析其基因结构。
1. 4 顺式作用元件分析
从Phytozome数据库中提取大麦TIFY基因的起始密码子上游2000 bp序列,利用PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)预测基因启动区域的顺式作用元件,最后用TBtools绘制顺式作用元件图谱(Chen et al.,2020)。
1. 5 系统发育及共线性分析
利用MEGA-X中的ClustalW模块对大麦TIFY蛋白进行多序列比对(Tamura et al.,2011),并采用邻接法(Neighbor-joining,NJ)构建系统发育进化树,其中Bootstrap设置为1000,其他参数采用默认值。采用同样方法,基于来自拟南芥、野生大豆、水稻、二穗短柄草和大麦的79条TIFY蛋白进行系统发育进化分析,利用Evolview v3(https://www.evolgenius.info/evolview/#login)绘制系统发育进化树。利用TBtools分别分析大麦与拟南芥、大麦与水稻之间TIFY基因的共线性。
1. 6 TIFY家族表达分析
基于Expression Atlas数据库(https://www.ebi.ac.uk/gxa/experiments)的大麦RNA-Seq表达数据(E-MTAB-2809和E-MTAB-4634)进行TIFY基因表达模式分析,其中E-MTAB-2809包含了TIFY基因在大麦的根、花序(1 cm)、花序(5 mm)、节间、颖果(15 d)、颖果(5 d)、幼苗和胚芽等8个组织中的RNA-Seq数据,从中获得了大麦TIFYs基因的FPKM值,然后使用Log2归一化FPKM值;E-MTAB-4634包含了TIFY基因在NaCl胁迫后在大麦根尖、根伸长区和根分生区基因表达的RNA-Seq数据,从中获得TIFY基因的Log2(Fold-change)值表示其表达量的倍数变化。最后,用TBtools绘制大麦TIFY基因的表达热图。
2 结果与分析
2. 1 大麦TIFY基因家族的鉴定及分析
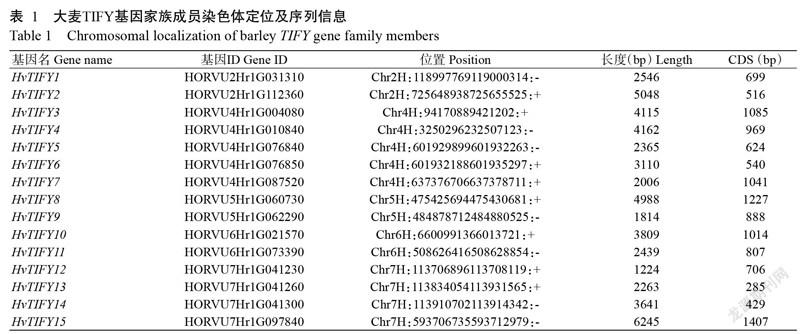
利用HMMER从大麦中鉴定出15个TIFY基因家族成员,根据其在染色体上的位置命名为HvTIFY1~ HvTIFY15,长度为1224~6245 bp,编码区(CDS)长度为285~1407 bp(表1)。这15个HvTIFYs基因不均匀地分布在5条大麦染色体上(图1),其中2H、5H和6H染色体各含有2个HvTIFYs基因,4H和7H染色体上分别含有5和4个HvTIFYs基因。大多HvTIFYs基因在染色体上成簇分布,每簇内包含2~3个HvTIFYs基因,如5H染色体上的HvTIFY8和HvTIFY9基因簇,7H染色体上的HvTIFY12、HvTIFY13和HvTIFY14基因簇。
上述HvTIFYs基因编码的15个HvTIFYs蛋白均具有典型的的特征结构域(TIFY)。15个HvTIFYs蛋白由94~468个氨基酸残基组成,相对分子量为10.0~50.3 kD;理论等电点(pI)为4.7~10.4,其中,大于7.0的蛋白有11个(表2),说明偏碱性蛋白居多;除HvTIFY7蛋白外,其余14个大麦TIFYs蛋白均为亲水性蛋白。蛋白二级结构预测结果(表2)显示,大麦TIFY蛋白的二级结构相似度较高,均含α-螺旋、β-转角和无规则卷曲,均不含β-折叠,除HvTIFY7蛋白外,其余蛋白的二级结构所占比例排序为:无规则卷曲>α-螺旋>β-转角。三级结构的预测结果(图2)表明,无规则卷曲和α-螺旋在维持三级结构稳定中发挥主要作用,β-转角主要起修饰作用。HvTIFY1、HvTIFY9和HvTIFY13蛋白中均具有1个典型的锌指结构,即1个α-螺旋与2条反向的β-转角平行。亚细胞定位预测结果显示,15个大麦TIFYs蛋白均被定位于细胞核中(表2),证实了TIFY蛋白是一类转录因子。
2. 2 大麦TIFY家族基因结构分析结果
由图3可知,相邻分支的HvTIFYs基因具有相似的基因结构。基因结构分析结果显示,JAZ亚族的11个HvTIFYs基因的内含子数为0~6,其中HvTIFY6、HvTIFY13和HvTIFY14基因不含任何内含子;ZML亚族的4个基因(HvTIFY3、HvTIFY4、HvTIFY10和HvTIFY15)的内含子数为6~7个(图3-B),推测这2個亚族HvTIFYs基因在进化过程中分别发生了内含子的丢失和插入;内含子数量越多,表示基因可形成剪接体的潜在能力越高,相对于JAZ亚族的HvTIFYs基因来说,ZML亚族的HvTIFYs基因可形成多种剪接体的潜在能力更高。另外,本研究还发现有7个HvTIFYs基因的TIFY保守域对应编码序列存在同一个外显子内,而另外8个HvTIFYs基因的TIFY保守域对应编码序列则跨越了由1个内显子隔开的两个外显子(图3-B)。HvTIFYs基因不仅内含子数量不同,而且内含子长度也有明显差异,导致这些基因的长度(1224~6245 bp)存在明显差异(图3-B和表1),比如HvTIFY15和HvTIFY2基因是2个最长的基因,其内含子的长度至少占基因总长度的70%以上。
2. 3 大麦TIFY蛋白结构域分析结果
保守基序分析结果(图4)显示,15个HvTIFYs蛋白中共鉴定出8个motif,且所有大麦TIFYs蛋白均含有motif 1和motif 2,其中motif 2含有TIFY结构域的核心基序(图5-A),motif 1则是缺少了末端保守的脯氨酸(P)和酪氨酸(Y)的Jas结构域(图5-B)。HvTIFY3、HvTIFY4、HvTIFY10和HvTIFY15還包含1个CCT结构域(motif 4)(图4和图5-C)和1个GATA锌指结构域(motif 3)(图4和图5-D)。根据所含保守结构域的不同,15个大麦TIFYs蛋白被分为ZML和JAZ亚族(表1),其中11个属于JAZ亚族,4个属于ZML亚族。
2. 4 大麦TIFY家族基因启动子顺式作用元件分析结果
启动子区域是起始密码子上游的一段DNA序列,包含多种顺式作用元件,可调控植物对外源激素和环境胁迫的响应(Wang et al.,2020)。为了探索HvTIFYs基因的转录调控模式及潜在功能,本研究利用PlantCARE对起始密码子(ATG)上游2000 bp序列进行顺式作用元件分析,结果显示,15个HvTIFYs基因启动子区域的顺式作用元件种类及分布呈多样性,至少含有2种以上顺式作用元件(图6),暗示HvTIFYs基因可能参与多种信号通路的调控,从而对环境胁迫作出应答。将鉴定出的顺式作用元件分为六大类:蛋白结合位点、调控元件、光响应元件、激素响应元件、生长发育相关元件和胁迫响应元件(表3和图6),其中,激素响应元件和胁迫响应元件数量最多,均为114个,故推测HvTIFYs基因在大麦的激素调控和逆境胁迫响应中发挥重要作用。此外,15个HvTIFYs基因启动子区域至少含有1个光响应元件,表明HvTIFYs基因的转录可能受光周期调控。
2. 5 进化分析和共线性分析结果
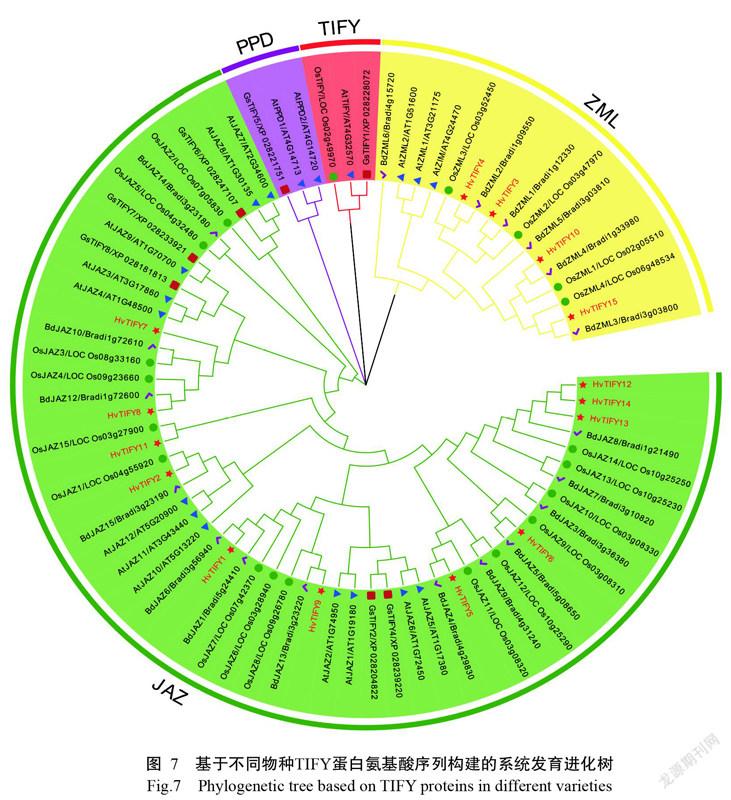
为深入了解不同TIFY蛋白间的系统发育关系,将拟南芥、野生大豆、水稻、二穗短柄草和大麦的TIFY蛋白序列(共79条)进行多序列比对,并构建了系统发育进化树,结果如图7所示。将这5个物种的79条TIFY蛋白可分为4个组,恰好与TIFY家族的4个亚族对应,其中,ZML、TIFY和JAZ亚族包含单、双子叶植物的TIFY蛋白,而PPD亚族仅含有双子叶植物的TIFY蛋白,不仅暗示TIFY基因在单、双子叶植物分化前已存在,还暗示该家族的ZML、TIFY和JAZ亚族可能在单、双子叶分化前就已存在。JAZ亚族成员数目最多,共56个TIFY蛋白可进一步分为3种类型:JAZ-Ⅰ(同时含单、双子叶植物的TIFY蛋白)、JAZ-Ⅱ(只含单子叶植物的TIFY蛋白)和JAZ-Ⅲ(仅含双子叶植物的TIFY蛋白),故推测JAZ-Ⅰ在单、双子叶分化之前就已存在;JAZ-Ⅱ和JAZ-Ⅲ则出现在单、双子叶分化之后分别在单、双子叶植物内单独进化。大麦和二穗短柄草的TIFY蛋白通常分布在相邻分支上,表明这两个物种的亲缘关系相对较近。15个HvTIFYs蛋白仅归属于ZML和JAZ两个亚族,与上述TIFY、Jas、CCT和GATA保守域的氨基酸序列比对结果一致,进一步验证本研究对大麦TIFY家族分类的可靠性。
共线性分析结果显示,在大麦和拟南芥基因组间虽然存在共线区块,但不存在共线性TIFY基因对(图8-A);在大麦和水稻基因组间存在4对共线性TIFY基因对(HvTIFY10与OsZML1、HvTIFY10与OsZML4、HvTIFY9与OsJAZ8、HvTIFY6与OsJAZ9)(图8-B),其中HvTIFY10与水稻中的2个基因(OsZML1和OsZML4)均存在共线性,表明大麦与水稻的亲缘关系比与拟南芥更近,与系统发育进化树分析结果一致。此外,大麦与水稻之间的这4对共线性TIFY基因在经历了物种分化等漫长的进化过程后仍维持了高度的保守,且具有相同或高度相似的功能,可根据水稻TIFY基因的功能来推测HvTIFYs基因的功能。
2. 6 大麦TIFY基因家族成员表达分析结果
基于Expression Atlas数据库中的大麦RNA-Seq表达数据(E-MTAB-2809),本研究对大麦HvTIFYs基因在8个不同组织或发育阶段的表达模式进行分析,结果图9所示。15个HvTIFYs基因在不同组织器官中的表达量存在明显差异,具有组织特异性。其中,HvTIFY1、HvTIFY2和HvTIFY8基因在8个组织中的表达量均较高,暗示这3个基因在8个组织的生长发育中起着重要作用;HvTIFY10和HvTIFY15基因在8个组织中的表达量中等;HvTIFY6基因在8个组织中的表达量均较低;HvTIFY11基因则在8个组织中均不表达。值得注意的是,3个高表达的基因(HvTIFY1、HvTIFY2和HvTIFY8)均属于JAZ亚族,而2个表达量中等的基因(HvTIFY10和HvTIFY15)则均属于ZML亚族。另外,还发现HvTIFY1、HvTIFY8和HvTIFY12基因在花序中的表达量随着花序的发育而降低,而HvTIFY10和HvTIFY15正好相反。HvTIFY9基因在颖果中的表达量随着颖果的发育而升高,而HvTIFY5、HvTIFY8和HvTIFY13基因则正好相反。可见,HvTIFYs基因在表达上具有时空特异性,暗示HvTIFYs基因在长期进化过程中发生了功能分化。
为了探究大麦在盐胁迫诱导下HvTIFYs基因的表达差异,利用Expression Atlas数据库下载的RNA-Seq表达数据(E-MTAB-4634)进行基因差异表达分析,结果如图10所示。15个基因在根的不同组织中对盐胁迫的敏感程度不同。例如,经盐胁迫诱导后,HvTIFY9基因的表达量在根尖上调,而在根分生区和根伸长区下调,尤其是在根的分生区的表达量减少50%;HvTIFY5基因虽然在根的3个组织中表达量均上调,但上调程度不同,尤其是在根尖中的表达量上调至1.62倍;HvTIFY6、HvTIFY12、HvTIFY13和HvTIFY14基因在根伸长区表达量上调,而在其他2个组织则无变化,特别是HvTIFY12基因在根伸长区中的表达量上调至1.52倍。HvTIFY11基因在根3个组织中不表达,经盐胁迫后仍无任何变化。
3 讨论
TIFY家族作为一类植物特有的转录因子,在植物生长发育及胁迫防御反应中发挥着重要作用。据Bai等(2011)研究发现,大多数二倍体植物中的TIFY基因家族成员通常为10~34个。本研究从大麦中鉴定出15个TIFY基因家族成员,在上述范围内。Zhao等(2016)从亚洲棉(二倍体)、雷蒙德氏棉(二倍体)和陆地棉(四倍体)中分别鉴定出21、28和50个TIFY基因家族成员。Xie等(2019)从乌拉尔图小麦(二倍体,AA)、粗山羊草(二倍体,DD)和普通小麦(六倍体,AABBDD)中分别鉴定出13、17和63个TIFY基因。赵晓晓等(2019)从四倍体柳枝稷中鉴定出48个TIFY成员。由此可见,植物中TIFY基因家族成员数可能与基因组倍数有关。
在植物进化过程中,基因复制是基因家族扩张的主要途径之一,有助于增加植物对环境的适应能力,同时基因冗余也使基因功能的分化成为可能(Moore and Purugganan,2005)。基因复制分为两种模式:全基因组重复和单基因重复(基因的串联重复与片段重复)。本研究发现,HvTIFYs基因通常在染色体上成簇分布,形成了多个基因簇,这与大麦SBP基因家族在染色体上成簇分布的特征非常相似(蔡倩等,2019)。其中,位于7H染色体上的1个基因簇包括HvTIFY12、HvTIFY13和HvTIFY14基因,三者彼此相邻,紧密排列,而且序列高度相似,进化关系非常近,属于串联重复基因,其编码蛋白理化性质、保守域、基因结构及表达模式等方面虽高度相似但稍有不同,暗示这些串联重复基因在进化中发生了亚功能化,共同行使祖先基因的功能。另外,HvTIFY12、HvTIFY13和HvTIFY14位于系统发育树的一个独立分支中,该分支内无其他物种的TIFY基因,故推测三者是在大麦中单独进化而来。相比之下,其他成簇分布的基因(如HvTIFY8和HvTIFY9)虽然在染色体上彼此相邻,但在系统发育进化树上分别处于不同的分支,而且各分支上均含其他物种(拟南芥、野生大豆、水稻和二穗短柄草)的基因,暗示HvTIFY8和HvTIFY9基因在单、双子叶植物分化之前已存在了。
TIFY基因家族分为TIFY、PPD、JAZ和ZML 4个亚族(White,2006;Chini et al.,2007;Yan et al.,2007;Cai et al.,2014;Tian et al.,2019)。本研究系统发育进化树分析结果显示,这4个亚族所包含的TIFY基因数目差别很大,其中,JAZ(56個)和ZML(17个)亚族成员数目最多,PPD(3个)和TIFY(3个)亚族成员较少,且PPD亚族分支中只含双子叶植物(拟南芥和野生大豆)的3个TIFY基因。前人研究认为,PPD亚族可能在维管束植物分化后出现,随后在单子叶植物谱系中消失,因此单子叶植物中不包含PPD亚族的TIFY基因(白有煌,2014),而TIFY亚族成员在进化过程中或许因基因复制等原因,获得了其他结构域(如GATA、CCT、PPD和Jas),进而演变成了JAZ或ZML亚族成员从而导致了TIFY亚族成员减少,而JAZ和ZML亚族成员增多。
TIFY保守域是TIFY家族的核心特征结构域,该保守域的核心基序通常由6个氨基酸组成。目前,该保守基序主要有9种类型:TIFYXG、TII[F/Y]XG、TIS[F/Y]XG、TLF[F/Y]XG、TLL[F/Y]XG、TLS[F/Y]XG、TLV[F/Y]XG、TMF[F/Y]XG和VIF[F/Y]XG,其中TIFYXG为最主要的核心基序,其他几种类型均是该基序内个别氨基酸发生变化产生的(Bai et al.,2011)。本研究发现,15个HvTIFYs蛋白仅含有其中的4种核心基序:TIFYXG、TLLFXG、TLSFXG和TMFYXG,且67% HvTIFYs蛋白的TIFY保守域核心基序是TIFYXG,27%的核心基序为TL[L/S]FQG。值得注意的是,所有HvTIFYs蛋白的TIFY保守域核心基序中首尾2个氨基酸(T和G)完全保守,说明大麦TIFY家族蛋白核心基序的保守性相对更高。
本研究发现,HvTIFYs基因的表达存在组织特异性,而且对盐胁迫的敏感程度也不相同,说明HvTIFYs基因存在一定的功能分化。15个大麦HvTIFYs基因上游启动区鉴定出的顺式作用元件种类和个数均不相同,推测其是导致HvTIFYs基因之间存在功能差异的主要原因(Yamaguchi-Shinozaki and Shinozaki,2005)。此外,15个大麦HvTIFYs基因均含有3~11个胁迫响应元件,主要包括低温胁迫、干旱胁迫、厌氧胁迫和缺氧胁迫等4种胁迫响应类型,推测HvTIFYs基因参与这些逆境胁迫的响应。HvTIFYs基因启动子区中的干旱胁迫响应元件数量最多,占胁迫响应元件的50%以上,暗示HvTIFYs基因在抵御干旱胁迫中发挥重要调控作用。目前有关TIFY基因在干旱胁迫中的调控作用已在棉花(Zhao et al.,2016)、柳枝稷(赵晓晓,2019)、西瓜(Yang et al.,2019)和小麦(Xie et al.,2019)等多种植物中得到证实。此外,HvTIFYs基因启动子区均含有茉莉酸相关元件,占激素相关元件的41%,与Chung和Howe(2009)报道的TIFY基因在茉莉酸信号传递中发挥重要调控作用相吻合。茉莉酸信号是生物胁迫与非生物胁迫反应的“中央控制器”,可激活与植物生长、发育和防御相关基因的表达,尤其在植物响应生物与非生物胁迫中发挥重要作用,在植物的防御过程中起调控全局的作用(Ismail et al.,2012)。由此可见,大麦TIFY基因家族成员可能在大麦逆境胁迫防御反应及激素信号调控等方面发挥重要作用。
4 结论
从大麦中鉴定出的15个HvTIFYs基因存在一定的功能分化,具有明显的组织和时空特异性,推测其在大麦逆境响应和激素调节中发挥重要调控作用。
参考文献
白有煌. 2014. 植物中编码基因TIFY家族和非编码基因IncRNA的生物信息学研究[D]. 杭州:浙江大学. [Bai Y H. 2014. Bioinformatics analysis of TIFY gene family and long noncoding RNA in plants[D]. Hangzhou:Zhejiang University.]
蔡倩,赵甜甜,刘梦迪,王艳芳,赵彦宏,马臣,董红霞. 2019. 大麦SBP转录因子的鉴定与表达分析[J]. 麦类作物学报,40(1):65-74. [Cai Q,Zhao T T,Liu M D,Wang Y F,Zhao Y H,Ma C,Dong H X. 2019. Bioinformatics identification of SBP genes and expression profiles in barley[J]. Journal of Triticeae Crops,40(1):65-74.] doi:10.7606/j. issn.1009-1041.2020.01.08.
龚谨. 2020. 我国大麦进口增长的原因、冲击及贸易政策研究[D]. 北京:中国农业科学院. [Gong J. 2020. Research on the causes,impacts and trade policies of barley import growth in China[D]. Beijing:Chinese Academy of Agricultural Sciences.]
郭铭. 2021. 大麦条纹病菌基因Pgr07060的功能研究[D]. 兰州:甘肃农业大学. [Guo M. 2021. Functional study of the Pgr07060 gene in Pyrenophora graminea[D]. Lanzhou:Gansu Agricultural University.]
刘俊,陈玉龙,刘燕,司富春. 2021. 杜仲TIFY转录因子鉴定与表达分析[J]. 中国实验方剂学杂志,27(19):165-174. [Liu J,Chen Y L,Liu Y,Si F C. 2021. Identification and expression analysis of TIFY transcription factor in Eucommia ulmoides[J]. Chinese Journal of Experimental Traditional Medical Formulae,27(19):165-174.] doi:10. 13422/j.cnki.syfjx.20211211.
罗冬兰,巴良杰,陈建业,陆旺金,单伟,邝健飞. 2017. 香蕉MaTIFY1转录因子特性及其在成熟过程中基因表达分析[J]. 园艺学报,44(1):43-52. [Luo D L,Ba L J,Chen J Y,Lu W J,Shan W,Kuang J F. 2017. Characterization and expression analysis of banana MaTIFY1 transcription factor during fruit ripening[J]. Acta Horticulturae Sinica,44(1):43-52.] doi:10.16420/j.issn.0513-353x.2016-0454.
溫东,王梦月,米要磊,马伟,孙伟,师玉华. 2020. 中药火麻仁基原植物大麻的TIFY基因家族鉴定及功能分析[J]. 中国实验方剂学杂志,26(24):134-143. [Wen D,Wang M Y,Mi Y L,Ma W,Sun W,Shi Y H. 2020. Genome-wide identification and characterization of TIFY gene family in medicinal plant Cannabis sativa[J]. Chinese Journal of Experimental Traditional Medical Formulae,26(24):134-143.] doi:10.13422/j.cnki.syfjx.2020.23.11.
张沪,肖翠,王贵元,廖晶晶,仝铸,何秀娟,邱文明,孙中海. 2020. 柑桔TIFY基因结构特征及响应低温表达分析[J]. 中国南方果树,49(2):34-39. [Zhang H,Xiao C,Wang G Y,Liao J J,Tong Z,He X J,Qiu W M,Sun Z H. 2020. Structural characteristics and expression analysis of TIFY gene in citrus at low temperature[J]. Fruit trees in South China,49(2):34-39.] doi:10.13938/j.issn.1007-1431.2019 0723.
赵晓晓,谢坤良,张舒梦,张超,奚亚军,孙风丽. 2019. 柳枝稷TIFY基因家族的鉴定与分析[J].草地学报,27(5):1126-1137. [Zhao X X,Xie K L,Zhang S M,Zhang C,Xi Y J,Sun F L. 2019. Identification and analysis of TIFY gene family in switchgrass[J]. Acta Agrestia Sinica,27(5):1126-1137.] doi:10.11733/j.issn.1007-0435.2019.05. 003.
趙晓晓. 2019. 柳枝稷幼穗分化分期与TIFY基因家族鉴定[D]. 杨陵:西北农林科技大学. [Zhao X X. 2019. Dvide differentiation process of switchgrass spike into stages and identify TIFY gene family in switchgrass[D]. Yangling:Northwest A & F University.]
Bai Y H,Meng Y J,Huang D L,Qi Y H,Chen M. 2011. Origin and evolutionary analysis of the plant-specific TIFY transcription factor family[J]. Genomics,98(2):128-136. doi:10.1016/j.ygeno.2011.05.002.
Cai Q,Yuan Z,Chen M J,Yin C S,Luo Z J,Zhao X X,Liang W Q,Hu J P,Zhang D B. 2014. Jasmonic acid regu-lates spikelet development in rice[J]. Nature Communications,5(1):1-13. doi:10.1038/ncomms4476.
Chen C J,Chen H,Zhang Y,Thomas H R,Xia R. 2020. TBtools:An integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant,13(8):1194-1202. doi:10.1016/j.molp.2020.06.009.
Chini A,Fonseca S,Fernandez G,Adie B,Chico J M,Lorenzo O,Casado G G,Vidriero L I,Lozano F M,Ponce M R. 2007. The JAZ family of repressors is the missing link in jasmonate signalling[J]. Nature,448(7154):666-671. doi:10.1038/nature06006.
Chung H S,Howe G A. 2009. A critical role for the TIFY motif in repression of jasmonate signaling by a stabilized splice variant of the JASMONATE ZIM-domain protein JAZ10 in Arabidopsis[J]. The Plant Cell,21(1):131-145. doi:10.1105/tpc.108.064097.
Ismail A,Riemann M,Nick P. 2012. The jasmonate pathway mediates salt tolerance in grapevines[J]. Journal of Experimental Botany,63(5):2127-2139. doi:10.1093/jxb/err426.
Liu S H,Zhang P Y,Li C C,Xia G M. 2019. The moss jasmonate ZIM-domain protein PnJAZ1 confers salinity to-lerance via crosstalk with the abscisic acid signalling pathway[J]. Plant Science,280:1-11. doi:10.1016/j.plantsci. 2018.11.004.
Moore R C,Purugganan M D. 2005. The evolutionary dyna-mics of plant duplicate genes[J]. Current Opinion in Plant Biology,8(2):122-128. doi:10.1016/j.pbi.2004.12.001.
Nishii A,Takemura M,Fujita H,Shikata M,Kohchi T. 2000. Characterization of a novel gene encoding a putative single zinc-finger protein,ZIM,expressed during the reproductive phase in Arabidopsis thaliana[J]. Bioscience,Biotechnology,and Biochemistry,64(7):1402-1409. doi:10. 1271/bbb.64.1402.
Shikata M,Matsuda Y,Ando K,Nishii A,Takemura M,Yokoto A,Kohchi T. 2004. Characterization of Arabidopsis ZIM,a member of a novel plant-specific GATA factor gene family[J]. Journal of Experimental Botany,55(397):631-639. doi:10.1111/j.1399-3089.2008.00478. x.
Tamura K,Peterson D,Peterson N,Stecher G,Nei M,Kumar S. 2011. MEGA5:Molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J]. Molecular Biology and Evolution,28:2731-2739. doi:10.1093/molbev/msr121.
Tian J Q,Cao L H,Chen X F,Chen N J,Zhang P,Cao L M,Persson S,Zhang D B,Yuan Z. 2019. The OsJAZ1 degron modulates jasmonate signaling sensitivity during rice development[J]. Development,146(4):dev173419. doi:10.1242/dev.173419.
Vanholme B,Grunewald W,Bateman A,Kohchi T. 2007. The tify family previously known as ZIM[J]. Trends in Plant Science,12(6):239-244. doi:10.1016/j.tplants.2007.04. 004.
Wang H Z,Leng X,Xu X M,Li C H. 2020. Comprehensive analysis of the TIFY gene family and its expression profiles under phytohormone treatment and abiotic stresses in roots of Populus trichocarpa[J]. Forests,11(3):315-336. doi:10.3390/f11030315.
White D W R. 2006. PEAPOD regulates lamina size and curvature in Arabidopsis[J]. Proceedings of the National Aca-demy of Sciences of the United States of America,103(35):13238-13243. doi:10.1073/pans.0604349103.
Xie S F,Cui L C,Lei X L,Yang G. 2019. The TIFY gene family in wheat and its progenitors:Genome-wide identification,evolution and expression analysis[J]. Current Genomics,20(5):371-388. doi:info:doi/10.2174/13892029 20666191018114557.
Yamaguchi-Shinozaki K,Shinozaki K. 2005. Organization of cis-acting regulatory elements in osmotic- and cold-stress-responsive promoters[J]. Trends in Plant Science,10(2):88-94. doi:10.1016/j.tplants.2004.12.012.
Yan Y X,Stolz S,Chételat A,Reymond P,Pagni M,Dubugnon L,Farmera E E. 2007. A downstream mediator in the growth repression limb of the jasmonate pathway[J]. The Plant Cell,19(8):2470-2483. doi:10.1105/tpc.107. 050708.
Yang Y,Ahammed G J,Wan C,Liu H,Chen R,Zhou Y. 2019. Comprehensive analysis of TIFY transcription factors and their expression profiles under jasmonic acid and abiotic stresses in watermelon[J]. International Journal of Genomics. doi:10.1155/2019/6813086.
Ye H Y,Du H,Tang N,Li X H,Xiong L Z. 2009. Identification and expression profiling analysis of TIFY family genes involved in stress and phytohormone responses in rice[J]. Plant Molecular Biology,71(3):291-305. doi:10. 1007/s11103-009-9524-8.
Zhang Y C,Gao M,Singer S D,Fei Z,Hua W,Wang W,Yang H. 2012. Genome-wide identification and analysis of the TIFY gene family in grape[J]. PLoS One,7:e44465. doi:10.1371/journal.pone.0044465.
Zhang L H,You J,Chan Z. 2015a. Identification and characterization of TIFY family genes in Brachypodium distachyon[J]. Journal of Plant Research,128(6):995-1005. doi:10.1007/s10265-015-0755-2.
Zhang Z B,Li X L,Yu R,Han M,Wu Z Y. 2015b. Isolation,structural analysis,and expression characteristics of the maize TIFY gene family[J]. Molecular Genetics and Genomics,290(5):1849-1858. doi:10.1007/s00438-015-1042-6.
Zhao G,Song Y,Wang C X,Butt H I,Wang Q H,Zhang C J,Yang Z R,Liu Z,Chen E Y,Zhang X Y,Li F G. 2016. Genome-wide identification and functional analysis of the TIFY gene family in response to drought in cotton[J]. Molecular Genetics and Genomics,291(6):2173-2187. doi:10.1007/s00438-016-1248-2.
Zhu D,Bai X,Luo X,Chen Q,Cai H,Ji W,Zhu Y M. 2013. Identification of wild soybean(Glycine soja) TIFY family genes and their expression profiling analysis under bicarbonate stress[J]. Plant Cell Reports,32(2):263-272. doi:10.1007/s00299-012-1360-7.
Zhu D,Li R T,Liu X,Sun M Z,Wu J,Zhang N,Zhu Y M,Wu K Q. 2014. The positive regulatory roles of the TIFY10 proteins in plant responses to alkaline stress[J]. PLoS One,9:e111984. doi:10.1371/journal.pone.0111984.
(責任编辑 陈 燕)

