鸡TRAF6和TIFA基因克隆及其组织表达特征分析
赵采芹 王燕碧 张福平 唐宏 周磊 韩一帆 段志强

摘要:【目的】分析腫瘤坏死因子受体相关因子6(TRAF6)及具有叉头相关结构域的TRAF相互作用蛋白(TIFA)基因在贵州黄鸡不同发育阶段各组织中的表达特点,为后续开展TRAF6和TIFA基因调控鸡的生长发育及二者相互作用调控鸡相关病毒复制打下基础。【方法】利用RT-PCR扩增鸡TRAF6和TIFA基因编码区(CDS)序列,通过ProtParam、SOPMA、SWISS-MODEL及MegAlign等在线软件进行生物信息学分析;同时采用实时荧光定量PCR分别检测TRAF6和TIFA基因在鸡胚发育第14 d(E14d)、鸡出壳第1 d(H1d)、第7 d(H7d)和第14 d(H14d)不同组织中的表达情况。【结果】鸡TRAF6和TIFA基因CDS序列全长分别为1638和564 bp,编码545和187个氨基酸残基,对应的编码蛋白分子量约62和22 kD,理论等电点(pI)为6.14和5.18。鸡TRAF6蛋白二级结构中无规则卷曲占47.08%、α-螺旋占38.14%、延伸链占11.86%、β-转角占2.92%,鸡TIFA蛋白二级结构中无规则卷曲占51.87%、α-螺旋占27.81%、延伸链占16.58%、β-转角占3.74%,二者均以无规则卷曲和α-螺旋为主。TRAF6和TIFA基因核苷酸序列同源比对分析均显示鸡与火鸡的相似性最高,分别为96.4%和92.2%,符合物种进化规律,也表明TRAF6和TIFA基因在禽类中具有一定的遗传保守性。TRAF6和TIFA基因在贵州黄鸡不同发育阶段的眼球、脑组织、心脏、肝脏、肺脏、肌胃、胸肌及腿肌中均有不同程度表达,且内脏组织的相对表达量普遍高于肌肉组织,以肺脏中的相对表达量最高,其次是肝脏和肌胃;从E14d发育到H14d,鸡TRAF6和TIFA基因在肺脏、肝脏和肌胃中的表达均呈先下降后上升的变化趋势,在眼球中则呈先上升后下降再上升的变化趋势,在脑组织、胸肌和腿肌的表达水平较低且趋于稳定。【结论】TRAF6和TIFA基因在鸡不同发育阶段各组织中均有表达,且以肺脏和肝脏中的表达水平相对较高,在脑组织、胸肌和腿肌中的表达相对较低,故推测TRAF6和TIFA基因相互作用对鸡的生长发育具有调控作用。
关键词: 贵州黄鸡;TRAF6基因;TIFA基因;组织表达特征;发育阶段
中图分类号: S831.1 文献标志码: A 文章编号:2095-1191(2022)02-0546-11
Cloning and analysis tissue expression of chicken
TRAF6 and TIFA genes
ZHAO Cai-qin, WANG Yan-bi, ZHANG Fu-ping, TANG Hong,
ZHOU Lei,HAN Yi-fan, DUAN Zhi-qiang
(College of Animal Sciences, Guizhou University/Key Laboratory of Animal Genetics, Breeding and Reproduction in the Plateau Mountains Region, Ministry of Education/Key Laboratory of Animal Genetics,Breeding and Reproduction in Guizhou, Guiyang 550025, China)
Abstract:【Objective】The aim of this study was to analyze the expression characteristics of the tumor necrosis factor (TNF), receptor-associated factor 6 (TRAF6) and TRAF6 interactions with a forkhead associated domain protein (TIFA) genes in tissues of Guizhou yellow chickens at different developmental stages, which provided a foundation for further studying the role of TRAF6 and TIFA genes in chicken growth and development and the replication of chicken associa-ted virus. 【Method】The coding DNA sequences (CDS) of chicken TRAF6 and TIFA genes were amplified by RT-PCR and then analyzed by bioinformatics software including ProtParam, SOPMA, SWISS-MODEL and MegAlign. Quantitative real-time PCR was used to detect the expression of TRAF6 and TIFA genes in different tissues in 14 day old (E14d) chicken embryos, and at 1 (H1d), 7 (H7d) and 14 (H14d) days after hatching. 【Result】The CDS regions of chicken TRAF6 and TIFA genes were 1638 and 564 bp in length, respectively, which encoded 545 and 187 amino acids. The corresponding proteins had molecular weights of about 62 and 22 kD, with theoretical isoelectric points of 6.14 and 5.18. The results of protein secondary structure analysis showed that chicken TRAF6 and TIFA proteins mainly contained irregular coil (47.08% and 51.87%), alpha helix (38.14% and 27.81%), extended chain (11.86% and 16.58%), and β -rotation (2.92% and 3.74%), respectively. Therefore, the two proteins were primarily composed of irregular coil and alpha helix. The nucleotide homology analysis of TRAF6 and TIFA genes revealed that chicken and turkey had the highest similarity at 96.4% and 92.2%, respectively, indicating a certain degree of genetic conservatism among avian species. The tissue expression analysis showed that TRAF6 and TIFA genes had different expression levels in the eyeball, brain tissue, heart, liver, lung, masticatory stomach, breast muscle and leg muscle of Guizhou yellow chickens at different developmental stages, but the relative expression level in visceral tissues was much higher than that in muscle tissues, of which the highest visceral expression level was in the lung, followed by the liver and masticatory stomach. From E14d to H14d, the expression of chicken TRAF6 and TIFA genes in lung, liver and masticatory stomach first showed a trend of decrease then an increase. The expression in the eye showed first a rising trend then falling and rising again. The expression in brain tissue, breast muscle and leg muscle was initially low then tended towardsa higher, stable level. 【Conclusion】TRAF6 and TIFA genes were expressed in all tissues of chicken at different developmental stages, with relatively high expression levels in the lung and liver, but relatively low in brain tissue, breast muscle and leg muscle, suggesting that the interaction between TRAF6 and TIFA genes could regulate the growth and development of chickens.
Key words: Guizhou yellow chicken; TRAF6 gene; TIFA gene; tissue expression; developmental stages
Foundation items:National Natural Science Foundation of China(31760732,31960698);Science and Technology Foundation of Guizhou Province(QKHJC〔2020〕1Y134);Joint Project of Local Poultry Industry in Guizhou Province(QCN〔2020〕175)
0 引言
【研究意義】肿瘤坏死因子受体相关因子6(Tumor necrosis factor receptor-associated factor 6,TRAF6)是白细胞介素1受体/Toll样受体(IL-1/TLR)超家族的一种关键衔接蛋白,具有E3泛素连接酶作用(Dou et al.,2018),可通过激活下游的细胞核因子κB(Nuclear factor-κB,NF-κB)及干扰素调节因子等信号通路,影响细胞的增殖、分化与凋亡,并调控细胞炎症反应、氧化应激和骨代谢等生物学过程(Jeong et al.,2007;Schnetzke et al.,2013)。具有叉头相关结构域的TRAF相互作用蛋白[TNF receptor associated factor(TRAF)-interacting protein with a Forkhead-associated (FHA) domain,TIFA]是一种在白细胞介素1(Interleukin 1,IL-1)信号通路上将TRAF6连接到IL-1受体相关激酶1的蛋白分子(Takatsuna et al.,2003),在细胞周期调控、细胞生长及信号转导等生物学过程中发挥关键作用(Pennell et al.,2010)。此外,TIFA与先天性免疫相关,通过调节TRAF6蛋白的泛素化和寡聚化激活NF-κB信号通路,促进细胞炎性因子产生(Ea et al.,2004)。目前,关于人类和小鼠TRAF6、TIFA在NF-κB信号通路中抵抗微生物感染的报道越来越多,但针对其组织表达特征的研究相对较少。因此,开展鸡TRAF6和TIFA基因生物信息学及其组织表达特征分析,可为后续探讨TRAF6和TIFA基因调控鸡相关组织生长发育及二者相互作用抵抗病毒感染的作用机制打下基础。【前人研究进展】TRAF6在人类和不同动物的组织中广泛表达,尤其在肝脏、肺脏、脾脏和心脏中高表达,在脑组织中也有表达(Jin et al.,2017;Guo et al.,2019)。已有研究证实,TRAF6白不仅介导信号转导,还在先天性免疫反应及淋巴结、中枢神经系统和胚胎发育等过程中发挥重要作用(Lomaga et al.,1999;Xiao et al.,2020)。此外,TRAF6是多种组织发育所必须的蛋白。Lomaga等(1999)研究发现,敲除小鼠TRAF6基因后一些神经区域出现神经管缺陷和裸脑产生的现象,从而引起小鼠的非正常死亡。Akiyama 等(2005)研究表明,TRAF6基因缺乏会导致小鼠的胸腺基质发育异常,从而改变其免疫耐受性。王晓芬(2019)通过生长发育定量和成蝇干扰投喂试验,发现TRAF6基因在家蝇不同发育阶段均有表达,且在蛹和成蝇阶段表达量较高,表明TRAF6参与家蝇蛹期生殖发育过程且在家蝇的发育中发挥免疫防御作用。另外,研究发现TRAF6和TIFA基因在肺癌患者的肺脏组织存在高表达,沉默TIFA基因可促进肺癌细胞的凋亡,并抑制肺癌细胞迁移(骆磊,2014;门万夫,2017)。Jin等(2017)研究发现,TRAF6基因在新城疫病毒(Newcastle disease virus,NDV)感染前的鸡肺脏和脾脏中高表达,而在感染后的鸡免疫器官(法氏囊、脾脏和胸腺)中高表达,表明TRAF6基因在宿主防御鸡病毒感染过程中发挥至关重要的作用。本课题组最近研究发现,M蛋白的细胞质定位能通过下调TIFA/TRAF6/NF-κB介导的细胞因子产生而促进NDV复制(Duan et al.,2020)。综上所述,TRAF6和TIFA基因在调控动物组织生长发育及TIFA/TRAF6/NF-κB信号通路抵抗微生物感染方面发挥重要作用。【本研究切入点】目前,TRAF6与TIFA相互作用在机体先天性免疫方面的报道仅涉及非洲爪蟾(Inoue et al.,2005)和人类(Huang et al.,2019),有关鸡TRAF6和TIFA相互作用及二者在鸡组织中的表达特征尚未明确。【拟解决的关键问题】以贵州黄鸡为研究对象,克隆鸡RAF6和TIFA基因的完整编码区(CDS)序列,运用在线软件进行生物信息学分析,进一步通过实时荧光定量PCR检测TRAF6和TIFA基因在鸡胚发育第14 d(E14d)、鸡出壳第1 d(H1d)、第7 d(H7d)和第14 d(H14d)不同组织中的表达情况,分析二者在鸡组织发育过程中的表达特点,为后续开展TRAF6和TIFA基因调控鸡的生长发育及二者相互作用调控鸡相关病毒复制打下基础。
1 材料与方法
1. 1 试验材料
鸡胚成纤维细胞(DF-1)和大肠杆菌(Escherichia coli)DH5α感受态细胞由贵州大学高原山地动物遗传育种与繁殖教育部重点实验室保存提供。贵州黄鸡受精鸡蛋由贵州大学种鸡场提供,在相同条件下进行孵化及饲养管理,取样屠宰后用高温灭菌的剪刀和镊子分别取E14d、H1d、H7d和H14d等4个阶段贵州黄鸡的眼球、脑组织、心脏、肝脏、肺脏、肌胃、胸肌及腿肌等8个组织,置于-80 ℃冰箱保存备用。主要试剂:TRIzol Regent提取试剂盒及胶回收试剂盒购自OMEGA公司;Star-Script Ⅱ逆转录试剂盒购自GenStar公司;SYBR Green qPCR Master Mix(No ROX)购自APE×BIO公司;2×Taq PCR StarMix及DNA Marker购自Thermo Fisher公司;pMD19-T载体、卡那霉素和氨苄青霉素购自TaKaRa公司;其他均为国产分析纯。
1. 2 引物设计与合成
根据GenBank已公布的鸡TRAF6基因(XM_015287208.2)和TIFA基因(XM_015276339.2),利用Primer Premier 5.0分别设计扩增鸡TRAF6和TIFA基因的特异性引物(表1),以GAPDH为内参基因。所有引物均委托北京擎科新业生物技术有限公司合成。
1. 3 鸡TRAF6和TIFA基因克隆
使用TRIzol法从DF-1细胞中提取总RNA并进行反转录,以特异性引物分别扩增鸡TRAF6和TIFA基因CDS序列。扩增程序:94 ℃预变性2 min;94 ℃ 30 s,60 ℃ 30 s,72 ℃ 90 s,进行25个循环;72 ℃延伸5 min,4 ℃保存。PCR扩增产物经1.0%琼脂糖凝胶电泳检测,按照胶回收试剂盒说明对目的条带进行胶回收,将胶回收产物与pMD19-T载体进行连接,金属浴16 ℃连接24 h,然后转化DH5α感受态细胞,挑取白斑单菌落扩大培养后进行菌液PCR鉴定,经1.0%琼脂糖凝胶电泳检测,将阳性菌液送至北京擎科新业生物技术有限公司测序。
1. 4 鸡TRAF6和TIFA蛋白生物信息学分析
利用生物信息学在线软件ProtParam(http://us.expasy.org/tools/protparam.html/)、SOPMA(http://npsapbil. ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_ sopma.html)和SWISS-MODLE(http://swissmodel.expasy.org/)分别对鸡TRAF6和TIFA基因编码蛋白进行理化性质、二级和三级结构预测(邓珊珊等,2017;康超等,2021);从GenBank下载不同物种的TRAF6和TIFA基因核苷酸序列,利用MegAlign进行多序列比对分析,并采用MEGA 6.0中的邻接法(Neighbor-joining,NJ)构建系统发育进化树。
1. 5 总RNA提取及cDNA第一链合成
根据TRIzol试剂盒说明,分别提取4个阶段的鸡眼球、脑组织、心脏、肝脏、肺脏、肌胃、胸肌和腿肌等组织总RNA,利用超微量紫外分光光度计对各组织总RNA进行浓度和OD测定,然后按照反转录试剂盒操作说明合成cDNA第一链。
1. 6 实时荧光定量PCR检测鸡TRAF6和TIFA基因表达
采用实时荧光定量PCR检测TRAF6和TIFA基因在鸡各组织中的表达情况。反应体系20.0 μL:2×SYBR Green qPCR Master Mix 10.0 μL,上、下游引物各0.5 ?L,cDNA模板0.5 ?L,ROX Refeience Dye 0.4 ?L,ddH2O 8.1 ?L。扩增程序:95 ℃预变性2 min;95 ℃ 15 s,52 ℃ 30 s,72 ℃ 30 s,进行39个循环。熔解曲线由仪器自动设置,每个样品设3个重复。获得的熔解曲线呈光滑的S形,可用于2-ΔΔCt法计算,且熔解曲线全部呈单峰,峰值较好,即引物特异性良好。
1. 7 统计分析
利用Excel 2016对试验数据进行整理后,采用2-ΔΔCt换算目的基因相对表达量(韩一帆等,2020),并通过SPSS 18.0进行统计分析。
2 结果与分析
2. 1 鸡TRAF6和TIFA基因PCR扩增结果
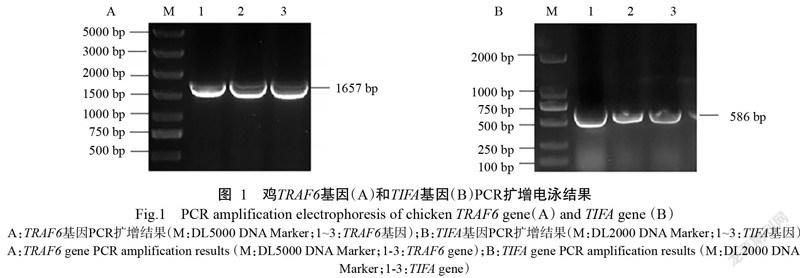
以DF-1细胞总RNA反转录合成的cDNA第一链为模板,分别对鸡TRAF6和TIFA基因CDS序列进行PCR扩增,经1.0%琼脂糖凝胶电泳检测PCR扩增产物,结果获得与预期结果相符的TRAF6基因(图1-A)和TIFA基因(图1-B)目的条带。
2. 2 鸡TRAF6和TIFA基因测序結果
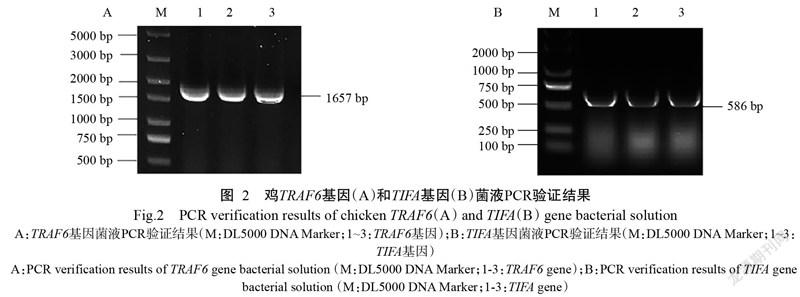
菌液PCR鉴定结果显示:重组质粒pMD19-TRAF6和pMD19-TIFA分别获得1657 bp(图2-A)和586 bp(图2-B)的目的条带。与GenBank已公布的鸡TRAF6和TIFA基因序列相比,送测序的9个样品中TRAF6基因CDS序列第148位核苷酸均存在碱基突变(T→C),导致苯丙氨基酸(F)突变为亮氨基酸(L),都属于疏水性氨基酸;TIFA基因CDS序列第265位核苷酸发生碱基突变(T→C),但未引起氨基酸改变。表明鸡TRAF6基因和TIFA基因重组质粒pMD19-TRAF6和pMD19-TIFA构建成功。
2. 3 鸡TRAF6和TIFA蛋白生物信息学分析结果
2. 3. 1 鸡TRAF6和TIFA蛋白理化性质 利用ProtParam对鸡TRAF6和TIFA蛋白进行理化性质分析,结果显示,鸡TRAF6基因CDS序列全长1638 bp,分子式为C2696H4233N779O819S52,共编码545个氨基酸残基,含量较高的氨基酸是亮氨酸(8.4%)、谷氨酸(8.4%)和精氨酸(6.0%),含量较低的是色氨酸(0.4%)和酪氨酸(2.2%),带负电荷的氨基酸残基(天冬氨酸+谷氨酸)有68个,带正电荷的氨基酸残基(精氨酸+赖氨酸)有58个;编码蛋白分子量约62 kD,理论等电点(pI)为6.14。鸡TIFA基因CDS序列全长564 bp,分子式为C962H1491N253O293S17,共编码187个氨基酸残基,含量较高的氨基酸是谷氨酸(10.2%)、异亮氨酸(8.6%)、丝氨酸(7.0%)和赖氨酸(7.0%),带负电荷的氨基酸残基(天冬氨酸+谷氨酸)有29个,带正电荷的氨基酸残基(精氨酸+赖氨酸)有21个;编码蛋白分子量约22 kD,pI为5.18。
2. 3. 2 鸡TRAF6和TIFA蛋白二、三级结构 采用SOPMA和SWISS-MODEL分别对鸡TRAF6和TIFA蛋白的二、三级结构进行预测,结果显示,鸡TRAF6蛋白由4种结构组成,其中无规则卷曲占47.08%、α-螺旋占38.14%、延伸链占11.86%、β-转角占2.92%;鸡TIFA蛋白也由4种结构组成,其中无规则卷曲占51.87%、α-螺旋占27.81%、延伸链占16.58%、β-转角占3.74%。蛋白三级结构预测结果(图3)表明,鸡TRAF6和TIFA蛋白的三级结构与二级结构相符,主要由无规则卷曲和α-螺旋构成。
2. 3. 3 鸡TRAF6和TIFA基因核苷酸序列相似性及系统发育进化树 采用MegAlign对鸡与火鸡、日本鹌鹑和绿头鸭等禽类的TRAF6和TIFA基因核苷酸序列进行同源比对分析,结果(图4-A)显示,禽类TRAF6基因核苷酸序列相似性在85.9%~96.4%,其中鸡与火鸡的相似性最高,相似性为96.4%,与短嘴鸦的相似性最低(仅为85.9%);将鸡与牛、褐家鼠及人类等非禽类的TRAF6基因核苷酸序列进行同源比对分析,结果显示鸡与马的相似性最高(72.7%),与石斑鱼的相似性最低(40.6%)。禽类TIFA基因核苷酸序列相似性在61.7%~92.2%(图4-B),其中鸡与火鸡的相似性最高,相似性为92.2%,与冠小嘴乌鸦的相似性最低(61.7%);将鸡与羊、褐家鼠及人类等非禽类的TIFA基因核苷酸序列进行同源比对分析,结果表明鸡与人类的相似性最高(45.1%),与穴兔的相似性最低(31.4%)。基于TRAF6和TIFA基因核苷酸序列相似性构建系统发育进化树,TRAF6基因的系统发育进化树(图5-A)显示,鸡与火鸡在同一分支上,其遗传距离最近,而与石斑鱼的遗传距离最远;TIFA基因的系统发育进化树(图5-B)显示,鸡与火鸡位于同一分支上,其遗传距离最近,而与穴兔的遗传距离最远。
2. 4 不同发育阶段鸡TRAF6基因的组织表达差异
如图6所示,鸡TRAF6基因在E14d、H1d、H7d和H14d等4个阶段各组织中均有表达,且不同发育阶段鸡TRAF6基因的组织表达水平存在明显差异。以脑组织为对照,TRAF6基因在肺脏中的相对表达量最高,且在E14d、H1d和H14d阶段存在极显著差异(P<0.01,下同)。在E14d阶段的各组织中,TRAF6基因在鸡肺脏、肝脏和肌胃中的相对表达量也极显著高于其他组织,在8个组织中的相对表达量排序为:肺脏>肝脏>肌胃>腿肌>脑组织>眼球>心脏>胸肌;在H1d阶段的各组织中,TRAF6基因在肺脏和肝脏的相对表达量较高,胸肌中的相对表达量最低,在8个组织中的相对表达量排序为:肺脏>肝脏>肌胃>眼球>腿肌>心脏>脑组织>胸肌;在H7d阶段的各组织中,TRAF6基因在肺脏和肝脏中的相对表达量也较高,且极显著高于其他组织,胸肌中的相对表达量最低,在肌胃、心脏和眼球中的相对表达量差异不显著(P>0.05,下同),在8个组织中的相对表达量排序为:肺脏>肝脏>肌胃>心脏>眼球>脑组织>腿肌>胸肌;在H14d阶段的各组织中,TRAF6基因在脑组织中的表达量最低,且在眼球、肌胃和腿肌中的相对表达量极显著高于脑组织,在8个组织中的相对表达量排序为:肺脏>肝脏>眼球>肌胃>腿肌>脑组织>心脏>胸肌。
2. 5 不同发育阶段鸡TIFA基因的组织表达差异
如图7所示,不同发育阶段鸡TIFA基因的组织表达水平存在明显差异。以脑组织为对照,TIFA基因在E14d、H1d、H7d和H14d等4个阶段的各组织中均能检测到,且以肺脏中的相对表达量最高,其次是肝脏。在E14d阶段的各组织中,TIFA基因在脑组织中的相对表达量最低,在肺脏中的相对表达量极显著高于其他组织,在8个组织中的相对表达量排序为:肺脏>肝脏>肌胃>心脏>眼球>腿肌>胸肌>脑组织;在H1d阶段的各组织中,TIFA基因在肝脏和肺脏中的相对表达量极显著高于其他组织,在眼球、心脏、肌胃、胸肌和腿肌中的相对表达量差异不显著,具体排序为:肺脏>肝脏>眼球>心脏>肌胃>胸肌>腿肌>脑组织;在H7d阶段的各组织中,TIFA基因在肺脏和肝脏中的相对表达量极显著高于其他组织,以胸肌中的相对表达量最低,在8个组织中的相对表达量排序为:肺脏>肝脏>肌胃>心脏>眼球>脑组织>腿肌>胸肌;在H14d阶段的各组织中,TIFA基因在肺脏中的相对表达量极显著高于其他组织,肌胃、心臟和眼球中的相对表达量也极显著高于脑组织,在8个组织中的相对表达量排序为:肺脏>肝脏>肌胃>心脏>眼球>脑组织>胸肌>腿肌。
2. 6 鸡TRAF6和TIFA基因在不同发育阶段各组织中的表达变化趋势
鸡TRAF6和TIFA基因在4个阶段各组织中的表达变化趋势如图8所示。从E14d发育到H14d,TRAF6基因在肺脏、肝脏和肌胃中的表达均呈先下降后上升的变化趋势,于H7d阶段降至最低值,整体上呈V形变化模式;脑组织中的TRAF6基因表达在整个试验周期内均趋于稳定;在眼球中,TRAF6基因的表达呈先上升后下降再上升的变化趋势。TIFA基因在肝脏、肺脏、心脏和肌胃中的表达也呈先下降后上升的变化趋势,但最低值出现的发育阶段不同,肝脏、肺脏和心脏在H7d阶段降至最低值,肌胃则在H1d阶段降至最低值;TIFA基因在眼球中的表达呈先上升后下降再上升的变化趋势,在其他组织中的表达整体上趋于稳定。
3 讨论
已有研究证实,在非洲爪蟾中TIFA与TRAF6相互作用后可激活NF-κB信号通路(Inoue et al.,2005)。Huang等(2019)研究发现,人类TRAF6蛋白TRAF结构域与TIFA蛋白FHA结构域相互作用是介导NF-κB激活的重要因素。近年来,关于TRAF6上游蛋白分子介导TRAF6/NF-kB信号通路的研究报道越来越多,包括TLR4/TRAF6/NF-κB、IRAK1/TRAF6/NF-κB和IL-17RA/TRAF6/NF-κB等(Edmonds et al.,2011;Ding et al.,2013;Ahmedy et al.,2020)。已有研究表明,志贺氏菌感染可抑制宿主的TRAF6/NF-κB信号通路,进而抑制炎症细胞因子产生,促进志贺氏菌复制(Ying and Ferrero,2019)。可见,TRAF6介导的NF-κB信号通路在揭示病毒和细菌感染机理方面具有重要意义。在NF-κB信号通路中,TIFA与TRAF6的相互作用在抵抗微生物感染方面也逐渐得到关注。TRAF6作为细胞内信号转导分子,在激活多种信号转导通路中发挥重要的衔接作用(Ishida et al.,1996)。TRAF6的组织分布是其功能的重要决定因素,且影响细胞在不同组织中监测不同微生物的能力(Cheng et al.,2015);TRAF6对正常的骨骼形成也至关重要(Naito et al.,2002)。Kobayashi等(2001)利用基因敲除技术在胚胎发育过程中敲除TRAF6基因,结果发现TRAF6基因缺陷型小鼠因信号转导缺陷而导致严重的骨质疏松和缺乏淋巴结,同时表现出汗湿性外胚层发育不良。Naito等(2002)研究发现,TRAF6对于X相关的外异蛋白A2受体(X-linked ectodysplasin-A2 receptor,XEDAR)介导的NF-κB激活必不可少,TRAF6能转导XEDAR发出与表皮发育有关的信号。Inoue等(2005)研究表明,在非洲爪蟾的胚胎期TRAF6基因不仅在神经节的头部区域和神经管中表达,还在咽及头部和神经管中表达。Zhou等(2015)研究认为,TRAF6基因在雌雄梭子蟹各组织中的表达量存在差异可能与其不同发育存在联系,即TRAF6在组织生长发育过程中发挥重要作用。此外,已有研究发现TRAF6基因在人类(Starczynowski et al.,2011)、病毒感染小鼠(Mason et al.,2004)和鸭(Zhai et al.,2015)的免疫相关组织中高表达,且TRAF6是负责病原体识别受体介导先天免疫应答的关键细胞质衔接分子,表明TRAF6在抗病毒先天免疫应答的激活中扮演重要角色。
TIFA是通过酵母菌双杂交技术首次筛选获得并被鉴定的一种TRAF6相互作用蛋白(Takatsuna et al.,2003)。在结构上,TIFA具有N末端叉形头相关结构域(Forkhead-associated,FHA)及C末端共同的TRAF6结合基序(Huang et al.,2019)。在國内,针对TIFA基因的研究报道相对较少,且主要集中在各类癌症中的表达分析。TIFA在肺腺癌中通过激活NF-κB信号通路并影响下游蛋白表达,而促进肿瘤细胞增殖(门万夫,2017)。TIFA基因在人体内的很多组织中均有表达,且以在脾脏中的表达量最高,而在骨骼肌中几乎不表达(Takatsuna et al.,2003)。Ding等(2013)研究发现,在创伤和失血性休克后,TIFA基因在肝脏早期显著上调而引发炎症反应。近年来,ALPK1控制TIFA/TRAF6/NF-κB依赖的先天免疫抵抗细菌感染也逐渐被报道(Milivojevic et al.,2017;Xue and Man,2018;Ying and Ferrero,2019),且在宿主抵抗微生物感染的免疫调控机制研究方面发现TIFA/TRAF6/NF-κB信号通路活化也是产生炎性细胞因子的关键通路。
本研究成功克隆获得鸡TRAF6和TIFA基因CDS序列并进行生物信息学分析,结果发现鸡TRAF6和TIFA蛋白二、三级主要以无规则卷曲和α-螺旋为主,为混合型蛋白。无规则卷曲是蛋白肽链中构成配体、受体结合的组成部分,会受侧链的影响而改变其空间结构。TRAF6和TIFA蛋白的无规则卷曲可能是通过影响蛋白肽链的结合活性而调节其功能。TRAF6和TIFA基因核苷酸序列同源比对分析结果均显示,鸡与火鸡的相似性最高,且分别与石斑鱼和穴兔的相似性最低,符合物种进化规律,表明TRAF6和TIFA基因在禽类中具有一定的遗传保守性。同时,利用实时荧光定量PCR检测TRAF6和TIFA基因在鸡发育过程中的表达规律,结果表明TRAF6和TIFA基因在贵州黄鸡不同发育阶段的眼球、脑组织、心脏、肝脏、肺脏、肌胃、胸肌及腿肌中均有不同程度表达,且内脏组织的相对表达量普遍高于肌肉组织,说明TRAF6和TIFA基因属于广谱表达基因,在肌肉组织中低表达暗示贵州黄鸡的TRAF6和TIFA基因可能已获得非免疫信号转导功能。鸡TRAF6和TIFA基因表达量受生长发育的影响,在不同发育阶段的肺脏、肝脏和肌胃中,TRAF6和TIFA基因的相对表达量均随贵州黄鸡日龄增长呈先下降后上升的变化趋势,表明TRAF6和TIFA基因在鸡生长发育过程的不同阶段可能发挥着不同的调控作用。此外,TRAF6和TIFA基因在贵州黄鸡4个发育阶段肺脏中的相对表达量最高,其次是肝脏,与骆磊(2014)、贾生美(2017)、Jin等(2017)、丁少青等(2019)的研究结果一致,即在E14d阶段肺脏中高表达,暗示TRAF6和TIFA基因可能参与鸡肺脏的发育及早期胚胎的形成;在H1d、H7d和H14d阶段肺脏中高表达,故推测TRAF6和TIFA基因还参与鸡肺脏中的先天免疫;在肝脏中高表达则表明TRAF6和TIFA基因可能与脂代谢及糖代谢等调控功能相关。此外,TRAF6基因在贵州黄鸡4个阶段脑组织、胸肌和腿肌中的表达量相对较低,与骆磊(2014)、Jin等(2017)研究发现TRAF6基因在白来航鸡和黑康三世蛋鸡中的表达规律一致,故推测TRAF6基因在中枢神经系统发育中也发挥重要作用。TRAF6基因在贵州黄鸡不同发育阶段肌胃中的表达呈中等水平,而Inoue等(2005)研究发现TRAF6基因在非洲爪蟾胃中的表达水平较低,可能是物种差异所致。TRAF6和TIFA基因在不同物种各组织和胚胎发育阶段的特异性表达,揭示二者在生命体的生长发育过程中发挥重要作用,尤其与生长发育、骨代谢和先天免疫等密切相关,但是否影响贵州黄鸡组织生长发育尚未明确。
4 结论
TRAF6和TIFA基因在鸡不同发育阶段各组织中均有表达,且以肺脏和肝脏中的表达水平相对较高,在脑组织、胸肌和腿肌中的表达相对较低,故推测TRAF6和TIFA基因相互作用对鸡的生长发育具有调控作用。
参考文献:
邓珊珊,嵇辛勤,段志强,熊建民,阮涌,赵佳福,胡焱,田宇杰. 2017. 鸡基质蛋白3基因的生物信息学分析[J]. 中国家禽,39(19):67-70. [Deng S S,Ji X Q,Duan Z Q,Xiong J M,Ruan Y,Zhao J F,Hu Y,Tian Y J. 2017. Bioin-formatics analysis of chicken matrix protein 3 gene[J]. China Poultry,39(19):67-70.] doi:10.16372/j.issn.1004-6364.2017.19.014.
丁少青,周泽斌,王雅倩,罗鑫,何缘圆,任建峰,李伟明,张庆华. 2019. 东北七鳃鳗TRAF6基因克隆、表达分析及亚细胞定位研究[J]. 水生生物学报,43(1):9-16. [Ding S Q,Zhou Z B,Wang Y Q,Luo X,He Y Y,Ren J F,Li W M,Zhang Q H. 2019. Molecular cloning,expression analysis and subcellular localization of tumor necrosis factor receptor-associated factor 6 in Korean lamprey,Lethente-ron morii[J]. Acta Hydrobiologica Sinica,43(1):9-16.] doi:10.7541/2019.002.
韓一帆,周磊,袁超,高洪波,王燕碧,赵采芹,唐宏,段志强. 2020. 鸡BRD2亚细胞定位及其组织表达特性分析[J]. 南方农业学报,51(8):1857-1863. [Han Y F,Zhou L,Yuan C,Gao H B,Wang Y B,Zhao C Q,Tang H,Duan Z Q. 2020. Subcellular localization of chicken bromodomain-containing protein 2(BRD2) and its expression characteristics[J]. Journal of Southern Agriculture,51(8):1857-1863.] doi:10.3969/j.issn.2095-1191.2020.08.009.
贾生美. 2014. 鲤鱼TLR通路中TRAF6的基因克隆、鉴定及差异表达分析[D]. 长春:吉林大学. [Jia S M. 2014. Cloning,characterization and expression analysis of common carp TRAF6[D]. Changchun:Jilin University.]
康超,荣娜,简思杰,孙薇,刘祥,陈琛,陈春琳. 2021. 嗜水气单胞菌外膜蛋白AH6169的生物信息学分析及其原核表达[J]. 河南农业大学学报,55(1):80-88. [Kang C,Rong N,Jian S J,Sun W,Liu X,Chen C,Chen C L. 2021. Bioinformatics analysis of outer membrane protein AH6169 of Aeromonas hydrophila and its prokaryotic expression[J]. Journal of Henan Agricultural University,55(1):80-88.] doi:10.16445/j.cnki.1000-2340.20210122. 017.
骆磊. 2014. 鸡TRAF3、TRAF6基因的克隆、表达及其在鸡抗病毒免疫应答过程中的作用研究[D]. 雅安:四川农业大学. [Luo L. 2014. Molecular cloning,tissue distribution of chicken TRAF3 and TRAF6 genes,and their roles in chickens antiviral immune response[D]. Yaan:Sichuan Agricultural University.]
门万夫. 2017. TIFA在肺腺癌中的异常表达及其促进肺腺癌恶性生物学行为机制的研究[D]. 沈阳:中国医科大学. [Men W F. 2017. The investigation of TIFA expression in lung adenocarcinoma and the mechanism in promoting malignant biological behavior[D]. Shenyang:China Me-dical University.]
王晓芬. 2019. TRAF6在家蝇免疫及生殖中的作用[D]. 保定:河北大学. [Wang X F. 2019. The role of TRAF6 in immunity and reproduction of house flies[D]. Baoding:Hebei University.]
Ahmedy O A,Ibrahim S M,Salem H H,Kandil E A. 2020. Antiulcerogenic effect of melittin via mitigating TLR4/TRAF6 mediated NF-κB and p38MAPK pathways in acetic acid-induced ulcerative colitis in mice[J]. Chemico-Biological Interactions,331(8):109276-109283. doi:10. 1016/j.cbi.2020.109276.
Akiyama T,Maeda S,Yamane S,Ogino K,Kasai M,Kajiura F,Matsumoto M,Inoue J I. 2005. Dependence of self-to-lerance on TRAF6-directed development of thymic stroma[J]. Science,308(5719):248-251. doi:10.1126/science. 1105677.
Cheng Y Q,Sun Y J,Wang H G,Yan Y X,Ding C,Sun J H. 2015. Chicken STING mediates activation of the IFN gene independently of the RIG-I gene[J]. The Journal of Immunology,195(8):3922-3936. doi:10.4049/jimmunol.1500638.
Ding N,Zhang Y,Loughran P A,Wang Q D,Tsung A,Billiar T R. 2013. TIFA upregulation after hypoxia-reoxygena-tion is TLR4- and MyD88-dependent and associated with HMGB1 upregulation and release[J]. Free Radical Bio-logy and Medicine,63:361-367. doi:10.1016/j.free-radbiomed.2013.05.029.
Dou Y,Tian X D,Zhang J,Wang Z,Chen G. 2018. Roles of TRAF6 in central nervous system[J]. Current Neuropharmacology,16(9):1306-1313. doi:10.2174/1570159X16 666180412094655.
Duan Z Q,Yuan C,Han Y F,Zhou L,Zhao J F,Ruan Y,Chen J Q,Ni M M,Ji X Q. 2020. TMT-based quantitative proteomics analysis reveals the attenuated replication mechanism of Newcastle disease virus caused by nuclear localization signal mutation in viral matrix protein[J]. Virulence,11(1):607-635. doi:10.1080/21505594.2020. 1770482.
Ea C K,Sun L J,Inoue J I,Chen Z J. 2004. TIFA activates IκB kinase(IKK) by promoting oligomerization and ubi-quitination of TRAF6[J]. Proceedings of the National Academy of Sciences of the United States of America,101(43):15318-15323. doi:10.1073/pnas.0404132101.
Edmonds R D,Vodovotz Y,Lagoa C,Dutta-Moscato J,Yang Y W,Fink M P,Levy R M,Prince J M,Kaczorowski D J,Tseng G C,Billiar T R. 2011. Transcriptomic response of murine liver to severe injury and hemorrhagic shock:A dual-platform microarray analysis[J]. Physiologcal Genomics,43(20):1170-1183. doi:10.1152/physiolgenomics.00020.2011.
Guo Y X,Xu Y,Kang X L,Meng C,Gu D,Zhou Y Y,Xiong D,Geng S Z,Jiao X N,Pan Z M. 2019. Molecular clo-ning and functional analysis of TRAF6 from Yangzhou great white goose Anser anser[J]. Developmental & Co-mparative Immunology,101(1):103435-103447. doi:10. 1016/j.dci.2019.103435.
Huang W C,Liao J H,Hsiao T C,Wei T Y W,Maestre-Reyna M,Bessho Y,Tsai M D. 2019. Binding and enhanced binding between key immunity proteins TRAF6 and TIFA[J]. ChemBioChem,20(2):140-146. doi:10.1002/cbic. 201800436.
Inoue J I,Yagi S G,Ishikawa K,Azuma S,Ikawa S,Semba K. 2005. Identification and characterization of Xenopus laevis homologs of mammalian TRAF6 and its binding protein TIFA[J]. Gene,358(7):53-69. doi:10.1016/j.gene.2005.05.016.
Ishida T,Mizushima S I,Azuma S,Kobayashi N,Tojo T,Suzuki K,Aizawa S,Watanabe T,Mosialos G,Kieff E,Yamamoto T,Inoue J. 1996. Identification of TRAF6,a novel tumor necrosis factor receptor-associated factor protein that mediates signaling from an amino-terminal domain of the CD40 cytoplasmic region[J]. Journal of Biological Chemistry,271(46):28745-28748. doi:10.1074/jbc.271.46.28745.
Jeong S,Cho I R,An W G,Jhun B H,Lee B S,Park K,Chung Y H. 2007. STP-A11,an oncoprotein of Herpesvirus saimiri augments both NF-κB and AP-1 transcription activity through TRAF6[J]. Experimental & Molecular Medicine,39(1):56-64. doi:10.1038/emm.2007.7.
Jin J,Ran J S,Yang C W,Jiang X S,Zhou Y G,Feng Z Q,Wang Y,Lan D,Ren P,Liu Y P. 2017. Molecular characterization,expression,and functional analysis of chicken TRAF6[J]. Genetics and Molecular Research,16(1):87-93. doi:10.4238/gmr16019138.
Kobayashi N,Kadono Y,Naito A,Matsumoto K,Yamamoto T,Tanaka S,Inoue J. 2001. Segregation of TRAF6-media-ted signaling pathways clarifies its role in osteoclastogenesis[J]. The EMBO Journal,20(6):1271-1280. doi:10.1093/emboj/20.6.1271.
Lomaga M A,Yeh W C,Sarosi I,Duncan G S,Furlonger C,Ho A,Morony S,Capparelli C,Van G,Kaufman S,van der Heiden A,Itie A,Wakeham A,Khoo W,Sasaki T,Cao Z D,Penninger J M,Paige C J,Lacey D L,Dunstan C R,Boyle W J,Goeddel D V,Mak T W. 1999. TRAF6 deficiency results in osteopetrosis and defective interleukin-1,CD40,and LPS signaling[J]. Genes & Development,13(8):1015-1024. doi:10.1101/gad.13.8.1015.
Mason N J,Fiore J,Kobayashi T,Masek K S,Choi Y,Hunter C A. 2004. TRAF6-dependent mitogen-activated protein kinase activation differentially regulates the production of interleukin-12 by macrophages in response to Toxoplasma gondii[J]. Infection and Immunity,72(10):5662-5667. doi:10.1128/IAI.72.10.5662-5667.2004.
Milivojevic M,Dangeard A S,Kasper C A,Tschon T,Emmenlauer M,Pique C,Schnupf P,Guignot J,Arrieumerlou C. 2017. ALPK1 controls TIFA/TRAF6-dependent innate immunity against heptose-1,7-bisphosphate of gram-negative bacteria[J]. PLoS Pathogens,13(1):6224-6236. doi:10.1371/journal.ppat.1006224.
Naito A,Yosahiro H,Nishioka E,Satoh M,Azuma S,Yamamoto T,Nishikawa S I,Inoue J I. 2002. TRAF6-deficient mice display hypohidrotic ectodermal dysplasia[J]. Proceedings of the National Academy of Sciences of the United States of America,99(13):8766-8771. doi:10. 1073/pnas.132636999.
Pennell S,Westcott S,Ortiz-Lombardia M,Patel D,Li J J,Nott T J,Mohammed D,Buxton R S,Yaffe M B,Verma C,Smerdon S J. 2010. Structural and functional analysis of phosphothreonine-dependent FHA domain interactions[J]. Structure,18(12):1587-1595. doi:10.1016/j.str.2010. 09.014.
Schnetzke U,Fischer M,Kuhn A K,Spies-Weisshart B,Zirm E,Hochhaus A,Müller J P,Scholl S. 2013. The E3 ubi-quitin ligase TRAF6 inhibits LPS-induced AKT activation in FLT3-ITD-positive MV4-11 AML cells[J]. Journal of Cancer Research Clinical Oncology,139:605-615. doi:10.1007/s00432-012-1362-4.
Starczynowski D T,Lockwood W W,Deléhouzée S,Chari R,Wegrzyn J,Fuller M,Tsao M S,Lam S,Gazdar A F,Lam W L,Karsan A. 2011. TRAF6 is an amplified oncogene bridging the RAS and NF-κB pathways in human lung cancer[J]. The Journal of Clinical Investigation,121(10):4095-4105. doi:10.1172/JCI58818.
Takatsuna H,Kato H,Gohda J,Akiyama T,Moriya A,Okamoto Y,Yamagata Y,Otsuka M,Umezawa K,Semba K,Inoue J I. 2003. Identification of TIFA as an adapter protein that links tumor necrosis factor receptor-associated factor 6(TRAF6) to interleukin-1 (IL-1) receptor-associa-ted kinase-1(IRAK-1) in IL-1 receptor signaling[J]. The Journal of Biological Chemistry,278(14):12144-12150. doi:10.1074/jbc.M300720200.
Xiao L,Zhong M D,Huang Y,Zhu J,Tang W K,Li D Y,Shi J D,Lu A Q,Yang H L,Geng D C,Li H,Wang Z R. 2020. Puerarin alleviates osteoporosis in the ovariectomyin-duced mice by suppressing osteoclastogenesis via inhibition of TRAF6/ROS- dependent MAPK/NF-κB signa-ling pathways[J]. Aging,12(21):21706-21729. doi:10. 18632/aging.103976.
Xue Y S,Man S M. 2018. ALPK1:Innate attraction to the sweetness of bacteria[J]. Cell Research,28:1125-1126. doi:10.1038/s41422-018-0100-0.
Ying L,Ferrero R L. 2019. Role of NOD1 and ALPK1/TIFA signalling in innate immunity against Helicobacter pylori infection[J]. Current Topics in Microbiology and Immunology,421:159-177. doi:10.1007/978-3-030-15138-6_7.
Zhai Y J,Luo F,Chen Y S,Zhou S S,Li Z L,Liu M,Bi D G,Jin H. 2015. Molecular characterization and functional analysis of duck TRAF6[J]. Developmental & Comparative Immunology,49(1):1-6. doi:10.1016/j.dci.2014.11. 006.
Zhou S M,Li M,Yang N,Liu S,Yuan X M,Tao Z,Wang G L. 2015. First description and expression analysis of tumor necrosis factor receptor-associated factor 6(TRAF6) from the swimming crab,Portunus trituberculatus[J]. Fish & Shellfish Immunology,45(2):205-220. doi:10.1016/j.fsi.2015.04.005.
(責任编辑 兰宗宝)

