PCNT基因SNP位点rs2070426多态性与食管癌的关系
乔佳欣,宋 昕,徐瑞华,赵学科,魏梦霞,王 苒,韩雪娜,陈亚杰,孙 琳,马琳琳,谢叶真,白合林,罗 宏,李爱丽,王立东
食管癌是上消化道常见的恶性肿瘤,每年新发50万例食管癌患者中一半以上发生在中国[1-2],患者的死亡率排在消化系统肿瘤的第一位[3]。食管癌早期无特异性症状,确诊时大多已达中晚期,5 a生存率仅约20%[4-7],严重威胁着人们的健康。食管癌病因复杂,尚未明确。普遍认为食管癌的发生与胃部食物的反流有关[8-9]。但目前国内外的食管癌因主要发生部位不同而对食管的研究方向也不相同,导致目前对食管癌的诊断没有明确的判断标准。最新研究表明,PCNT SNP位点rs2070426多态性不同方向的突变表达,其对应的蛋白表达量也不同,不同的蛋白表达量食管癌患者的生存也不同,因此蛋白表达在肿瘤发生、进展和侵袭中发挥重要作用。总结得出食管癌中基因的改变与食管癌的预后有一定的相关性[10]。
以上方法的进行需要外显子技术的参与,外显子组测序(whole exome sequencing,WES)是利用探针杂交富集外显子区域的DNA序列,通过高通量测序,发现与蛋白质功能变异相关遗传突变的技术手段。相比于全基因组测序,外显子组测序深度更高,更加经济、高效,能直接对蛋白编码序列进行测序,找出影响蛋白结构的变异。同时,WES能高深度测序,可发现常见变异、低频变异及罕见变异,是非常有价值的一种方法。
综上,本研究提出以下研究目的:探究PCNT SNP位点rs2070426不同基因型患者的生存状况,探究PCNT SNP位点rs2070426不同基因型患者的蛋白表达量。
1 材料与方法
1.1 样本来源
本研究纳入的199例患者均来自于郑州大学第一附属医院省部共建食管癌防治国家重点实验室50万例食管癌患者临床信息数据库,患者来自河南省食管癌高发区,经过确诊并行手术治疗的人群,确诊时间为2019年11月至2020年1月,其中男132例(66.33%),女67例(33.67%)。
1.1.1 样本采集收集199位食管癌患者的癌组织和癌旁组织。均为新鲜的组织样本,在进行手术切除后30 min内进行宏观解剖,保存在-80 ℃下。在样本采集前病人未接受放化疗等其他治疗,以半定量的方式对肿瘤细胞进行视觉评分,仅选择恶性细胞>70%的肿瘤组织进行全基因组外显子测序。癌旁正常组织为距肿瘤边缘至少2 cm远的邻近的上皮正常组织(生殖系对照)。本研究所有患者均签署了独立的知情同意书,均已获得相关机构的审查和批准。
1.1.2 随访本研究通过打电话、家访或联系村医等方法与患者或患者家属直接联系,或者通过新合作医疗数据库、医疗保障局数据库和公民死亡信息登记管理等系统查询联系方式进行随访,每半年进行一次随访,记录死亡时间和主要死因等。本研究随访时间从手术结束开始,截止时间为全因死亡时间,为期2 a,即从随访成功199例开始,全部死亡结束。
1.1.3 样本制备将199例食管癌患者的癌组织和癌旁组织进行配对,分装到准备好的已经标记的冻存管内,样本编号为001~199号。在样本类型中,食管癌组织用字母T标记(tumor),癌旁正常组织用字母N标记(normal)。收集到的样品在液氮中快速冷冻并保存,同时保证每份样品有足够的DNA可供提取。
1.2 WES测序分析
1.2.1 DNA样品检测利用琼脂糖凝胶电泳分析DNA样本是否有蛋白,RNA污染及其降解程度,并用Qubit 3.0测出DNA样品浓度,选择0.6 μg以上的样品进行后续建库。
1.2.2 建库捕获及上机测序用Agilent液相芯片捕获系统对人全外显子区的DNA进行富集,Agilent SureSelect Human All Exon V6试剂盒进行建库及捕获,后在Illumina平台上进行高通量测序。
1.2.3 数据质量控制高通量测序获得的原始图像数据,经过转化后形成原始Raw Data测序序列。将原始Raw Data数据进行过滤,并且控制平均碱基位置测序错误率低于0.1%,样本的测序reads达到95%以上的比对率,且碱基覆盖深度达到10×以上时该点检测的SNP(single nucleotide polymorphisms)比较可信。
1.2.4 SNP检测和功能注释对部分家系成员DNA进行WES测序(由诺禾致源测序公司进行) ,基于WES数据中所检测到的SNP位点,将正常与癌组织中都有的SNP进行比对,使用bcftools软件识别SNP,并利用ANNOVAR软件对多态性位点进行功能注释。
1.3 蛋白质组学分析与鉴定
采用液相色谱—质谱(liquid chromatography-tandem mass spectrometry,LC-MS/MS)技术对199例食管癌的癌组织和癌旁组织样本进行分析。
1.4 SNP与蛋白质组作用关系分析
采用WES测序,将基因中的突变位点和突变位点对应靶点引起的蛋白表达量之间联系起来,进行一定的探讨。
2 结果
2.1 患者rs2070426位点的SNP型
基因分析结果显示,199例食管癌患者中,rs2070426 GG型51例(25.63%);GC型64例(32.16%);CC型84例(42.21%)。
2.2 SNP型与生存和蛋白表达的关系
2.2.1 不同突变位点生存结果收集199例患者的标本,并行WES测序探讨PCNT基因SNP位点rs2070426多态性的3种不同基因型(GG、GC、CC)与生存的关系。在研究中发现是第21号染色体上的一个位点发生了突变,即由原来基因位点上的G变为C,因此可以得出若为CC型则为纯合突变、GC型为杂合突变、而若为GG型则为野生型。通过采用SPSS软件中的Kaplan-Meier分析画图比较,得出在2 a的生存随访中,相同的时间点3种(GG、GC、CC)基因型生存率不同,见表1。发生纯合基因突变的食管癌病人的生存人数最好,杂合突变次之,野生型最差(P=0.033)。但到随访的最终都死亡,均为致癌基因,但发生了纯合突变的人群存活率在相同的时间段内生存率相对较多。由于PCNT的改变与其它疾病或癌的发生之间有一定的关系[11-15],是一个敏感点。但其它文中无对SNP位点rs2070426多态性与食管癌之间的关系进行探讨,因此在本文中对PCNT基因SNP位点rs2070426多态性与癌症之间关系也变得比较有价值。见图1。
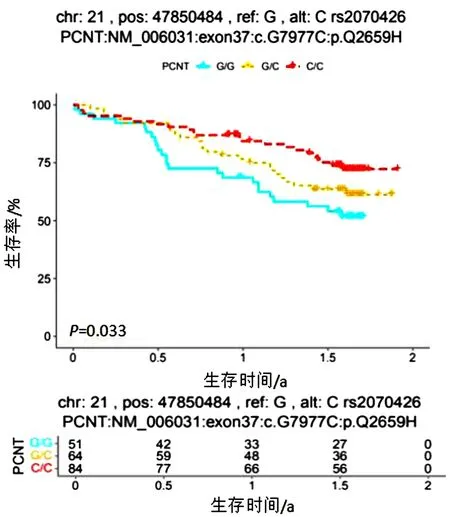
图1 不同突变食管癌患者的生存图
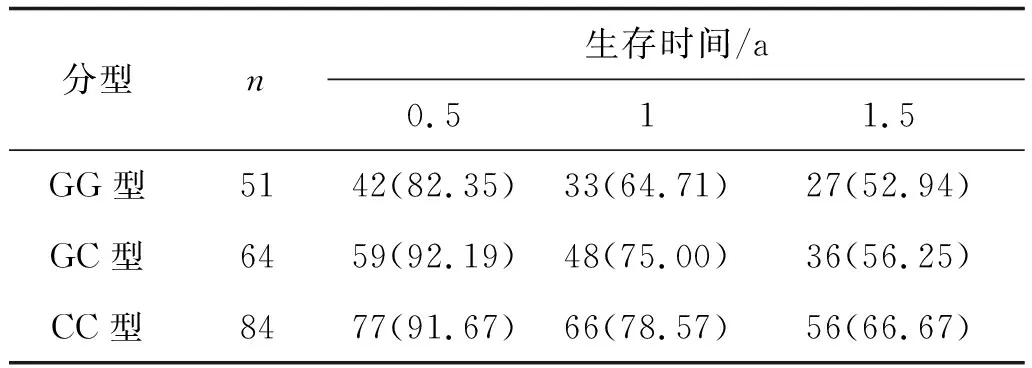
表1 PCNT种基因型与生存率的比较 199 例(%)
2.2.2 不同基因型蛋白表达量的对比SNP位点rs2070426多态性与肿瘤的发生密切相关。由于SNP位点 rs2070426不同基因型的改变,导致了对应的基因型的蛋白表达量也不相同(beta=0.267)。完全突变型CC的蛋白表达平均水平最高,GC型的蛋白表达平均水平次之,而GG型的蛋白表达平均水平最低(P<0.001)。见图2。
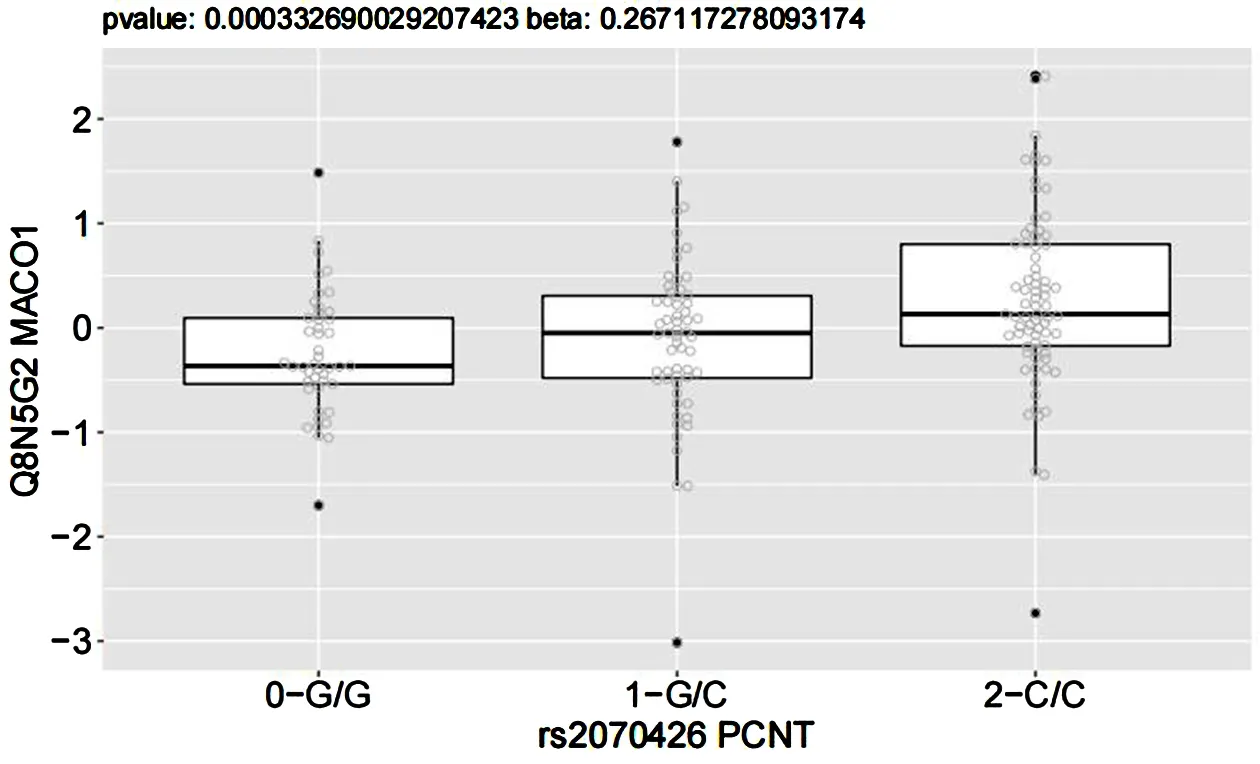
图2 rs2070426位点3种基因型食管癌患者的MACO1蛋白表达
3 讨论
食管癌是常见的一种消化道恶性肿瘤。因其发生率高存活率低,目前提高病人的存活率非常重要,但这一问题至今没有精准的解决方法。
已有研究发现,食管癌的预后生存率,除了与饮食有关系以外,还与内部的基因的改变存在着非常大的关系。因此作者对内部基因方面进行了一定的探讨。本文采用同一批手术相同的时间段内的食管癌患者的SNP位点rs2070426的不同突变基因型存活率多少进行研究。采用WES技术能直接对蛋白编码序列进行测序,找出影响蛋白结构的变异。为下一步的研究提供了帮助。在基因突变探讨的过程中,发现PCNT基因SNP rs2070426位点发生了改变。通过图1探讨中了解到食管癌患者2 a生存率CC型高于GC型与GG型患者,突变等位基因越多,2 a时间段的分析中存活率反而越高。从图2中了解到纯合突变型CC的蛋白表达平均水平最高,GC型的蛋白表达平均水平次之,而GG基因型的蛋白表达平均水平最低(P<0.001)。从两个图中了解到PCNT基因位点的突变是一个好的突变,但其他文章中并未对PCNT SNP rs2070426位点与食管癌生存之间关系进行研究,只探讨了PCNT与其它病的关系[16],是一个敏感点。
因此本文这一研究是非常有意义的。这一基因位点突变引起的蛋白表达改变进而影响生存是通过影响对应的MACO1靶点引起的,虽然其他文章也研究了MACO1与肿瘤的发生有关[17]。本研究对阐明PCNT基因SNP位点rs2070426多态性对应靶点MACO1与蛋白表达量的关系非常有意义。本文针对SNP位点rs2070426,探讨基因位点的改变与食管癌预后生存差异的关系,进而了解其可能治疗靶点,对以后食管癌的研究具有一定意义。是否可以通过此基因的改变延长病人的生命,还需要进一步的研究跟进。
综上所述,食管癌预后的存活率与位点突变数有关。PCNT基因突变的3种位点与蛋白表达量呈现正相关性。从本文中可知这是一个正向突变生存位点。作者将在下一步的研究中深入了解PCNT基因SNP位点rs2070426多态性与食管癌预后的存活关系,为以后的个体化治疗提供一个新的线索,进而开启食管癌研究的新途径,延长食管癌患者的生存时间,这将是一个有价值的研究思路。

