CRISPR/Cas技术在病原体检测中的应用
陈逸凡 综述,尹利民审校
1.昆明医科大学,云南昆明 650000; 2.昆明市第一人民医院检验科,云南昆明 650000
1987年在大肠埃希菌基因组中首次发现了成簇间隔短回文重复序列(CRISPR),具有切割外来入侵病毒核酸的获得性免疫功能[1]。CRISPR及其相关蛋白(CRISPR/Cas)系统是由编码Cas蛋白基因和由启动子加上重复序列与入侵核酸同源的间隔序列交替而构成[2]。该系统的获得性免疫机制可以分为3个阶段:获得/适应、表达、干扰[3]。获得阶段,首次入侵的外来核酸被Cas1-Cas2复合物选择并将其整合到CRISPR序列中,产生免疫记忆。随后表达阶段中外来核酸再次入侵,CRISPR序列被转录成前体CRISPR RNA(pre-crRNA),再由Cas蛋白或以RNA为底物的RNA水解酶(RNases)进一步加工成短的成熟的CRISPR RNAs (crRNA)。最后的干扰阶段,成熟的crRNA引导Cas蛋白进入外来核酸,在其与靶序列结合时,Cas蛋白识别靶序列上的原型间隔序列毗邻基序(PAM)并切割入侵核酸[3],例如Cas9和Cas12。Cas蛋白按照其功能可以分为两大类(Class1、Class2)、六型、四十八个亚型[4]。Class1蛋白主要基于多亚基蛋白复合物负责采集病毒核酸,并产生免疫记忆,而Class2则是单效应蛋白,根据免疫记忆在guideRNA(gRNA)或crRNA引导下切割再次入侵的病毒核酸,从而抵御外源性病毒入侵[5]。Class2包括Cas9、Cas12、Cas13和Cas14,这4种Cas蛋白的性质见表1。其中Cas9作为强大的基因编辑工具被广泛使用,其他则可应用于核酸检测[6]。Cas12、Cas13和Cas14主要凭借它们的“附带切割”能力进行核酸检测,即在crRNA与靶序列结合后激活Cas蛋白,从而对非靶向的核酸片段进行切割使得检测信号放大,达到检测目的。

表1 Class2 Cas蛋白的性质
目前经常应用于临床的病原体检测方法多为基于聚合酶链反应(PCR)的分子检测方法,但是在临床实际应用中仍存在费用昂贵、对环境设备要求高、操作人员专业性要求高等问题。因此,开发出价格低廉、快速准确的病原体检测方法,是当前病原体诊断的主要挑战[7]。基于CRISPR/Cas的病原体检测技术不断发展,以其灵敏度和特异度高、可操作性强等优点,有望成为理想的病原体核酸检测技术。
1 基于靶向DNA的CRISPR/Cas技术
1.1Cas12 Cas12a属于Class2中的Ⅴ型蛋白,也被称为Cpf1,在一个富含T PAM的crRNA引导下识别裂解双链DNA(dsDNA)或单链DNA(ssDNA)[8]。而Cas12a有RuvC结构可以切割靶标和非靶标dsDNA[9],与Cas13一样具有“附带切割”能力。2018年,CHEN等[10]利用LbCas12a与重组酶聚合酶扩增(RPA)技术结合开发出DETECTR平台,成功在阿摩尔灵敏度水平快速检测出人乳头瘤病毒(HPV)16型和18型。该平台不需要将扩增后的DNA产物再转录为RNA,因此,将检测时间缩短到1 h内。同年,LI等[11]利用Cas12a的“附带切割”能力识别并切割非靶向ssDNA,开发出HOLMES核酸检测平台,该平台检测乙脑病毒虽然具有较高的灵敏度和特异度,但其核酸扩增和靶向检测是两个独立的步骤,增加了交叉污染的风险。次年,为改善这一问题,LI等[12]开发出了HOLMESv2平台,将嗜热蛋白Cas12b与环介导等温扩增(LAMP)技术结合,实现了核酸扩增和靶向检测一步化,从而达到简化步骤、避免污染的目的。TENG等[13]同样利用Cas12b开发出了CDetection平台,研究表明该方法可以在1阿摩尔浓度人血浆中检测出HPV DNA的存在,达到亚阿摩尔水平灵敏度和单碱基分辨特异度。WANG等[14]将RPA技术与Cas12a相互分隔集成到一个反应容器中,开发出Cas12aVDet可视化核酸检测平台,先进行RPA反应再加入Cas12a,从而实现一步式检测,可检测出细胞培养中的支原体污染。
除了与各种核酸扩增技术结合,CRISPR/Cas12还可以与各种传感器相结合,进一步拓宽了CRISPR的应用。DAI等[15]开发出了一个基于Cas12a的电化学生物传感器(E-CRISPR),由于该平台为Cas12a在体外反向裂解创造了优越的电化学环境,没有任何酶扩增,所以在HPV16和细小病毒B19检测中检测限达到了皮摩尔水平。E-CRISPR有望成为精准、便携的即时检测诊断系统。
1.2Cas14 2018年有研究发现了Cas14蛋白,并将其作为DETECTR的一个辅助工具,开发出了Cas14-DETECTR[16]。Cas14蛋白由400~700个氨基酸组成,体积只有其他Cas蛋白的一半。它同Cas12、Cas13一样具有“附带切割”能力,能够裂解ssDNA,且它不需要PAM激活。Cas14-DETECTR平台可应用于高保真单核苷酸多态性(SNP)基因分型和高灵敏度单链DNA病原体检测方法的研发,但目前还没有用Cas14-DETECTR平台进行检测病原体的报道[6]。
2 基于靶向RNA的CRISPR/Cas技术
Cas13a属于Class2中的Ⅵ型蛋白,也被称为C2c2,只需要在crRNA的引导下靶向切割靶标单链RNA(ssRNA)或非靶标ssRNA[5]。研究表明,Cas13a还有双重酶活性,能够加工crRNA前体使其成为成熟的crRNA[17]。2017年,GOOTENBERG等[18]将Cas13a与RPA技术结合开发出了SHERLOCK核酸检测系统。该系统原理是dsDNA通过RPA技术进行扩增,扩增后产物用T7聚合酶转录成靶ssRNA,ssRNA与crRNA互补配对,并激活Cas13a进行附带切割报告基团,使其释放荧光信号,见图1。SHERLOCK在检测寨卡病毒和登革热病毒特异株中表现出较高的灵敏度和特异度,标志着CRISPR/Cas诊断技术的正式建立。为改善该系统缺乏量化、依赖荧光检测设备读取结果等局限性,进一步开发出了SHERLOCKv2[19]。首先,SHERLOCKv2同时运用LwaCas13a、CcaCas13b、AsCas12a、PsmCas13b实现四通道多重分析。根据不同crRNA与相应的Cas蛋白结合,切割不同的核酸,建立了4个颜色不同的荧光通道。其次,找到了合适的引物水平,并引入Csm6与Cas13耦联,使灵敏度达到阿摩尔水平,检测信号增强。最后,将结果读取方式改为金标层析格式,将荧光素和淬灭剂换成FAM和生物素(biotin)。当报告RNA没有被切割时,形成金纳米颗粒-FAM抗体-FAM-RNA-biotin耦合物,只出现第一条线。而报告RNA被切割时,形成金纳米颗粒-FAM抗体-FAM释放,进一步出现第二条线。这种方式类似于人绒毛膜促性腺激素(HCG)早孕试纸,检测方便、快速,有助于该系统在非医院环境中的使用。2018年,HUDSON技术被发明[20]。HUDSON技术通过加热法和化学还原法先灭活核酸酶再灭活病毒颗粒,从而使SHERLOCK可直接从患者体液标本中分析采集读取信号。“HUDSON+SHERLOCK”的设计实现了所有反应均在一个容器内进行,不需要提取和纯化,并且结果读取使用荧光或金标均可。这对于病原体实现即时检测有着重要意义。
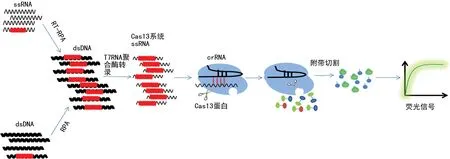
图1 SHERLOCK检测原理示意图
随后,基于Cas13的病原体检测方法的研究被大量报道。LIU等[21]利用LwCas13a的纳米机制和便携式荧光探测器成功快速检出甲型禽流感病毒。QIN等[22]运用Cas13a识别并切割埃博拉病毒RNA后,引入微流控技术开发出了基于CRISPR的自动化多通路微流控检测平台。由于该平台体积小,灵敏度高,没有复杂的操作步骤,因此很适合用于埃博拉病毒流行、资源匮乏的发展中国家。ACKERMAN等[23]将微流体技术引入Cas13检测系统开发出CARMEN-Cas13高通量多通路病原体检测平台。该平台可同时检测169种不同的人类相关病毒和十几种人类免疫缺陷病毒耐药突变株,灵敏度可达阿摩尔水平,与SHERLOCK和基于PCR的方法灵敏度相当。
3 基于靶向DNA或RNA的CRISPR/Cas技术
Cas9蛋白属于Class2的Ⅱ型蛋白,在由反式激活crRNA(tracrRNA)和成熟crRNA整合形成的单向导RNA(sgRNA)引导下,识别靶dsDNA上的PAM并特异性切割dsDNA[24-25]。当靶ssDNA或ssRNA存在起PAM作用的寡核苷酸时,sgRNA/Cas9同样可以切割ssDNA和ssRNA[7]。2016年,PARDEE等[26]将CRISPR/Cas9与核酸依赖性扩增检测(NASBA)技术结合开发出了NASBACC平台,这也是CRISPR/Cas技术在核酸检测中的首次亮相,用于检测非洲和美国寨卡病毒变异体,该平台利用Cas9对寨卡病毒RNA经NABSA扩增并反转录而产生的dsDNA进行特异性切割,再通过纸基传感器实现结果可视化判读。该研究表明NASBACC平台具有检测基因突变的能力,可适应病毒的不断变异[26]。HUANG等[27]利用Cas9切割和NEase介导的核酸扩增开发出一种新的CRISPR/Cas9触发等温指数扩增(CAS-EXPAR)反应,结合SYBR GREEN荧光显色,用于快速、位点特异性核酸检测。该平台能够在阿摩尔水平单碱基分辨率下成功在1 h内检测出李斯特菌。
没有切割活性的Cas9(dCas9)依然具有识别能力,同样可应用于病原体检测。2016年,ZHANG等[28]在成对的两个dCas9上连接荧光素酶,分别位于一个dCas9的N端和另一个dCas9一半的C端。这对dCas9在sgRNA的引导下,识别并结合到靶DNA上下游,使荧光酶重组发出荧光信号。该方法在结核分枝杆菌的检测中表现出良好的灵敏度及特异度。2017年,GUK等[29]采用CRISPR介导DNA-FISH方法检测耐甲氧西林金黄色葡萄球菌,该方法使用dCas9/sgRNA复合物作为靶向材料,SYBRGreenI(SGI)为荧光探针。由于不需要细胞裂解进行基因分离步骤,所以该方法可以在30 min内灵敏地检测靶基因,而且通过改变sgRNA序列从而适用于耐甲氧西林金黄色葡萄球菌以外的任何靶点。2019年,HAJIAN等[30]将CRISPR/Cas9与石墨烯场效应晶体管(gFET)技术结合,开发出了CRISPR-Chip平台。该平台将dCas9固定在石墨烯晶体管上,并与sgRNA形成dRNP复合物,识别结合靶序列并产生电化学信号。该方法不需要扩增技术,简化了检测使用的仪器和操作步骤。
4 总结与展望
上述基于CRISPR/Cas的病原体检测方法,证明了该系统不仅是强大的基因编辑工具,还对病原体分子诊断方法的发展进步作出了巨大贡献。截至2021年6月22日,全球报告新型冠状病毒肺炎病例数量超过1.77亿,死亡人数超过385万[31]。实现COVID-19的快速准确诊断成为控制疫情的关键,但目前基于PCR的检测方法的准确度和效率依赖于标本采集部位是否存在足够数量的病毒,因此,急需开发出一种精确、快速、简便的新型冠状病毒(SARS-CoV-2)检测方法[32]。研究表明,基于CRISPR/Cas方法在病原体核酸检测方面表现出色,一定程度上可以弥补传统PCR检测方法的缺陷,有望成为理想的诊断方法[33]。AZHAR等[34]应用Cas9的同源物FnCas9开发了FELUDA平台,该平台拥有与金标准qRT-PCR相同的准确度,可广泛应用于病原体的单核苷酸变体和核酸序列检测。BROUGHTON等[35]将DETECTR与RT-LAMP技术结合开发出了一种快速可视化的实时RT-PCR替代方法,总用时只需30~40 min,检测限低至10 copy/μL。JOUNG等[36]将SHERLOCK Cas12b同样与LAMP结合开发出STOP平台,用于SARS-CoV-2的检测,被称为STOPCovid。该平台的优势是不需要进行样品提取,并且可以像HCG试纸一样简单可视化读取结果,从而简化流程,减少污染,精准检测。在疫情暴发后,ACKERMAN等[23]在CARMEN-Cas13的冠状病毒板中加入了SARS-CoV-2,该平台用单个mChip可检测400多份标本。这种可扩展的高通量多通路病原体检测平台极大地帮助了传染性疾病的诊断和疫情的防控。
CRISPR/Cas系统在保证其灵敏度和特异度的同时,还具有不需要复杂的核酸提取、检测时间短、易于操作、不需要昂贵的设备和专业的实验环境等优点。尽管CRISPR/Cas系统在病原体检测中表现优秀,但该系统仍然存在有待解决的问题:第一,脱靶效应可导致假阴性结果。脱靶效应指设计的sgRNA会与非靶点DNA序列错配引起非预期的基因突变[37]。脱靶效应主要发生在基于Cas9的检测方法中,通过优化sgRNA的设计和开发高保真的Cas9变体可以有效减少甚至避免脱靶效应。第二,识别靶位点依赖于PAM序列。不同的Cas蛋白识别不同的PAM序列,从而在增加检测系统特异性的同时降低了sgRNA设计的灵活性,增加了实验设计的难度。第三,大多数基于CRISPR/Cas的检测方法都需要核酸扩增技术。有的结合RPA技术,有的运用LAMP技术,虽然较PCR技术简单但是仍阻碍了即时检测的实现。第四,sgRNA易被核酸酶缩短或降解。第五,目前大多数运用该系统的检测方法还无法实现像SHERLOCKv2一样的高通量多病原体检测。
未来应进一步研究和发现更多适用于病原体分子诊断的新Cas蛋白,从而使CRISPR工具更加多样化,同时也可以加入新材料或进一步优化信号放大系统真正实现一步化检测。CRISPR/Cas系统还应进一步与其他技术平台结合,如与计算机领域结合开发出适用于个人通信设备的软件或系统,方便数据的储存与及时上传,从而步入完全的即时检测时代。
总之,基于CRISPR/Cas系统的病原体检测方法虽然存在着一些有待解决的问题,但凭借其方便、快速、准确等优势,它仍然是目前最具潜力的病原体分子诊断工具。

