辣椒基因组密码子使用偏性分析
刘潮 韩利红 吴丽芳 代小波 刘婕









摘要:为揭示辣椒基因组密码子偏性形成的影响因素,了解密码子偏性对基因表达的影响,以辣椒基因组和转录组数据为研究内容,利用CodonW和EMBOSS等软件对基因组密码子使用偏性进行分析。分析结果显示,辣椒编码基因平均GC含量为42.27%,偏好A/T作为密码子的第3位核苷酸,基因平均ENC值偏大;GC3与GC12相关性较弱,大部分基因远离ENC-plot曲线,近50%的基因ENC比值分布在GC3s的0.05~0.15组中;发现21个最优密码子,均以T、A或G结尾,大部分最优密码子在组织器官表达数据中得到验证。表明辣椒基因密码子使用偏性较低,除碱基突变外,较多地受到物种进化和人工选择等因素的影响。研究结果可为辣椒的分子進化和遗传育种研究提供理论依据。
关键词:辣椒;基因组;最优密码子;基因表达
中图分类号: S641.301文献标志码: A
文章编号:1002-1302(2022)05-0016-07
收稿日期:2021-10-16
基金项目:国家自然科学基金(编号:32060710、31860005);云南省地方本科高校基础研究联合专项(编号:202001BA070001-017)。
作者简介:刘 潮(1980—),男,河北景县人,博士,副教授,主要从事植物逆境生物学研究。E-mail:liuchao_80@163.com。
通信作者:韩利红,博士,副教授,主要从事植物谱系地理学研究。E-mail:hanlihong9527@126.com。
密码子使用偏性(codon usage bias,简称CUB)广泛存在于基因组中,反映了密码子在基因编码中的不均匀使用,在基因调控中起重要作用。遗传密码的兼并性不改变蛋白质氨基酸的构成,但同义突变通常会对表型和适应性产生影响。最优密码子的偏好在特定物种基因组中存在整体一致性,往往对应丰度较高的tRNAs,高表达基因的高频密码子比其他密码子丰富度更高,表明CUB与翻译过程存在关联[1-2]。通过对2个果蝇(Drosophila melanogaster)种群同义密码子的偏性分析,发现 10%~20%的高频密码子受到较强的选择作用影响,同时CUB相关的密码子多型性以及可变剪切和转录因子都与选择作用有关[3]。南方菟丝子(Cuscuta australis)的基因组编码基因高频密码子第3位偏好A/T,而最优密码子偏好G/C,这种高频密码子和最优密码子的不一致可能与其寄生生活有关[4]。抗辐射细菌基因组编码基因密码子第3位碱基偏好G和C,这与细菌抗辐射能力有直接关系[5]。农业病原真菌柄锈菌属(Puccinia)的高表达基因偏好使用富含GC的最优密码子[6]。基因组尺度密码子使用性分析将有助于理解物种的进化及其对环境的适应性,对研究物种进化、mRNA翻译和新基因发现都有重要意义[7]。
辣椒(Capsicum annuum)作为全球范围内重要的蔬菜和调味品,具有广阔的开发利用价值。随着辣椒基因组数据的公布[8],有关辣椒抵御胁迫环境的转录组和代谢组研究[9-11] 被大量开展,尤其一些重要功能基因的研究为辣椒品种选育提供了参考[12-13]。利用分子生物学方法开展辣椒关键功能基因的研究具有重要的应用价值,这就需要了解基因组水平上的密码子使用性。然而,目前未见辣椒基因组密码子使用偏性的报道。本研究以公布的辣椒基因组和转录组数据为基础,对辣椒基因组密码子使用偏性及其影响因素进行分析,以了解基因的表达潜力,为辣椒种质资源开发和遗传育种提供借鉴。
1 材料与方法
1.1 数据获取
基因组数据由辣椒基因组数据库(http://peppersequence.genomics.cn/)下载。编码序列(coding sequence,简称CDS)的筛选遵循以下原则:(1)以ATG开始,以终止密码子结束;(2)序列长度大于300 bp[14]。从NCBI网站GEO DataSets数据库下载相应转录组数据(GEO登录号GSE45037)[15]。选取遵辣(ZL1)的根、茎、叶、花蕾和花,以及果实发育的9个时期(F-Dev1~F-Dev9)的基因表达数据。
1.2 分析方法
1.2.1 密码子使用偏性分析 应用CodonW软件和EMBOSS网站的CUSP程序获得CDS序列的密码子使用偏性参数。包括有效密码子数(effective number of codons,简称ENC)、GC含量、A3s、T3s、G3s、C3s、GC3s(第3位同义密码子上碱基的出现频率)和相对同义密码子使用性(relative synonymous codon usage,简称RSCU)。应用Mega X[16]对辣椒基因序列进行核苷酸和氨基酸组成分析。
1.2.2 中性绘图 分别以GC12和GC3作纵坐标和横坐标绘制图,分析密码子使用偏性的影响因素。GC12与GC3相关性越强,密码子使用性受碱基突变的影响越大,反之,受选择压力影响越大。
1.2.3 ENC-plot绘图 ENC期望值计算公式为ENC=2+GC3s+29/[GC3s2+(1-GC3s)2]。ENC比值=(ENC期望值-ENC观察值)/ENC期望值。ENC值越靠近理论曲线,GC3s决定密码子使用性的影响越大,反之,自然选择对密码子使用偏性影响越大[17]。ENC比值越接近零,表明基因密码子受碱基突变的影响越大[18]。
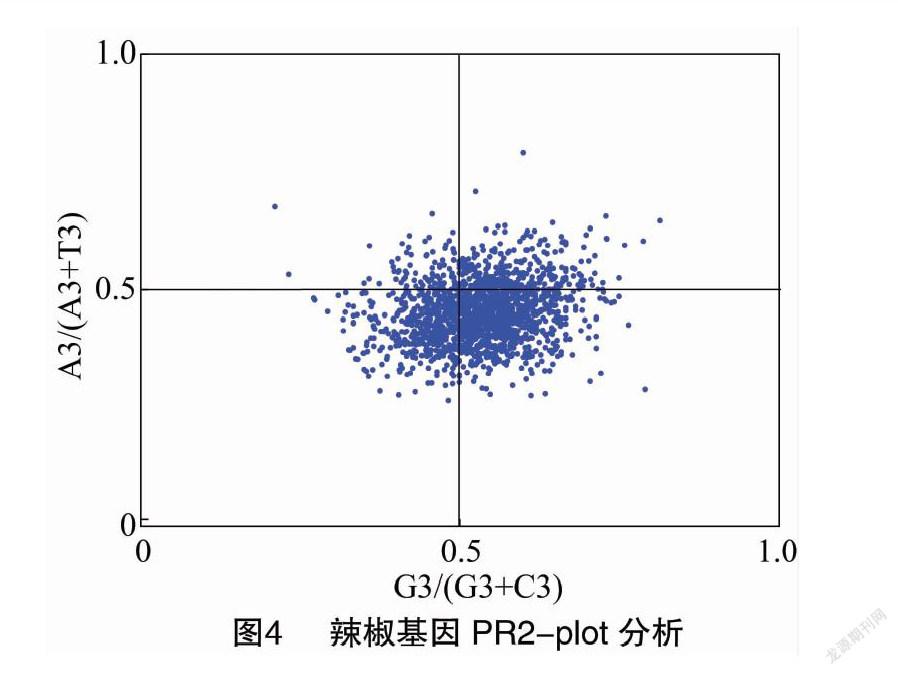
1.2.4 奇偶偏性分析(Parity Rule 2,简称PR2) 分别计算G3/(G3+C3)和A3/(A3+T3)的比值,绘制PR2-plot。数值点的分布中心的坐标靠近05,表明碱基的使用不存在突变或选择偏性 [19]。
1.2.5 对应性分析(correspondence analysis,简称COA) 以59个密码子(除了起始密码子、终止密码子和Trp密码子之外)的RSCU值作为变量,得到每个基因的超维空间分布。不同向量代表每个基因密码子使用模式的相关性。
1.2.6 最优密码子分析 ENC值最低和最高2端各选取5%的基因,分别构成高表达基因(high expression genes,简称HEG)和低表达基因(low expression genes,简称LEG),基因组基因RSCU值≥120时,该密码子定义为高频密码子,如果同时满足ΔRSCU(高表达组RSCU值-低表达组RSCU 值)≥0.20,该密码子定义为最优密码子。
1.2.7 密码子使用性与基因表达的相关性 以NCBI基因表达数据(GEO登录号GSE45037)为依据,从以下2个方面了解CUB与基因表达的关系[4]:(1)序列水平,以ENC值50为界限,将所有序列分为2组;(2)密码子水平,根据密码子第3位的偏好性,将所有序列分为4组。使用各组基因的器官FPKM(fragments per kilobase of transcript per million)的平均值代表基因表达丰度。
2 结果与分析
2.1 密码子使用性
辣椒(Zunla 1 Ref_v1.0)基因组大小为 2 909.15 Mb,GC含量为35.30%[8],包含35 336个基因序列,经筛选后得到34 986个CDS序列用于后续分析。分析显示,辣椒编码基因平均GC含量为42.27%,高于基因组GC含量,而GC3s值与基因组GC含量非常接近,均为35.30%左右(表1)。4种核苷酸中,T3s最高,其次为A3s,而C3s最低,说明辣椒基因偏好A/T作为密码子的第3位核苷酸。编码序列中,平均ENC值为50.79,其中ENC值小于35.00的有233个序列,占比0.67%,大于55的有21 549个序列,占比61.59%,表明辣椒基因密码子使用偏性较低,大部分基因表达潜力较低。
辣椒基因组编码的氨基酸中,亮氨酸、丝氨酸所占比例较高,半胱氨酸、色氨酸所占比例较低(图1)。
2.2 密码子使用性影响因素
中性绘图显示,GC3值介于2.78%~88.89%,GC12值介于14.09%-82.33%,二者分布范围均较广泛(图2)。回归斜率较低(0.06),说明GC3与GC12相关性很弱,突变对密码子第1、2位和第3位碱基使用性的影响不同。表明碱基突变在辣椒基因密码子偏性形成中作用较小。
ENC-GC3s关联分析显示,辣椒基因分布较广泛(图3)。大部分基因分布在标准曲线下方,有部分GC3s值较高和较低基因的ENC值较低,这部分基因远离标准曲线,密码子使用偏性较强。分析显示,近50%的基因ENC比值分布在GC3s的0.05~0.15组中,其次是-0.05~0.05组(表2),表明多数基因ENC观察值与期望值偏差较大,辣椒密码子偏性受GC3s影响较小,密码子偏性受到碱基突变以外的其他因素的影响较大。
PR2-plot用于分析密码子第3位嘧啶和嘌呤的比例关系[19]。多数基因偏向分布在第4象限(图4),说明密码子第3位T的使用频率高于A,G的使用频率高于C,与基因组编码基因第3位上4种核苷酸的分布一致,表明辣椒基因密码子除了受突变影响外,也受到了选择等其他因素的影响。
对应性分析显示,数值较高的第1轴和第2轴分别解释了6.89%和4.00%的变异,各轴占比均较小,各基因均远离0.5的中心位置(图5),表明密码子偏性受到多种因素的影响。A和T结尾的密码子主要分布在第1和第4象限且靠近坐标轴的位置,C结尾的密码子主要分布在第3象限,G结尾的密码子主要分布在第1和第2象限,表明除碱基突变外,密码子的使用偏性更多的受到自然选择等其他因素的影响。
2.3 最优密码子分析
根据ENC值大小,分别选取5%基因构建HEG库和LEG库,并对辣椒全基因组基因、HEG组、 LEG组和各组织器官中高表达基因(分别选取5%比例)的RSCU进行分析(表3)。共发现22个高频密码子,其中21个(除甘氨酸密码子GGA)为最优密码子,分别以T、A或G结尾,其中以T、A和G结尾的分别占66.7%、23.8%和9.5%。分析发现,最优密码子中丙氨酸密码子GCA、半胱氨酸密码子TGT、苯丙氨酸密码子TTT、组氨酸密码子CAT、天冬氨酸密码子AAT、苏氨酸密码子ACA和络氨酸密码子TAT,在各组织器官和发育时期高表达基因中RSCU值略低于基因组总体密码子RSCU值,尤其TGT、TTT和TAT的RSCU值低于1.20,表明这些最优密码子使用偏性相对较弱。而甘氨酸密码子GGA在各组织器官高表达基因和基因组编码基因中RSCU值均大于1.20,然而通过ENC值建库的方法却未能识别为最优密码子。说明仅通过ENC值来鉴定最优密码子的方法存在一定的缺陷,有必要结合转录组数据进行核实和验证。
2.4 密码子使用性与基因表达的关系
基因序列水平上,ENC值小于50的基因有 13 432 个,基因平均表达量为29.89,ENC值大于50的基因有21 554个,基因平均表達量为33.59,这2组基因表达量无显著差异(图6-A),一些ENC值较高的基因,其组织表达水平并不低,表明基于ENC值划分高表达基因和低表达基因存在一定的缺陷。密码子水平上,T3、C3、A3、G3偏好基因所占比例分别为83.0%、1.2%、12.3%和3.5%,其中C3偏好基因的平均表达水平显著较高,T3偏好基因的平均表达水平显著较低(图6-B),这与最优密码子分析结果不一致,可能是4类碱基第3位偏好基因的比例差异导致的,说明最优密码子只解释基因组编码基因的总体表达水平,而不代表单个基因的表达水平。T3偏好基因的平均表达水平较低,可能是大量不表达的假基因拉低了基因的平均表达水平。
3 讨论与结论
密码子使用偏性是生物体偏爱使用某些同义密码子的现象,不同物种甚至同一物种的不同家族基因的密码子使用性存在较大差异[20]。随着大量物种基因组测序项目的完成,密码子使用模式分析的研究引起了全球科学家的极大兴趣[7]。本研究对辣椒编码基因的密码子使用偏性及其影响因素
进行分析,以期为辣椒种质资源的开发及遗传育种提供参考。
碱基组成在植物的进化过程中起着非常重要的作用。生物体因内在代谢过程、外部环境条件以及进化过程事件等的不同形成了不同的核苷酸组成特征[7]。基因GC含量可以影响密码子偏性和氨基酸的组成,对多种植物基因组密码子使用性分析发现,单细胞藻类的GC含量最高,双子叶植物的GC含量最低,密码子使用偏性的程度随物种进化而降低[7]。辣椒基因组编码基因GC含量为4227%,与多数双子叶植物一致,明显低于藻类、禾本科和卷柏等植物,验证了物种间的关系越密切,GC含量越相似的结果[7]。大部分辣椒基因ENC值较大,说明辣椒密码子使用偏性较低。碱基突变和自然选择是基因密码子偏性变异的主要进化力量。在某种核苷酸含量极高的基因组中,突变压力是影响同义密码子使用模式的重要因素[21]。辣椒 ENC-plot、中性绘图、PR2-plot和COA等密码子使用性影响因素分析表明,辣椒基因密码子偏性受到较小的碱基突变影响,可能更多地受到物种进化和人工选择作用等因素的影响。
基因组中,高表达基因优先使用最优密码子以提高翻译效率,而低表达基因使用最优密码子以外的密码子以降低翻译效率[22]。密码子碱基组成影响宿主细胞对外源基因的表达,通过密码子优化,基因表达水平可提高1 000倍以上[23]。不同物种中最优密码子的种类有较大差异,水稻(Oryza sativa)、玉米(Zea mays)等单子叶植物偏好以G或C结尾,而拟南芥(Arabidopsis thaliana)、烟草(Nicotiana tabacum)等主要以A或T结尾,有的也以G或C结尾[24],寄生植物南方菟丝子高表达基因倾向于使用G或C结尾的密码子[4]。随着物种的进化,基因密码子偏性降低,低等植物更需要优化密码子,而高等植物需要较少的密码子优化[7],这是因为与低等植物相比,高等植物的基因表达受到顺式作用元件、转录后调控、mRNA稳定性、选择性剪切等多种因素的调节[25]。辣椒平均ENC值为50.79,显示大部分辣椒基因在序列水平上具有较低的表达潜力,然而,也发现一些ENC值较高的基因,其组织表达水平并不低,说明在高等植物中,基因表达受到多种因素的调节,仅以ENC值高低预测基因表达水平存在较大缺陷。辣椒中21个最优密码子分别以T、A或G结尾,结果与大部分双子叶植物一致。研究也发现,鉴定的最优密码子TGT、TTT和TAT在各組织器官高表达基因的RSCU值并未达到1.20的阈值。因此,对辣椒进行基因工程操作时,密码子优化还需结合转录组数据进行应用。
参考文献:
[1]Qian W F,Yang J R,Pearson N M,et al. Balanced Codon usage optimizes eukaryotic translational efficiency[J]. PLoS Genetics,2012,8(3):e1002603.
[2]Novoa E M,de Pouplana L R. Speeding with control:codon usage,tRNAs,and ribosomes[J]. Trends in Genetics,2012,28(11):574-581.
[3]Machado H E,Lawrie D S,Petrov D A.Pervasive strong selection at the level of codon usage bias in Drosophila melanogaster[J]. Genetics,2020,214(2):511-528.
[4]Liu X Y,Li Y,Ji K K,et al. Genome-wide Codon usage pattern analysis reveals the correlation between Codon usage bias and gene expression in Cuscuta australis[J]. Genomics,2020,112(4):2695-2702.
[5]Dilucca M,Pavlopoulou A,Georgakilas A G,et al. Codon usage bias in radioresistant bacteria[J]. Gene,2020,742:144554.
[6]Roy A,van Staden J.Insights into the riddles of codon usage patterns and codon context signatures in fungal genus Puccinia,a persistent threat to global agriculture[J]. Journal of Cellular Biochemistry,2019,120(12):19555-19566.
[7]Li N,Li Y Y,Zheng C C,et al. Genome-wide comparative analysis of the codon usage patterns in plants[J]. Genes & Genomics,2016,38(8):723-731.
[8]Kim S,Park M,Yeom S I,et al. Genome sequence of the hot pepper provides insights into the evolution of pungency in Capsicum species[J]. Nature Genetics,2014,46(3):270-278.
[9]Kang W H,Sim Y M,Koo N,et al. Transcriptome profiling of abiotic responses to heat,cold,salt,and osmotic stress of Capsicum annuum L.[J]. Scientific Data,2020,7(1):17.
[10]Chiaiese P,Corrado G,Minutolo M,et al. Transcriptional regulation of ascorbic acid during fruit ripening in pepper (Capsicum annuum) varieties with low and high antioxidants content[J]. Plants,2019,8(7):206.
[11]Zhang Y Y,Bouwmeester H J,Kappers I F.Combined transcriptome and metabolome analysis identifies defence responses in spider mite-infested pepper (Capsicum annuum)[J]. Journal of Experimental Botany,2019,71(1):330-343.
[12]Lu B Y,Cheng G X,Zhang Z,et al. CaMYC,A novel transcription factor,regulates anthocyanin biosynthesis in color-leaved pepper (Capsicum annuum L.)[J]. Journal of Plant Growth Regulation,2019,38(2):574-585.
[13]Fan F F,Liu F W,Yang X A,et al. Global analysis of expression profile of members of DnaJ gene families involved in capsaicinoids synthesis in pepper (Capsicum annuum L.)[J]. BMC Plant Biology,2020,20(1):326.
[14]Sharp P M,Cowe E. Synonymous codon usage in Saccharomyces cerevisiae[J]. Yeast,1991,7(7):657-678.
[15]Qin C,Yu C S,Shen Y O,et al. Whole-genome sequencing of cultivated and wild peppers provides insights into Capsicum domestication and specialization[J]. PNAS,2014,111(14):5135-5140.
[16]Kumar S,Stecher G,Li M,et al. MEGA X:molecular evolutionary genetics analysis across computing platforms[J]. Molecular Biology and Evolution,2018,35(6):1547-1549.
[17]Novembre J A.Accounting for background nucleotide composition when measuring codon usage bias[J]. Molecular Biology and Evolution,2002,19(8):1390-1394.
[18]尚明照,劉 方,华金平,等. 陆地棉叶绿体基因组密码子使用偏性的分析[J]. 中国农业科学,2011,44(2):245-253.
[19]Sueoka N.Translation-coupled violation of Parity Rule 2 in human genes is not the cause of heterogeneity of the DNA G+C content of third Codon position[J]. Gene,1999,238(1):53-58.
[20]Haig D,Westoby M.Genomic imprinting in endosperm:its effect on seed development in crosses between species,and between different ploidies of the same species,and its implications for the evolution of apomixis[J]. Philosophical Transactions:Biological Sciences,1991,333(1266):1-13.
[21]Zhao S,Zhang Q,Chen Z H,et al. The factors shaping synonymous codon usage in the genome of Burkholderia mallei[J]. Journal of Genetics and Genomics,2007,34(4):362-372.
[22]Kotlar D,Lavner Y.The action of selection on codon bias in the human genome is related to frequency,complexity,and chronology of amino acids[J]. BMC Genomics,2006,7(1):67.
[23]Gustafsson C,Govindarajan S,Minshull J.Codon bias and heterologous protein expression[J]. Trends in Biotechnology,2004,22(7):346-353.
[24]Kawabe A,Miyashita N T.Patterns of Codon usage bias in three dicot and four monocot plant species[J]. Genes & Genetic Systems,2003,78(5):343-352.
[25]Shrestha A,Khan A,Dey N.Cis-trans engineering:advances and perspectives on customized transcriptional regulation in plants[J]. Molecular Plant,2018,11(7):886-898.

