基于BSA-seq方法定位贵州高粱抗炭疽病害关键遗传区段
陈满静 任艳 彭秋 李青风 高杰 邓小锋



摘要:高粱炭疽病是威胁高粱生长的主要病害之一,挖掘高粱抗炭疽病相关基因能够为高粱抗性品种育种打下基础。利用前期田间试验鉴定出的高粱高抗材料F41和高感材料B57进行杂交构建F2:3代分离群体,挑选高梁高抗炭疽病植株和高感炭疽病植株各30株,分别构建2个极端性状的DNA混合池,利用高通量测序技术与集团分离分析法相结合的方法(BSA-seq)进行全基因组重测序和關联分析,定位和抗性性状相关的基因组区段。通过SNP-index及InDel-index方法进行关联分析及对候选区域进行基因注释,共注释到基因143个,其中非同义突变基因49个,移码突变基因16个。研究结果为高粱抗炭疽病分子机制及抗性相关基因的克隆奠定了理论基础。
关键词:高粱;炭疽病;抗性;候选区段;BSA-seq
中图分类号:S435.14 文献标志码: A
文章编号:1002-1302(2022)05-0028-07
收稿日期:2021-09-08
基金项目:国家科技支撑计划(编号:2014BAD07B02-2-4);黔农科院青年基金( 编号:[2018]57号)。
作者简介:陈满静(1991—),女,贵州三穗人,硕士,助理研究员,主要从事高粱作物栽培技术研究。E-mail:nkycmj@yeah.net。
通信作者:邓小锋,博士,助理研究员,主要从事高粱育种研究。E-mail:dixifor@yeah.net。
高粱[Sorghum bicolor (L.) Moench]作为全球重要的禾谷作物,其耐旱、耐瘠薄的生长优势深受种植人的喜爱。一直以来,高粱主要用于粮食、饲料和酿造加工业,近年来,以高粱为原料开发绿色能源的研究也正在兴起[1]。白酒酿造作为贵州省的主打产业,其原料就是有机酒用高粱,据统计,全贵州省在2018年种植的高粱面积不少于8.67万hm2,其中有机高粱面积占总面积的50%[2]。高粱炭疽病是危害高粱生长的主要病害之一,侵染源是禾生炭疽菌(Colletotrichum sublineolum P. Henn.,Kabát and Bubák)[3-4],它能够侵染高粱所有地上部分组织,包括高粱叶片、茎秆、花序等,病害严重时可发生植株干枯死亡,感病品种生物学产量减产可达67%,在我国北方甚至能减产78%,严重影响高粱的产量和品质[5-7]。该病害易在高温潮湿地区发生,但近年来其流行区域不断扩展,在贵州省的高粱栽种区均有发生。在炭疽病的防治措施中最稳定可靠的方法是选育抗炭疽病高粱品种,但因炭疽病病株存在高度变异的生理小种,增加了抗性资源筛选与抗性选育的难度[8-9]。
为研究高粱抗炭疽病机制,定位抗性相关基因,获得稳定的抗性品种,众多学者对各类高粱抗性材料进行了分子连锁标记和抗性基因定位分析。相关的研究显示,控制高粱抗炭疽病性状的基因有呈显性,也有呈隐性,并且许多材料如 SC414-12E、SC748-5、HC136、Bk7的抗性都受主效数量性状座位(QTL)控制[7,10-15]。通过对这些材料进行遗传分析和连锁分析后得出抗性主效位点,如利用扩增片段长度多态性(AFLP)方法和简单重复序列(SSR)方法对材料SC748-5进行关联标记,得到1个主效位点,位于5号染色体上,基因长度为1.8 cM;又如利用随机扩增多态性DNA(RAPD)方法对抗性材料HC136进行关联标记,得到1个主效位点;再如单核苷酸多态性(SNP)法标记的Bk7中的抗性性状主效位点有2个,位于7号染色体48.7 Mb区间,接近着丝粒;SC414-12E 同样利用SNP标记方法进行关联后,得到3个主效位点,分别位于4、5、7号染色体上。此外,许多学者利用全基因组关联分析(GWAS)方法将抗性位点进行基因组定位[16-18]。如Cuevas等通过GWAS方法分析材料群体SAP,在染色体1号与5号上挖掘出5个与抗性性状相关的位点,位点注释分别为Sobic01G37720葡萄糖醛酸转移酶、Sobic01G379400过氧化物酶、obic05G172300:F-box结构域、Sobic05G182400蛋白酪氨酸激酶、obic05G228400咪唑甲合成[16];Cuevas等对NPGS Sudan core collection群体进行GWAS分析,得到4个抗性为点,位于2号染色体上[17];而在2019年,Cuevas等对NPGS Ethiopian sorghum collection进行分析后得到3个抗性位点,位于9号染色体上,位点注释为Sobic09G012500复制蛋白A:DNA结合亚基内含子区、Sobic09G012900:亮氨酸重复序列、Sobic09G013300:R基因[18]。
集团分离分析法(BSA)技术是能够快速准确地寻找与质量性状基因连锁分子标记的主要途径之一,其方法在不同作物农艺性状相关的基因挖掘上取得了较大进展。曾维英等利用高通量测序技术与集团分离分析法相结合(BSA-seq)的方法挖掘到12个大豆抗豆卷叶螟候选基因[19];张之昊等基于BSA-seq技术定位到了大豆突变体中控制黄622的多小叶的1对不完全显性基因[20]。本研究在前期试验结果的基础上,利用BSA技术快速挖掘贵州高粱抗炭疽病害关键遗传区段,为进一步分子连锁标记开发和育种应用及研究高粱对植物真菌叶部病害的抗性机制奠定基础。
1 材料与方法
1.1 试验材料
利用2019年田间鉴定出来的高抗炭疽病高粱材料F41与高感炭疽病高粱材料B57杂交,得到F2:3群体种子,群体共约500个株系。炭疽菌菌株收集于贵州省惠水县好花红乡试验地,在实验室内分离培养,配制炭疽菌侵染的高粱种子。
1.2 试验方法
1.2.1 构建极端混合池 将F41与B57杂交得到的F2:3群体种子全部播种于贵州省黔南州惠水县好花红乡(26°0′39.29″N,106°34′25.80″E),该区域常年温度高、雨水充沛,适合真菌病害的接种试验。播种时群体内的每个株系种植为1个小区,在拔节期后进行炭疽病病菌接种,2次重复。接种时在每个单株的喇叭口中投4~5粒种子,同时田间种植高感材料作为诱导行,2周后田间观察鉴定,确定高感和高抗材料的纯合株小区,从纯合小区各取30株极端抗性单株叶片和极端感性单株叶片构建子代高抗混合池和高感混合池。对2个亲本和2个混合池进行重测序和关联分析,定位与抗性性状相关联的基因组区段。
1.2.2 混合池DNA提取与测序 采用十六烷基三甲基溴化铵(CTAB)提取法将2个亲本的叶片样品及2个极端混合池的叶片样品的DNA提取出来,将不同群体的DNA逐一等量混合后構建4个混合池,分别是2个亲本(编号为F41、B57)的高抗混合池和高感混合池,待DNA浓度和质量检测合格之后,交由北京百迈克生物科技有限公司进行样品文库构建及上机测序,利用Illunima Casava 1.8进行碱基识别分析,测序参数为双端测序,读长为150 bp,参考基因组为Sorghum_bicolor_JGI_v3.1.1版本的高粱基因组,具体操作流程见图1。
1.2.3 信息分析 将构建的样品文库上机测序得到原始测序数据(raw reads),过滤后得到待分析测序数据(clean reads),将clean reads通过BWA软件与高粱基因组进行比对,根据clean reads的比对结果,利用GATK软件检测和过滤得到检测样品与参考基因组之间高质量的可信SNP位点和序列插入与缺失(InDel)位点。
利用欧式距离法(Euclidean distance,简称ED)和SNP-index法计算与性状关联的候选区域。欧式距离是通过混池间存在的显著差异标记来评估与性状关联区域的方法[21]。SNP-index是通过混池间的基因型频率存在的显著差异标记来进行关联分析的方法[21-22],用Δ(SNP-index)值统计。
通过BLAST软件[23]对SNP和InDel结果的交集区域中可编码基因进行深度注释,准确挖掘高粱抗炭疽病选候选基因区段。
2 结果与分析
2.1 数据质量评估
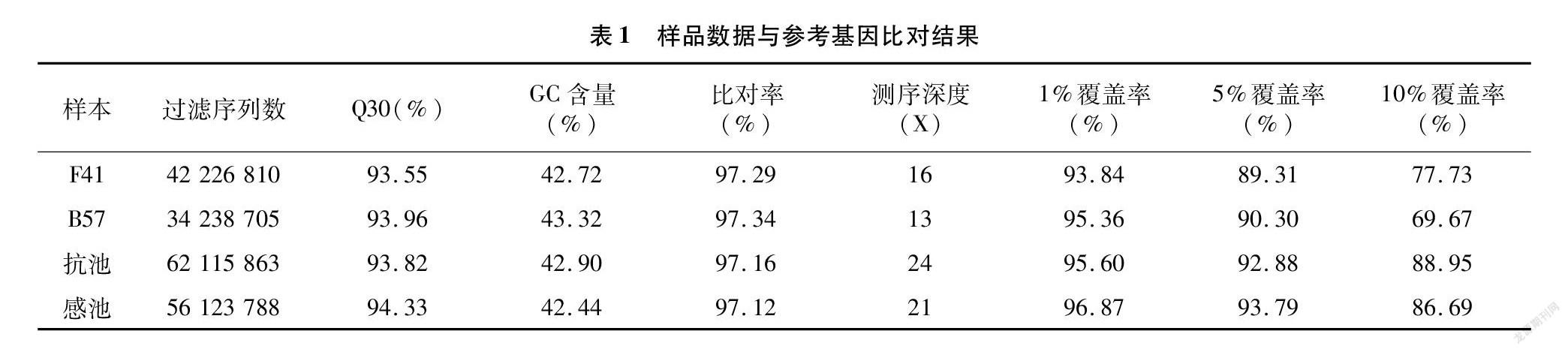
利用Illunima HiSeq高通量测序平台对2个子代极端混合池和2个亲本进行全基因组重测序,过滤后得到40 Gbp高质量的clean reads数据,Q30≥93.55%,GC含量在42.44%~43.32%之间,插入片段长度呈正态分布,样品与参考基因组平均比对效率为97.23%,基因组平均覆盖深度约为18.50X,基因组覆盖率约为95.42%(至少覆盖1X)(表1)。研究结果表明,该数据符合分析标准。
2.2 SNP和InDel变异检测与注释
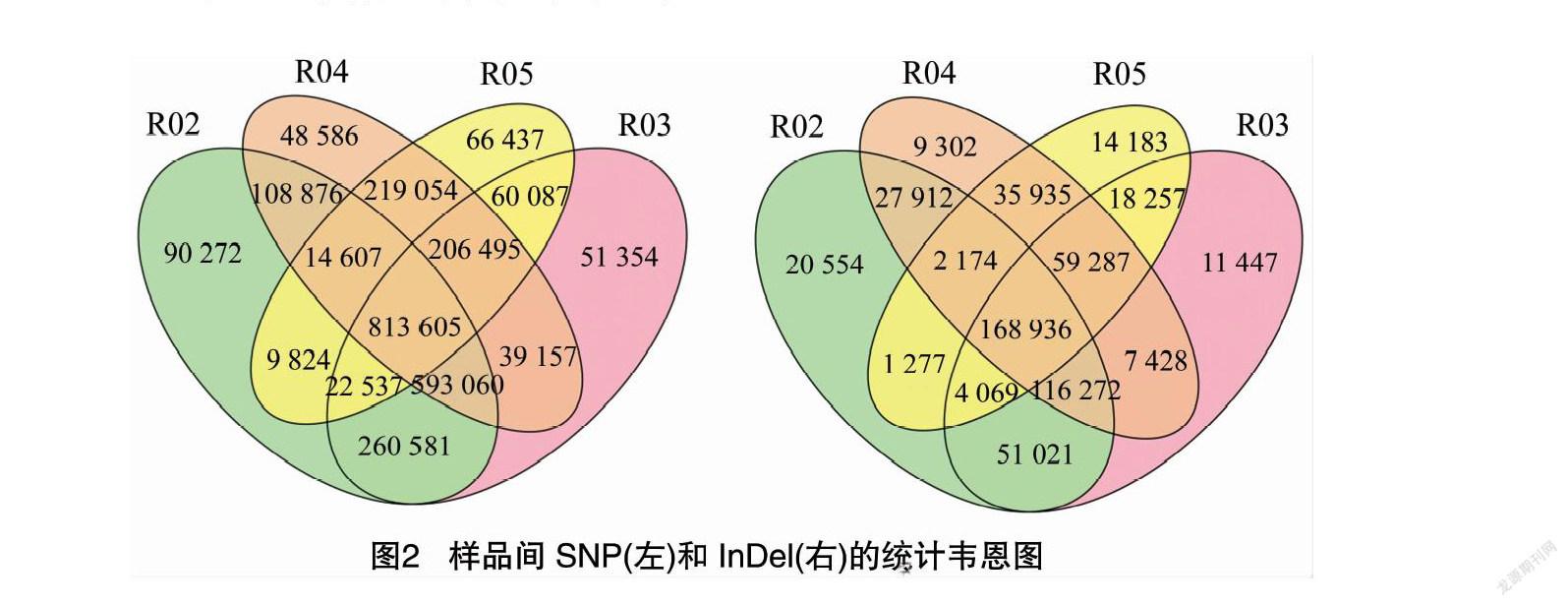
通过SNP检测分析,亲本之间共获得1 520 997个SNP,其中非同义突变的SNP共29 661个,混合池之间共获得1 095 653个SNP,引起非同义突变的SNP共19 086个;通过InDel检测,亲本之间共获得323 821个小片段插入与缺失(small InDel);混合池之间共获得229 420个small InDel(图2)。
2.3 SNP关联分析
对SNP进行过滤之后得到质量高的可信SNP位点754 261个。采用ED算法和SNP-index算法对这些位点数据进行关联分析。
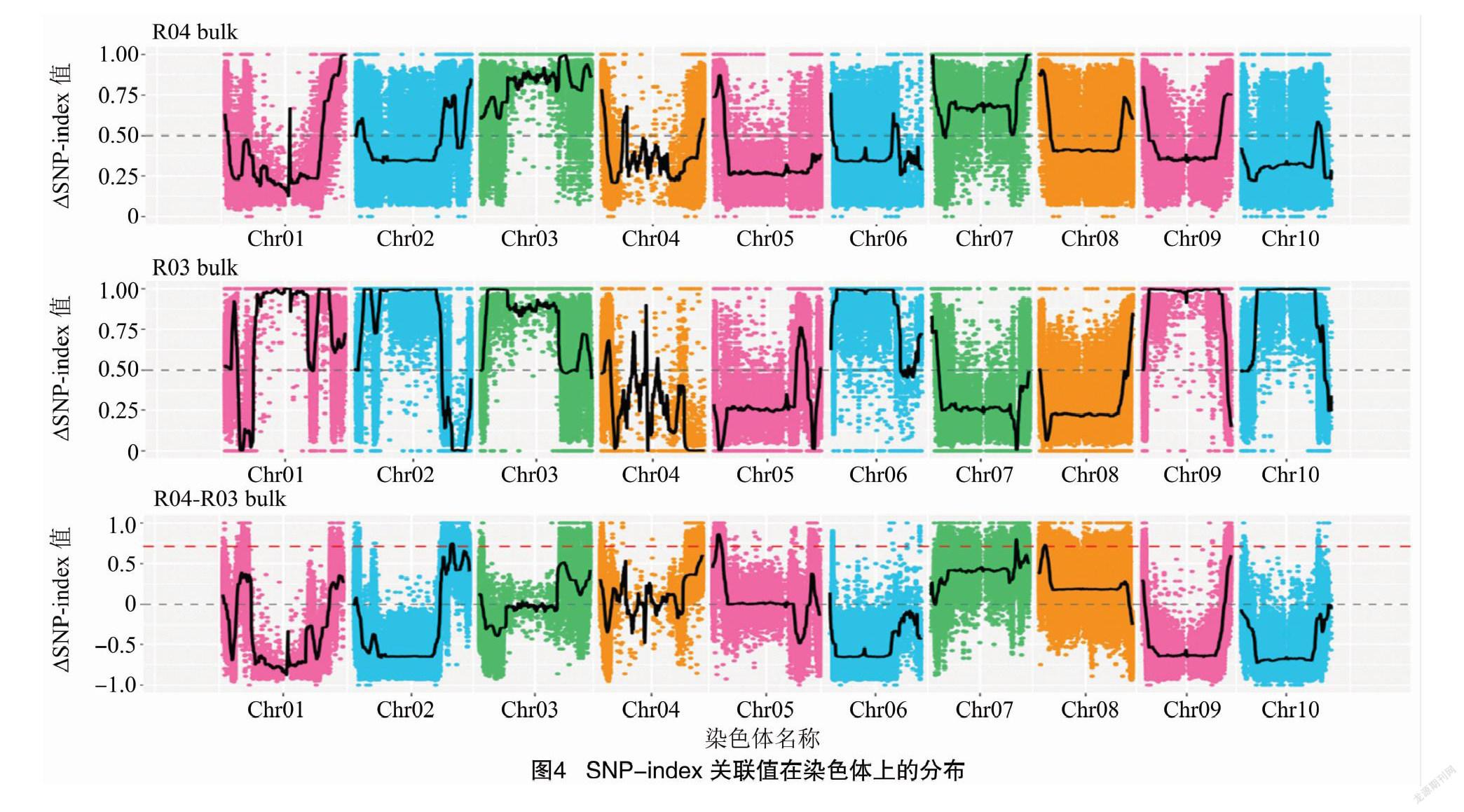
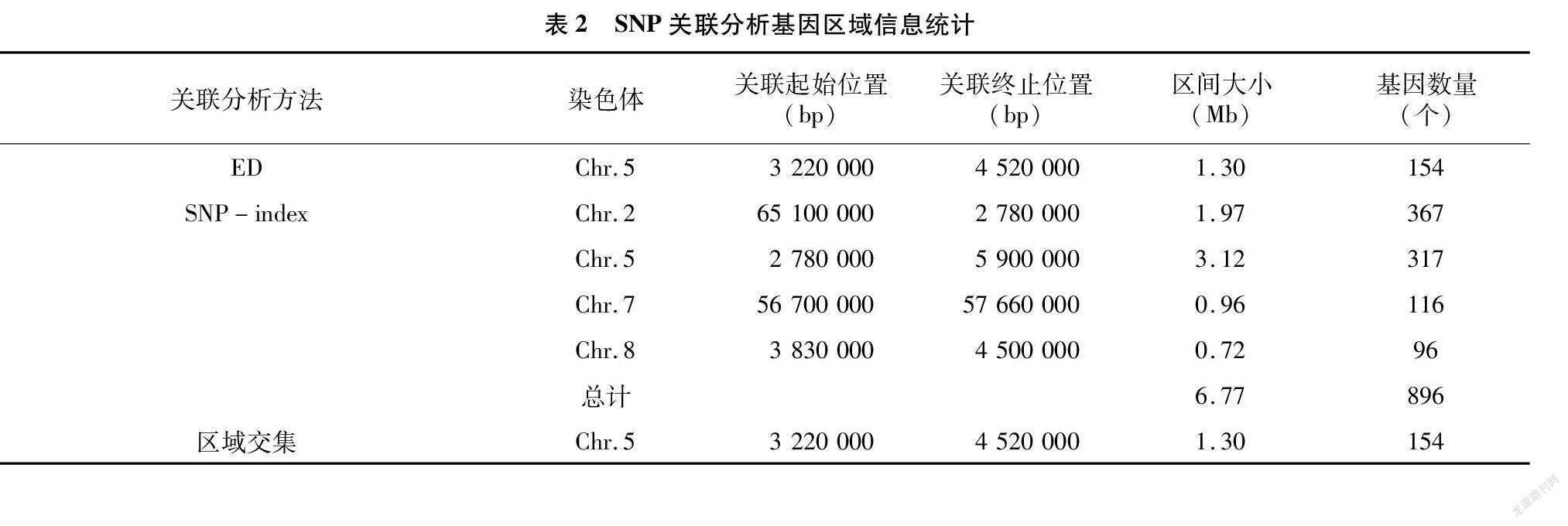
根据ED算法关联阈值判定,共得到2个与高粱抗炭疽病相关联的染色体区域,关联值分布见图3,区域所注释到的基因总长度为1.30 Mb,共包含154个基因,其中非同义突变位点的基因共49个;根据SNP-index算法关联阈值判定,共得到4个与高粱抗炭疽病相关联的染色体区域,关联分布见图4,区域注释到的基因总长度为6.77 Mb,共包含896个基因,其中非同义突变位点的基因共179个。2种关联分析方法取交集,得到1个交集区域,区域总长度为1.30 Mb,共包含154个基因(表2)。
2.4 InDel关联分析
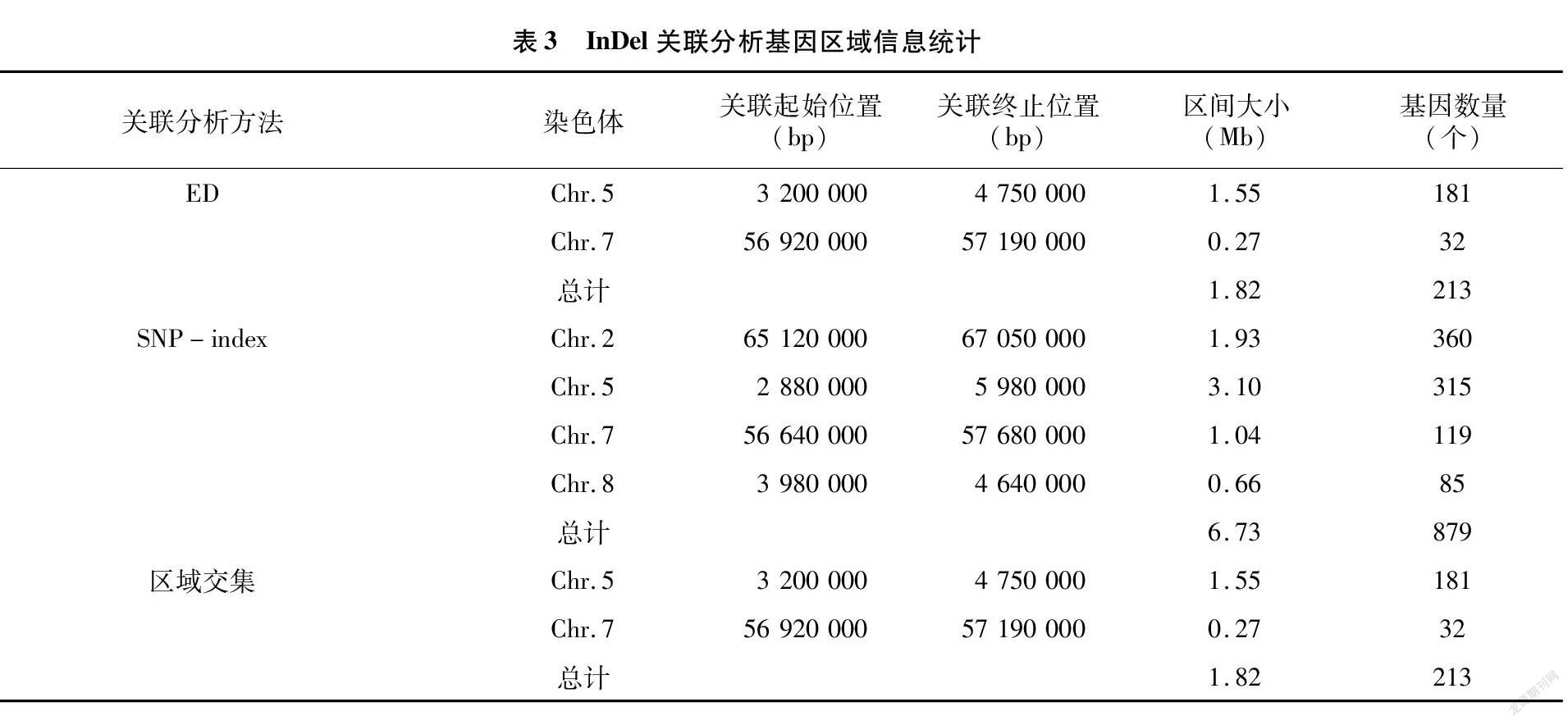
对InDel位点进行过滤,得到质量高的可信InDel位点170 725个。同样采用ED算法和SNP-index算法对这些位点数据进行关联。根据ED算法和 SNP-Index 算法分析得到的结果取交集,共得到2个区域,区域总长度为1.82 Mb,共包含基因213个(表3)。
2.5 候选区段筛选与基因注释
本研究将SNP关联分析结果与InDel关联结果进行分析统计后取交集,最终锁定高粱抗炭疽病的候选区域在5号染色体上,候选区域基因全长为 1.30 Mb,共有基因154个。利用GO[24]、KEGG[25]等数据库对候选区间内的基因进行注释后,共143个基因被注释,其中非同义突变基因有49个,移码突变基因有16个。
2.5.1 GO数据库分析 Gene Ontology(GO)数据库能够将全世界所有与定位基因有关的研究结果进行汇总,利用GO数据库可以在生物学进程、分子功能和细胞组分3个方面对基因和基因产物进行分类注释。为明确候选基因在细胞上的位置、分子的功能和参与生物学进程,将候选区域154个基因进行GO功能注释,有86个基因分别被注释到10个细胞组分、7个分子功能和14个生物学进程中,分类结果见图5。基因在细胞组分中主要存在于细胞膜以及细胞器膜中;基因在分子功能中主要发挥催化、转录以及信号传递等功能;在基因生物学进程中主要参与免疫过程、代谢过程与生物调节等。结合以上结果,说明高粱炭疽病胁迫下高粱的抗病响应可能有生物膜修复、逆境中相关的催化代谢、细胞调节与虫害信息的传递等。
2.5.2 KEGG数据库分析 生物体中的生物学功能是由基因之间的相互协调来完成的,代谢通路(Pathway)代表不同基因间相同的作用通路,而KEGG是收集Pathway的公共数据库。为了进一步解读候选基因的功能,利用KEGG数据库对候选基因区段进行Pathway富集分析,154个候选基因被注释到的基因数为30个,这30个基因所参与的代谢通路主要为植物激素信号转导、核糖体合成、核苷酸合成、RNA降解与碳代谢等(图6)。表明当高粱受到炭疽病病菌侵害时,植物会出现传导病害信号的响应,体内合成代谢增加,产生各种蛋白酶与核酸等物质,同时进行RNA等分解代谢抵御炭疽病病菌的入侵。
3 结论与讨论
本试验利用BSA-seq方法快速挖掘到高粱抗炭疽病关键遗传区段,通过对材料全基因组重测序共挖掘得到1个与抗性性状相关的候选区域,位于5号染色体上,关联区域总长度为1.30 Mb,对候选区域内的基因进行生物信息学分析后,共注释到基因143个,经过多个数据库的分析发现,这一关键遗传区段内的基因可能在高粱抗炭疽病害过程中发挥着重要作用,为后续抗性基因的克隆与分子研究奠定了基础。
前人通过连锁分析研究显示,高粱抗炭疽病的主效位点位于4、5、7、9号染色体上[25-27]。如高抗材料SC748-5的抗性主效位点位于5号染色体上约42.5 cM(60.77 Mbp)的位置,材料SC112-14的主效位点位于5号染色体上55.0~56.1 cM区间内,材料SC414-12E的1个抗性相关位点位于5号染色体上116.90~118.10 cM区间内。此外全基因组关联分析也显示高粱抗炭疽病的一些抗性基因位于5号染色体上65 193 948、66 491 767、71 578 176、65 136 879~65 137 882 bp位置上[18,28]。
本研究结果显示,高粱抗性材料F41的抗性位点位于5号染色体上3 200 000~4 750 000 bp区间内,不同于以上任何抗性位点或基因的位置。同时,已报道的高粱5号染色体上抗炭疽病的抗性基因富集区域位于53.81~62.16、64.53~66.87 Mbp区间[27,29],因此F41的位点是一个新发现的炭疽病抗性位点,其中的相关基因是新发现的抗性相关基因。该新位点的发现为研究炭疽病抗性机制及新抗性基因聚合奠定了基础。
本研究利用BSA-seq方法挖掘到了与高粱抗炭疽病相关的遗传基因区段,但未进一步对候选区段进行基因筛选。后续试验中,将结合转录组测序及大群体连锁分析对候选区段基因进行鉴定,定位到具体抗性基因。
参考文献:
[1]邓小锋,陈满静,曹绍书,等. 贵州糯质高粱GBSSI基因型的鉴定[J]. 贵州农业科学,2020,48(5):13-18.
[2]王小波. 贵州酱香型白酒用粱状况及对策分析[C]//谷粮商务信息网,贵州省农业科学院旱粮研究所.谷粮网2019年第六届中国高粱产业高峰论坛会刊集.仁怀:中国高粱产业高峰论坛,2019:34-39.
[3]徐秀德,刘志恒. 高粱病虫害原色图鉴[M]. 北京:中国农业科学技术出版社,2013.
[4]Erpelding J E,Prom L K. Variation for anthracnose resistance within the Sorghum germplasm collection from Mozambique,Africa[J]. Plant Pathology Journal,2006,5(1):28-34.
[5]Thomas M D. Development of leaf anthracnose and its effect on yield and grain weight of Sorghum in west Africa[J]. Plant Disease,1996,80(2):151.
[6]徐 婧,姜 鈺,胡 兰,等. 高粱抗炭疽病资源筛选及病情与产量损失的关系[J]. 中国农业科学,2019,52(22):4079-4087.
[7]邓小锋,彭 秋,李青风,等. 高粱炭疽病抗性机理研究进展[J]. 贵州农业科学,2019,47(11):68-74.
[8]Chala A,Tronsmo A M,Brurberg M B. Genetic differentiation and gene flow in Colletotrichum sublineolum in Ethiopia,the centre of origin and diversity of Sorghum,as revealed by AFLP analysis[J]. Plant Pathology,2011,60(3):474-482.
[9]Felderhoff T J,McIntyre L M,Saballos A,et al. Using genotyping by sequencing to map two novel anthracnose resistance loci in Sorghum bicolor[J]. Genes Genomes Genetics,2016,6(7):1935-1946.
[10]Erpeldi J E,Prom L K. Evaluation of Malian Sorghum germplasm for resistance against anthracnose[J]. Plant Pathology Journal,2004,3(2):65-71.
[11]Erpelding J E,Wang M L. Response to anthracnose infection for a random selection of Sorghum germplasm[J]. Plant Pathology Journal,2007,6(2):127-133.
[12]Prom L K,Erpelding J E,Montes-Garcia N. Chinese Sorghum germplasm evaluated for resistance to downy mildew and anthracnose[J]. Communications in Biometry and Crop Science,2007,2(1):26-31.
[13]Erpelding J E. Field evaluation of anthracnose disease response for the Sorghum germplasm collection from the Kayes region of Mali[J]. Tropical and Subtropical Agroecosystems,2008(8):291-296.
[14]Erpelding J E. Anthracnose disease response for photoperiod-insensitive Ethiopian germplasm from the US sorghum collection[J]. World Journal of Agricultural Science,2009,5(6):707-713.
[15]Singh M,Chaudhary K,Boora K S.RAPD-based SCAR marker SCA 12 linked to recessive gene conferring resistance to anthracnose in sorghum[Sorghum bicolor (L.) Moench][J]. Theoretical and Applied Genetics,2006,114(1):187-192.
[16]Cuevas H E,Prom L K,Rosa-Valentin G.Population structure of the NPGS Senegalese sorghum collection and its evaluation to identify new disease resistant genes[J]. PLoS One,2018,13(2):e0191877.
[17]Cuevas H E,Prom L K,Cruet-Burgos C M. Genome-wide association mapping of anthracnose (Colletotrichum sublineolum) resistance in NPGS Ethiopian sorghum germplasm[J]. Genes Genomes Genetics,2019,9(9):2879-2885.
[18]Cuevas H E,Prom L K.Evaluation of genetic diversity,agronomic traits,and anthracnose resistance in the NPGS Sudan sorghum core collection[J]. BMC Genomics,2020,21(1):88.
[19]曾維英,赖振光,孙祖东,等. 基于BSA-Seq和RNA-Seq方法鉴定大豆抗豆卷叶螟候选基因[J]. 作物学报,2021,47(8):1460-1471.
[20]张之昊,王 俊,刘章雄,等. 基于BSA-Seq技术挖掘大豆中黄622的多小叶基因[J]. 作物学报,2020,46(12):1839-1849.
[21]Hill J T,Demarest B L,Bisgrove B W,et al. MMAPPR:mutation mapping analysis pipeline for pooled RNA-seq[J]. Genome Research,2013,23(4):687-697.
[22]Fekih R,Takagi H,Tamiru M,et al. MutMap+:genetic mapping and mutant identification without crossing in rice[J]. PLoS One,2013,8(7):e68529.
[23]Altschul S F,Madden T L,Schffer A A,et al. Gapped BLAST and PSI-BLAST:a new generation of protein database search programs[J]. Nucleic Acids Research,1997,25(17):3389-3402.
[24]Ashburner M,Ball C A,Blake J A,et al. Gene Ontology:tool for the unification of biology[J]. Nature Genetics,2000,25(1):25-29.
[25]Kanehisa M,Goto S,Kawashima S,et al. The KEGG resource for deciphering the genome[J]. Nucleic Acids Research,2004,32:277-280.
[26]Cuevas H E,Prom L K,Erpelding J E.Inheritance and molecular mapping of anthracnose resistance genes present in sorghum line SC112-14[J]. Molecular Breeding,2014,34(4):1943-1953.
[27]Burrell A M,Sharma A,Patil N Y,et al. Sequencing of an anthracnose-resistant sorghum genotype and mapping of a major QTL reveal strong candidate genes for anthracnose resistance[J]. Crop Science,2015,55(2):790-799.
[28]Cuevas H E,Prom L K,Cooper E A,et al. Genome-wide association mapping of anthracnose (Colletotrichum sublineolum) resistance in the US sorghum association panel[J]. The Plant Genome,2018,11(2):99-112.
[29]Patil N Y,Klein R R,Williams C L,et al. Quantitative trait loci associated with anthracnose resistance in sorghum[J]. Crop Science,2017,57(2):877-890.

