铅黄肠球菌CMCC(B) 32220的鉴定和全基因组分析*
石继春,陈驰,梁丽,杜宗利,龙新星,郑锐,孙文媛,徐颖华,叶强,(.中国食品药品检定研究院 国家卫生健康委员会生物技术产品检定方法及其标准化重点实验室,北京069;.中国医学细菌保藏管理中心,北京069)
铅黄肠球菌最早由Mundt和Graham于1968年分离获得,并依据其形态和生长特性命名为屎链球菌铅黄亚种[1],后经DNA分子杂交证实不同于屎链球菌,建议新分类为铅黄肠球菌[2]。可能由于以前命名不规范和鉴定手段限制等原因,在中国医学细菌保藏管理中心早期根据生长形态和生化反应等传统方法鉴定而收录的几株屎肠球菌标准菌株中,发现1株菌株CMCC(B) 32220的16SrRNA分析比对结果不符合屎肠球菌。为了更加全面鉴定该菌株,本研究应用全自动生化微生物鉴定、16SrRNA基因序列分析、质谱分析以及全基因组测序方法对CMCC(B) 32220进行分析和鉴定,并探讨分析其基因组基本特征,预测基因功能、耐药性及致病性。
1 材料和方法
1.1菌株来源 待鉴定菌株CMCC(B) 32220和对照菌株铅黄肠球菌CMCC(B) 32240(ATCC编号为700327)来源于中国食品药品检定研究院中国医学细菌保藏管理中心。
1.2主要仪器和试剂 细菌培养箱(北京中仪国科科技有限公司),Vitek全自动微生物鉴定仪(法国生物梅里埃公司),Bruker MALDI Biotyper系统(德国Bruker Daltonics公司)。2%含糖牛肉及其琼脂培养基(北京三药公司),DNA提取试剂盒(德国Qiagen公司),其他试剂均为国产分析纯。
1.3形态学与生化鉴定 将待鉴定菌株CMCC(B) 32220和阳性对照菌株CMCC(B) 32240接种于2%含糖牛肉培养基复苏后[3],再转至琼脂平板传代培养12 h,观察菌落形态,取新鲜培养物染色镜检并应用Vitek全自动生化鉴定仪进行分析。
1.4基质辅助激光解吸电离飞行时间质谱(MALDI-TOF MS)分析 参考文献[3],选取待鉴定菌株CMCC(B) 32220和阳性对照菌株CMCC(B) 32240新鲜培养的菌落,直接涂抹于MALDI靶板上,晾干后滴加1 μL 70%甲酸,待晾干后滴加1 μL基质液,然后待靶板晾干后上机检测,最终用仪器自带Biotyper软件对结果谱图分析。
1.516SrRNA基因序列分析 按照试剂盒说明书将CMCC(B) 32220菌株新鲜过夜培养物进行全基因组DNA提取,并使用通用引物27F(5′-AGAGTTTGATCMTGGCTCAG-3′) 和1492R (5′-GGYTACCTTGTTACGACTT-3′)进行扩增和测序(由北京中美泰和公司完成),应用DNAstar软件将菌株16SrRNA基因序列拼接结果与美国NCBI数据库保存肠球菌属不同种的菌株序列进行Blast比对分析和聚类分析。
1.6细菌全基因组序列测定与生物信息学分析 参考文献[3],使用Illumina公司Hiseq 2500测序仪进行全基因组测序,对获得下机数据应用软件velvet V1.2.03进行拼装,并结合软件glimmer 3.02进行基因预测、KEGG蛋白数据库构建代谢通路以及SEED蛋白数据库进行基因功能注释与直系同源簇(COG)分类。同时CMCC(B) 32220拼接全基因组序列与已发表的铅黄肠球菌EC20(NCBI登录号:NC_020995.1)、屎肠球菌ATCC 8459(NCBI登录号:ASM33640v1)、鸡肠球菌(NCBI登录号:ASM1764196v1)和粪肠球菌(NCBI登录号:ASM331981v1)代表性菌株基因组进行配对基因组的平均核苷酸一致性(ANI)和同源蛋白匹配比对分析。参考文献[4-5],应用CARD耐药抗性数据库在线工具(https://card.mcmaster.ca/)和 PathogenFinder软件对CMCC(B) 32220基因组进行细菌耐药性和致病性预测分析。
2 结果
2.1菌株的形态学和生化鉴定 CMCC(B) 32220在含糖琼脂平板上菌落呈现透明、边缘整齐、中间略隆起。革兰染色为阳性球菌。Vitek全自动生化鉴定仪鉴定待鉴定菌株CMCC(B) 32220和阳性对照菌株CMCC(B) 32240均为铅黄肠球菌。
2.2MALDI-TOF MS鉴定 待鉴定菌株CMCC(B) 32220和阳性对照菌株CMCC(B) 32240经MALDI-TOF MS分析呈现相似的质谱特征峰,鉴定分析结果均为铅黄肠球菌,系统分值分别为1.8和2.0,均大于1.7,表明分析结果可靠。
2.316SrRNA分析 将CMCC(B) 32220菌株16SrRNA测序结果进行拼接,然后应用DNAstar软件比对分析发现,与NCBI数据库中收录的2株代表性铅黄肠球菌(NCBI登录号为LC375241和AJ301832)的16SrRNA序列相似度分别为99.4%和98.9%,而与鸡肠球菌(MN055932)的同源性也高达98.7%,与其他肠球菌菌株之间的16SrRNA序列相似度也超过94%。例如,与屎肠球菌、粪肠球菌和E.columbae的16SrRNA序列相似度分别为96.4%、96.9%和94.8%。将其与NCBI收录其他16种肠球菌属菌株进行系统发育进化分析,结果显示CMCC(B) 32220与另1株铅黄肠球菌以及E.flavescens与鸡肠球菌菌株共属于一个亚分支。而与屎肠球菌、粪肠球菌的进化稍有不同。见图1。
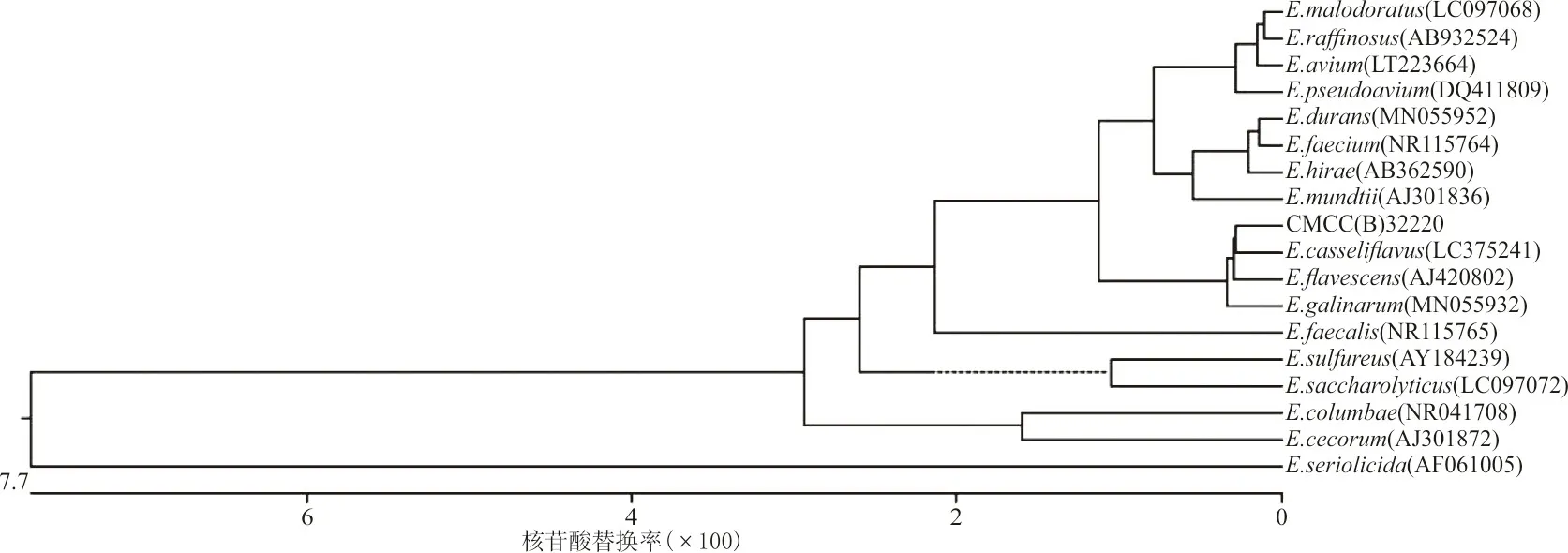
图1 铅黄肠球菌CMCC(B) 32220与NCBI数据库中不同肠球菌属菌株的16S rRNA基因序列系统进化分析
2.4菌株全基因组的特征 高通量测序分析显示,CMCC(B) 32220菌株共获得4 433 333个读长片段,覆盖其基因组大小416倍,拼装后获得32个contig片段,N50大小为254 987 bp。经拼接比对分析发现CMCC(B) 32220染色体全基因组序列全长为3 195 952 bp,预测共有3 054个基因,平均基因大小为905 bp编码基因,占整个基因组序列的86.5%,其平均GC含量为43.8%。而非编码基因间区域大小为430 669 bp,平均GC含量为36.8%,同时鉴定出50个tRNA与3个rRNA序列。
2.5基因组功能分析 COG数据库注释分析结果证实,CMCC(B)32220菌株基因组中具有COG功能注释的蛋白质有2 453个,进一步分析发现负责细菌氨基酸与碳水化合物转运与代谢的蛋白质数量高达366个,占14.9%,其次为负责细菌能力产生和转化的蛋白质为225个。此外,仍有282个(占11.5%)未知功能蛋白质无法获得功能注释。
微生物基因组的KEGG功能分类包括涉及细胞内、环境信息处理以及遗传信息处理过程、代谢和有机系统。KEGG数据库构建代谢通路,分析表明CMCC(B) 32220菌株中1 622个蛋白质具有KEGG的ortholog。其中,代谢类基因数量最多,占KEGG注释总基因数的54.8%;其次为环境信息处理过程基因数,占18.2%。
2.6比较基因组学分析 将CMCC(B)32220菌株基因组的预测蛋白质进行同源蛋白质匹配分析,结果发现相匹配的同源蛋白质来源于95个不同细菌菌株,多数(97.2%)同源蛋白质集中在肠球菌,但其中以铅黄肠球菌特异性同源蛋白质最高,为2 772个(93.7%),进一步分析证实其与铅黄肠球菌EC30菌株同源性蛋白质比例最高,为37.9%。
基于全基因组序列的ANI分析发现,CMCC(B)32220菌株与铅黄肠球菌代表性EC20菌株的基因组的ANI数值为95.8%,但与屎肠球菌及粪肠球菌基因组的ANI数值仅分别为72.4%和71.3%,而与鸡肠球菌基因组的ANI数值为75.3%。
2.7耐药性和致病性预测分析 与CARD耐药抗性数据库收录数据比较分析显示,CMCC(B) 32220基因组仅含有vanRC和vanXYC2个耐药基因,预测分析显示与糖肽类耐药抗性相关。
细菌的致病性预测分析结果发现,CMCC(B) 32220基因组含有5种对人致病性相关蛋白质,并获得匹配蛋白质详细的种属来源以及相似度,见表1。

表1 铅黄肠球菌CMCC(B) 32220菌株致病性预测分析
3 讨论
微生物菌种保藏管理中心提供的标准菌种,是保证微生物实验室检验结果准确、有效的物质基础。本研究应用多种不同原理的微生物鉴定方法,证实CMCC(B) 32220为铅黄肠球菌。传统的生化反应不容易将铅黄肠球菌和屎肠球菌区分,而随着技术的发展,Vitek 2全自动细菌鉴定分析系统可通过高达30项生化反应分析,自动给出最终鉴定报告。但该分析系统要求细菌为新鲜培养物,耗时较长,约5 h[6]。尽管本研究应用该系统将铅黄肠球菌成功鉴定,但研究报道Vitek 2系统通常对部分细菌,例如沙门菌、部分肠球菌等,仅能鉴定到属水平[6]。而MALDI-TOF MS是基于细菌菌体内蛋白质特征性的模式峰进行菌株的鉴定。从准备菌株到鉴定结束, 不到5 min即可获得结果,鉴定时效最强[5]。近期研究人员应用MALDI-TOF MS技术直接从300份清洁尿沉渣样本中,成功鉴定出包括2株粪肠球菌、15株大肠埃希菌和1株产气肠杆菌等25种细菌,与细菌培养法结果100%符合[7]。
由于16SrRNA基因序列在细菌基因组中的保守性、存在的普遍性与稳定性,被广泛用于细菌微生物分类鉴定和检测[6,8]。在肠球菌属中,部分群组内的菌种16SrRNA基因序列的同源性非常高,本研究发现CMCC(B) 32220与其他肠球菌的16SrRNA序列可高达98.7%,建议肠球菌属16SrRNA基因序列分析仅能达到属的水平,用于种的鉴定时需要结合其他分析结果。随着生物技术的发展,近10年测序成本显著降低,应用全基因组序列进行微生物鉴定与分类比单个特征基因序列分析具有更高准确性。大量研究证实当2株细菌之间的ANI值≥95%,通常认为这2株为同种细菌[9]。本研究发现CMCC(B)32220与NCBI收录铅黄肠球菌代表性EC20菌株基因组的ANI值为95.8%,而与其他肠球菌基因组ANI值均低于80%,这些结果也进一步支持基于全基因序列的ANI分析能将细菌鉴定到种的水平[9]。同时,同源蛋白质比对分析结果显示,基因组中约93.7%的同源蛋白质均集中在铅黄肠球菌,这些结果也为全基因序列的ANI分析结果提供了蛋白质水平的数据支持。
此外,本研究通过分析铅黄肠球菌的全基因组序列,发现其基因组中存在5种潜在致病相关蛋白,但这些预测蛋白是否能表达,表达后是否会对人产生致病性等均有待研究。同时,发现铅黄肠球菌基因组不同于屎肠球菌或粪肠球菌[10],仅含有2个耐药基因,这也部分解释了在临床分离的铅黄肠球菌耐药菌株远远低于屎肠球菌和粪肠球菌等肠球菌的原因。
综上,本研究应用多种不同原理方法对CMCC(B)32220系统鉴定和分析,并以此了解其全基因组的基本特征、基因功能、耐药性及致病性,不仅为细菌标准菌株提供了更多背景数据,提高了其质量控制手段,同时为后续相关分析提供范例。
致谢:感谢上海人类基因组研究中心郑华军和朱永强给予的帮助。

