基于TCGA在宫颈癌肿瘤微环境中挖掘免疫疗效相关的预后基因
吴南昌 王静 卢东林 韦云宝 卢海娉 何雪芬 陈升才




【摘要】 目的 結合生物信息学方法,使用肿瘤基因组图谱(TCGA)数据库中宫颈癌表达谱数据,通过ESTIMATE算法分析肿瘤微环境挖掘有价值的预测免疫治疗疗效相关的预后基因。
方法 从TCGA数据库下载宫颈癌数据,运用R软件ESTIMATE包将宫颈癌基因表达谱数据分为高低免疫/高低基质评分组,通过limma包计算各组差异基因,最终两组的交集基因将用于生存分析、功能富集分析及蛋白互作网络构建。使用R Survival软件包分析高低免疫/高低基质评分组与预后的关系,同时使用Wilcox检验分析高低免疫/高低基质评分组与临床特征之间的关系。
结果 高免疫评分组与预后呈正相关(P=0.035);高基质评分组中位总生存期延长,但无统计学意义(P=0.391)。高免疫评分组、高基质评分组与M分期呈正相关(P<0.05)。设定阈值为|log2(FC)|>2且FDR<0.05,共鉴定出485个交集差异基因(上调475个,下调10个)。差异基因生存分析显示157个基因与预后有关。基因本体分析(GO)和京都基因与基因组百科全书途径(KEGG)共富集788个基因本体术语和36条通路(FDR<0.05)。差异基因构建PPI网络并利用Cytoscape提取3个关键核心网络,共包括66个基因,其中12个也与预后相关。
结论 基于TCGA数据库分析肿瘤微环境获得的免疫预后相关基因可能是潜在的免疫治疗疗效预测的生物标志物,值得深入研究。
【关键词】 宫颈癌;肿瘤微环境;免疫基质评分;ESTIMATE算法;癌症基因组图谱
中图分类号:R737.33 文献标志码:A DOI:10.3969/j.issn.1003-1383.2022.01.002
Exploration on prognosis genes related to immunization efficacy based on TCGA in cervical cancer tumor microenvironment
WU Nanchang, WANG Jing, LU Donglin, WEI Yunbao, LU Haiping, HE Xuefen, CHEN Shengcai
(Department of Gynecology, Affiliated Hospital of Youjiang Medical University for Nationalities, Baise 533000, Guangxi, China)
【Abstract】 Objective To combine the bioinformatics method and use the Cancer Genome Atlas (TCGA) database cervical cancer expression profile data to analyze the tumor microenvironment by using the ESTIMATE algorithm to explore valuable prognostic genes related to the prediction of immunotherapy efficacy.
Methods Cervical cancer data were downloaded from the TCGA database. The cervical cancer gene expression profile data were divided into high and low immune/high and low matrix score groups by R software ESTIMATE package. The differential genes were calculated by limma package. And the final two sets of intersection genes would be used in survival analysis, functional enrichment analysis, and protein interaction network construction. R Survival package was used to analyze the relationship between the high and low immune/high and low matrix score groups and the prognosis, and the relationship between the high and low immune/high and low matrix score groups and clinical features was analyzed by Wilcox test.
Results The high immune score group was positively correlated with prognosis (P=0.035); the median overall survival was prolonged in the high matrix score group, but difference was not statistically significant (P=0.391). The high immune score group and the high matrix score group were positively correlated with M stage (P<0.05). The threshold was set to |log2(FC)|>2 and FDR<0.05. A total of 485 intersection difference genes were identified (475 up-regulation and 10 down-regulation). Differential gene survival analysis showed that 157 genes were associated with prognosis.The gene ontology (GO) analysis and the Kyoto Encyclopedia of Genes and Genome Pathways (KEGG) enriched 788 gene ontology terms and 36 pathways (FDR<0.05). The differential gene constructed PPI network and used Cytoscape to extract 3 key core networks, including 66 genes, 12 of which were also associated with prognosis.
Conclusion Immune prognosis-related genes obtained from tumor microenvironment based on TCGA database may be a potential biomarker for predicting the efficacy of immunotherapy, which is worthy of further study.
【Key words】 cervical cancer; tumor microenvironment; immune matrix score; ESTIMATE algorithm; TCGA
宫颈癌是威胁我国女性健康最常见的妇科恶性肿瘤之一。全球每年有超过50万新发的宫颈癌患者,约30万例死于宫颈癌,其发病率及病死率均居女性恶性肿瘤第4位[1]。手术为主、放化疗为辅的传统治疗对早期宫颈癌治疗效果不错,但对于晚期、转移和复发宫颈癌5年生存率低于20%[2~3]。近年来,免疫治疗是肿瘤治疗的新手段,其机制是通过重新激活自身的抗肿瘤免疫系统对肿瘤细胞强且持续性的杀伤,显著提高生存时间。免疫治疗目前研究最全面的是免疫检查点抑制剂:其代表药为程序性死亡蛋白1(PD-1)抑制剂(帕姆单抗),在多种癌症中被证明有效,但总体客观有效率仅为20%~30%[4]。目前用于指导免疫治疗的分子靶标主要局限于程序性死亡蛋白配体1(PD-L1)表达水平、高度微卫星不稳定(MSI-H)[5]、错配修复系统缺陷(dMMR)[6]、肿瘤突变负荷(TMB)[7~8],虽然取得一些进展,但仍存在很大争议,因此,通过全面分析肿瘤微环境筛选更合理的分子标志物以指导免疫治疗显得尤为重要。肿瘤的发生发展是致癌因素持续存在刺激基因突变与肿瘤细胞经免疫逃逸后形成微环境的共同结局[9]。肿瘤微环境是指肿瘤细胞与肿瘤局部浸润的免疫细胞、基质细胞等共同构成的局部病理环境。既往研究提示,肿瘤微环境中高免疫、基质细胞浸润水平往往伴随良好预后。最近,YOSHIHARA等[10]开发了一种通过分析肿瘤内部免疫和基质细胞的特定基因表达特征来计算免疫和基质评分进而预测肿瘤纯度的算法。该算法在前列腺癌[11]、乳腺癌[12]和结肠癌[13]中得到应用并被证实有效,然而在宫颈癌领域尚未见报道。本研究利用肿瘤基因组图谱(TCGA)数据库中宫颈癌队列和ESTIMATE算法衍生的免疫评分[10],探索了与宫颈癌肿瘤微环境免疫预后相关的基因,为挖掘有价值的预测免疫治疗疗效生物标志物提供新思路。
1 材料与方法
1.1 基因表达数据集
宫颈癌患者的3级基因表达谱数据来源于TCGA数据门户网站(https://tcga-data.nci.nih.gov/tcga/)。另外,我们还下载了包括年龄、组织学类型、生存时间和分期等临床特征信息。下载的数据使用ESTIMATE算法来计算免疫评分和基质评分[10]。本研究严格遵循TCGA批准的出版指南,因此不需要伦理委员会批准。
1.2 差异表达基因分析
所有宫颈癌样本根据中位值分为高、低免疫评分组和高、低基质评分组后分别进行差异表达计算。将Log2|FC|>2且FDR<0.05设定为选择阈值,通过limmar包筛选显著差异基因,并使用R软件的pheatmap包生成热图。使用Venn包选择出高、低免疫评分组和高、低基质评分组交集的基因。
1.3 生存分析
为表明免疫/基质评分以及差异基因与患者预后之间的关系,根据Kaplan-Meier曲线法在R中运行Survival软件包绘制Kaplan-Meier图。
1.4 功能分析
为了解差异基因的潜在功能,免疫基质组交叉DEG的功能富集分析采用基因本体分析(GO)和京都基因与基因组百科全书途径(KEGG)。FDR<0.05作为截止值。
1.5 免疫、基质评分与临床特征的关系
使用Wilcox检验分析免疫、基质评分与临床特征之间的关系,包括T期[(T3 + T4) vs (Tis + T1 + T2)]、N期(N1 vs N0)、M期(M1 vs M0)、FIGO临床期(ⅠA~ⅡA2 vs ⅡB~ⅣB)、年龄、淋巴血管侵犯(YES vs NO)和肿瘤组织学分级(G1~2 vs G3~4)。P<0.05为差异有统计学意义。
1.6 PPI网络的构建
进一步寻找基因之间的相互作用,利用STRING数据库[14]构建蛋白质-蛋白质相互作用(PPI)网络,并借助Cytoscape[15]平台重建,只有包含超过10个节点的单个网络用于进一步分析。计算网络的每个节点的连接程度,然后使用MCODE插件基于拓扑找到簇以定位密集连接的区域。
2 结 果
2.1 差異表达的免疫相关基因分析
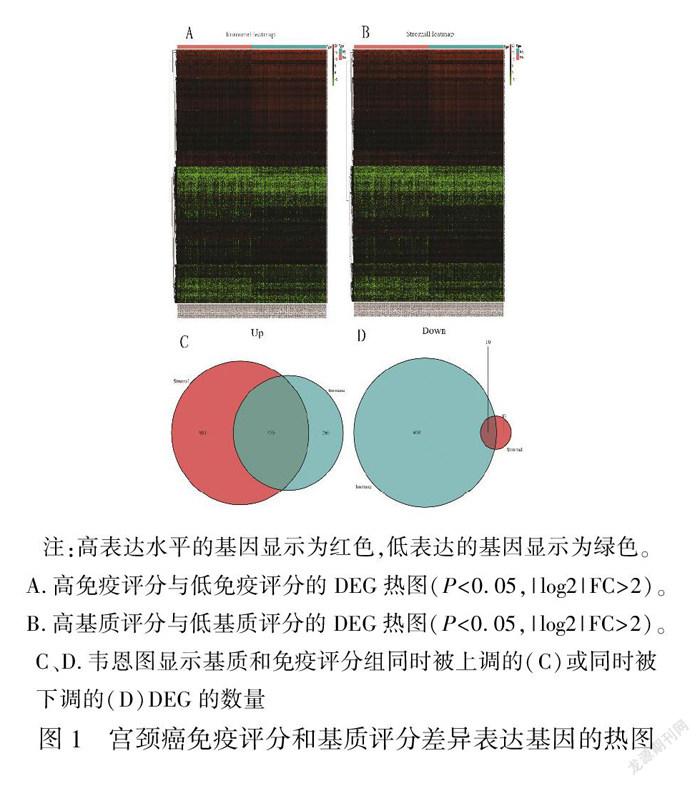
将TCGA数据库307例宫颈癌样本的表达谱数据分成高、低免疫评分组和高、低基质评分组,并比较选择差异表达的免疫相关基因。高免疫评分组有735个上调基因,412个下调基因;高基质评分组有1156个上调基因和20个下调基因(图1A、1B)。此外,为提高结果的可靠程度,韦恩图显示,免疫与基质变化方向一致的交集差异基因共485个(475个上调,10个下调)。见图1C、1D。
2.2 免疫基质评分与临床特征的关系
当设定P<0.05时,结果提示免疫、基质评分与M分期有统计学意义。低免疫、低基质肿瘤细胞与转移相关,高免疫、高基质肿瘤细胞不易发生转移(图2A、2B)。
2.3 免疫和基质评分与宫颈癌预后的关系
为了解免疫/基质评分是否与患者预后相关,根据307例宫颈癌患者的免疫/基质评分分别纳入高分组和低分组并构建Kaplan-Meier生存曲线。结果提示,高免疫评分组病例的中位总生存期(overall survival,OS)高于低免疫评分组,免疫评分与OS呈正相关(P=0.035);高基质评分组较低基质评分组中位生存期更长,但差异无统计学意义(P=0.391)。见图2C、2D。
2.4 交叉基因生存分析
485个交叉基因在通过R的Survival进行Kaplan-Meier曲线分析,结果157个基因与总生存率相关(P<0.05),其中参与构建核心网络的有12个(P<0.05,图3)被认为是潜在的免疫相关的预后基因。
2.5 功能富集分析
GO通路和KEGG共富集788个GO术语和36条通路(FDR<0.05,如补充S1、S2)。结果显示前5条KEGG通路为:细胞因子-细胞因子-受体相互作用、趋化因子信号通路、造血细胞系、原发性免疫缺陷、细胞黏附分子(图4A)。前五个GO术语为:T细胞激活、淋巴细胞活化的调节、白细胞-细胞黏附、T细胞活化的调节、白细胞-细胞黏附的调节(图4B)。
2.6 PPI核心网络构建
将485个基因导入String并结合Cytoscape构建PPI网络,揭示不同基因之间可能存在功能上的互作,包括239个节点和1267个边缘。包含15个节点以上的三个模块核心网络被选出进一步分析(图5A~C)。模块A(图5A)由33个节点及528个边缘构成,C3AR1、GNG8、GNGT2和FPR2与其他基因紧密关联。模块B(图5B)具有18个节点和141个边缘,ITGB2、FCER1G和GPR84具有更高的连通度值。模块C有15个节点和70个边缘(图5C),重要节点包括CD3E、CD3D和CD3G。3个模块共包含66个基因,其中12个与预后密切相关,提示可以作为免疫相关的预后基因。
3 讨 论
除贝伐单抗联合化疗一线治疗外,缺乏有效的二线治疗方案是晚期、复发及转移性宫颈癌患者死亡的主要原因。目前,新兴的免疫治疗在多种实体瘤中取得良好疗效,尤其是免疫检查点(PD-1/PD-L1)抑制剂,包括宫颈癌。KEYNOTE 028(第一阶段研究)[16]和KEYNOTE 158(第二阶段研究)[17]分别以10 mg/(kg·2W)和200 mg/3W剂量的帕姆单抗评估了复发性、转移性宫颈癌,结果提示帕姆单抗具有持久的抗肿瘤活性和可控制的安全性,FDA于2018年加速批准帕姆单抗用于化疗期间或之后疾病进展且PD-L1阳性的晚期宫颈癌患者。随后NCCN妇瘤指南指出MSI-H/dMMR/PD-L1阳性的晚期/复发和耐药宫颈癌患者二线用药推荐使用帕姆单抗。虽然免疫治疗在个别晚期宫颈癌收到奇效,但总体有效率仅为10%,寻找更加合适的标志物筛选受益人群是急需解决的难题。因此,我们试图通过研究宫颈癌肿瘤微环境鉴定可以预测宫颈癌免疫治疗疗效的分子靶标。本研究下载TCGA数据库宫颈癌307例样本的转录数据,经ESTIMATE算法计算,人为将它们分为高、低免疫评分组和高、低基质评分组进行差异表达分析。我们的研究结果再次证明了肿瘤微环境免疫及基质占比高低与患者预后相关,并且最终鉴定了485个基因与宫颈癌肿瘤微环境免疫显著相关。
近年,在直肠癌中建立的免疫评分研究提示,免疫评分高,生存时间长,预后较好,可作为预后的独立影响因素[18]。本研究分析每个样本免疫和基质的评分分别与个体预后及临床病理特征的关系,发现肿瘤微环境高免疫、高基质评分与良好预后及肿瘤病理M分期呈正相关,然而基质细胞与预后无统计学意义。该结果与既往结论基本一致,进一步说明肿瘤微环境对肿瘤的恶性增殖、免疫逃逸及逃避凋亡等有促进作用。
将样本分为高、低免疫评分和高、低基质评分的差异基因交集结果作为最终的免疫相关基因,通过GO、KEGG分析,结果显示大部分基因参与肿瘤微环境的众多关键环节(图4),如GO中的T细胞激活、淋巴细胞活化的调节、T细胞活化的调节及KEGG中细胞因子-细胞因子-受体相互作用、趋化因子信号通路、细胞黏附分子。这与先前的报道一致,即免疫细胞与细胞外基质细胞的分子功能在构建宫颈癌肿瘤微环境中有重要作用[19]。
为了进一步确定预后免疫相关基因,我們对485个差异基因进行总体生存分析,其中168个与宫颈癌患者的预后有关。此外,我们选取了包含节点大于15个基因的3个PPI蛋白互作网络进一步分析,发现网络中12个核心基因同时具备预后特征,因此,我们对它们特别感兴趣。6个核心基因已经被报道(CCL5、CXCL9、CD3D、CD3E、CD8A、IL2RB)参与肿瘤微环境的构建,甚至影响免疫治疗的效果,这表明我们基于TCGA数据库分析肿瘤微环境相关免疫基因具有重大意义。其余6个基因在肿瘤微环境领域尚未见报道,包括CD3G、CLEC4C、GNG8、GNGT2、P2RX1、P2RY13,可作为宫颈癌免疫治疗潜在的疗效评价生物标志物。研究表明在b16-f10肿瘤中靶向大自噬/自噬基因becn1/beclin1激活mapk8/jnk-jun/c-jun信号通路并释放CCL5细胞因子诱导NK细胞浸润抑制肿瘤生长[20]。ALLEN等[21]在卵巢癌中发现Th17细胞可通过IL-17与干扰素γ的协同作用,刺激CXCL9和CXCL10趋化因子产生,使肿瘤浸润效应T细胞向肿瘤微环境中募集,进而提高抗肿瘤免疫能力。SHI等[22]通过分析4个肌肉浸润性膀胱癌(MIBC)公共数据集,发现CD3D/CD4比值在MIBC中是一个稳定的独立预后因子,高CD3D/CD4比值有较好的生存率,CD4表达明显高于CD3D提示细胞具有免疫抑制作用,可用于免疫治疗疗效评估的潜在指标。CHEN等[23]基于肿瘤基因组图谱数据库对软组织肉瘤(STS)分析,发现IL-33和ST2的表达水平与CD3E、CD8A和趋化因子的表达水平呈正相关,提示IL-33/ST2轴可能在1型极化CD8+T细胞的免疫应答中起重要作用,提示这些分子可作为免疫治疗的预后标志物。ZHU等[24]分析了92例ICC组织中PD-L1表达与CD8+T细胞浸润的关系,发现肿瘤IFN-γ与PD-L1和CD8A基因表达显著相关,此外,用CD8+T细胞分泌的重组IFN-γ刺激HCCC9810和RBE细胞可在体外快速诱导PD-L1的上调,表明高表达CD8A可能是选择抗PD-L1/PD-1治疗的潜在标志物。人类IL-2rβ链(IL2RB)基因缺陷是致命的免疫失调的原因,CAMPBELL等[25]研究采用野生型IL2RB干细胞移植改善了1例患者T淋巴细胞的IL-2反应性,为使用IL-2疗法治疗免疫性疾病和癌症提供了新观点。
總之,本研究基于TCGA数据库的宫颈癌表达谱数据通过ESTIMATE算法得到高低免疫、基质评分组,将两者的交集差异基因进行生存分析、GO及KEGG富集,同时利用String构建PPI网络,最终提取了一份宫颈癌肿瘤微环境免疫相关的预后基因。肿瘤微环境由多种因素相互作用,极其复杂,目前对于免疫治疗疗效预测和评估的生物标志物有限,准确筛选获益人群仍是难题,我们的研究通过分析宫颈癌TME中主要成分免疫、基质细胞免疫评分进而获得免疫相关的基因列表,它们可能是先前被忽略的潜在有力的免疫治疗疗效预测及预后评估的生物标志物,值得进一步全面深入研究。
参 考 文 献
[1] BRAY F,FERLAY J,SOERJOMATARAM I,et al.Global cancer statistics 2018:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J].CA:A Cancer J Clin,2018,68(6):394-424.
[2] PFAENDLER K S,TEWARI K S.Changing paradigms in the systemic treatment of advanced cervical cancer[J].Am J Obstet Gynecol,2016,214(1):22-30.
[3] TEWARI K S,SILL M W,PENSON R T,et al.Bevacizumab for advanced cervical cancer:final overall survival and adverse event analysis of a randomised,controlled,open-label,phase 3 trial (Gynecologic Oncology Group 240)[J].Lancet,2017,390(10103):1654-1663.
[4] IWAI Y,HAMANISHI J,CHAMOTO K,et al.Cancer immunotherapies targeting the PD-1 signaling pathway[J].J Biomed Sci,2017,24(1):26.
[5] LE D T,URAM J N,WANG H,et al.PD-1 blockade in tumors with mismatch-repair deficiency[J].N Engl J Med,2015,372(26):2509-2520.
[6] LE D T,DURHAM J N,SMITH K N,et al.Mismatch repair deficiency predicts response of solid tumors to PD-1 blockade[J].Science,2017,357(6349):409-413.
[7] GOODMAN A M,KATO S,BAZHENOVA L,et al.Tumor mutational burden as an independent predictor of response to immunotherapy in diverse cancers[J].Mol Cancer Ther,2017,16(11):2598-2608.
[8] YARCHOAN M,HOPKINS A,JAFFEE E M.Tumor mutational burden and response rate to PD-1 inhibition[J].N Engl J Med,2017,377(25):2500-2501.
[9] FIDLER I J.The pathogenesis of cancer metastasis:the 'seed and soil' hypothesis revisited[J].Nat Rev Cancer,2003,3(6):453-458.
[10] YOSHIHARA K,SHAHMORADGOLI M,MARTNEZ E,et al.Inferring tumour purity and stromal and immune cell admixture from expression data[J].Nat Commun,2013,4:2612.
[11] SHAH N, WANG P, WONGVIPAT J, et al.Regulation of the glucocorticoid receptor via a BET-dependent enhancer drives antiandrogen resistance in prostate cancer[J].Elife, 2017,6:e27861.
[12] PRIEDIGKEIT N,WATTERS R J,LUCAS P C,et al.Exome-capture RNA-sequencing of decade-old breast cancers and matched decalcified bone metastases identifies clinically actionable targets[J].bioRxiv,2017.
[13] ALONSO M H,AUSS S,LOPEZ-DORIGA A,et al.Comprehensive analysis of copy number aberrations in microsatellite stable colon cancer in view of stromal component[J].Br J Cancer,2017,117(3):421-431.
[14] SZKLARCZYK D,FRANCESCHINI A,WYDER S,et al.STRING v10:protein-protein interaction networks,integrated over the tree of life[J].Nucleic Acids Res,2015,43(database issue):D447-D452.
[15] SHANNON P,MARKIEL A,OZIER O,et al.Cytoscape:a software environment for integrated models of biomolecular interaction networks[J].Genome Res,2003,13(11):2498-2504.
[16] OTT P A,BANG Y J,PIHA-PAUL S A,et al.T-cell-inflamed gene-expression profile,programmed death ligand 1 expression,and tumor mutational burden predict efficacy in patients treated with pembrolizumab across 20 cancers:KEYNOTE-028[J].J Clin Oncol,2019,37(4):318-327.
[17] CHUNG H C,ROS W,DELORD J P,et al.Efficacy and safety of pembrolizumab in previously treated advanced cervical cancer:results from the phase II KEYNOTE-158 study[J].J Clin Oncol,2019,37(17):1470-1478.
[18] MLECNIK B,BINDEA G,ANGELL H K,et al.Integrative analyses of colorectal cancer show immunoscore is a stronger predictor of patient survival than microsatellite instability[J].Immunity,2016,44(3):698-711.
[19] DENKO N,SCHINDLER C,KOONG A,et al.Epigenetic regulation of gene expression in cervical cancer cells by the tumor microenvironment[J].Clin Cancer Res,2000,6(2):480-487.
[20] NOMAN M Z,BERCHEM G,JANJI B.Targeting autophagy blocks melanoma growth by bringing natural killer cells to the tumor battlefield[J].Autophagy,2018,14(4):730-732.
[21] ALLEN F,BOBANGA I D,RAUHE P,et al.CCL3 augments tumor rejection and enhances CD8+ T cell infiltration through NK and CD103+ dendritic cell recruitment via IFNγ[J].Oncoimmunology,2018,7(3):e1393598.
[22] SHI M J,MENG X Y,WU Q J,et al.High CD3D/CD4 ratio predicts better survival in muscle-invasive bladder cancer[J].Cancer Manag Res,2019,11:2987-2995.
[23] CHEN H,CHEN Y,LIU H,et al.Integrated expression profiles analysis reveals correlations between the IL-33/ST2 axis and CD8+T cells,regulatory T cells,and myeloid-derived suppressor cells in soft tissue sarcoma[J].Front Immunol,2018,9:1179.
[24] ZHU Y,WANG X Y,ZHANG Y,et al.Programmed death ligand 1 expression in human intrahepatic cholangiocarcinoma and its association with prognosis and CD8+ T-cell immune responses[J].Cancer Manag Res,2018,10:4113-4123.
[25] CAMPBELL T M,BRYCESON Y T.IL2RB maintains immune harmony[J].J Exp Med,2019,216(6):1231-1233.
(收稿日期:2021-08-03 修回日期:2021-11-16)
(編辑:潘明志)

