酿酒酵母表面展示技术表达酸性蛋白酶
黄 蓉, 董 超, 余鸿飞, 秦 义, 刘延琳, 宋育阳
(西北农林科技大学葡萄酒学院 陕西杨凌712100)
酸性蛋白酶是一种具有丰富的化学和物理性质的天冬氨酸蛋白酶, 其活性位点中心含有一个或更多的羧基,能够在酸性介质(pH<4.5)中有效地水解蛋白质[1]。 这类酶具有极强的耐酸性,在水解过程中不会因微生物的滋生繁殖而腐败变质,普遍应用于食品、 酿造等各个工业的蛋白水解过程中[1-3]。大量的真菌可分泌产生酸性蛋白酶,有研究表明担子菌也可分泌产生酸性蛋白酶[4],分泌酸性蛋白酶的原核生物比较罕见[5]。依据活性位点上官能团的差异,蛋白酶通常被分为4 种类型:含硫类氨基酸Cys 的蛋白酶, 富含羟基类氨基酸Ser的蛋白酶, 富含酸性氨基酸Asp 的蛋白酶和金属蛋白酶[6]。对于天冬氨酸(Asp)蛋白酶而言,其等电点大致为3.0~5.0 且分子质量大约是30~40 ku,最大的酶活性体现位于酸性介质中[7-8],被称为酸性蛋白酶。
啤酒、 白酒和酱油等的酿造常用酸性蛋白酶作为蛋白澄清剂。在啤酒发酵糖化阶段,利用酸性蛋白酶可降解底物麦芽中的蛋白质, 通过添加适量的酸性蛋白酶阻止啤酒遇冷浑浊的现象[9]。有研究报道在白酒酿造过程中, 酸性蛋白酶可以溶解发酵原料和辅料的成分,促进酒精发酵,同时降解微生物残体,赋予白酒香气物质[10-11]。 蛋白质与其它物质形成的复合物是组成葡萄酒的物理、 化学特性的重要指标[7,11],然而白葡萄酒中来自发酵原料和辅料的非稳定蛋白质, 会对葡萄酒的香气和感官特性造成不良影响[12]。 针对白葡萄酒蛋白沉淀问题, 传统方法是在发酵或陈酿过程使用明胶和膨润土,虽可去除蛋白沉淀,但下胶过程会将葡萄酒中部分稳定性蛋白去除[12],影响葡萄酒的香气质量。 使用蛋白质酶解法可将葡萄酒中蛋白质水解成小肽[9],同时对酒液补充部分氨基酸,使葡萄酒获得良好的澄清度和稳定性, 提高葡萄酒的营养价值[10,13]。 目前可利用分子生物学发展的平台,通过基因工程改造酵母菌,使其分泌酸性蛋白酶,在发酵过程中水解不稳定的蛋白质,达到改善白葡萄酒的感官品质,解决蛋白沉淀的目的。
本研究以宇佐美曲霉(Aspergillus usamii)为酸性蛋白酶基因(pepA)供体,采用酵母细胞表面展示技术,将宇佐美曲霉的酸性蛋白酶基因pepA整合到酿酒酵母的基因位点, 以期在酿酒酵母发酵的过程中, 将不稳定蛋白质水解成氨基酸和小肽, 旨在解决白葡萄酒中蛋白浑浊的问题同时为酵母的生长提供可吸收性氮源。
1 材料与方法
1.1 材料
1.1.1 菌株与质粒 本研究所用的宿主菌为酿酒酵母单倍体BY4741,武汉淼灵生物科技有限公司(his3Δ1,实验室改造后为SD-Ura 缺陷性酵母)和酿酒酵母二倍体82-9-35(实验室前期分离得到,his3Δ1,为SD-Ura 缺陷性酵母)。生物工程公司合成含宇佐美曲霉的酸性蛋白酶基因pepA 的大肠杆菌,利用来自宝生物官网Phanta@Turbe Super-Fidelity DNA Polymerase 扩增体系, 进行PCR 扩增目的片段pepA 基因, 后利用Takala 公司生产的试剂盒直接纯化回收。E.coli DH5α 购自于宝生物, 出发质粒为本实验室保存的大肠杆菌的质粒PUC-GAP-α-factor-HQM-SED1(GAP 为启动子,α-factor-为信号肽,SED1 为酿酒酵母细胞壁蛋白)。
1.1.2 工具酶与主要试剂 0.4 mol/L 碳酸钠溶液、乳酸缓冲液(pH 3.0)、0.4 mol/L 三氯乙酸、0.5 mol/L 氢氧化钠、10.00 mg/mL 酪素溶液、100 μg/mL 酪氨酸标准溶液、1 mol/L Tris-HCl (pH 8.0)、0.5 mol/L 乙二胺四乙酸 (EDTA)(pH 8.0)、TE 缓冲液、氨卡青霉素(Amp)、1 mol/L 山梨醇、酵母感受态制备处理液、酵母全营养培养基(YPD)、尿嘧啶缺陷型合成葡萄糖基础培养基 (SD-URA)、Luria-Bertain(LB)培养基和LB-Amp 培养基的配制方法参照Zhang 等[14]的方法。 一步克隆试剂盒、DNA 凝胶纯化试剂盒、缺失尿嘧啶的DO 添加剂、Taq 酶、DNA Markers 和DNA 限制性内切酶(Cla I、Sph I、Sma I),宝生物(大连)有限公司;DNA 高保真聚合酶,南京诺唯赞生物科技有限公司;DNA提取液 (V酚∶V氯仿∶V异戊醇=25∶4∶1)、1 mol/L 二硫苏糖醇(DTT)、DNA 纯化试剂盒和酵母基础氮源(YNB),北京索莱宝科技有限公司;引物的合成和DNA 测序交由上海生物工程有限公司完成。
1.1.3 引物与pepA 序列 本试验所用引物如表1 所示。
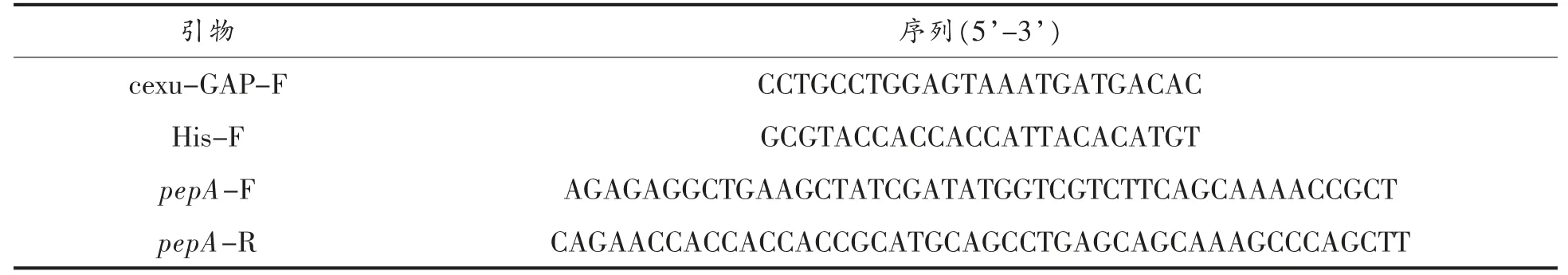
表1 引物序列Table 1 Primer sequences
1.1.4 仪器与设备 FB-K20 恒温金属浴,上海枫岭生物技术有限公司;MJPS-250 生化培养, 上海精宏实验设备有限公司;SW-CJ-2FD 超净工作台,苏州安泰空气技术有限公司;KQ-300DE 超声波清洗机, 昆山市超声仪器有限公司;PB-10 pH计, 北京赛多利斯仪器系统有限公司;LDZX-50KBS 灭菌锅, 上海申安医疗器械厂;DYY-10C电泳仪, 北京市六一仪器厂;BG-sub M2DI 水平电泳槽,百晶生物技术有限公司;FRESCO17 低温高速离心,Thermo;C1000 PCR 仪,Biorad 公 司;GBOX-EF 凝胶成像系统,Syngene 公司;ND-1000微量紫外分光光度计,Nanodrop 公司;DYY-6C 电穿孔仪,北京六一生物科技有限公司;AUW-220D十万分之一天平,岛津公司。
1.2 方法
1.2.1 pepA 基因的合成和TFP6 因子表达载体构建 pepA 基因通过NCBI 查找相应序列并进行优化, 由生工生物工程公司合成, 利用宝生物的DNA 聚合酶扩增体系扩增pepA 基因片段, 上游引 物 pepA -F: AGAGAGGCTGAAGCTATCGAT ATGGTCGTCTTCAGCAAAACCGCT; 下游引 物pepA-R: CAGAACCACCACCACCGCATGCAGCC TGAGCAGCAAAGCCCAGCTT,后用试剂盒直接纯化回收。 TFP6 载体PUC-GAP-α-factor-HQMSED1 用快切酶Sph I 和Cla Ⅰ双酶切,试剂盒直接纯化回收。用一步克隆的方法(操作方法参照宝生物In-Fusion RHD Cloning Kit 说明书)连接目的基因片段和线性化质粒载体。 具体操作步骤参照OMEGA 质粒提取试剂盒和天根的DNA 纯化回收试剂盒。
1.2.2 大肠杆菌转化 一步克隆连接的表达载体转化入大肠杆菌DH5α, 涂布在LB-Amp 固体平板上,37 ℃培养12~14 h, 挑取单菌落用Taq 酶体系(参考宝生物)进行菌落PCR 验证,引物为pepA-F 和pepA-R。 将验证成功的大肠杆菌单菌落接种在LB-Amp 液体培养基中,37 ℃、180 r/min振荡培养12 h。
1.2.3 目标酶基因插入酵母基因组 OMEGA 质粒提取试剂盒提取大肠杆菌中含有pepA 的重组质粒,用DNA 限制性快切酶Sma Ⅰ单酶切后,进行试剂盒纯化回收。 将回收后的DNA 片段,转化至SD-Ura 缺陷性酿酒酵母单倍体BY4741 和二倍体82-9-35(目标酶片段依靠His3 组氨酸同源臂与酵母基因组发生同源重组),后涂布在SD-Ura 平板上,30 ℃恒温培养48 h,挑取酵母单菌落进行酵母菌落PCR 验证重组酵母。 Taq 酶体系PCR验证引物: 上游引物为His-f: GCGTACCACCACCATTACACATGT, 下游引物pepA-R:CAGAACCACCACCACCGCATGCAGCCTGAGCAGCA AAGCCCAGCTT。
1.2.4 pepA 信号肽预测 在网址http://www.cbs.dtu.dk/services/Signal P/进行pepA 的氨基酸序列的信号肽预测。
1.2.5 酸性蛋白酶酶活的测定 酶活力单位的定义和酶活的测定方法和计算公式参照文献[15]的方法。
2 结果与分析
2.1 重组质粒的构建
根据NCBI 网站中宇佐美曲霉的酸性蛋白酶基因(pepA)序列,用Vector NTI 软件设计扩增pepA 目的基因的引物, 引物为pepA-F 和pepAR。pepA 基因由生工生物工程公司合成,进行高保真酶PCR 后,直接用试剂盒纯化回收,得到片段为1 182 bp 的目的片段(图1)。 扩增后pepA 测序结果与NCBI 网站目的基因pepA 序列一致。 出发质粒PUC-GAP-α-factor-HQM-SED1 用快切酶Sph Ⅰ和Cla Ⅰ双酶切,直接用试剂盒纯化回收,得到TFP 线性质粒载体(图2);用一步克隆的方法(操作方法参照宝生物In-Fusion RHD Cloning Kit 说明书) 连接目的基因片段pepA 和线性化质粒载体(图3)。具体操作方法参照OMEGA 质粒提取试剂盒和天根的DNA 纯化回收试剂盒。提取大肠杆菌重组质粒测序, 引物为cexu-GAP-f 和pepA-R,测序结果与预期一致。
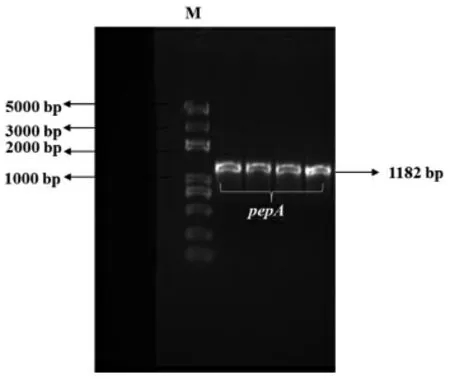
图1 目的片段电泳图片Fig.1 Electrophoresis picture of the target fragment

图2 PUC-GAP-α-factor-HQM-SED1 质粒双酶切Fig.2 Double digestion of PUC-GAP-α-factor-HQM-SED1 plasmid
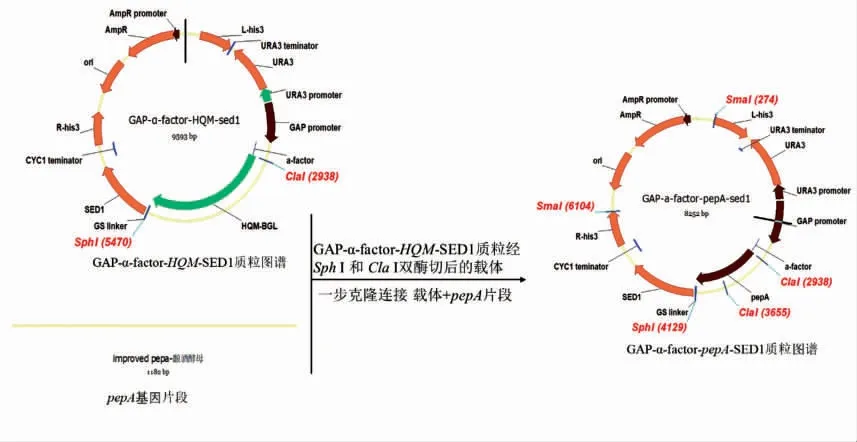
图3 质粒PUC-GAP-α-factor-pepA-SED1 构建流程Fig.3 Construction process of plasmid PUC-GAP-α-factor-pepA-SED1
2.2 电激转化酿酒酵母
参考文献[13]的方法制备酵母感受态并进行酵母电转化。 将转化子划线在YPD 和SD-Ura 平板上,恒温30 ℃,培养48 h 后,进行菌落PCR 验证,验证引物为cexu-GAP-f、his-f 和pepA-R。 图4 所示的电泳结果表明,pepA 基因插入位点正确。
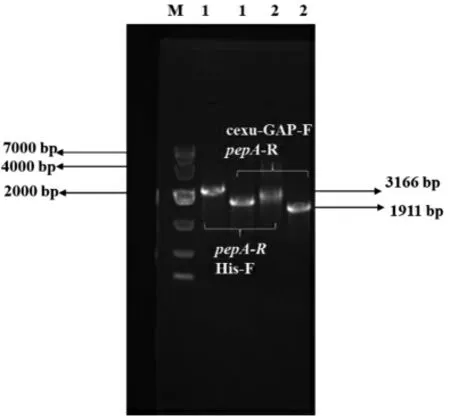
图4 重组菌PCR 扩增Fig.4 Recombinant bacteria PCR amplification
2.3 酸性蛋白酶酶活测定
图6 为在培养温度30 ℃条件下,酿酒酵母表面展示的酸性蛋白酶96 h 内的酶活曲线图。 在72 h 左右出现最高酶活,72 h 前随着酵母培养时间增加,酶活呈现上升的趋势,而在72 h 后酸性蛋白酶活性随时间呈下降的趋势,并且82-9-35-8 的酶活96 h 内基本高于BY4741-i。 推测其酶活下降的原因可能是酵母培养基中的营养物质被酵母消耗减少, 酵母自身生长能力下降所致。 其在72 h 前酶活上升的原因与酵母在72 h 内繁殖增长相关。

图6 BY4741-i 和82-9-35-8 在YPD 培养基上的酶活变化Fig.6 Changes of enzyme activity of BY4741-i and 82-9-35-8 on YPD medium
2.4 监测阳性重组菌的生长
重组酵母和宿主菌 (BY4741 和82-9-35)分别在SD-ura 和PD 上划线培养,48 h 后挑取单菌落接种到50 mL 的SD-URA 选择性液体培养基和YPD 液体培养基中,30 ℃、200 r/min 培养48 h进行活化后,用血球计数板进行活菌计数,以1×106CFU/mL 的接种量接种至150 mL 的YPD 液体培养基中,培养条件30 ℃、200 r/min,每隔12 h 取样, 用酶标仪测定其在波长600 nm 处的吸光度值。 以生长时间为横坐标,OD600nm为纵坐标,绘制重组酵母菌株和对照菌株的生长曲线。

表2 酪氨酸标准曲线Table 2 The standard curve of tyrosine

图5 酪氨酸标准曲线Fig.5 The standard curve of tyrosine
用YPD 液体培养基培养重组酵母和原菌株,每隔6 h 取样600 nm 处测定吸光度,绘制重组酵母与对照菌株的生长曲线,如图7 所示。 0~24 h,所有菌株处于生长延滞期。 24 h 后所有菌株迅速繁殖,30~54 h 是所有酵母的生长对数期, 在对数前期,二倍体菌株的生长速度比单倍体快;在对数后期,单倍体的生长速度超过二倍体,最终单倍体的生长量高于二倍体。表面展示pepA 的二倍体重组酵母在稳定期达到的生长量明显比原菌株高,表明单倍体和二倍体酵母表面展示pepA 并未对菌株的生长有不良影响, 且显著增强了菌株生长能力。
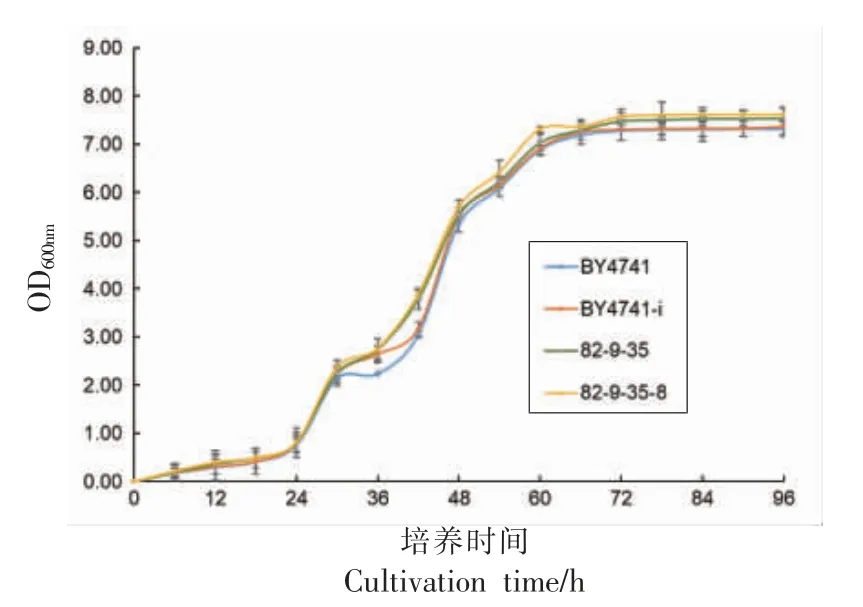
图7 酵母在YPD 培养基上的生长曲线Fig.7 Growth curve of recombinant yeast on YPD medium
如图8 所示,当阳性转化子BY4741-i 和82-9-35-8 在YPD 培养基上的生长进入平衡期前,细胞数量积累很多,酶活剧烈上升,并在细胞生长平衡期时酶活基本达到最大值。 72 h 左右酶活达到最大值,之后酶活呈下降的趋势。推测由于培养基营养物有限,细胞相互间竞争,死亡的细胞数和新生的细胞数持平,因此酶活曲线呈下降趋势。
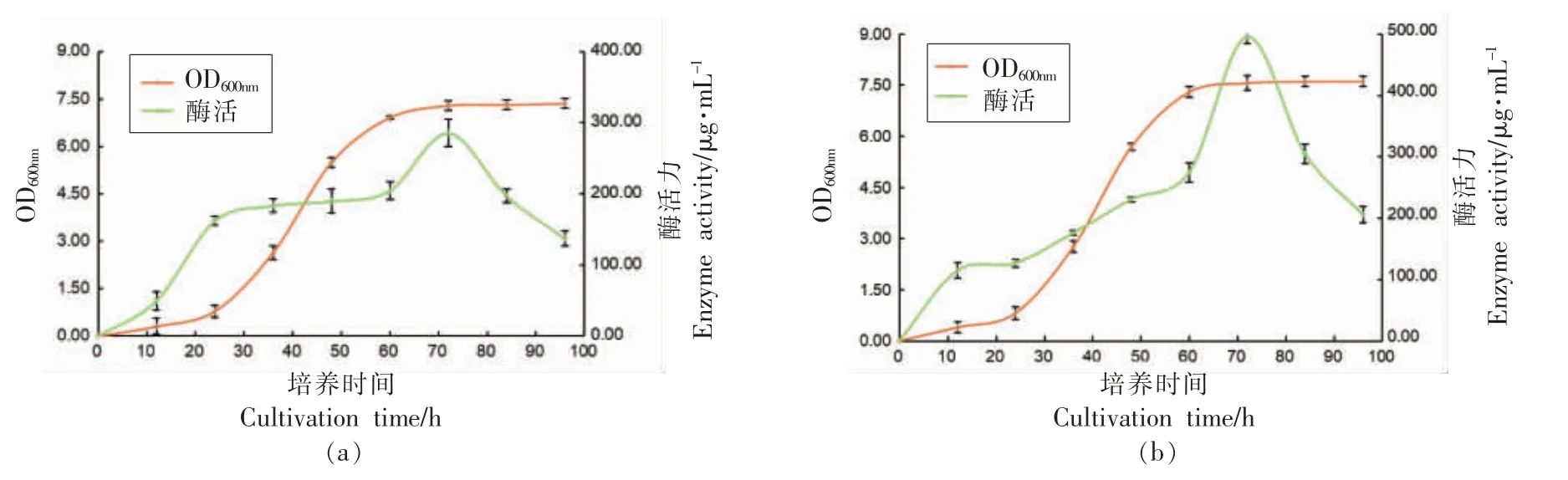
图8 BY4741-i 和82-9-35-8 在YPD 培养基上的生长曲线与酶活关系图Fig.8 The relationship between the growth curve and enzyme activity of BY4741-i and 82-9-35-8 on YPD medium
3 讨论
目标蛋白转录翻译的过程受代谢途径的影响[16-17]。 在酿酒酵母中,异源蛋白的分泌和表达过程与其它真核生物基本相同。 从核糖体开始mRNA 被翻译成新的肽链, 并被转运至高尔基体,高尔基体对多肽链存在进一步加工和修饰的作用[17-19],最终通过囊泡运输作用被分泌到细胞外部。 若蛋白质折叠错误, 则内质网将重新折叠或开通蛋白质降解途径使其分解[20]。 当错误的蛋白质不能及时修复并积累时,会导致宿主细胞的凋亡[21]。 在分泌表达过程中,正确的蛋白若未被及时送出胞外,则会在胞内积累,导致其活性降低甚至被降解,因此囊泡运输过程是影响蛋白分泌表达效率的重要限制性因素之一[22-23]。当前研究主要使用融合伴侣包括反式伴侣和顺式伴侣来增强目标蛋白的折叠运输效率, 如依靠筛选CIS 伴侣蛋白可提升蛋白的正确折叠程度[24]。 信号肽引导目标蛋白的具有的作用, 其编码区由17 到30 个氨基酸残基序列组成,一般是10 个带正电荷的非极性氨基酸[25-26]。大多数表面展示的宿主细胞的目标蛋白的表达通常以包涵体的形式存在,并非直接分泌表达,但包涵体不具有蛋白活性[27-28]。 因此,信号肽被用来解决包涵体重折叠的影响, 引导外源蛋白在细胞特定位置的分泌[24]。 目标蛋白的信号肽与宿主细胞的融合性存在一定的差异[29-30],其不恰当使用会对目标蛋白的分泌途径造成负面影响[26,31]。有研究报道当宿主细胞的二氨基肽酶对信号肽去除不完整时,会影响目标蛋白的活性[32]。 基于以上因素,为了确保表达盒产生的包含酸性蛋白酶的融合蛋白能够通过细胞机制正确地定向到细胞表面, 本研究引入酿酒酵母自身的翻译融合伴侣和信号肽α-factor。
连接肽的设计在确保得到正确构象的融合蛋白时, 也要保证融合蛋白能保持正常的分泌和表达[33-34],否则会影响目的蛋白的活性。 鉴于存在不利影响,本研究选择具有柔韧性、亲水性、易折叠性和小侧链氨基酸的接头肽序列【(G4S)3】[35],在一些酵母展示载体(例如,pYD1 和pYD5)中具有编码这种类型连接肽的序列。
展示效率同时应考虑要展示的蛋白质的分子质量, 其会影响融合蛋白在细胞内外的转运和分泌[36]。 Fan 等[32]在分析解纤梭菌DSM 5812 支架II的几个重复单元的试验中, 发现随着174 个氨基酸重复序列的增加, 暴露Aga2-C 端融合蛋白的细胞的百分比下降。 这是由于每个细胞的表面积是有限的, 所以在展示较大蛋白的菌株中存在重组质粒不稳定的现象。 Yang 等[33]使用Pir 蛋白作为锚定蛋白时, 发现展示800 个以上氨基酸的目的蛋白效率很低。 锚定蛋白存在与酵母细胞壁不同的结合方式, 会影响目标蛋白的展示量和表达活性[27]。 因此,在构建细胞表面展示表达盒时,应科学选择目标蛋白, 同时选择其与锚定蛋白适合的融合方式[27,37]。 Yamada 等[38]报道,当表面展示纤维素酶的工程酵母菌株通过交配进行二倍化并评估其乙醇发酵性能时 , 观察到乙醇产率增加约2.5 倍。 本研究进行宿主二倍体优化,阳性二倍体菌株酸性蛋白酶酶活是阳性单倍体菌株酶活1.7倍左右,这是由于与单倍体菌株相比,二倍体菌株具有更高的细胞生长速率、 细胞产量以及对各种胁迫的耐受性。
4 结论
本研究通过克隆宇佐美曲霉的酸性蛋白酶基因(pepA),以SED1 为锚定蛋白,GAP 为启动子成功搭建了酿酒酵母a-凝集素表面展示系统,获得可通过酵母细胞表面展示技术表达酸性蛋白酶的两株酿酒酵母,分别是单倍体BY4741-i 和二倍体82-9-35-8。 监测重组酵母的生长,发现表面展示pepA 未对宿主菌株造成代谢压力,对其生长有不利影响, 重组酵母的最高酶活性表达时间与其生长进入稳定期时间基本一致。 依据重组酿酒酵母酸性蛋白酶的活性表现, 证明酿酒酵母表面展示技术可作为葡萄酒介质低pH 值条件下的全细胞生物催化剂, 在改善白葡萄酒的蛋白稳定性方面的具有可行性。

