大白猪ENO3基因多态性及其与生长性状的关联分析
赵玉强,陈 鑫,李清春,马继林,董银河,汪德明,郭凌云,黄 涛
(1.乌鲁木齐正大畜牧有限公司博士后科研工作站,乌鲁木齐 830000;2.石河子大学动物科技学院,石河子 832000)
烯醇化酶(enolase,ENO),又称为磷酸丙酮酸脱氢酶,是一种金属酶,负责催化糖酵解途径中磷酸烯醇丙酮酸和2-磷酸甘油酸的相互转化[1]。哺乳动物细胞中ENO存在3种亚型:α或非神经元性烯醇化酶(NNE,ENO1)、γ或神经元特异性烯醇化酶(NSE,ENO2)和β或肌肉特异性烯醇化酶(MSE,ENO3),它们是参与糖酵解或异生的细胞质酶[2]。ENO1在许多组织中都有表达,ENO2只在神经系统的细胞中表达,而ENO3定位于肌肉组织[3]。猪ENO3基因(Gene ID:692156)位于12号染色体上[4],含有12个外显子。Fougerousse等[5]研究表明,ENO3在增殖的成肌细胞和分化的肌管中表达,是人肌源性分化的最早标志物之一。在肌管的功能成熟阶段,ENO3的数量增加[6];ENO3缺乏会导致远端糖酵解代谢性肌病[7],提示ENO3可能参与调控肌肉的生长过程。Wu等[8]研究表明,ENO3基因的表达量在梅山猪和大白猪之间存在差异,且ENO3基因多态性与猪脂肪率、平均背膘厚、大理石纹及肌内脂肪有关。以上研究提示,ENO3基因可能与肌肉的生成及猪的生长有关。目前,ENO3基因多态性与大白猪生长性状的关联分析鲜见报道。鉴于此,本试验以大白猪为研究对象,应用混池PCR扩增目的区段,通过CE测序挖掘多态位点,结合GenoPlexs分型技术辨别试验个体的基因型,并与试验群体的校正体重达100 kg时的日龄、日增重、背膘厚、眼肌面积、眼肌厚共5个性状进行关联分析,挖掘与生长性状显著关联的SNP位点,以期为大白猪的分子标记辅助选育提供数据支持。
1 材料与方法
1.1 样品采集
从新疆某公司下属的种猪场选取316头生长表现正常且表型记录完整的经产大白母猪,用耳缺钳收集大白母猪的耳缘组织于装有1 mL 75%酒精的1.5 mL离心管中,母猪耳标号与离心管编号依次对应记录,低温运输回实验室,-20 ℃保存备用。
1.2 主要试剂及仪器
琼脂糖购自Biowest公司;血液/细胞/组织基因组DNA提取试剂盒(离心柱型)购自天根生化科技(北京)有限公司。梯度PCR仪购自SensoQuest公司;电泳仪(DDY-8C型)购自北京六一生物科技公司;NanoDrop 2000型超微量分光光度计购自Thermo Fisher公司。
1.3 基因组DNA提取
用眼科剪在离心管中剪碎耳组织,按照血液/细胞/组织基因组DNA提取试剂盒(离心柱型)操作步骤提取DNA,分别用1.0%琼脂糖凝胶电泳和超微量分光光度计进行DNA完整性、浓度和纯度检验,选择条带完整、明亮且浓度、纯度均符合要求的样本用于后续试验。
1.4 引物设计与合成
从GenBank数据库中下载猪ENO3基因全长序列(登录号:NC_010454.4),利用Primer Premier 5.0软件设计引物,用于扩增ENO3基因的各个外显子及其附近区域,引物序列见表1。引物均由生工生物工程(上海)股份有限公司合成。

表1 引物信息
1.5 混池建库与PCR测序
从试验群体DNA样本中随机选取40个样本,每个样本用移液器各取5 μL至同一个1.5 mL无菌离心管中,轻微振荡混匀,利用表1引物对混合DNA样进行PCR扩增。PCR反应体系10 μL:2×EsTaqMasterMix(Dye) 5 μL,去离子水2 μL,Primer Mix(10 μmol/L) 1 μL,DNA模板 2 μL。PCR反应程序:94 ℃预变性5 min;94 ℃变性30 s,退火(退火温度见表1)30 s,72 ℃延伸30 s,共35个循环;72 ℃延伸3 min;4 ℃保存。PCR产物经1.5%琼脂糖凝胶电泳鉴定。鉴定合格后将引物和混合DNA样本送至生工生物工程(上海)股份有限公司,利用ABI测序仪进行CE测序,利用Chromas DNA分析软件筛选多态位点。
1.6 GenoPlexs分型
筛选出基因多态位点后,利用多重PCR的靶向基因捕获技术(GenoPlexs)对每个个体的目标SNP及其附近区域设计引物,通过PCR扩增富集目的片段,结合二代测序技术对每个样本进行高深度测序,通过生物信息学分析对目标个体进行基因分型[9]。
1.7 数据统计分析
在育种场测定并记录316头大白猪的表型信息:结测日龄、结测体重、结测背膘厚、结测眼肌面积和结测眼肌厚。利用电子过道秤测定活体体重,待读数稳定后记录数值;利用B超仪测定大白猪正常站立情况下倒数3-4肋间左侧距背中线5 cm处的活体背膘、眼肌厚及眼肌面积,参照《种猪生产性能测定规程》(NY/T 822-2004)计算以上表型的校正数据。利用Excel 2016对数据进行整理,利用SPSS 19.0软件进行单因素方差分析,采用LSD进行多重比较,分析各SNP位点与大白猪体重达100 kg时的日龄、日增重、背膘厚、眼肌面积和眼肌厚的关联性。结果用平均值±标准差表示,以P<0.05为差异显著性判断标准。
2 结 果
2.1 PCR扩增产物
对大白猪ENO3基因外显子1~10进行PCR扩增,扩增产物经1.5%琼脂糖凝胶电泳鉴定,结果显示,7对引物PCR扩增产物条带长度分别为675、801、499、633、420、684和706 bp(图1),均符合预期大小,可用于后续试验。

1~7,ENO3基因外显子PCR扩增产物;M,DL5000 DNA Marker1-7,PCR amplification products of ENO3 gene exons;M,DL5000 DNA Marker图1 ENO3基因PCR扩增结果Fig.1 PCR amplification results of ENO3 gene
2.2 突变位点测序峰图
由图2可知,通过混池PCR直接测序发现9个SNPs位点,均为已发现的内含子突变位点,分别为:rs325279236(A/G)、rs342598032(A/G)、rs341541240(C/T)、rs329283992(G/C)、rs327882211(G/A)、rs345530479(G/C)、rs324943047(T/C)、rs786427749(A/-)和rs196953768(A/G)。其中rs196953768、rs324943047、rs327882211、rs341541240、rs325279236、rs342598032为转换;rs345530479和rs329283992为颠换;rs786427749为缺失。

rs325279236、rs342598032、rs341541240、rs327882211和rs324943047是由反向测序获得,其余位点均是由正向测序获得rs325279236,rs342598032,rs341541240,rs327882211 and rs324943047 were obtained by reverse sequencing,and the remaining sites were obtained by forward sequencing图2 ENO3基因突变位点测序峰图Fig.2 Sequencing peak map of mutation sites of ENO3 gene
2.3 分型结果及群体遗传学分析
送样个体中有2个样本由于严重降解导致分型失败,其余样本分型成功,基因分型结果见表2。由表2可知,9个SNPs位点中,rs196953768、rs324943047、rs345530479、rs329283992、rs342598032和rs786427749的优势基因型分别为AG、TC、GC、GC、AG和A/-,均为杂合型,rs327882211、rs341541240和rs325279236位点的优势基因型分别为GG、CC和AA,均为纯合型;9个SNPs位点均处于Hardy-Weinberg平衡状态(P>0.05)。ENO3基因遗传多样性分析结果见表3。由表3可知,rs196953768、rs324943047、rs345530479、rs329283992和rs342598032位点杂合度较高,rs786427749、rs327882211、rs341541240和rs325279236位点纯合度较高;多态信息含量(PIC)均属于中度多态,说明这些位点所受到的选择压力较小,可用作候选分子标记。

表2 ENO3基因的基因型频率及基因频率

表3 ENO3基因遗传多样性分析
2.4 连锁不平衡分析
由图3可知,rs196953768与rs324943047、rs345530479、rs329283992间完全连锁(D’=1,r2=1),与rs342598032呈强连锁(D’=1,r2=0.99);rs327882211与rs341541240、rs325279236呈强连锁(D’=1,r2>0.7),其余多态位点间呈弱连锁。
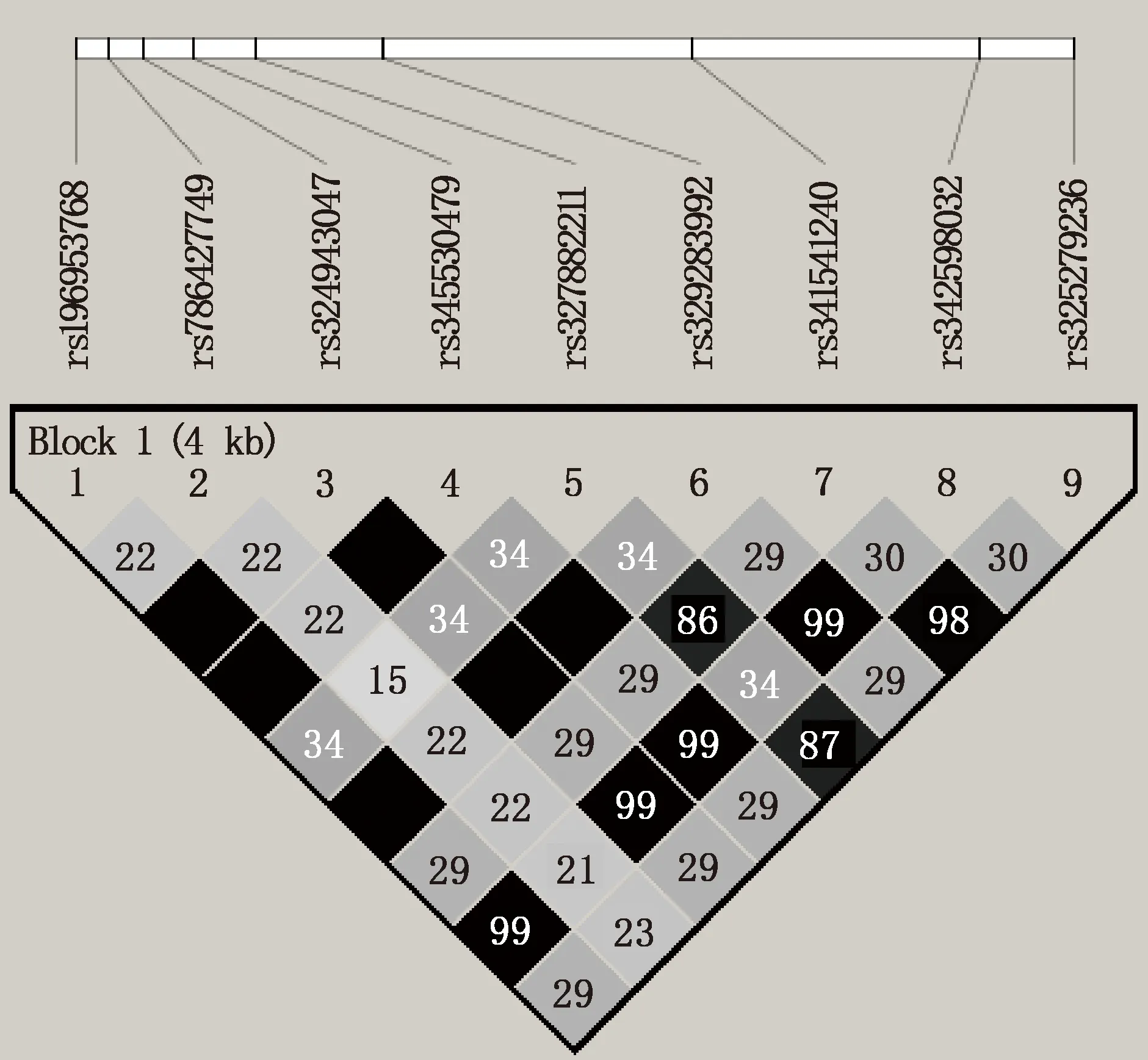
浅灰色表示D’<0.3;灰色表示0.3≤D’<0.6;深灰色表示0.6≤D’<1;黑色表示D’=1;数字表示r2值的小数点后两位,不显示数字表示完全连锁(r2=1,D’=1)Light gray indicates D’<0.3;Gray means 0.3≤D’<0.6;Dark grey indicates 0.6≤D’<1;Black indicates D’=1;And a number indicates the r2 value to two decimal places,no number indicates full linkage (r2=1,D’=1)图3 ENO3基因SNP连锁分析Fig.3 SNP linkage analysis of ENO3 gene
2.5 ENO3基因多态性与大白猪生长性状的关联分析
由表4可知,rs196953768位点GG基因型个体体重达100 kg时的日龄显著小于AG基因型(P<0.05),日增重显著高于AG基因型(P<0.05),其余性状各基因型间差异均不显著(P>0.05);rs327882211位点AA基因型个体体重达100 kg时的眼肌面积和眼肌厚均显著小于GA和GG基因型(P<0.05),其余性状各基因型间差异均不显著(P>0.05);rs341541240位点CC基因型个体体重达100 kg时的眼肌面积和眼肌厚均显著高于TT基因型(P<0.05),其余性状各基因型间差异均不显著(P>0.05);rs325279236位点AA基因型个体体重达100 kg时的眼肌面积显著高于GG基因型(P<0.05),其余性状各基因型间差异均不显著(P>0.05);rs786427749、rs342598032位点各性状在各基因型间差异均不显著(P>0.05)。

表4 ENO3基因SNPs与大白猪生长性状的关联分析
3 讨 论
生猪育种在中国的发展相对较晚,虽然近年来逐渐受到重视,但仍有待完善。随着分子生物学技术的发展,动物育种技术已进入分子水平,分子育种技术对猪的育种和生产产生了巨大影响[10],其中,分子标记辅助选择结合常规育种方法加速了遗传改良进程[11]。因此,挖掘与性状相关的SNPs作为辅助选择的分子标记位点,对提高生猪养殖企业的经济效益、增强中国育种能力有着重要意义。多重PCR靶向捕获技术是在单一PCR反应体系中加入2对或2对以上引物,用于富集目的片段,其反应原理、反应试剂盒操作过程与一般PCR相同[9],且MPprimer[12]、MultiPLX[13]等软件均可用于多重PCR引物的设计。同时结合二代测序技术对富集到的目的片段构建测序文库,上机进行深度测序[14],用以获得高质量的目的序列。通过数据质控、过滤及质量评估,比对到参考基因组,对目标区域序列进行基因组定位和功能注释,分析SNP、InDel后对样品进行准确分型。该技术的优势是支持针对任一位点设计引物,其特异性、测序深度及覆盖度均较高,能够明显提高效率,降低经济成本[15]。本试验中获得了较好的基因分型结果,送测316个样本,成功检测314个样本,多态位点检出率为99.37%,充分说明了该技术的可靠性。
ENO3基因在猪肌肉发育不同阶段存在明显的表达差异,但其多态性对猪生长性状的影响鲜见报道。 ENO3定位于肌肉组织,α-烯醇化酶在肌肉发育过程中转换成β-烯醇化酶[16]。在个体发育过程中,横纹肌分化伴随着β-烯醇化酶的增加和α-烯醇化酶的减少[5,17]。提示ENO3基因可能参与调控肌肉的生长发育。本研究结果发现,ENO3基因存在9个SNPs位点,均符合Hardy-Weinberg平衡定律,处于中度多态,且与大白猪生长性状有一定的关联。
生物群体中,由于2个位点在染色体上的距离足够近,导致这些位点上的等位基因经常出现在同一染色体上,使得遗传时位点上的等位基因出现非随机组合的现象,就是连锁不平衡(linkage disequilibrium,LD),又称基因的连锁遗传[18]。位点间的距离与LD程度呈明显的负相关,即距离越近,LD程度越高。SNP的连锁性在遗传中普遍存在,彭娅鑫等[19]、李常红等[20]研究均发现存在完全连锁或强连锁的SNP。本试验中,rs196953768与rs324943047、rs345530479、rs329283992间完全连锁(D’=1,r2=1),与rs342598032呈强连锁,进一步分析发现,rs342598032位点SNP与其余SNPs间没有完全连锁,可能是由于测序错误造成该位点判定的基因型与真实基因型产生偏离,也可能是由于该位点与其余位点距离较远使得LD程度降低。
Li等[21]通过分析猪编码基因区公共SNP发现,猪ENO3基因BV726865_302位点与剪切力呈显著相关。研究发现,人ENO3基因的2个错义突变与一种罕见的代谢性肌病有关[22];ENO3蛋白与绵茵安琼奶牛的肌肉嫩度和大理石纹密切相关[23];ENO3的转录及蛋白水平在瘦肉型猪和脂肪型猪间存在显著差异[8,24-25]。上述研究均提示,ENO3基因可能对动物骨骼肌的发育有着重要的影响。本研究通过对大白猪ENO3基因的9个SNPs位点与生长性状关联分析发现,rs196953768、rs324943047、rs345530479、rs329283992与体重达100 kg时的日龄和日增重呈显著相关,rs327882211、rs341541240与体重达100 kg时的眼肌面积和眼肌厚呈显著相关,rs325279236与体重达100 kg时的眼肌面积呈显著相关,说明这些位点均可作为对应性状的候选分子辅助标记。而rs786427749和rs342598032与上述表型的相关性均不显著,可能不是影响这些性状的重要标记,不适用于本试验大白猪群体生长性状选育。综上所述,ENO3基因多态性显著影响大白猪部分生长性状,但这些位点能否作为大白猪生长性状相关的遗传标记,还需要在更多、更大的群体中验证;同时,还需要从ENO3基因的功能及其SNP的作用机制出发加以验证。
4 结 论
本研究共鉴定到9个SNPs,其中rs196953768与rs324943047、rs345530479、rs329283992完全连锁,与大白猪体重达100 kg时的日龄和日增重均呈显著相关,rs327882211、rs341541240与大白猪体重达100 kg时的眼肌面积和眼肌厚均呈显著相关,rs325279236与大白猪体重达100 kg时的眼肌面积呈显著相关;共筛选到7个与生产性状显著相关的SNPs,可为大白猪的分子标记辅助选育提供数据支撑。

