畜禽粪便堆放地土壤中抗生素抗性基因和细菌群落的垂直分布特征*
韩婉雪,王凤花,柏兆海,李文彦,王新珍,马 林
(1.中国科学院遗传与发育生物学研究所农业资源研究中心/中国科学院农业水资源重点实验室/河北省土壤生态学重点实验室 石家庄 050022; 2.河北师范大学地理科学学院/河北省环境演变与生态建设实验室/河北省环境变化遥感识别技术创新中心/河北省地理科学实验教学示范中心 石家庄 050024; 3.中国科学院大学 北京 100049)
长期以来,抗生素在预防和治疗动物传染性疾病、促进动物生长以及提高饲料转化率等方面发挥着重要作用。随着集约化养殖进程的加快,畜禽养殖业中抗生素的使用量也急剧增加。2013年中国抗生素的使用量为16.2 万t,其中一半以上为兽用抗生素。但抗生素通常不能完全被动物吸收,约30%~90%的抗生素以母体或代谢产物的形式随粪尿进入环境,对环境和人类健康产生潜在威胁。由于抗生素的长期滥用,诱导动物体内抗生素抗性基因(以下简称“抗性基因”)的产生,造成动物病原菌耐药性的升高,加速了养殖环境中抗性基因的产生和扩散。畜禽粪便已成为抗生素、抗性基因及抗性细菌的重要“储蓄库”。此外,随着养殖业的不断发展,随之而来的环境问题也日益凸显,尤其是畜禽粪便排放量逐年增加,从2016年的31.6 亿t增加到2021年的38 亿t。然而,大量的畜禽粪便在农业利用前未经处理直接堆放在养殖场周边,其残留的抗生素和抗性基因会进一步污染周边土壤和水体,对生态环境和人类健康造成潜在危害。
目前关于我国养殖场畜禽粪便中抗生素和抗性基因残留已开展了大量的研究。Qiao 等研究发现四环素类、磺胺类和喹诺酮类抗生素在畜禽粪便中具有较高的检出率和检出浓度,四环素类和磺胺类抗性基因也是施用畜禽粪便土壤中检出频率较高的两类抗性基因。同其他污染物一样,抗生素和抗性基因在土壤中会随降水、灌溉等途径,向下层土壤迁移。Tang 等研究表明,长期施畜禽粪便的土壤中、、和基因丰度随土壤深度的增加而降低。綦峥等在畜禽养殖场周边不同深度土壤中检测到多种抗性基因,其中磺胺类抗性基因随着土壤深度的增加逐渐降低,而-内酰胺类抗性基因随土层深度的增加出现了上升的趋势。这就说明畜禽粪便的施用会不同程度地污染下层土壤。值得注意的是,长期堆放畜禽粪便的土壤受畜禽粪便中残留的抗生素和抗性基因等污染物的影响要高于施畜禽粪便的农田土壤。但有关长期堆放畜禽粪便土壤中抗性基因的垂直分布特征的研究相对较少。
微生物作为抗性基因的宿主,其群落结构的变化与抗性基因的分布紧密相关。土壤微生物对土壤环境的变化非常敏感,有研究表明,有机肥施用通过改变土壤环境因子,引起土壤微生物群落结构的变化,进而影响土壤中抗性基因的分布。此外,不同深度土壤的化学性质存在差异,为微生物生存提供的生态位也不同,导致不同深度土壤中微生物群落结构有显著差异。除微生物群落结构外,基因水平转移机制(Horizontal Gene Transfer,HGT)也是影响抗性基因传播的重要因素。畜禽粪便中残留大量的可移动基因元件,如质粒、整合子和转座子等,粪源抗性基因能与可移动基因元件相结合,通过HGT 转移到土著微生物中且能够长期存活。已有研究将整合酶基因作为表征抗性基因传播潜力的重要指标之一。Gou 等研究发现,基因与、和基因具有显著正相关,促进了抗性基因在环境中的传播。
本研究采集了长期堆放猪粪和鸡粪的0~100 cm土壤,解析畜禽粪便堆放地土壤中抗性基因、基因和微生物群落结构的垂直分布特征; 同时结合土壤化学性质和微生物测序结果,探讨影响畜禽粪便堆放地土壤中抗性基因分布的主导因子,以期为控制养殖场中畜禽粪便堆放地土壤中抗性基因污染提供科学依据,为环境中抗性基因生态风险评价提供参考。
1 材料与方法
1.1 土壤样品采集及处理
本研究分别采集了河北省石家庄市栾城区(37°53′N,114°14′E)养猪场中猪粪(PM)堆放地(约20年)和养鸡场中鸡粪(CM)堆放地(约20年)0~100 cm 土壤,同时采集未堆放畜禽粪便的土壤样品作为对照(CK)。土壤分层为0~10 cm、10~30 cm、30~50 cm、50~70 cm、70~100 cm。每个样点采集3个重复。样品采集完后,置于冰上运回实验室。土壤样品过2 mm 筛后,一部分风干后用于测定土壤化学性质; 另一部分保存于−80 °C 超低温冰箱用于后续分子生物学实验。
1.2 土壤化学性质分析
土壤pH 和有机质、总碳、总氮含量的测定参照《土壤农业化学分析方法》。利用pH 计(PHS-3C,Shanghai INESA)测定土壤pH,土水比为1∶2.5(/); 土壤有机质(soil organic matter,SOM)含量采用低温外热重铬酸钾氧化法,利用分光光度计进行测定(UV-6100S,Shanghai); 土壤总碳(total carbon,TC)和土壤总氮(total nitrogen,TN)含量利用元素分析仪(Vario PYRO cube,Elementar,Germany)测定。
1.3 土壤微生物DNA 提取和实时荧光定量PCR
采用FastDNA Spin Kit for Soil 试剂盒(MP Biomedicals,United States)依照说明提取土壤微生物总DNA。利用超微量分光光度计(NanoDrop One,ThermoFisher Scientific,United States)测定DNA 的纯度和浓度。将DNA 保存于−20 ℃冰箱用于后续分析。
采用SYBR Green 染料法分别定量磺胺类抗性基因(和)、四环素类抗性基因(、、、)和基因的丰度。PCR 反应体系(25 μL)如 下:12.5 μL 2×SYBR Premix Ex Taq(Takara Biotech,Dalian,China),0.25 μL前引物/后引物(10 μmol∙L),1 μL DNA (约20 ng∙μL)和11 μL ddHO。扩增条件:预变性95 ℃ 3 min,35 个循环变性95 ℃ 30 s,退火30 s (55.9 ℃;60.8 ℃;、和55 ℃;和60 ℃),72 ℃延伸3 min,83 ℃采集荧光信号10 s。采用TaqMan 探针法定量细菌16S rRNA 基因丰度。反应体系为25 μL;12.5 μL 2 × Premix Ex Taq(Probe qPCR) (Takara Biotech,Dalian,China),0.5 μL 前引物/后引物(10 μmol∙L),0.6 μL 探针(10 μmol∙L),1 μL DNA (约20 ng∙μL)和9.9 μL ddHO。扩增条件:预变性95 ℃ 2 min; 40个循环变性95 ℃ 15 s,退火56 ℃ 60 s。
1.4 细菌16S rRNA 基因高通量测序与生物信息学分析
为探究土壤细菌群落组成,使用515F/806R 引物对16S rRNA 基因V4 区域进行扩增。扩增产物送往上海派森诺公司,利用Illumina Miseq 平台(Illumina,San Diego,CA,United States)对扩增产物进行测序。
测序数据通过QIIME2 2019.7 版本(Quantitative Insights Into Microbial Ecology)进行分析。利用DADA2 对序列进行如下操作:去除引物、质量过滤、去噪、拼接和去嵌合体,最终生成特征表。使用Silva 数据库对特征表进行注释,经物种过滤、重采样后得到物种不同分类水平上的相对丰度,同时计算细菌群落结构的α 多样性和β 多样性。
1.5 统计分析
采用IBM SPSS Statistic 20.0 软件对土壤化学性质和Shannon 指数进行方差分析。利用R 语言corrplot 包对基因丰度、抗性基因丰度和土壤化学性质进行Pearson 相关性分析。箱线图、条形图和散点图由SigmaPlot 12.5 生成。采用R 语言的pheatmap 包绘制热图。使用R 语言的vegan 包进行方差分解分析(variation partitioning analysis,VPA)、主坐标分析(principal coordinate analysis,PCoA)和置换多元方差分析(permutational multivariate analysis of variance,PERMANOVA)。
2 结果与分析
2.1 土壤中化学性质分析
在0~10 cm 表层土中,猪粪堆放地土壤中pH 和SOM、TC 含量显著高于对照土壤(<0.05),鸡粪堆放地土壤中TC 含量和C/N 值显著高于对照土壤(<0.05)(表1)。猪粪堆放地土壤pH 和SOM 含量显著高于鸡粪堆放地,说明畜禽粪便种类不同,对堆放地土壤化学性质的影响也不同。此外,土壤化学性质随土壤深度呈现不同的变化趋势。猪粪堆放地土壤SOM 和TN 含量、鸡粪堆放地土壤TC 含量随土壤深度增加呈递减趋势,而猪粪堆放地土壤C/N 值随土壤深度增加呈递增趋势。畜禽粪便堆放对表层土壤(0~10 cm)化学性质的影响高于底层土壤(10~100 cm)。

表1 不同畜禽粪便堆放地的土壤化学性质Table 1 Soil chemical properties in different treatments
2.2 土壤中抗性基因和intI1 基因的分布特征
猪粪和鸡粪堆放地0~100 cm 土壤样品中、、、、、以及基因均有检出,而在对照土壤样品中只有和基因在0~100 cm 土层中有检出。猪粪和鸡粪堆放地土壤中、、、、、以及基因的相对丰度与对照土壤样品中显著不同,分级聚类结果显示,所有土壤样品的基因丰度分为两枝,一枝为对照土壤样品,另一枝为堆放猪粪和鸡粪的土壤样品(图1)。猪粪堆放地土壤0~100 cm 样品中抗性基因和基因的相对丰度分别为7.55×10~1.37×10和1.71×10~6.87×10基因拷贝数/16S rRNA 基因拷贝数,鸡粪堆放地土壤0~100 cm 样品中抗性基因和基因的相对丰度分别为1.94×10~1.55×10和1.70×10~3.15×10基因拷贝数/16S rRNA 基因拷贝数,对照土壤0~100 cm 样品中抗性基因和基因的相对丰度分别为未检出~2.07×10和未检出~1.84×10基因拷贝数/16S rRNA 基因拷贝数,其检出丰度均显著低于猪粪和鸡粪堆放地土壤。此外,对照土壤、猪粪和鸡粪堆放地土壤中抗性基因和基因丰度随土壤深度增加呈递减趋势。聚类结果表明,猪粪和鸡粪堆放地土壤中抗性基因和基因丰度也有差异,但基因丰度均主要集中在0~30 cm。土壤样品中不同种类的抗性基因丰度也有差异,其中磺胺类抗性基因和的相对丰度(8.10×10~1.55×10基因拷贝数/16S rRNA 基因拷贝数)高于四环素类抗性基因、、和的相对丰度(1.18×10~2.94×10基因拷贝数/16S rRNA 基因拷贝数)。

图1 不同处理不同深度土壤tetW、tetC、tetG、tetL、sulI、sulII 和intI1 基因的相对丰度(基因拷贝数/16S rRNA 基因拷贝数)Fig.1 Relative abundances (gene copy number/16S rRNA gene copy number) of tetW,tetC,tetG,tetL,sulI,sulII and intI1 genes in different soil depths of different treatments
2.3 土壤中细菌群落结构和多样性分析
通过对54 个样品的测序结果进行质量筛选后得到3 322 558 条序列,平均61 529 条特征序列(Amplicon Sequence Variants,ASVs)。猪粪和鸡粪堆放对土壤细菌群落结构组成产生了不同的影响(图2)。对照0~10 cm 表层土壤中的细菌主要由变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、酸杆菌门(Acidobacteria)、浮霉菌门(Planctomycetes)、绿弯菌门(Chloroflexi)和拟杆菌门(Bacteroidetes)组成,约占细菌群落的68.64%。而猪粪和鸡粪堆放地0~10 cm表层土壤中的细菌主要由变形菌门、放线菌门、绿弯菌门、拟杆菌门和厚壁菌门(Firmicutes),分别约占细菌群落的81.76%和92.71%。与对照土壤相比,猪粪堆放表层土壤中变形菌门、拟杆菌门和厚壁菌门相对丰度分别增加至39.14%、9.29%和12.46%,而鸡粪堆放表层土壤中变形菌门、放线菌门和拟杆菌门相对丰度分别增加到30.32%、32.64%和14.60%。此外,土壤细菌群落组成也受土壤深度的影响。例如,在对照土壤中,浮霉菌门和拟杆菌门相对丰度随土壤深度呈递减趋势,而放线菌门和芽单胞菌门呈递增趋势。在猪粪和鸡粪堆放地土壤中,变形菌门、放线菌门、厚壁菌门等随土壤深度呈现不同的变化趋势。

图2 不同处理不同土层细菌优势门分类水平的相对丰度Fig.2 Relative abundances of the dominant bacterial phyla in different soil layers of different treatments
畜禽粪便堆放和土壤深度影响了土壤细菌的α多样性和β 多样性(图3A 和图3B)。从图3A 可以看出,在0~10 cm 和10~30 cm 土层,猪粪和鸡粪堆放地降低了土壤细菌α 多样性,且猪粪堆放地土壤细菌α 多样性要显著高于鸡粪堆放地土壤。在30~50 cm土层,猪粪和鸡粪堆放地土壤细菌α 多样性与对照土壤无显著差异。而在70~100 cm 土层,只有猪粪堆放土壤中细菌α 多样性与对照土壤相比显著降低。PCoA 主坐标分析结果(图3B)表明,前两轴共解释了34.3%的群落变异。总体上是,对照0~10 cm 和10~30 cm 土壤样品聚在一起(Ⅰ),猪粪堆放地0~10 cm和10~30 cm 样品以及鸡粪堆放地0~10 cm、10~30 cm和30~50 cm 土壤样品聚在一起(Ⅱ),而其余的土壤样品聚在一起。这说明猪粪堆放地0~30 cm 土壤和鸡粪堆放地0~50 cm 土壤与对照土壤样品细菌群落结构的差异相较于深层土壤更为明显。
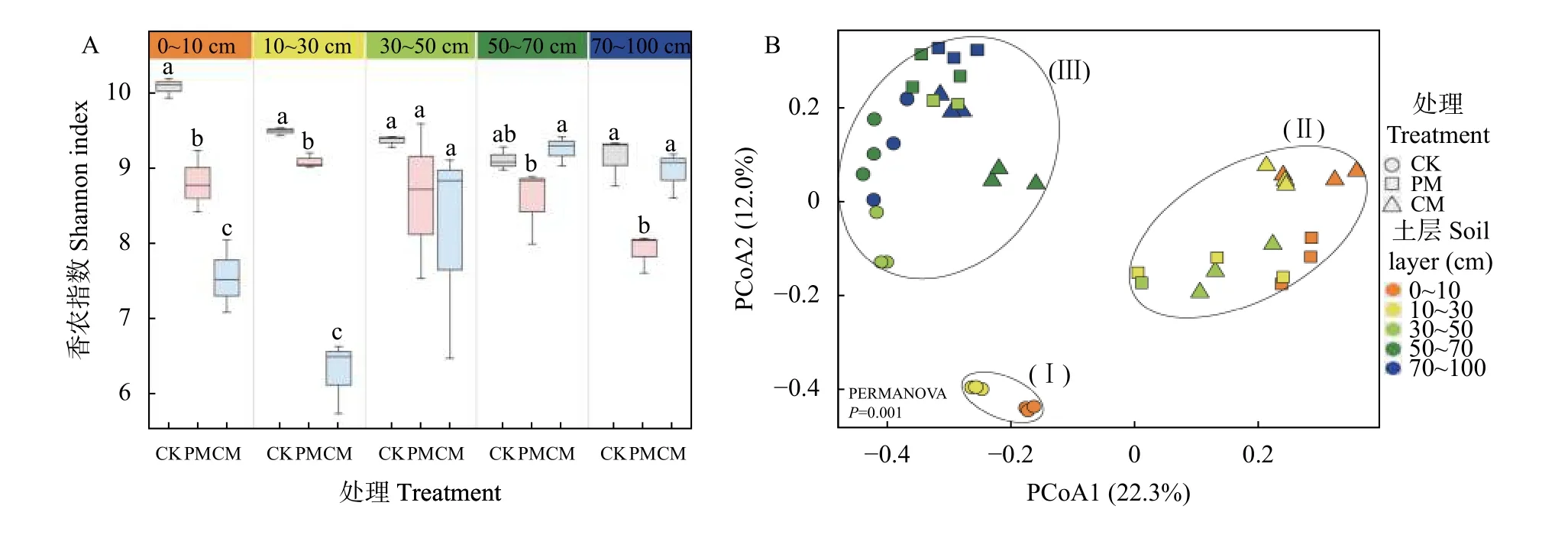
图3 不同处理不同土层细菌α 多样性(A)和基于Bray-Curtis 距离的主坐标分析(PCoA)的细菌群落结构组成(B)Fig.3 Soil bacterial α diversity (A) and principal coordinate analysis (PCoA) based on the Bray-Curtis distance (B) showing the profile of bacterial community structure in different soil layers of different treatments
2.4 影响土壤中抗性基因分布特征的因素
VPA 分析结果表明,土壤化学性质和细菌群落结构对土壤中抗性基因分布的总解释量为45.93%(图4A)。其中,土壤化学性质和细菌群落结构对抗性基因分布的解释量分别是15.91%和37.47%,表明细菌群落结构是影响土壤中抗性基因分布的主要因素(图4A)。进一步通过Pearson 相关性分析发现(图4B),SOM、TC 和TN 分别与、、、、、以及基因丰度呈显著正相关(<0.05),而土壤pH 与、、及基因丰度呈显著正相关(<0.05),这也说明了土壤化学性质的变化影

图4 细菌群落结构和土壤化学性质对抗性基因分布的方差分解分析(VPA) (A)以及intI1 基因丰度、抗性基因丰度和土壤化学性质间的Pearson 相关性分析(B)Fig.4 Effects of bacterial community and soil chemical properties on the variations of resistance genes (A),and Pearson’s correlation among the intI1 gene abundance,resistance gene abundances,and soil chemical properties (B)
响了土壤中抗性基因的变化。值得注意的是,基因丰度分别与(=0.97,<0.001)、(=0.99,<0.001)、(=0.67,<0.01)、(=0.91,<0.001)、(=0.77,<0.001)和(=0.73,<0.01)基因丰度呈显著正相关关系(<0.05),说明基因在抗性基因的传播过程中也起着重要的作用。
3 讨论和结论
本研究在长期堆放猪粪和鸡粪土壤中检测到较高丰度的四环素类抗性基因(、、、)和磺胺类抗性基因(、),说明畜禽粪便直接堆放加剧土壤中抗性基因的污染程度。已有研究表明,畜禽粪便中含有高丰度的抗性基因,其中四环素类抗性基因和磺胺类抗性基因也是猪粪和鸡粪等畜禽粪便中检出率较高的两类抗性基因。Ji 等分析了养猪场、养鸡场和养牛场粪便中抗性基因的丰度,四环素类抗性基因和磺胺类抗性基因的相对丰度分别是10~10基因拷贝数/16S rRNA基因拷贝数和10~10基因拷贝数/16S rRNA 基因拷贝数。Cheng 等定量分析了杭州不同规模养猪场、养鸡场、养鸭场和养羊场粪便中的10 种四环素类抗性基因(、、、、、、、、和)和2 种磺胺类抗性基因(和),结果表明、和基因的相对丰度分别可达7.12×10、1.99×10和3.99×10基因拷贝数/16S rRNA 基因拷贝数,且猪粪中磺胺类抗性基因丰度低于鸡粪。这可能是因为养猪场和养鸡场的养殖密度较大,使得抗生素的使用量相应增加,进而导致猪粪和鸡粪中抗生素的残留和抗性基因的丰度也相应增加。尽管本研究中猪粪和鸡粪堆放地土壤中四环素类和磺胺类抗性基因的丰度与已有研究不尽相同,但总体维持在一个较高的水平,同样会对周边土壤和水体造成潜在危害。
猪粪和鸡粪堆放土壤中抗性基因的分布特征与土壤化学性质和细菌群落结构密切相关,对土壤中抗性基因分布的解释量为45.93%,其中细菌群落结构是影响土壤中抗性基因分布的主要因素,这与前人的研究结果一致。长期堆放猪粪和鸡粪改变了土壤的化学性质,例如pH、SOM 等,进而改变了土壤细菌的群落组成。有研究表明土壤微生物对土壤化学性质的变化非常敏感,不同环境因子在构建细菌群落中的贡献不同,其中pH 和SOM 被认为是影响微生物群落组成的重要因素。土壤化学指标的变化也会造成土壤微生物群落组成的差异。例如,猪粪堆肥和鸡粪堆肥土壤均增加了变形菌门、放线菌门、厚壁菌门和拟杆菌门的相对丰度。Zhang 等研究发现变形菌门、放线菌门、厚壁菌门和拟杆菌门是抗性基因的主要宿主,这些细菌门也被认为是多重耐药类抗性基因的重要宿主。此外,在垂直分布上,猪粪和鸡粪堆放地土壤中四环素类抗性基因和磺胺类抗性基因均随土壤深度呈降低的趋势,但抗性基因的检出率和相对丰度都要高于对照土壤,说明畜禽粪便堆放会导致抗性基因向下层土壤迁移,进而污染下层土壤。Tang 等研究发现,施用猪粪有机肥增加了土壤中和基因的丰度,且抗性基因丰度与土壤深度呈负相关。猪粪和鸡粪有机肥堆放地土壤中抗性基因主要集中在0~10 cm 和10~30 cm 土层,这可能与土壤细菌群落结构相关,因为猪粪和鸡粪堆放地土壤中细菌群落结构在0~10 cm和10~30 cm 土层较为相似。
除土壤化学指标和细菌群落结构外,基因水平转移机制也是影响抗性基因分布的重要因素,其中基因常作为表征抗性基因在环境中传播潜力的指示基因。在本研究中,猪粪和鸡粪堆放地土壤中基因相对丰度比对照土壤高4 个数量级,且基因丰度与、、、呈显著正相关(<0.05),表明基因可能促进了抗性基因在土壤中的传播和扩散。Wang 等在施加畜禽粪便的土壤中同样检测到了较高丰度基因,这可能是残留的基因通过畜禽粪便被引入到土壤中。有机肥堆放显著增加了土壤中基因丰度,可能是有机肥中大量的可移动基因元件被引入土壤中。畜禽粪便堆放可能使得土壤微生物通过基因水平转移机制,加速了抗性基因在土壤中的传播扩散,并对土壤生态系统造成潜在的环境风险。此外,基因丰度随土壤深度增加呈递减趋势,说明表层土壤中抗性基因发生基因水平转移的可能性更大,抗性基因传播扩散的潜力要高于深层土壤。
综上所述,长期堆放猪粪和鸡粪增加了土壤中四环素类和磺胺类抗性基因的丰度,且增加了抗性基因向下层土壤迁移的风险。猪粪和鸡粪堆放地土壤中抗性基因主要集中在0~10 cm 和10~30 cm 土层。土壤化学性质和细菌群落结构是影响抗性基因垂直分布特征的重要因素。此外,猪粪和鸡粪堆放增加了土壤中基因的丰度,基因在抗性基因传播中起着重要的作用。本研究结果为评估畜禽粪便堆放对周边环境抗性基因污染风险提供了重要理论依据。后续研究也应积极探索养殖场中畜禽粪便产生、处置和利用各环节的畜禽粪便管控方式,降低抗性基因和可移动基因元件在养殖场内向周围环境传播扩散的风险。

