表观“阅读器”BET蛋白家族对哺乳动物发育和iPSC重编程的调控
张祉靖,乔钰,孙宇晨,雷蕾
表观“阅读器”BET蛋白家族对哺乳动物发育和iPSC重编程的调控
张祉靖,乔钰,孙宇晨,雷蕾
哈尔滨医科大学组织学与胚胎学教研室,哈尔滨 150081
溴结构域和超末端结构域(bromodomain and extra-terminal, BET)蛋白家族作为表观“阅读器”,在哺乳动物发育过程中起着至关重要的作用。其家族内的各成员通过识别各种表观修饰并募集相应的功能复合物,对相关基因进行精密调控,促使早期胚胎向特定方向分化和发育。另外,随着诱导性多潜能干细胞(induced pluripotent stem cell, iPSC)重编程技术发展,越来越多的研究发现BET蛋白家族在体细胞重编程中可能也占据着核心地位。本文总结了BET蛋白家族在哺乳动物发育和iPSC重编程中的作用,并对BET家族调控重编程的新机制进行了展望。
BET蛋白;哺乳动物发育;诱导性多潜能干细胞
哺乳动物的发育始于精子和卵子结合,两个高度分化的生殖细胞结合成为具有全能性的合子[1]。随后一系列发育的关键事件相继发生,包括卵母细胞活化、母源–合子转换(maternal-to-zygotic transition, MZT)、合子基因激活(zygotic gene activation, ZGA)、第一次细胞命运决定和谱系特异性分化等[2~6]。这些复杂且重要的事件是在精密“网络”的调控下快速且有序地发生,而表观调控正是这一“网络”中的关键内容。在其调控下各基因依次开放或关闭,使胚胎朝着特定方向生长发育。通常将参与表观调控的关键蛋白称为“编辑器”、“清除器”和“阅读器”。“编辑器”和“清除器”一般负责沉积和去除翻译后修饰(post-translation modification, PTMs),而“阅读器”则负责识别这些标记[7]。含溴结构域(bromodomains, BDs)的蛋白分子是“阅读器”中最具代表性的一类,能够快速且特异性的识别基因组中乙酰化的位点[8]。
目前将含有BDs的61种分子分为8个不同的家族[9],溴结构域和超末端结构域(bromodomain and extra-terminal, BET)蛋白家族属于BDs家族第二类亚家族,其成员包括:BRD2、BRD3、BRD4以及BRDT。BET蛋白家族通过BDs能够高效且准确地识别并结合其天然配体—乙酰化H3、H4,并发挥不同的功能来维持胚胎正常发育。除此之外,目前越来越多的证据提示在诱导性多潜能干细胞(induced pluripotent stem cell, iPSC)重编程中,BET蛋白家族可能也以类似的机制促进重编程的进行。iPSC重编程自Yamanaka首次提出以来,经历了迅猛发展,目前已快速地应用于多个研究领域。然而,其中详细的机制至今还不是十分清楚。因此,本文对BET蛋白家族在胚胎发育和iPSC重编程中的功能进行了综述,以期帮助人们更加深入地理解BET蛋白家族的功能和作用,并为BET蛋白家族在iPSC重编程中的研究带来新的启示。
1 翻译后修饰与表观“阅读器”
表观遗传的概念最早由奥地利发育生物学家Conrad Waddington提出,主要用于描述基因及其产物间的相互作用,以及对生物表型的影响[10]。后来,这一概念扩展为在不改变DNA序列的情况下,基因表达的可遗传变化[11]。位于组蛋白上的各种PTMs是表观遗传学研究的重要内容[12]。几乎每一种组蛋白修饰,即“组蛋白密码”,都可以影响染色质结构。这些修饰相互交织,共同决定染色质的整体状态[8]。
组蛋白的PTMs在维持基因组完整性、调控转录以及形成表观记忆等方面发挥重要作用[13]。组蛋白上的PTMs大体分为两类:位于球状结构域的修饰和位于组蛋白尾的修饰[14,15]。球状结构域的PTMs能够直接影响基因的转录和核小体的结构。例如,核心组蛋白上的乙酰化修饰能够减弱组蛋白和DNA间的相互作用,促进核小体解离[16]。位于组蛋白尾的PTMs则依赖表观“阅读器”识别并募集各种复合物来发挥作用[17,18]。而表观“阅读器”通过特异性识别并结合各种PTMs,使自身能够更加精准并持久地靶向、维持和调控染色体修饰,同时帮助其他调控蛋白到达特定的基因位点并发挥相应作用。依靠这些功能和特征,表观“阅读器”调控着众多基因的表达。例如,DNA上组蛋白H3K9三甲基化(H3K9me3)形成后,人源沉默中心(human silencing hub, HUSH)复合物识别并与之结合。随后,HUSH进一步募集甲基转移酶SETDB1,促进H3K9me3沉积,从而抑制靶基因的表达[19,20]。表观“阅读器”具有不同结构特征,但每种蛋白质都至少含有一个或多个在进化上保守的效应元件。通过这些效应元件,它们能够识别基因组上的各种共价修饰[8]。在过去几十年里,通过生物化学和生物物理学分析已经发现了大量的这类元件,它们特异性结合组蛋白PTMs并发挥相应作用[21]。例如:BDs、PHD finger结构域、PWWP结构域、Tudor结构域等。其中,BDs是唯一能够特异性识别e-N乙酰的赖氨酸位点的效应元件[8]。
2 BET蛋白家族在哺乳动物发育中的作用
2.1 BDs蛋白与BET蛋白家族
BDs最初是在染色体相关蛋白(例如组蛋白乙酰转移酶),以及某些在转录活化和染色质重塑中起关键作用的重塑复合物中发现的[22]。BDs结构保守,共含有120个氨基酸,具有4个左手α螺旋(αZ、αA、αB和αC)和2个分别连接αZ和αA (ZA环)以及αB和αC (BC环)的loop环。作为BDs亚家族,BET蛋白家族拥有两个串联的BDs、一个超末端结构域(ET)以及仅BRD4和BRDT末端含有的羧基区域(carboxyl-terminal repeat domain, CTD)[8](图1)。BET蛋白家族利用BDs识别并结合组蛋白上乙酰化的赖氨酸,调控众多基因的表达和功能,作用于哺乳动物的生长发育。
2.2 BET蛋白家族对胚胎发育的影响
BRDT仅在睾丸组织中表达,调控精原细胞的减数分裂及其后续的基因组重组。在敲除基因的雄性小鼠中,精子数量显著减少同时伴有异常形态[23,24]。BRDT是BET蛋白家族中唯一拥有与BRD4相似CTD的蛋白[25],BRDT能够和BRD4一样募集正性转录延伸因子b (positive transcription elongation factor b, P-TEFb),促进多种生精基因的表达(图2A)。因此,也将BRDT称为“BRD4样的组织特异性类似物”[26~29]。减数分裂开始后,BRDT识别并结合睾丸特异性基因的转录起始位点(transcription start site, TSS)区域。通过募集P-TEFb,推动靶基因进入转录延伸阶段,促进基因表达[27]。之后,精子为了达到受精的目的,会进行结构上的特化,例如过渡蛋白(transition protein, TP)和鱼精蛋白替换组蛋白[30]。BRDT识别并结合到高乙酰化的组蛋白上,随后相邻BRDT分子“挤压”染色体,促使组蛋白脱落并被过渡蛋白和鱼精蛋白替换[27]。BRDT通过辅助鱼精蛋白对组蛋白替换,促进精子细胞核中染色体浓缩,增强精子的灵活性并保护内部遗传物质。
BRD4是BET家族蛋白中研究最清晰的一个成员。BRD4含有1362 aa,属于长亚型,其结构主要包括2个BDs、1个CTD和1个ET (图1)。通过可变剪接还可产生1个722 aa的短亚型a。该亚型保留了2个BDs,但缺少CTD。目前还发现第3种亚型,即短亚型b,其结构类似于短亚型a,但另外含有76 aa的CTD[31,32]。基因早在植入前胚胎中就开始表达,维持着早期胚胎正常的结构和功能[33~35]。在植入前胚胎中抑制BRD4后,尽管仍然能够发育到囊胚但表现出严重的发育迟缓[33]。在植入后胚胎中,基因完全敲除小鼠胚胎,由于内细胞团(inner cell mass, ICM)的退化,导致胚胎在E6天时死亡;半敲除小鼠胚胎虽然可以出生,但在出生前后均表现出严重的生长缺陷,同时伴有颅骨畸形、皮下脂肪缺失、白内障等器官病变[34]。
最初发现BRD2时,认为其是人类细胞中的核激酶,通过与多个核转录因子E2Fs结合调控细胞周期基因表达[36~38]。目前发现BRD2还可以调控胚胎发育和神经系统发育相关基因。在E9.5天小鼠胚胎的前脑、中脑以及后脑中均可检测到高水平的BRD2[39]。而基因缺失的小鼠胚胎表现出神经管闭合缺陷、神经系统发育迟缓以及细胞增殖能力下降等缺陷,并在E12.5天左右死亡[40]。BRD2通过与E2Fs相互作用,还可以对细胞周期基因、和进行调控,促进G1期向S期过渡[41]。作为BET家族成员,BRD2和BRD4在功能上具有相似之处。LeRoy等[42]研究发现,BRD2通过引导RNA聚合酶II(Pol II)定位于高乙酰化的转录位点,也可以对mRNA、microRNA的转录进行调控[42](图2B)。

图1 BET蛋白家族4个主要成员的分子结构
数字代表每个已知结构域的氨基酸位点。BD1、BD2为两个溴结构域,ET为超末端家族,CTD代表BRD4和BRDT末端的羧基区域。
在BET蛋白家族成员中,BRD2和BRD3具有更高的相似性。都通过不依赖P-TEFb的方式与Pol II结合,调控胚胎干细胞(embryonic stem cell, ESC)相关基因的表达[39,43](图2B)。在造血系统发育过程中,BRD3与GATA1结合,并被募集到多个红系成熟相关基因上,促进红系分化[43]。干扰BRD3和GATA1结合后,不仅会显著降低这两种蛋白在红系基因上的富集,还会影响GATA1介导的红系成熟[44]。然而,大量负向转录因子和染色体重构蛋白,如核小体重构复合体(nucleosome remodeling complex, NuRD),也会优先同BRD3进行结合[43]。BRD3与NuRD间的相互作用,甚至对GATA1介导的红系成熟也会产生影响[44,45]。因此,BRD3虽然能够和GATA1一起促进造血系统发育,但也可以与许多负向调控因子结合,干扰GATA1介导的红系发育。综上所述,BRD3在胚胎发育过程中具有复杂的功能,一方面维持着造血系统的正常发育;另一方面,可能也干扰多种基因的表达,抑制细胞增殖。
3 BRD4对胚胎发育的调控机制
3.1 BRD4与靶基因转录
作为表观“阅读器”,BRD4通过BDs识别并结合乙酰化位点。随后,BRD4发挥组蛋白乙酰转移酶(HAT)活性,与其他表观修饰酶一起促进核小体解聚[46,47]。随着核小体解离,BRD4进入到“开放”的染色质中,同增强子、启动子、TSS处乙酰化的组蛋白结合并发挥支架作用。在这些组蛋白位点,BRD4募集大量功能复合物,例如转录中介体(mediator, MED)、转录因子以及Pol II等,进而维持基因的正常表达[48,49]。BRD4利用CTD结合P-TEFb,磷酸化Pol II的第五位丝氨酸(Ser5),促进转录启动[50,51]。在转录因子的辅助下,Pol II向TSS移动。到达TSS下游约100 bp处时,停滞于此处[52,53]。此时,BRD4结合并活化P-TEFb,磷酸化Pol II的第二位丝氨酸(Ser2),促进Pol II释放,使转录进入延伸阶段[53](图2A)。BRD4以这样的方式活化多能性基因,维持ESCs的自我更新能力和多能性。相反,干扰表达后,小鼠植入前胚胎中以为代表的多能性基因表达显著下降。这些干扰了表达的胚胎尽管仍可以发育到囊胚且保持较为正常的形态特征,但胚胎发育迟缓且ICM的大小较正常显著缩小[33]。总之,BRD4对多能性基因的转录具有强大的调控作用,对维持哺乳动物胚胎的正常发育不可或缺。
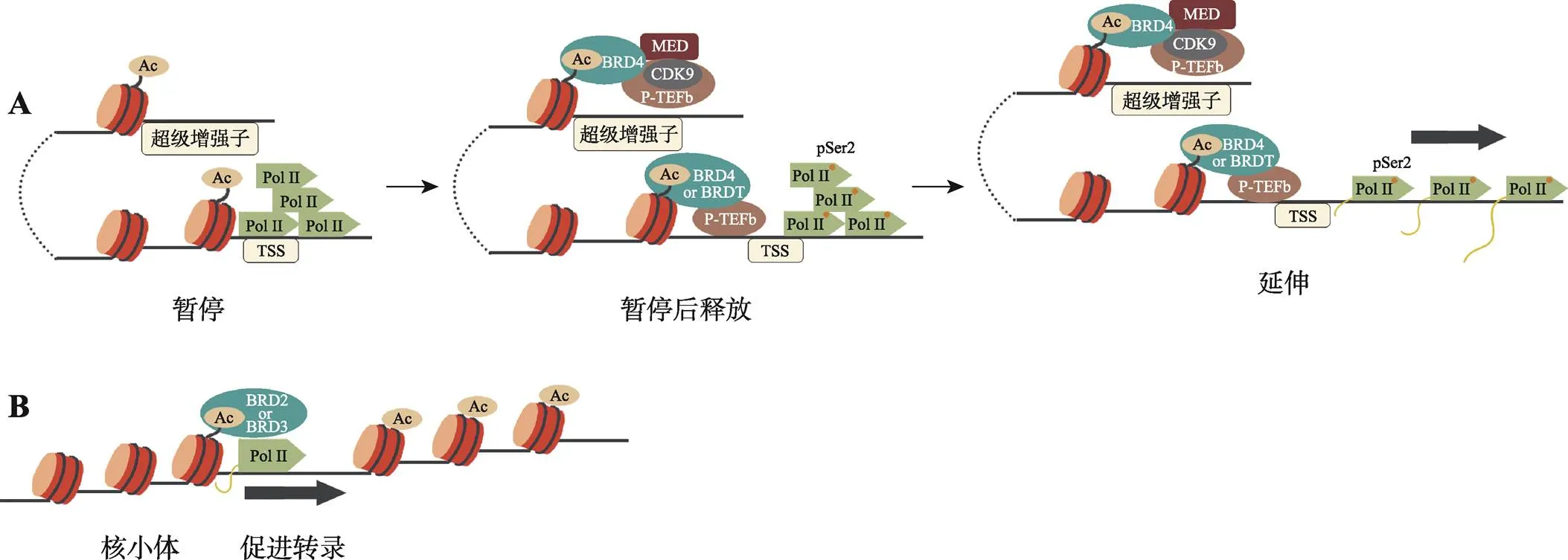
图2 BET蛋白推进转录模式图
A:BRD4和BRDT推进转录模式图。当Pol II在TSS附近暂停后,BRD4和BRDT依靠分子末端的羧基结构域(CTD)募集并结合正性转录延长因子b (P-TEFb),促进Pol II中第二位丝氨酸(Ser2)磷酸化。与此同时,BRD4还可以与超级增强子(super enhancer, SEs)结合,并募集转录中介体(mediator, MED)和P-TEFb的催化亚基CDK9。BRD4和BRDT通过以上方式推动Pol II释放,并使转录进入延长阶段;B:BRD2和BRD3推进转录模式图。BRD2和BRD3作为Pol II伴侣,识别基因组上乙酰化的组蛋白并帮助Pol II结合到基因组上促进转录。
3.2 BRD4与超级增强子
BRD4还可以结合在基因组中的一些“特殊区域”上。这些区域跨越的长度以及含有的转录因子密度远超一般增强子。一般增强子长度约为100 bp,而这些区域增强子的长度可达到近50 kb。因此,将这些功能远超一般增强子的“特殊区域”称作超级增强子(super enhancer, SEs)[54]。SEs内部含有大量由BRD4、Mediator、转录因子、乙酰化组蛋白等成员组装而成的增强子元件,因此可以辅助靶基因产生更高的转录水平[55]。Whyte等[56]最初在ESC中的多能性基因上发现SEs结构,这些SEs通过促进多能性基因表达,维持ESC的干性特征。
在ESC中,BRD4结合到多能性基因(例如和)的SEs内部,发挥“支架”作用并募集Mediator来辅助干性基因的转录。除此之外,当BRD4位于SEs中时,它还可以募集并结合P-TEFb的成员—细胞周期依赖蛋白激酶9 (CDK9),促进靶基因进入转录延伸阶段[57,58](图2A)。当抑制BRD4后,其不再同核心多能性基因的SEs结合,造成大量Mediator (例如MED1和MED12)无法结合到SEs上,引起多能性基因转录受限。除此之外,抑制BRD4后,ChIP分析还发现,启动子和SEs中的CDK9大量丢失,Pol II在TSS区域停滞,基因体中的Pol II数量显著下降。此时,这些多能性基因无法进入到转录延伸阶段,最终导致ESC干性丢失,且易于分化[57]。
3.3 BRD4与有丝分裂
BRD4通过促进早期胚胎细胞有丝分裂来维持胚胎正常的体积和重量。在有丝分裂过程中,大量基因被暂时沉默。此时,转录因子从染色体上解离,而BRD4是少数仍可与染色体结合的蛋白分子。在小鼠胚胎成纤维细胞(mouse embryonic fibroblasts, MEF)有丝分裂中,BRD4识别并结合M/G1期基因[59,60],诱导BRD4结合区域周围的染色体解聚,促进DNA复制,推动细胞从G1期进入M期[61]。当细胞进入有丝分裂后,基因转录被关闭,但大量的H4K5ac作为有丝分裂后重建转录的基因“标签”而被保留下来。有丝分裂结束后,子代细胞中的BRD4识别并结合基因组上的H4K5ac,促使结合的基因位点“开放”。之后BRD4进一步募集相应的转录复合物,例如CDK9、Pol II等,使靶基因以更高效的水平恢复转录[26]。因此,该过程也被称为BRD4介导的“转录记忆”。
在其他一些细胞系中,例如HeLa细胞以及NIH3T3细胞,还发现BRD4通过负向调控SPA-1促进G2期向M期的转变,或者促进Aurora B激酶的表达来推动有丝分裂后期染色体分离和胞质分裂[62,63]。在HeLa细胞和NIH3T3细胞中敲除基因后,不仅会使细胞阻滞在G1期,还导致细胞衰老和凋亡[34,64,65]。虽然该机制在ESC中还未得到验证,但当部分敲除基因后,小鼠胚胎在E10天时,其平均体重显著低于野生型小鼠,多种器官的重量和MEF增殖率也显著降低和下降[34]。表明BRD4可以通过调控有丝分裂,维持胚胎正常的生长发育。
4 BET家族与iPSC重编程
BET蛋白家族对生物体的生长发育具有广泛的调控作用,iPSC重编程作为正常分化发育的一个逆向过程,BET蛋白在该过程中可能也占据着重要地位。iPSC由Yamanaka于2006年提出,通过过表达、、、(OSKM)可以实现将体细胞重编程为iPSC。然而从体细胞跨越到iPSC不是一蹴而就的,其内部包含着多个事件依次有序的发生以及不同分子间的相互作用[66,67]。目前对于BET蛋白家族在iPSC重编程中的研究还处于探索阶段,且主要局限于BRD4。在ESC中,BRD4介导的Pol II释放对多能性基因的转录至关重要[48]。在OSKM诱导的重编程中也存在类似机制,体细胞重编程依赖着众多多能性基因的重新表达,例如、、、等。这些多能性基因在转录过程中,Pol II同样会在TSS附近停滞。BRD4通过CTD募集并活化P-TEFb,促进Pol II释放,有利于多能性基因表达[68]。当在重编程晚期使用JQ1 (BET蛋白家族抑制剂)抑制BRD4后,具有完全多能性的iPSC克隆数量显著减少,同时伴有多能性基因表达下降[68]。相反,在MEF中过表达BRD4后,iPSC的建系效率明显增加,表明BRD4调控着iPSC重编程的正常进行。
除了促进Pol II释放,BRD4通过与众多ESC特异性基因的SEs (ES-SEs)结合也能够推动重编程的进行。在C/EBPα和OSKM介导的高效重编程中,BRD4、MED1、CDK9的表达水平显著升高,并形成BRD4-MED1-CDK9复合物[69]。C/EBPα将其募集到ES-SEs上,通过促进染色体重构、推动Pol II释放等作用,增强多能性基因转录并促进iPSC重编程[69]。相反,如果在重编程前用JQ1和C/EBPα同时处理细胞,或在随后的重编程中同时添加OSKM和JQ1,重编程效率将显著降低[69]。其原因可能是JQ1使BRD4从染色体上脱落,导致C/EBPα无法顺利将BRD4募集到多能性基因上。
除了上调多能性基因的表达,BRD4可能还通过抑制谱系分化基因来维持多能性。受BRD4调控的不仅能够维持多能性,还可以阻遏神经外胚层基因的表达,将ESC维持在未分化状态[70]。抑制BRD4后,表达相应下调,此时ESCs易于向神经外胚层细胞分化。相反,在接受BET抑制剂处理后的ESCs中过表达,可以再次抑制上皮间质转化(epithelial-mesenchymal transition, EMT)和神经外胚层标记的诱导[57]。这些结果表明BRD4一方面维持着多能性基因的表达,另一方面还抑制一些促分化基因或谱系特异性基因的表达,维持ESC的未分化状态。敲除基因后,未分化细胞减少,同时伴有滋养层标志基因和上调[33]。虽然目前只在ESCs中验证了这一现象,还未在iPSC中得到直接证据,但iPSC和ESCs极为相似,因此我们有理由相信,这可能也是BRD4维持iPSC多能性的潜在机制。另外,还有研究发现在重编程早期,通过使用JQ1抑制BET蛋白可以帮助许多体细胞基因关闭,从而促进重编程效率[71]。然而,目前并无直接证据确定是何种BET蛋白调控这一过程。因此,我们猜测在iPSC重编程过程中,早期抑制BRD4可以辅助体细胞基因关闭,而在中晚期过表达BRD4则有利于抑制谱系基因转录,促进多能性基因表达,提高重编程效率。
除了BRD4,还发现其他一些BET蛋白在重编程中也发挥着重要作用。Yamanaka[72]曾提出重编程只会发生在细胞群体中少数具有特定倾向的细胞中,这些具有特定倾向的细胞被称为“精英细胞”。因此,重编程效率的高低一定程度上取决于“精英细胞”的数量。BRD3R是BRD3的一个亚型,在重编程早期阶段,BRD3R可以上调有丝分裂相关基因的表达,显著增加“精英细胞”的数量从而促进重编程[73]。有丝分裂活跃的细胞具有更高的重编程效率[74,75],可能是因为在有丝分裂过程中众多体细胞相关转录因子从染色体上解聚,为多能性转录因子提供了更多与靶基因结合的机会。BRD3R通过促进有丝分裂可能也为相关转录因子提供了结合位点,促进多能性基因表达。
5 结语与展望
BET蛋白家族含有4个成员:BRD2、BRD3、BRD4和睾丸特异性的BRDT,它们都拥有相似的BDs。通过BDs,BET蛋白识别并结合基因组上乙酰化位点。BET蛋白家族具有广泛的生物学功能,参与生物体多种不同的生理和病理过程。尽管每个成员调控的过程不同,但每种蛋白都发挥着不可替代的作用。BRD4是目前研究最广泛的一个成员,通过多种机制确保胚胎稳定而有序地发育。当BRD4缺失或者功能异常时,将导致胚胎早期致死。除此之外,目前发现BRD3同rDNA可能也有潜在关系。核糖体是细胞蛋白质合成的主要场所,其功能依赖于rDNA的表达。一旦rDNA的表达受到抑制,会严重影响蛋白质的合成,威胁细胞的生存和增殖。当使用BET抑制剂JQ1后,BRD3同rDNA结合,抑制rRNA的转录[43],干扰蛋白质合成。Leroy等[43]研究表明BRD3具有抗增殖作用,很可能与其对rDNA的抑制相关。BRD3具体是如何调控哺乳动物的胚胎发育?各通路之间是如相互拮抗或协同?还需要更多研究。
BRD4可通过促进Pol II暂停后释放、结合多能性基因的SEs等来推动重编程。但目前并未探究过BRD4是否可以通过调控细胞周期来促进重编程。已有研究证实活跃的有丝分裂能够有力地推动重编程[74,75],且已发现同为BET蛋白家族成员的BRD3R可以通过活化有丝分裂来促进重编程。那么,同样具有活化有丝分裂作用的BRD4是否也可以通过这一机制来促进重编程呢?另外,在许多肿瘤细胞中发现,上游结合因子(upstream binding factor 1, UBF)结合到rDNA上后,会与LYAR形成复合物并募集BRD4。通过BRD4的乙酰化作用,促进RNA Pol I介导的rDNA转录[76]。该结果表明,BRD4除了已知的功能外,其对rDNA可能也具有调控作用。本实验室以往的研究表明,rDNA的转录活性对核移植胚胎的早期发育以及iPSC重编程过程均有重要影响[77,78]。因此,我们猜测在iPSC诱导过程中,BRD4也许能够通过活化rDNA来促进重编程过程。对BET家族在体细胞重编程中作用的探索不仅对再生医学、药物研究等众多领域的发展有着重要意义,同时也为iPSC在临床上的应用奠定基础。
[1] Rivera RM, Ross JW. Epigenetics in fertilization and preimplantation embryo development., 2013, 113(3): 423–432.
[2] Amdani SN, Yeste M, Jones C, Coward K. Sperm factors and oocyte activation: current controversies and considerations., 2015, 93(2): 50, 1–8.
[3] Minami N, Suzuki T, Tsukamoto S. Zygotic gene activation and maternal factors in mammals., 2007, 53(4): 707–715.
[4] Tadros W, Lipshitz HD. The maternal-to-zygotic transition: a play in two acts., 2009, 136(18): 3033– 3042.
[5] Mihajlović AI, Bruce AW. The first cell-fate decision of mouse preimplantation embryo development: integrating cell position and polarity., 2017, 7(11): 170210.
[6] Yao CM, Zhang WH, Shuai L. The first cell fate decision in pre-implantation mouse embryos., 2019, 8(2): 51–57.
[7] Zaware N, Zhou MM. Chemical modulators for epigenome reader domains as emerging epigenetic therapies for cancer and inflammation., 2017, 39: 116–125.
[8] Filippakopoulos P, Knapp S. The bromodomain interaction module., 2012, 586(17): 2692–2704.
[9] Filippakopoulos P, Picaud S, Mangos M, Keates T, Lambert JP, Barsyte-Lovejoy D, Felletar I, Volkmer R, Müller S, Pawson T, Gingras AC, Arrowsmith CH, Knapp S. Histone recognition and large-scale structural analysis of the human bromodomain family., 2012, 149(1): 214–231.
[10] Waddington CH. Preliminary notes on the development of the wings in normal and mutant strains of., 1939, 25(7): 299–307.
[11] Holliday R. The inheritance of epigenetic defects., 1987, 238(4824): 163–170.
[12] Soshnev AA, Josefowicz SZ, Allis CD. Greater than the sum of parts: complexity of the dynamic epigenome., 2016, 62(5): 681–694.
[13] Jenuwein T, Allis CD. Translating the histone code., 2001, 293(5532): 1074–1080.
[14] Lawrence M, Daujat S, Schneider R. Lateral thinking: how histone modifications regulate gene expression., 2016, 32(1): 42–56.
[15] Villaseñor R, Baubec T. Regulatory mechanisms governing chromatin organization and function., 2021, 70: 10–17.
[16] Ura K, Kurumizaka H, Dimitrov S, Almouzni G, Wolffe AP. Histone acetylation: influence on transcription, nucleosome mobility and positioning, and linker histone- dependent transcriptional repression., 1997, 16(8): 2096–2107.
[17] Musselman CA, Lalonde ME, CôtéJ, Kutateladze TG. Perceiving the epigenetic landscape through histone readers., 2012, 19(12): 1218–1227.
[18] Patel DJ. A structural perspective on readout of epigenetic histone and DNA methylation marks., 2016, 8(3): a018754.
[19] Villaseñor R, Pfaendler R, Ambrosi C, Butz S, Giuliani S, Bryan E, Sheahan TW, Gable AL, Schmolka N, Manzo M, Wirz J, Feller C, Von Mering C, Aebersold R, Voigt P, Baubec T. ChromID identifies the protein interactome at chromatin marks., 2020, 38(6): 728–736.
[20] Tchasovnikarova IA, Timms RT, Matheson NJ, Wals K, Antrobus R, Göttgens B, Dougan G, Dawson MA, Lehner PJ. GENE SILENCING. Epigenetic silencing by the HUSH complex mediates position-effect variegation in human cells., 2015, 348(6242): 1481–1485.
[21] Taverna SD, Li HT, Ruthenburg AJ, Allis CD, Patel DJ. How chromatin-binding modules interpret histone modifications: lessons from professional pocket pickers., 2007, 14(11): 1025–1040.
[22] Zeng L, Zhou MM. Bromodomain: an acetyl-lysine binding domain., 2002, 513(1): 124–128.
[23] Matzuk MM, Mckeown MR, Filippakopoulos P, Li QL, Ma L, Agno JE, Lemieux ME, Picaud S, Yu RN, Qi J, Knapp S, Bradner JE. Small-molecule inhibition of BRDT for male contraception., 2012, 150(4): 673–684.
[24] Berkovits BD, Wolgemuth DJ. The first bromodomain of the testis-specific double bromodomain protein Brdt is required for chromocenter organization that is modulated by genetic background., 2011, 360(2): 358–368.
[25] Bisgrove DA, Mahmoudi T, Henklein P, Verdin E. Conserved P-TEFb-interacting domain of BRD4 inhibits HIV transcription., 2007, 104(34): 13690–13695.
[26] Zhao R, Nakamura T, Fu Y, Lazar Z, Spector DL. Gene bookmarking accelerates the kinetics of post-mitotic transcriptional re-activation., 2011, 13(11): 1295–1304.
[27] Gaucher J, Boussouar F, Montellier E, Curtet S, Buchou T, Bertrand S, Hery P, Jounier S, Depaux A, Vitte AL, Guardiola P, Pernet K, Debernardi A, Lopez F, Holota H, Imbert J, Wolgemuth DJ, Gérard M, Rousseaux S, Khochbin S. Bromodomain-dependent stage-specific male genome programming by Brdt., 2012, 31(19): 3809–3820.
[28] Wang L, Wolgemuth DJ. BET protein BRDT complexes with HDAC1, PRMT5, and TRIM28 and functions in transcriptional repression during spermatogenesis., 2016, 117(6): 1429–1438.
[29] Manterola M, Brown TM, Oh MY, Garyn C, Gonzalez BJ, Wolgemuth DJ. BRDT is an essential epigenetic regulator for proper chromatin organization, silencing of sex chromosomes and crossover formation in male meiosis., 2018, 14(3): e1007209.
[30] Yanagimachi R. Male gamete contributions to the embryo., 2005, 1061: 203–207.
[31] Devaiah BN, Gegonne A, Singer DS. Bromodomain 4: a cellular Swiss army knife., 2016, 100(4): 679–686.
[32] Floyd SR, Pacold ME, Huang QY, Clarke SM, Lam FC, Cannell IG, Bryson BD, Rameseder J, Lee MJ, Blake EJ, Fydrych A, Ho R, Greenberger BA, Chen GC, Maffa A, Del Rosario AM, Root DE, Carpenter AE, Hahn WC, Sabatini DM, Chen CC, White FM, Bradner JE, Yaffe MB. The bromodomain protein Brd4 insulates chromatin from DNA damage signalling., 2013, 498(7453): 246– 250.
[33] Liu W, Stein P, Cheng X, Yang W, Shao NY, Morrisey EE, Schultz RM, You J. BRD4 regulates Nanog expression in mouse embryonic stem cells and preimplantation embryos., 2014, 21(12): 1950–1960.
[34] Houzelstein D, Bullock SL, Lynch DE, Grigorieva EF, Wilson VA, Beddington RS. Growth and early postimplantation defects in mice deficient for the bromodomain- containing protein Brd4., 2002, 22(11): 3794–3802.
[35] Nagashima T, Maruyama T, Furuya M, Kajitani T, Uchida H, Masuda H, Ono M, Arase T, Ozato K, Yoshimura Y. Histone acetylation and subcellular localization of chromosomal protein BRD4 during mouse oocyte meiosis and mitosis., 2007, 13(3): 141–148.
[36] Beck S, Hanson I, Kelly A, Pappin DJ, Trowsdale J. A homologue of thefemale sterile homeotic (fsh) gene in the class II region of the human MHC., 1992, 2(4): 203–210.
[37] Florence B, Faller DV. You bet-cha: a novel family of transcriptional regulators., 2001, 6: D1008– D1018.
[38] Denis GV, Green MR. A novel, mitogen-activated nuclear kinase is related to adevelopmental regulator., 1996, 10(3): 261–271.
[39] Shang EY, Wang XY, Wen DC, Greenberg DA, Wolgemuth DJ. Double bromodomain-containing gene Brd2 is essential for embryonic development in mouse., 2009, 238(4): 908–917.
[40] Gyuris A, Donovan DJ, Seymour KA, Lovasco LA, Smilowitz NR, Halperin AL, Klysik JE, Freiman RN. The chromatin-targeting protein Brd2 is required for neural tube closure and embryogenesis., 2009, 1789(5): 413–421.
[41] Denis GV, Mccomb ME, Faller DV, Sinha A, Romesser PB, Costello CE. Identification of transcription complexes that contain the double bromodomain protein Brd2 and chromatin remodeling machines., 2006, 5(3): 502–511.
[42] Leroy G, Rickards B, Flint SJ. The double bromodomain proteins Brd2 and Brd3 couple histone acetylation to transcription., 2008, 30(1): 51–60.
[43] Lambert JP, Picaud S, Fujisawa T, Hou HY, Savitsky P, Uuskula-Reimand L, Gupta GD, Abdouni H, Lin ZY, Tucholska M, Knight JDR, Gonzalez-Badillo B, St-Denis N, Newman JA, Stucki M, Pelletier L, Bandeira N, Wilson MD, Filippakopoulos P, Gingras AC. Interactome rewiring following pharmacological targeting of BET bromodomains., 2019, 73(3): 621–638 e617.
[44] Lamonica JM, Deng WL, Kadauke S, Campbell AE, Gamsjaeger R, Wang HX, Cheng Y, Billin AN, Hardison RC, Mackay JP, Blobel GA. Bromodomain protein Brd3 associates with acetylated GATA1 to promote its chromatin occupancy at erythroid target genes., 2011, 108(22): E159–168.
[45] Kulikowski E, Rakai BD, Wong NCW. Inhibitors of bromodomain and extra-terminal proteins for treating multiple human diseases., 2021, 41(1): 223–245.
[46] Wu T, Kamikawa YF, Donohoe ME. Brd4's bromodomains mediate histone H3 acetylation and chromatin remodeling in pluripotent cells through P300 and Brg1., 2018, 25(7): 1756–1771.
[47] Greer CB, Tanaka Y, Kim YJ, Xie P, Zhang MQ, Park IH, Kim TH. Histone deacetylases positively regulate transcription through the elongation machinery., 2015, 13(7): 1444–1455.
[48] Jang MK, Mochizuki K, Zhou MS, Jeong HS, Brady JN, Ozato K. The bromodomain protein Brd4 is a positive regulatory component of P-TEFb and stimulates RNA polymerase II-dependent transcription., 2005, 19(4): 523–534.
[49] Yang ZY, Yik JHN, Chen RC, He NH, Jang MK, Ozato K, Zhou Q. Recruitment of P-TEFb for stimulation of transcriptional elongation by the bromodomain protein Brd4., 2005, 19(4): 535–545.
[50] Zhang WS, Prakash C, Sum C, Gong Y, Li YH, Kwok JJ, Thiessen N, Pettersson S, Jones SJM, Knapp S, Yang H, Chin KC. Bromodomain-containing protein 4 (BRD4) regulates RNA polymerase II serine 2 phosphorylation in human CD4+ T cells., 2012, 287(51): 43137– 43155.
[51] Sims RJ 3rd, Belotserkovskaya R, Reinberg D. Elongation by RNA polymerase II: the short and long of it., 2004, 18(20): 2437–2468.
[52] Itzen F, Greifenberg AK, Bösken CA, Geyer M. Brd4 activates P-TEFb for RNA polymerase II CTD phosphorylation., 2014, 42(12): 7577–7590.
[53] Rasmussen EB, Lis JT.transcriptional pausing and cap formation on threeheat shock genes., 1993, 90(17): 7923–7927.
[54] Hnisz D, Abraham BJ, Lee TI, Lau A, Saint-AndréV, Sigova AA, Hoke HA, Young RA. Super-enhancers in the control of cell identity and disease., 2013, 155(4): 934–947.
[55] Pott S, Lieb JD. What are super-enhancers?, 2015, 47(1): 8–12.
[56] Whyte WA, Orlando DA, Hnisz D, Abraham BJ, Lin CY, Kagey MH, Rahl PB, Lee TI, Young RA. Master transcription factors and mediator establish super-enhancers at key cell identity genes., 2013, 153(2): 307–319.
[57] Di Micco R, Fontanals-Cirera B, Low V, Ntziachristos P, Yuen SK, Lovell CD, Dolgalev I, Yonekubo Y, Zhang GT, Rusinova E, Gerona-Navarro G, Cañamero M, Ohlmeyer M, Aifantis I, Zhou MM, Tsirigos A, Hernando E. Control of embryonic stem cell identity by BRD4-dependent transcriptional elongation of super-enhancer-associated pluripotency genes., 2014, 9(1): 234–247.
[58] Lovén J, Hoke HA, Lin CY, Lau A, Orlando DA, Vakoc CR, Bradner JE, Lee TI, Young RA. Selective inhibition of tumor oncogenes by disruption of super-enhancers., 2013, 153(2): 320–334.
[59] Dey A, Nishiyama A, Karpova T, Mcnally J, Ozato K. Brd4 marks select genes on mitotic chromatin and directs postmitotic transcription., 2009, 20(23): 4899–4909.
[60] Mochizuki K, Nishiyama A, Jang MK, Dey A, Ghosh A, Tamura T, Natsume H, Yao HJ, Ozato K. The bromodomain protein Brd4 stimulates G1 gene transcription and promotes progression to S phase., 2008, 283(14): 9040–9048.
[61] Devaiah BN, Case-Borden C, Gegonne A, Hsu CH, Chen QR, Meerzaman D, Dey A, Ozato K, Singer DS. BRD4 is a histone acetyltransferase that evicts nucleosomes from chromatin., 2016, 23(6): 540–548.
[62] You JX, Li Q, Wu C, Kim J, Ottinger M, Howley PM. Regulation of aurora B expression by the bromodomain protein Brd4., 2009, 29(18): 5094–5103.
[63] Farina A, Hattori M, Qin J, Nakatani Y, Minato N, Ozato K. Bromodomain protein Brd4 binds to GTPase-activating SPA-1, modulating its activity and subcellular localization., 2004, 24(20): 9059–9069.
[64] Maruyama T, Farina A, Dey A, Cheong J, Bermudez VP, Tamura T, Sciortino S, Shuman J, Hurwitz J, Ozato K. A mammalian bromodomain protein, brd4, interacts with replication factor C and inhibits progression to S phase., 2002, 22(18): 6509–6520.
[65] Tasdemir N, Banito A, Roe JS, Alonso-Curbelo D, Camiolo M, Tschaharganeh DF, Huang CH, Aksoy O, Bolden JE, Chen CC, Fennell M, Thapar V, Chicas A, Vakoc CR, Lowe SW. BRD4 connects enhancer remodeling to senescence immune surveillance., 2016, 6(6): 612–629.
[66] Maherali N, Sridharan R, Xie W, Utikal J, Eminli S, Arnold K, Stadtfeld M, Yachechko R, Tchieu J, Jaenisch R, Plath K, Hochedlinger K. Directly reprogrammed fibroblasts show global epigenetic remodeling and widespread tissue contribution., 2007, 1(1): 55–70.
[67] Mikkelsen TS, Hanna J, Zhang XL, Ku MC, Wernig M, Schorderet P, Bernstein BE, Jaenisch R, Lander ES, Meissner A. Dissecting direct reprogramming through integrative genomic analysis., 2008, 454(7200): 49–55.
[68] Liu LQ, Xu Y, He MH, Zhang M, Cui FG, Lu LN, Yao MZ, Tian WH, Benda C, Zhuang Q, Huang ZJ, Li WJ, Li XC, Zhao P, Fan WX, Luo ZW, Li Y, Wu YS, Hutchins AP, Wang DY, Tse HF, Schambach A, Frampton J, Qin BM, Bao XC, Yao HJ, Zhang BL, Sun H, Pei DQ, Wang HT, Wang J, Esteban MA. Transcriptional pause release is a rate-limiting step for somatic cell reprogramming., 2014, 15(5): 574–588.
[69] Di Stefano B, Collombet S, Jakobsen JS, Wierer M, Sardina JL, Lackner A, Stadhouders R, Segura-Morales C, Francesconi M, Limone F, Mann M, Porse B, Thieffry D, Graf T. C/EBPalpha creates elite cells for iPSC reprogramming by upregulating Klf4 and increasing the levels of Lsd1 and Brd4., 2016, 18(4): 371–381.
[70] Wang Z, Oron E, Nelson B, Razis S, Ivanova N. Distinct lineage specification roles for NANOG, OCT4, and SOX2 in human embryonic stem cells., 2012, 10(4): 440–454.
[71] Shao ZC, Yao CP, Khodadadi-Jamayran A, Xu WH, Townes TM, Crowley MR, Hu KJ. Reprogramming by de-bookmarking the somatic transcriptional program through targeting of BET bromodomains., 2016, 16(12): 3138–3145.
[72] Yamanaka S. Elite and stochastic models for induced pluripotent stem cell generation., 2009, 460(7251): 49–52.
[73] Shao ZC, Zhang RW, Khodadadi-Jamayran A, Chen B, Crowley MR, Festok MA, Crossman DK, Townes TM, Hu KJ. The acetyllysine reader BRD3R promotes human nuclear reprogramming and regulates mitosis., 2016, 7: 10869.
[74] Lorthongpanich C, Solter D, Lim CY. Nuclear reprogramming in zygotes., 2010, 54(11–12): 1631– 1640.
[75] Egli D, Birkhoff G, Eggan K. Mediators of reprogramming: transcription factors and transitions through mitosis., 2008, 9(7): 505–516.
[76] Izumikawa K, Ishikawa H, Yoshikawa H, Fujiyama S, Watanabe A, Aburatani H, Tachikawa H, Hayano T, Miura Y, Isobe T, Simpson RJ, Li L, Min JR, Takahashi N. LYAR potentiates rRNA synthesis by recruiting BRD2/4 and the MYST-type acetyltransferase KAT7 to rDNA., 2019, 47(19): 10357–10372.
[77] Zhao QS, Wu YS, Shan ZY, Bai GY, Wang ZD, Hu J, Liu L, Li T, Shen JL, Lei L. Serum starvation-induced cell cycle synchronization stimulated mouse rDNA transcription reactivation during somatic cell reprogramming into iPSCs., 2016, 7(1): 112.
[78] Zheng Z, Jia JL, Bou G, Hu LL, Wang ZD, Shen XH, Shan ZY, Shen JL, Liu ZH, Lei L. rRNA genes are not fully activated in mouse somatic cell nuclear transfer embryos., 2012, 287(24): 19949–19960.
Epigenetic “reader” BET proteins regulate mammalian development and iPSC reprogramming
Zhijing Zhang, Yu Qiao, Yuchen Sun, Lei Lei
As an epigenetic “reader”, bromodomain and extra-terminal (BET) proteins play a vital role in mammalian development. Each member in the BET family regulates precisely the related genes and promotes the differentiation and development of early embryos specifically through recognizing a variety of epigenetic modifications and recruiting corresponding functional complexes. In addition, with the development of induced pluripotent stem cell (iPSC) technology, accumulating evidence have found that BET family proteins may also play a pivotal role in the iPSC reprogramming. In this review, we summarize the function of the BET family proteins in mammalian development and iPSC reprogramming from recent literatures and speculate new mechanisms of the BET family proteins in regulating reprogramming.
BET proteins; mammalian development; induced pluripotent stem cell (iPSC)
2021-06-30;
2021-08-23;
2021-11-08
国家自然科学基金项目(编号:31671545)资助[Supported by the National Natural Science Foundation of China (No. 31671545)]
张祉靖,在读硕士研究生,专业方向:人体解剖学与组织胚胎学。E-mail: cdzzj1998@163.com
雷蕾,博士,教授,研究方向:干细胞与胚胎发育。E-mail: lei086@ems.hrbmu.edu.cn
10.16288/j.yczz.21-232
(责任编委: 李海涛)

