20种溪蟹线粒体COI基因比较分析及系统发育研究
刘东东,高佳怡,张璐瑶,龚理
(浙江海洋大学海洋科学与技术学院/海洋生物种质发掘与利用国家地方研究中心,浙江舟山 316022)
0 引言
【研究意义】线粒体基因组作为细胞核外能独立进化的遗传系统,具有结构简单、呈母系遗传、进化速率快且不同结构进化速率不一致等特点,已广泛应用于物种鉴定、群体遗传结构、适应性进化和系统发育进化分析等研究领域(石林波等,2013;Wang et al.,2019;罗纯等,2021;Zhang et al.,2021)。DNA条形码首次由Hebert等(2003a,2003b)提出,经过近20年的发展现已普遍应用于物种分类鉴定和生物多样性评估等领域,在海关、法医鉴定、市场监督及生物多样性保护中心等行业部门发挥着重要作用(张瑞玲等,2017;刘娟等,2019;熊娟等,2020)。溪蟹科(Potamidae)是短尾次目(Brachyura)中物种十分丰富的类群,由于溪蟹生境复杂、近缘种形态接近、研究起步较晚,导致其分类及系统进化关系长期存在争议(邹节新等,2013;Zhang et al.,2020)。因此,建立溪蟹科DNA条形码数据库,辅以形态学特征,有望解决溪蟹物种长期存在的鉴定问题,为溪蟹科系统进化研究及其他类群物种鉴定提供参考依据。【前人研究进展】自2003年Hebert等(2003a,2003b)首次提出DNA条形码概念以来,作为分类鉴定的有效技术手段在从低等的真菌、藻类和无脊椎动物到高等的两栖爬行类、鱼类和哺乳类动物均已得到证实(孔晓瑜等,2001;孟玮等,2010;王淑英等,2014;张瑞玲等,2017;徐岩等,2019)。孔晓瑜等(2001)通过比较分析黄河口和珠江流域中华绒螯蟹与日本绒螯蟹间的COI基因序列差异,结果发现黄河口和珠江流域的中华绒螯蟹COI基因序列完全一致,但二者与日本绒螯蟹的COI基因序列差异较明显,支持存在中华绒螯蟹和日本绒螯蟹或二者为不同地理亚种的观点;Ortman等(2010)基于COI基因序列分析1种立方水母、10种钵水母和84种水螅水母的遗传距离,结果证实了DNA条形码间隔的存在,并提出COI基因可作为水母亚门种类鉴定的分子标记,类似方法在根口水母科(Rhizostomatidae)物种鉴定中同样有效(李玉龙等,2017);王淑英等(2014)在研究鲽形目鳎科(Soleidae)鱼类物种鉴定时发现,COI基因在种内和属内种间的平均遗传距离分别为0.33%和19.91%,二者的遗传差异约60倍(个别最小相差19倍),符合Hebert等(2003a,2003b)提出的物种鉴定标准;张瑞玲等(2017)基于线粒体COI基因序列,通过遗传距离和系统发育进化树对蚊、蝇、蜱、蟑螂等主要医学媒介生物进行鉴定,证实了DNA条形码技术在这些媒介生物鉴定中的可行性;徐岩等(2019)基于COI基因序列对中国沿海相手蟹(Sesarmidae)进行分子系统发育分析,结果表明相手蟹为多系起源,且日本的无齿螳臂相手蟹(Chiromantes dehaani)与我国长江口的无齿螳臂相手蟹(C.neglectum)虽然在形态上存在一定差异,但分子数据显示二者为同种异名。此外,孟玮等(2010)将线粒体COI基因序列作为基因条形码运用于三种鲑科鱼类(亚东鲑、大西洋鲑和红点鲑)鉴定,其系统发育进化树显示亚东鲑和大西洋鲑首先聚为一类,再与红点鲑聚类,表明线粒体COI基因序列对鲑科鱼类物种鉴定具有一定的可行性和有效性,类似的方法在刀鲚(Coilia nasus)、湖鲚(C.nasus taihuensis)、短颌鲚(C.branchygnathus)、七丝鲚(C.grayii)、凤鲚(C.mystus)(杨巧莉,2012)及裂腹鱼亚科(Schizothoracinae)(周建设等,2019;迪丽娜·茹斯坦木,2021)等物种鉴定中也得到证实。【本研究切入点】我国溪蟹物种资源丰富,但研究起步较晚,且主要集中在形态学分类、生态习性及生长繁殖等方面(戴爱云和陈国孝,1981;楚克林等,2018)。虽然目前针对溪蟹科已有分子方面的相关研究,但涉及的物种不多(郑芳等,2006;石林波等,2013;Ji et al.,2016),关于溪蟹科物种分类及其内部亲缘关系至今尚未得到较全面系统研究。【拟解决的关键问题】通过扩增测定2种溪蟹代表种[长安龙溪蟹(Longpotamon changanense)和龙溪蟹未定种(Longpotamonsp.)]的COI基因全序列,结合GenBank中已公布的18种溪蟹科COI基因序列,比较分析20种溪蟹的线粒体COI基因序列,评估其序列多态性,探究COI基因作为分子标记在溪蟹物种鉴定中的适用性;同时基于COI基因序列构建溪蟹科系统发育进化树,分析不同种类间的亲缘关系,为溪蟹科的物种鉴定及系统发育研究提供理论依据。
1 材料与方法
1.1 试验材料
2种溪蟹样品分别采自陕西洛南(东经110°07′41.36″,北纬34°06′17.43″)和湖南新化(东经111°20′23.68″,北纬27°43′40.19″)附近溪流,将新鲜样品用酒精固定后运回实验室;根据形态学特征并辅以NCBI中的线粒体COI基因序列进行鉴定,其中,陕西洛南采集到的样品鉴定为长安龙溪蟹,湖南新化采集的样品可能为龙溪蟹属新纪录种。由于该新纪录种样品采集数量有限,故暂命名为龙溪蟹未定种。
1.2 基因组DNA提取及检测
取2种溪蟹螯足肌肉组织约30 mg,剪碎后放入离心管中,利用盐析法提取2种溪蟹的基因组。以1.0%琼脂糖凝胶电泳检测基因组完整性,并采用核酸定量仪(NanoDrop)检测其纯度,-20℃保存备用。
1.3 PCR扩增及测序
基于NCBI已公布的蟹类线粒体基因组COI序列,设计扩增2种溪蟹线粒体COI基因序列的通用引物(F-COI:5'-ATGCAACGATGATTTTTTTCYA-3';R-COI:5'-TARCGGTTGATHARGAGT-3')。PCR扩增反应体系33.0 μL:PCR Mix酶(北京溪洋汇智科技有限公司)15.0 μL,ddH2O 13.5 μL,正、反向引物各1.5 μL,DNA模板1.5 μL。扩增程序:95℃预变性3 min;95℃30 s,48~54℃50 s,68℃1~3 min,进行35个循环;68℃延伸10 min。PCR扩增产物采用1.2%琼脂糖凝胶电泳进行检测,电泳缓冲液为1×TAE(pH 8.0)、电压为4 V/cm,以EB核酸染料染色,于凝胶成像系统下观察并拍照,回收目的片段送至生工生物工程(上海)股份有限公司进行双向测序;无法直接测序的片段则通过分子克隆后再测序。
1.4 序列分析及系统进化树构建
运用CodonCode Aligner 5.1.5将各目的基因序列进行拼接,并辅以手工校对;再以Sequin v15.10(http://www.ncbi.nlm.nih.gov/Sequin/)对线粒体COI基因全序列进行注释,利用ClustalX对测定的2种溪蟹和GenBank已公布的其他溪蟹线粒体COI基因核苷酸及其推导氨基酸序列进行比较分析(Larkin et al.,2007),利用MEGA X计算其碱基组成、保守位点和遗传距离(Kumar et al.,2018),采用MatGAT 2.02进行多序列相似性比较分析(Campanella et al.,2003),并以PhyloSuite构建贝叶斯树(BI)和最大似然树(ML)(李加爱等,2021),探究溪蟹科物种内部亲缘关系。
2 结果与分析
2.1 20种溪蟹COI基因全序列比较分析结果
长安龙溪蟹和龙溪蟹未定种的线粒体COI基因序列全长分别为1534和1539 bp,均以ATG为起始密码子,分别连续编码511和512个氨基酸残基,不存在碱基插入或缺失;长安龙溪蟹以不完全终止密码子T为终止密码子,龙溪蟹未定种以常见的TAA为终止密码子。2种溪蟹的碱基含量差异不明显,4种碱基的含量分别为36.7%~38.3%(T)、18.4%~19.2%(C)、28.1%~28.6%(A)和15.3%~15.4%(G)。为了比较溪蟹科线粒体COI基因序列多态性,从GenBank下载18种溪蟹科COI基因全序列,对这20种溪蟹的COI基因序列差异进行比较分析。结果(表1)显示,COI基因序列全长1534~1539 bp,连续编码511~512个氨基酸残基;所有物种均以ATG为起始密码子,6种以不完全终止密码子T为终止密码子,其余均以TAA为终止密码子(图1)。20种溪蟹的碱基含量略有不同(表1),分别为35.9%~40.7%(T)、16.4%~20.2%(C)、26.8%~28.9%(A)和14.7%~17.2%(G),呈明显的AT偏向性。核苷酸序列比较分析结果(图1)显示,共有962个保守位点,577个变异位点(约占37.3%);基因序列变异主要发生在密码子的第3位密码子上(452个变异位点),第1位密码子存在100个变异位点,第2位密码子非常保守,仅有25个变异位点。将核苷酸翻译成氨基酸序列再进行比较分析,结果(图2)显示仅有98个变异位点,变异位点率仅占19.0%,远低于核苷酸序列变异率。可见,线粒体COI基因第3位密码子具有摇摆性,但第3位密码子变异并不一定改变氨基酸种类,该现象称为遗传密码简并性。
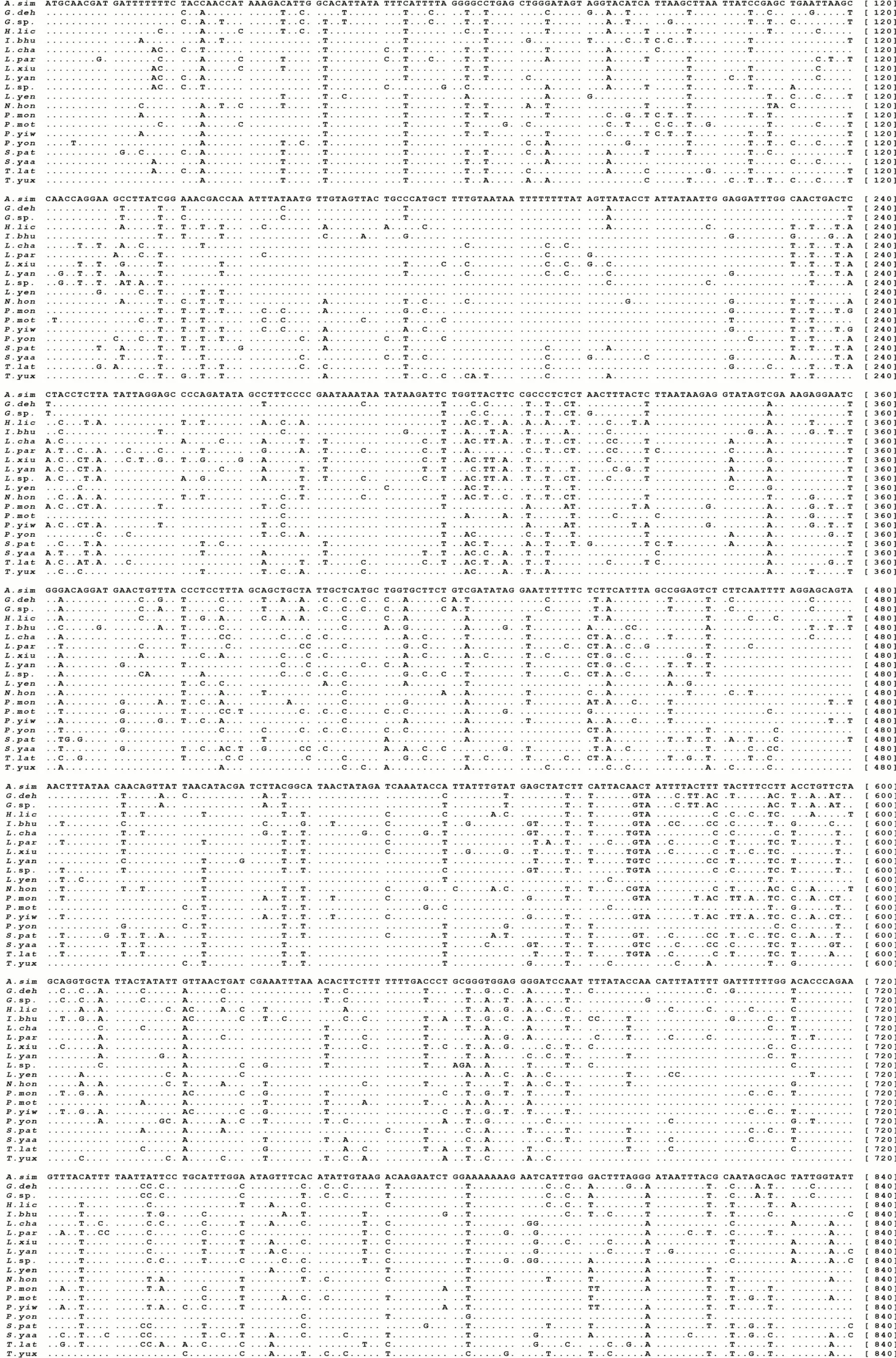
图1 20种溪蟹线粒体COI基因核苷酸序列比较分析结果Fig.1 Alignment of nucleotide sequence of mitochondria COI gene of 20 Potamidae species

图2 20种溪蟹线粒体COI基因推导氨基酸序列比较分析结果Fig.2 Alignment of deduced amino acid sequence of mitochondria COI gene of 20 Potamidae species

表1 构建系统发育进化树的物种信息Table 1 List of the species to build phylogenetic tree
2.2 遗传距离及相似分析结果
基于COI基因核苷酸序列计算20种溪蟹科物种的遗传距离及其序列相似性(表2),结果显示,近溪蟹属(Potamiscus)的易武近溪蟹(P.yiwuensis)与山区近溪蟹(P.montosus)间的遗传距离最小(0.007),二者对应的序列相似性最高(99.4%);其次是泽蟹属(Geothelphusa)的汉氏泽蟹(G.dehaani)与泽蟹未定种(Geothelphusasp.),二者的遗传距离为0.028,对应的序列相似性为97.3%;最大遗传距离出现在龙溪蟹未定种(Longpotamonsp.)和泰王中印溪蟹(Indochinamon bhumibol)间(0.234),对应的序列相似性最低(80.7%)。值得注意的是:小石蟹属(Tenuilapotamon)中的宽腹小石蟹(T.latilum)与龙溪蟹属中的小龙溪蟹(L.parvum)遗传距离最小(0.110),序列相似性最高(89.6%),而宽腹小石蟹与玉溪小溪蟹(Tenuipotamon yuxiense)间的遗传距离为0.199(大于0.110),序列相似性仅为83.0%(低于89.6%),小龙溪蟹与同属其他物种的遗传距离最小为0.122(与长安龙溪蟹),序列相似性最高为88.7%(与长安龙溪蟹)。类似现象也存在于近溪蟹属物种中,近溪蟹属中的永胜近溪蟹(P.yongshengense)与小溪蟹属(Tenuipotamon)的玉溪小溪蟹(T.yuxiense)间的遗传距离(0.135)低于永胜近溪蟹与同属的易武近溪蟹和山区近溪蟹间的遗传距离(均为0.188);永胜近溪蟹与玉溪小溪蟹间的COI基因序列相似性(88.1%)高于永胜近溪蟹与易武近溪蟹(84.2%)和山区近溪蟹(84.3%)间的相似性。此外,长安龙溪蟹和龙溪蟹未定种间的遗传距离最小(0.088),序列相似性最高(91.5%)。
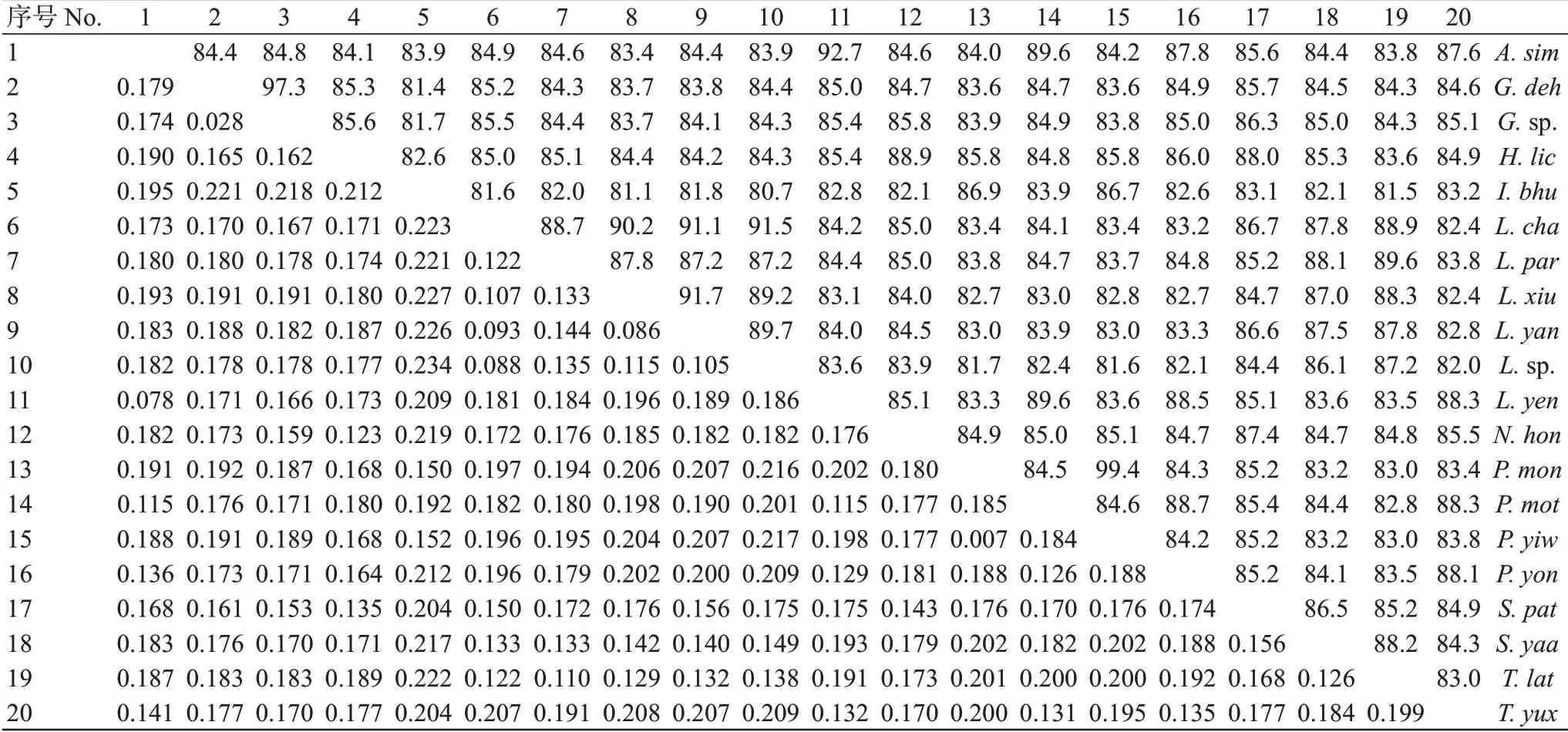
表2 20种溪蟹科物种的线粒体COI基因遗传距离及其序列相似性Table 2 The genetic distance and sequence similarity based on mitochondria COI gene in 20 Potamidae species
2.3 系统发育进化分析结果
基于线粒体COI基因及线粒体基因组中的13个蛋白编码基因序列,以束腹蟹科(Parathelphtlsidae)中的波阳束腰蟹(Somanniathelphusa boyangensis)和坝王束腰蟹(S.bawangensis)为外类群,同时构建溪蟹科贝叶斯树和最大似然树,结果显示,基于COI基因构建的2种系统发育树(图3和图4)略有不同,而基于13个蛋白编码基因构建的贝叶斯树和最大似然树拓扑结构(图5)完全一致,且基于COI基因构建的系统发育进化树与基于蛋白编码基因构建的系统发育进化树也存在一定差异。虽然采用不同方法基于不同数据集构建的系统发育进化树在拓扑结构上有所不同,但所有树型均显示长安龙溪蟹与同属的龙溪蟹未定种亲缘关系最近,除了小龙溪蟹与宽腹小石蟹聚在一起外,其余4种龙溪蟹属物种聚为一支,表明龙溪蟹属为非单系群;近溪蟹属4个物种中,易武近溪蟹与山区近溪蟹的亲缘关系最近,聚为一支,而其他2个物种[永胜近溪蟹和墨脱近溪蟹(P.motuoensis)]分散在系统发育进化树不同分支中,且永胜近溪蟹与玉蟹小溪蟹间的亲缘关系明显比永胜近溪蟹与墨脱近溪蟹间的亲缘关系近,说明近溪蟹属也是非单系群;泽蟹属中的汉氏泽蟹和泽蟹未定种聚在一起,二者存在较近的亲缘关系。

图3 基于线粒体COI基因序列构建的溪蟹科贝叶斯树Fig.3 Bayesian tree of Potamidae species based on mitochondria COI gene sequence
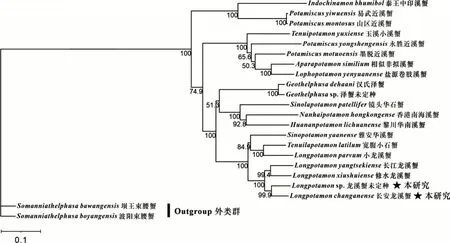
图4 基于线粒体COI基因序列构建的溪蟹科最大似然树Fig.4 The maximum likelihood tree of Potamidae species based on mitochondria COI gene sequence

图5 基于13种蛋白编码基因核苷酸序列构建的溪蟹科的贝叶斯树和最大似然树Fig.5 Bayesian tree and the maximum likelihood tree of Potamidae species based on 13 protein coding genes nucleotide sequence
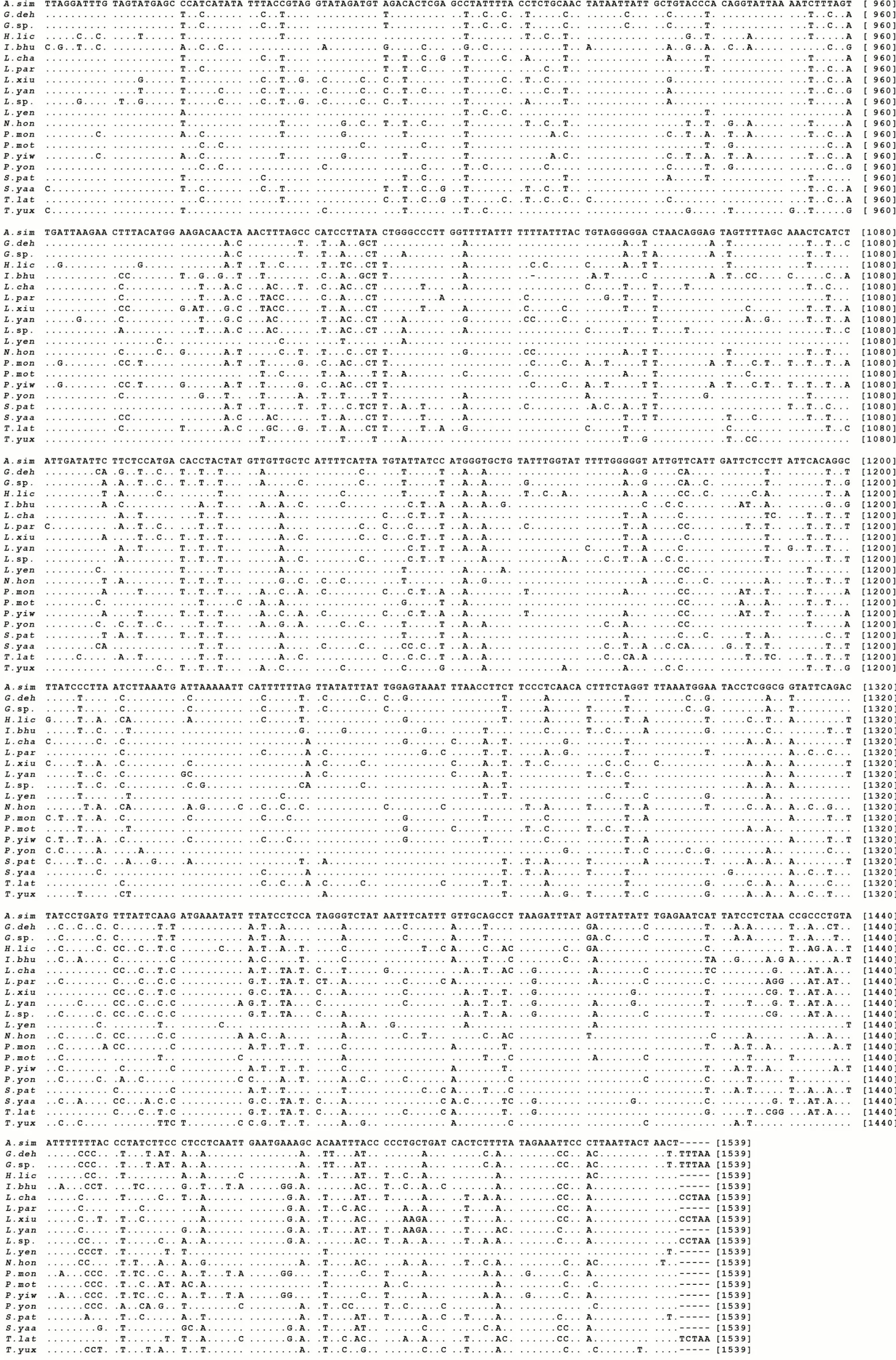
续图1 20种溪蟹线粒体COI基因核苷酸序列比较分析结果Continued Fig.1 Alignment of nucleotide sequence of mitochondria COI gene of 20 Potamidae species
3 讨论
线粒体是细胞核外独立的遗传系统,能自主进行DNA复制、转录和翻译。线粒体基因组具有分子量小、结构简单、编码效率高、进化速度快等优点,在物种鉴定和分类、遗传多样性分析及系统进化研究等领域发挥着重要作用(Wang et al.,2019;罗纯等,2021;Zhang et al.,2021;Sultana et al.,2022)。在众多线粒体基因中,COI基因因具有易扩增、进化速度适中等特点已广泛应用于不同类群生物分类和鉴定,很大程度上弥补了形态学分类的不足,尤其为形态特征极其相似的隐存种及形态界限模糊不清或形态破损不易区分的近缘种鉴定提供了有效依据(范蔓桦等,2020;唐富江等,2020;张格等,2020)。Hebert等(2003a)最早比较分析了13320种不同门类动物的COI基因序列,提出COI基因用于物种鉴定的标准是种内遗传距离小于2%,且种间遗传距离显著大于种内遗传距离(通常在10倍以上)。目前该标准在大量生物类群(甲壳类、两爬类、鱼类和哺乳类等)中已得到验证(Hebert et al.,2003b)。唐富江等(2020)通过比较分析乌苏里江260个鱼卵样本的线粒体COI基因序列,发现其中有213条序列与Gen-Bank已知物种参考序列的相似性超过99%,能有效鉴定到种;7条序列与已知物种参考序列的相似度为95%~99%,能鉴定到属;进一步分析还发现乌苏里江中下游漂流性卵鱼类以银鮈(Squalidus argentatus)、银鲴(Xenocypris argentea)、贝氏(Hemiculter bleekeri)和花斑副沙鳅(Parabotia fasciatus)等小型鱼类为主,资源量较大,而大中型鱼类中以翘嘴鲌(Culter alburnus)的资源量最丰富,该结论为鱼类资源开发和利用提供了便捷有效的分子参考。同样的方法在长江口海域夏季鱼卵、仔稚鱼种类组成及多样性分析中也得到广泛应用。张格等(2020)研究表生长、代谢过程及生物调节等功能条目上,而KEGG信息通路富集分析结果表明主要富集在过氧化物酶、RNA降解、轴突指导、神经活性配体—受体相互作用等通路上。根据GO功能注释及KEGG信号通路富集分析结果,最终选定TOB2、CRAT、CCT6、KLF4共4个基因为凡纳滨对虾生长相关候选基因,具体基因信息见表5。通过为期100 d的凡纳滨对虾养殖试验,结果(表6)发现试验组凡纳滨对虾的终末体质量、终末体长、日增重率、体长日增长率及特定生长率均显著高于对照组凡纳滨对虾(P<0.05,下同),表明耐低盐世代选育凡纳滨对虾群体的生长情况优于常规的凡纳滨对虾群体。
我国溪蟹物种资源丰富,是全球多样性最丰富的国家,很多种类还是我国的特有物种(楚克林等,2018)。但由于溪蟹研究起步相对较晚,长期以来溪蟹科分类及系统进化一直存在较大争议(石林波等,2012;邹节新等,2013;Ma et al.,2019;Zhang et al.,2020)。本研究同时基于线粒体COI基因和线粒体基因组13个蛋白编码基因序列构建至今为止涵盖物种最多的溪蟹科系统发育进化树,结果显示,基于2个数据集构建得到的溪蟹科系统发育进化树拓扑结构具有一定差异,主要表现在基于蛋白编码基因构建的系统发育进化树支持率(Bootstrap valve)和后验概率(Posterior probability)普遍高于基于COI基因构建的系统发育进化树,表明前者具有更高的可信度。究其原因是单个基因携带的有效遗传信息明显少于多基因串联序列,也是越来越多研究趋向于利用线粒体基因组全序列进行系统发育进化分析的原因。虽然采用不同方法基于不同数据集构建的系统发育进化树在拓扑结构上有所不同,但所有树型均显示长安龙溪蟹与同属的龙溪蟹未定种亲缘关系最近,同时暗示龙溪蟹属和近溪蟹属为非单系群,今后可基于更多的物种数量及更多基因序列进行重点验证。此外,本研究扩增的COI基因序列包含有效信息位点太少,对于进化地位尚有争议的物种解析度略显不足;同时比较分析的溪蟹代表种较少,某些属(卷肢溪蟹属Lophopotamon)仅包括1个物种,因此今后需进一步扩大取样范围,同时筛选更多的分子标记以厘清溪蟹科系统发育进化关系。
4 结论
20种溪蟹的线粒体COI基因序列平均种间遗传距离为0.173,均具有区别于其他种类的特异位点,即线粒体COI基因序列可作为溪蟹科物种鉴定的分子标记。

