基于DNA分子生物学食性研究领域的文献计量分析
张宇洋,董建宇,孙 昕,张秀梅
( 1.中国海洋大学,海水养殖教育部重点实验室,山东 青岛 266003; 2.浙江海洋大学 水产学院,浙江 舟山 316022; 3.青岛海洋科学与技术(试点)国家实验室,海洋渔业科学与食物产出过程功能实验室,山东 青岛 266072 )
食物网及其动态变化研究有助于理解群落生态学和生态系统的功能机制,而运用准确的技术手段来确定消费者的生态位宽度和食物组成对该领域研究至关重要[1]。目前关于食性分析方法主要分为3类:传统方法(直接观察、消化道内容物分析和显微镜镜检等)、生化方法(脂肪酸组成分析和稳定同位素分析等)和分子方法(同工酶电泳、单克隆抗体与多克隆抗体的免疫组化分析和DNA分子技术等)。3类食性分析方法针对不同的研究对象和研究范围各有其优缺点[2-4]。传统方法操作灵活、无硬性设备要求、直观快捷,但存在人为鉴定误差、辨识度低、耗时耗力等缺点;生化分析方法测定步骤简单,适用范围广,不足之处是难以确定食物组成,同一类群特征值存在偏差;早期关于野生动物食性的分子分析方法,通常采用的是同工酶电泳和单克隆抗体检测。同工酶电泳方法廉价、易操作,但灵敏度和特异性低[5]。单克隆抗体灵敏度和特异性高,但特异性抗体的制作是一个困难且耗时的过程[6]。此外,上述这两种方法仅能检验一对物种的营养关系,且往往需要先验知识,难以研究食物网中消费者的完整食谱[7]。
20世纪90年代,随着分子生物学的发展,遗传信息载体DNA开始应用于食性分析中的猎物鉴定[8]。基于DNA的食性分析方法分为两种:PCR鉴定和基于序列的鉴定。PCR鉴定是通过扩增猎物特定长度的DNA片段,使用电泳技术将其分离和可视化以实现猎物鉴定的一种分析方法。1992年Höss等[9]首次将PCR鉴定技术运用于棕熊的食性研究。随后,Asahida等[10]使用该方法发现了砂虾(Crangonaffinis)可捕食石鲽 (Kareiusbicoloratus) 幼体;Agusti等[11]在小盲蝽(Dicyphustamaninii)肠道内容物中检测到了猎物棉铃虫(Helicoverpaarmigera)的DNA。至此,基于PCR鉴定的食性研究方法初露锋芒,逐渐成为21世纪初期最常用的猎物鉴定技术[12]。基于序列的猎物鉴定技术本质上是DNA条形码技术,这是一种基于自然界生物基因序列唯一性原理,通过一段或者几段特异性基因序列实现物种的快速准确鉴定的技术方法[13]。到2003年,DNA条形码技术迎来发展的黄金期[14],其简便、高效、准确的检测方法,使其成为生命科学领域发展最快的前沿技术之一[15]。近年来,随着分子生物学数据库的完善和高通量测序技术(二代测序技术,NGS)的出现,整合了DNA条形码理念和二代测序技术的DNA宏条形码技术应运而生。宏条形码技术不受猎物种类限制,可以在分类学物种水平上识别猎物,尤其适合研究难以观测或者有特殊习性的广食性动物的食物谱[16],且允许对大量样本进行并行处理和测序[17]。鉴于DNA物种鉴定技术的巨大应用潜力,基于DNA测序的食性分析研究领域正经历着大规模流行的热潮[18],已逐渐成为分子摄食生态学研究的主流方法。而国内基于DNA分子生物学的食性研究起步相对较晚,尚未引起足够的重视,文献多为该领域研究方法可行性的讨论,多聚焦于食性定性分析研究[19],研究内容多为猎物种类鉴定研究与方法可行性探索[19]。但随着测序成本的降低与生物信息分析方法的日渐成熟,基于DNA分子生物学的食性研究技术引发了国内诸多学者的关注,近期涌现了多篇相关文献[20-23]。为了促进国内DNA物种鉴定技术在摄食生态学中的应用,笔者采用文献计量法,对基于DNA分子生物学食性分析的相关文献进行定量与定性的可视化分析。旨在直观、清晰地剖析该领域的发展脉络、研究趋势和研究热点等,以期为未来基于DNA的食性分析研究提供借鉴和启示。
1 分析方法与数据来源
本研究的数据来源于Web of Science(WOS)核心合集,通过编写检索式来限定检索范围。根据相关论文中的“条形码”、“猎物鉴定”和“分子鉴定”3个常用主题词的英文书写,编制检索式:TS=[(*barcod* OR “prey identification” OR “molecular dietary analysis ” OR “prey DNA”) AND (diet* OR “predator-prey” OR “trophic ecology” OR “feeding ecology”)],检索时间为2020年1月19日,共检索到716篇文献。
CiteSpace是美国德雷塞尔大学陈超美教授开发的,用来呈现学科知识结构、规律和分布状况的引文可视化分析软件[24]。HistCite是Thomson Reuters 公司开发的,一款用来绘制引文关系图谱,定位领域重要文献的引文编年可视化分析软件[25]。利用WOS数据库检索得到的数据,进行文献计量学分析。使用WOS的“分析检索结果”功能初步分析文献,利用HistCite软件检索学科基础知识文献,探究学科发展历史。在前两者的基础上,使用CiteSpace 5.6.R2软件对文献数据进行可视化分析,运用关键词共现知识图谱聚类分析和突现词检验,体现该领域的研究热点和研究趋势。通过科研合作网络,从作者、机构和国家3种层次分析该领域研究的相互合作关系。
2 研究结果分析
2.1 发文与引文量分析
WOS自带的“分析检索功能和引文报告功能”分析显示,1997—2019年,该领域共发表文献716篇,共被引频次17 864次,去除自引后的引用频次为13 373次,每篇平均引用次数24.88,H指数(H-index)为65。文献数量随时间的变化能够反映该领域的发展趋势,1996—2019年间,关于DNA分子生物学的食性研究发文量呈现缓慢增长—平稳增长—快速增长3个阶段(图1)。分析发现1996—2004年关于该领域的年平均发文数量在3篇以下,研究处于探索阶段;2005—2013年间不断摸索改进了基于DNA的食性分析方法,发文量呈现平稳增长,研究处于发展阶段;2014—2019年由于研究方法的革新与条形码数据库的完善,该领域研究的关注度迅速上升。在众多学者的不断摸索下提出了相应的技术路线和优化措施,形成了一套系统的基于DNA手段的食性分析方法,涌现出大量食性分析相关的研究文献,从而推动该领域研究进入到快速发展阶段。虽然基于DNA的分子生物学食性分析研究体系日渐成熟,但是目前整体刊文量仍较少,研究前景广阔。

图1 1997—2019年年度发表论文数量Fig.1 Annual number of publications from 1997 to 2019
2.2 发文期刊排名
WOS的检索文献期刊统计显示,发文量排名前10的期刊共发文277篇(表1),占总发文量的38.85%。其中,《PLoS ONE》刊文量最多为58篇,《Molecular Ecology》次之,然后是《Molecular Ecology Resources》和《Ecology and Evolution》。总体而言,Wiley旗下的分子生态学旗舰期刊《Molecular Ecology》和《Molecular Ecology Resources》是该领域发文量和影响力较高的核心期刊。此外,由于基于DNA的分子生物学食性分析技术具有综合性和学科交叉的特点,刊文期刊广泛涉及分子生态学、保护生态学、渔业、昆虫学、海洋和淡水生物学、生物技术与应用微生物学等相关学科。

表1 发文量前10位的期刊Tab.1 Top ten journals in the number of published articles from 1997 to 2019
2.3 基于HistCite的学科基础知识分析
使用HisCite软件,对WOS检索所得数据集进行的本地引用得分(LCS)分析。LCS即某一文献在本地数据集中的被引用次数。由于WOS检索数据与检索词有直接关系,可以认定基于WOS检索的文章多为同一领域,基于LCS的引文关系文献分析可以得到该研究领域内的重要文献或开创性文章(表2)。

表2 基于LCS分析前10位关键文献Tab.2 Top ten key literatures based on LCS analysis
根据时间线分析,Symondson[7]2002年在《Molecular Ecology》上刊文详细阐述了一系列分子生物学食性分析技术的研究背景,并比较了各自技术的优劣,指出基于DNA检测的猎物鉴定技术的高效性和通用性,其很有可能会取代其他分子生物学分析技术,该报道对DNA食性分析方法产生了巨大的推动作用。2005年Deagle等[30]利用克隆文库和定量PCR (qPCR)方法检测了圈养海狮 (Eumetopiasjubatus) 粪便中不同鱼种的比例,结果显示,从粪便DNA样本中检测到的鱼种比例与已知投喂的鱼种比例吻合,证实了基于DNA的食性分析方法能够进行定量分析。尽管当时该方法在摄食生态学领域开展了一系列应用实践,但尚未形成具体的试验操作流程与规范指南。King等[26]2008年在《Molecular Ecology》上刊文,总结了DNA食性分析技术的主要流程和最佳实践指南,成为引领该领域发展的标志性文献。随着高通量测序技术的兴起,Deagle等[28]应用宏条形码技术分析了澳大利亚3个不同繁殖区域内海豹(Arctocephaluspusillusdoriferus)的食性,提出该技术极大地加强了研究食物网的能力,适用于对各种动物类群进行大规模食性分析研究,这也是宏条形码技术第一次应用于大规模野生动物食物网研究。随后,Deagle等[29]运用DNA宏条形码技术分析了企鹅(Eudyptulaminor)的食性,并采用人工饲喂对比,着重强调了基于高通量测序的食性分析可以用于半定量食性分析。2009年,Valentini等[27]设计了能够扩增植物叶绿体tRNA(trnL)区域的通用引物,这是首次将宏条形码技术运用于植食性动物的食性研究,使得植食性动物的食物组成和资源分配研究成为可能。Valentini等[15]2010年在顶级期刊《Trends in Ecology & Evolution》上提出,在高通量测序技术支持下,DNA条形码能为生态学家提供更加便捷、高效的研究方法,由此推动了DNA宏条形码技术的兴起。2011年,Zeale等[31]在原有COⅠ通用引物基础上设计了长度157 bp适用于节肢动物的通用引物,该通用引物被证实可以准确识别被3种蝙蝠摄食的节肢动物类群。由于该引物的高效性和特异性,使之成为检验节肢动物(被捕食者)的常用通用引物[33]。2012年,法国学者Pompanon等[16]在《Molecular Ecology》发文,回顾了DNA食性分析技术的研究历程,总结了宏条形码食性分析方法的优缺点,并指出受污染物扩增、PCR错配等问题的限制,即便一个精心设计的研究也可能只提供一个物种饮食的半定量数据,强调了该领域相关研究需要重视数据的处理与分析。这篇文章也成为LCS最高的论文,其为后续研究提供了重要的指导经验[34]。Piol等[32]2015年也对定量分析提出了质疑,认为使用COⅠ通用引物和阻断引物(与宿主DNA目的基因片段结合阻碍其扩增的特异性引物)时,宏条形码技术的定量分析能力有限,仅定性结果可靠,为后续研究提供了警示[35]。
综合分析,LCS高的文章可被认为是与该领域发展历史、技术创新息息相关的学科基础文献。它们通常为高被引文献,研究内容更加侧重于对基于DNA食性分析技术的优化和对该领域研究进程的总结。特别是前3篇论文(表2)贯穿该学科的发展,从基于PCR诊断的食性分析方法的出现,到采用DNA条形码和DNA宏条形码技术研究物种食性,详细地阐述了该学科研究进程中相关分析理念和应用技术的革新。
2.4 研究热点与趋势
对标题或摘要、关键词的提取能够提炼文章研究的主要内容。笔者依据关键词共现知识图谱聚类分析,通过主题词频揭示了该领域的研究趋势(图2)。通过共词分析和突现检测,分析了该领域的研究热点(表3)。笔者将数据导入CiteSpace 5.6.R2中,时间切片为1 a,节点类型为关键词,其余采用默认设置。其中模块值(Q值)和平均轮廓值(S值)表征网络结构与聚类清晰度。Q>0.3意味着网络结构显著,S>0.7意味着聚类高效令人信服[24]。关键词突现强度越高代表该词汇在某时间段内的关注度越高;同质性为衡量聚类成员相似程度,数值越趋近于1,相似性越高。
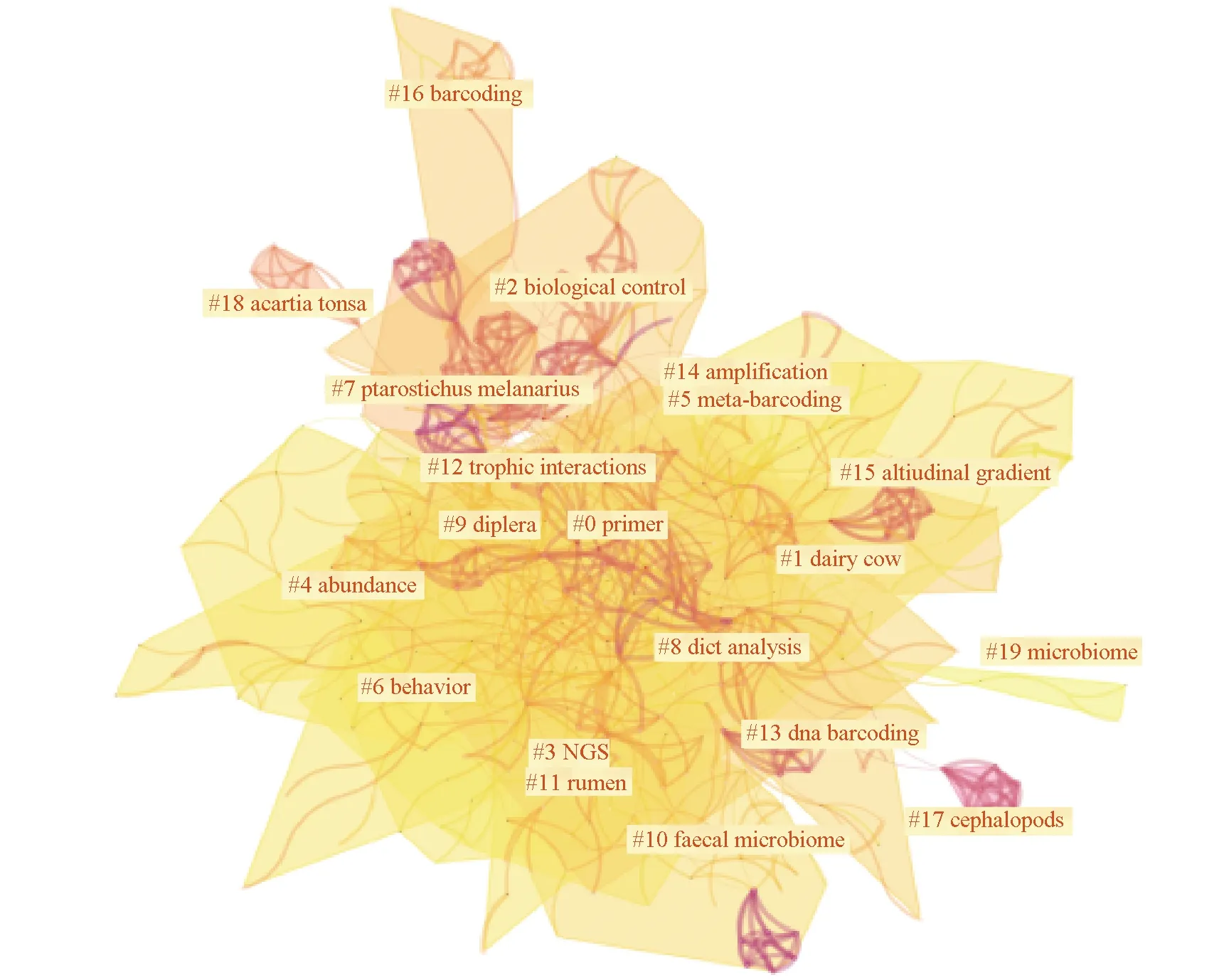
图2 关键词聚类知识图谱Fig.2 Keyword clustering knowledge map

表3 突现词检测检索表Tab.3 The table of burst words
关键词聚类分析结果显示,Q值为0.7675,S值为0.3934,表明关键词共现图谱构建合理。由于聚类的关键词是依照频率统计,聚类标签中的奶牛、通缘步甲虫(Pterostichusmelanarius)、汤氏纺锤水蚤(Acartiatonsa)和头足类是研究的热门物种,与研究前沿、研究趋势无关,故舍弃。笔者依据聚类分析结果,选取前10聚类,绘制了基于DNA食性分析研究的共词聚类分析的延伸内容(表4),同质性均超过0.8,表明聚类相似程度较高。按照时间线分布:
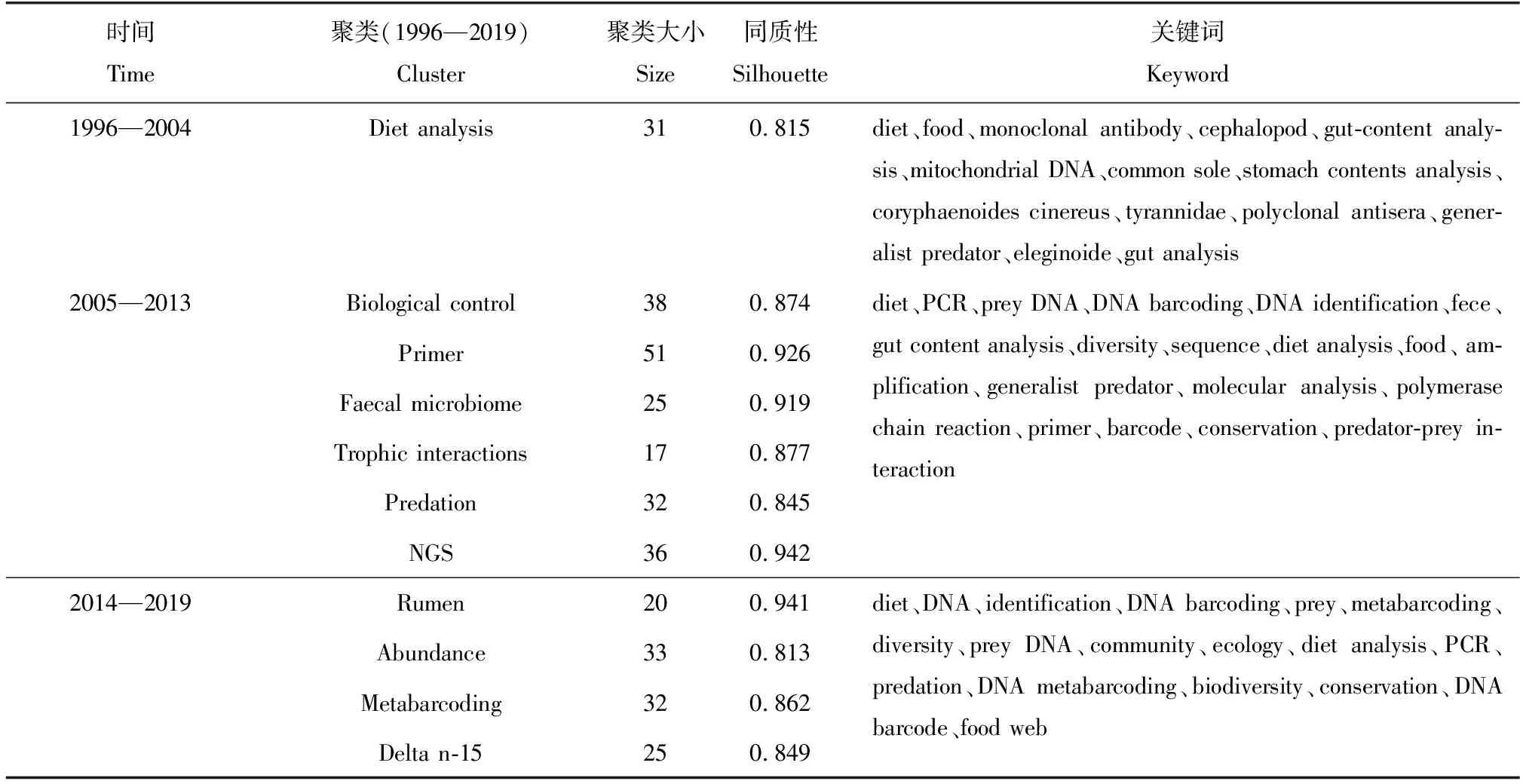
表4 关键词聚类主要信息Tab.4 The main information of keywords clustering
(1) 探索阶段(1996—2004年):伴随着分子生物学技术的革新,DNA食性分析手段由于其便捷、高效的特性引发了生态学研究的重视。食性、单克隆抗体是该阶段频次最高的词汇,表明基于免疫学的食性研究方法依然占据主流。但该阶段研究热点(表3)除单克隆抗体外还有聚合酶链反应。该阶段基于DNA食性分析方法为PCR鉴定,且往往结合肠道内容物分析、胃含物分析等手段[36-37]。其中COⅠ(线粒体DNA)为物种鉴定常用选择区域,食性分析多应用于海洋广食性物种营养关系的确认,如头足类、灰突吻鳕(Coryphaenoidescinereus)等。
(2)发展阶段(2005—2013):该阶段基于DNA食性分析手段日趋完善,涌现了大量的高频词汇和突现词汇,表明基于DNA食性分析方法成为当时的研究热点。DNA条形码概念的提出使得猎物可以通过其DNA的特异性序列实现DNA物种鉴定。PCR、猎物DNA、DNA条形码为该阶段高频词汇,意味着PCR鉴定技术依然是该阶段的核心研究内容[26]。但自Hebert等[14]2003年提出DNA条形码理念后,条形码技术开始兴起,基于DNA食性分析手段逐渐开始由PCR鉴定向条形码技术过渡。聚类“NGS”表明高通量技术的出现,宏条形码技术开始应用于广食性物种食性多样性分析。该阶段的热点研究内容为猎物—捕食者关系和食物网,研究侧重于为生物防治和生物保护等应用领域服务[38]。此外PCR、线粒体DNA、引物和测序等热点研究内容表明,引物选择、PCR体系与循环条件、测序方法、目的基因的选择、基因区域的选择等诸多因素制约着DNA食性分析方法的使用,聚类“Primer”表明该阶段围绕引物展开了一系列研究。简言之,PCR鉴定技术依然是该阶段的热点研究内容,但基于测序技术的食性分析开始展露锋芒。DNA食性分析方法的研究对象由捕食者和被捕食者相互作用拓展到了食物网的动态研究,从个体食性扩展到了种群规模的食性研究,并开始将其应用于更实际的生物防治和保护生物学等领域。
(3)高速发展阶段(2014—2019):DNA宏条形码为该阶段的高频词汇,基于DNA食性分析方法由PCR鉴定技术向DNA宏条形码技术过渡。该技术可以通过计算测序产生序列的种类数与数据库比对实现食物组成的定性研究,还可以比较不同猎物序列的相对丰度,来定量分析食物网内的种间关系[1,33],实现食性研究的半定量分析。由表4可见,研究趋势为植食性有蹄类物种(瘤胃)食性研究[39]。随着宏条形码技术日渐成熟化和系统化[40],并通过与其他研究方法的交叉融合,使得研究层次更加深入。例如,基于DNA的物种多样性调查也依赖DNA宏条形码技术,宏条形码技术的应用能够从分子角度分析群落多样性和物种食性,可以准确分析食物选择性,揭示广食性捕食者复杂的摄食生态[41]。DNA宏条形码技术与稳定同位素食性分析方法(delta n-15)结合,可以探究食物网中不同物种的营养关系,完善食物网的物质循环和能量流动过程[42]。目前,该领域的研究规模逐渐扩大,由种群生态学向保护生物学和群落生态学等研究方向拓展。
综上,随着DNA食性分析方法日趋完善,逐渐解决了全面分析食物网过程中面临的技术难点,也使其成为分子生态学领域的一个十分活跃的方向。通过研究热点和研究趋势分析发现,各阶段研究的规模逐步扩大,更加侧重于在保护生物学领域的应用。研究方法逐渐由单克隆抗体、PCR凝胶电泳分析向DNA条形码、DNA宏条形码等技术过渡发展,研究方法不断进步与完善,这均与分子生物学和测序技术的发展密切相关。目前,基于DNA宏条形码技术的分子生态学食性分析是热点研究内容。
2.5 研究力量
CiteSpace提供了宏观的国家或地区的合作网络、中观的机构合作网络以及微观的学者合作网络分析。笔者使用CiteSpace绘制分子生物学食性分析研究的合作网络知识图谱,依次从国家、机构和作者3种层次评价了学术研究贡献度。其中节点大小代表发文量的多少。笔者将数据导入CiteSpace 5.5.R2中,时间切片为2 a,节点类型依次为国家、机构和作者,国家合作网络知识图谱采用寻径网络法,网络辅助裁剪策略采用合并网络裁剪,其余图谱采用默认设置。图谱中节点用圆圈表示,连线粗细代表节点合作关系强弱。由3个指标衡量节点的重要性,即发文量、突发性、中介中心性。突发性高表明该节点某一时间段内关注度高,中介中心性代表节点在该领域知识结构中关联节点的重要性。
2.5.1 主要研究国家和地区
检索发现有39个国家应用了DNA食性分析技术开展相关研究,其中美国的发文量居世界第一(222篇),表明美国在分子生物学食性研究领域居于领导地位(表5)。然而,基于突现值和中介中心性的分析可知,相较于美国,英国和法国在该领域的研究具有更高的核心影响力。综上,法国关于分子生物学的食性研究最为深入,综合影响力较高,是该领域研究的核心国。而我国开始关注该领域的时间较晚,迄今,国际发文量居于第六位(41篇)。虽然科研水平和发文量在逐年上升,但目前我国在该领域研究的综合影响力仍落后于欧美等发达国家(图3)。
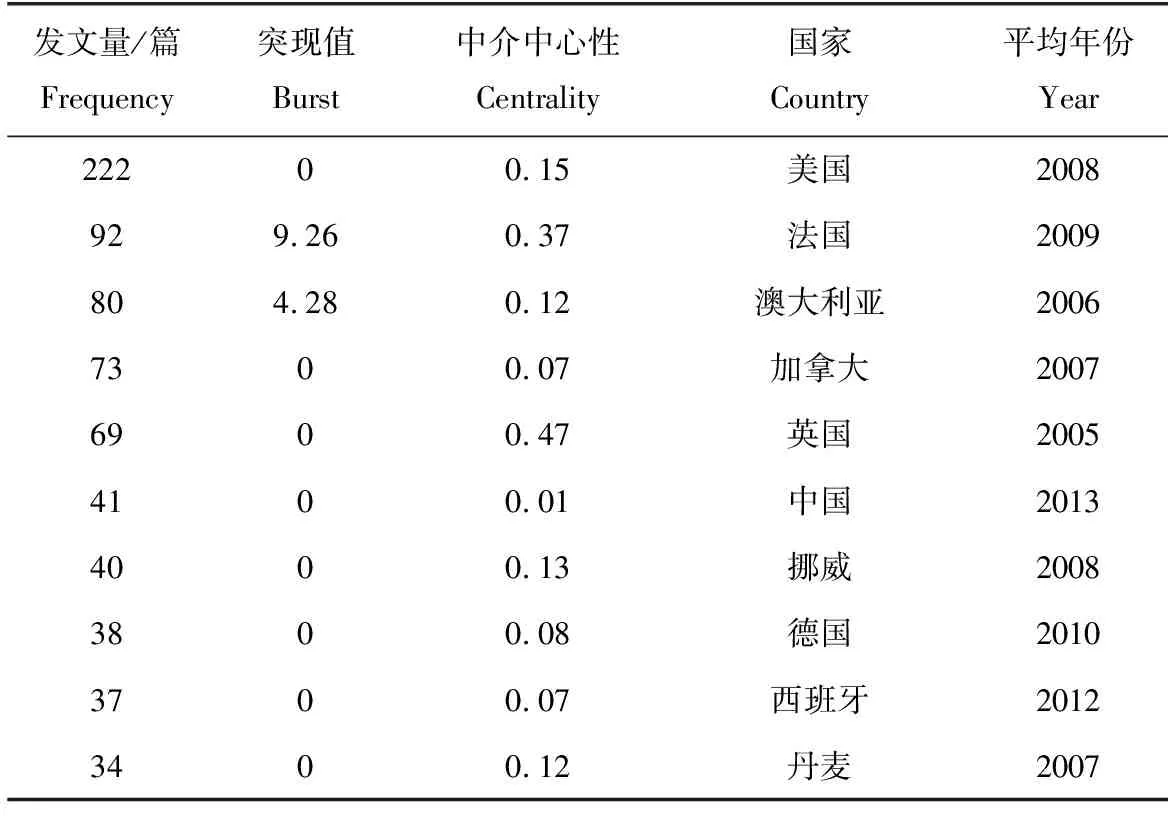
表5 合作发文量前10的国家Tab.5 Top ten countries in the number of cooperative papers

图3 国家合作共现图谱Fig.3 The network of country cooperation
2.5.2 主要合作研究机构
根据机构合作发文量来看(表6),第一名为法国格勒诺布尔—阿尔卑斯大学(31篇);英国卡迪夫大学位居第二位,发文量24篇;第三名澳大利亚南极分部和加拿大圭尔夫大学(22篇)。合作发文量居于前10的机构有半数在欧洲,2家在美洲,4家在澳大利亚。通过中介中心性比较和发文量比较,法国格勒诺布尔—阿尔卑斯大学、英国卡迪夫大学、丹麦哥本哈根大学在发文量和中介中心性上较高,说明这3家机构联系紧密,在分子生物学的食性研究方面具有较高的学术影响力。而法国格勒诺布尔—阿尔卑斯大学突现值为9.23,表明该机构引领基于DNA分析食性研究的前沿,是该领域的核心研究机构。我国中科院有11篇,位居第14位,综合影响力与西方发达国家尚有一定差距(图4)。

表6 合作发文量前10的机构Tab.6 Top ten institutions in the number of cooperative papers

图4 机构合作共现图谱Fig.4 The network of institution cooperationUniv Tasmania.澳大利亚塔斯马尼亚大学; Univ British Columbia.英属哥伦比亚大学; Curtin Univ.澳大利亚科廷大学; Univ Turku.芬兰图尔库大学; Australian Antarctic Div.澳大利亚南极分部; Univ Porto.葡萄牙波尔图大学; Univ Helsinki.芬兰赫尔辛基大学; Cardiff Univ.英国卡迪夫大学; Swedish Univ Agr Sci.瑞士农业科技大学; Univ Bristol.英国布里斯托大学; Uniy Copenhagen.丹麦哥本哈根大学; Smithsonian Inst.美国史密森学会; Univ Innsbruck.澳大利亚因斯布鲁克大学; INRA.法国农业科学研究院; Univ Guelph.加拿大圭尔夫大学; Univ Oslo.挪威奥斯陆大学; Chinese Acad Sci.中国科学院; Univ Grenoble 1.法国格勒诺布尔—阿尔卑斯大学.
2.5.3 主要发文作者
作者的合作网络知识图谱可以深入分析挖掘学者间的合作关系,以及不同团队间的团体效应。数据检索发现,发文量最多的5位分别为法国学者Pierre Taberlet(24篇)、法国学者Eric Coissac(16篇)、澳大利亚学者Michael Traugott(15篇)、丹麦学者Thomas Gilbert(11篇)、英国学者Elizabeth L Clare(10篇),该领域共有177名学者合作交流,且发文量均在2篇以上,说明该领域内研究者相互引用、相互合作较为频繁(表7)。学术团队主要分为:Francois Pompanon、Alice Valentini、Pierre Taberlet和Eric Coissac等所在的法国格勒诺布尔—阿尔卑斯大学高地生态学实验室,其研究聚焦于山地森林生态系统功能和生物多样性,依靠长期的观察和试验,利用研究结果来模拟和预测生物多样性和生态系统对全球变化的响应;Michael Traugott、Bettina Thalinger和Johannes Oehm等所在的澳大利亚因斯布鲁克大学的生态学研究所,其主要研究是通过分子手段分析不同生态系统食物网间的相互作用,侧重于研究农田生态系统昆虫与鸟类之间的相互作用[43-44];Bruce E Deagle、Simon Neil Jarman(澳大利亚)等学术团队主要应用分子生物学分析方法解析海洋生物的摄食生态[45-47];Elizabeth L Clare(英国)、Eero Juhani Vesterinen(瑞典)、Aitor Arrizabalaga(西班牙)等学术团队则是聚焦于农业和森林生态中蝙蝠和虫蛾之间的营养相互作用[48-50]。从作者合作图谱(图5)来看,大多数的学术合作来源于同一研究机构,但不同学术团队间也存在着密切的联系,该领域学术交流合作居多。
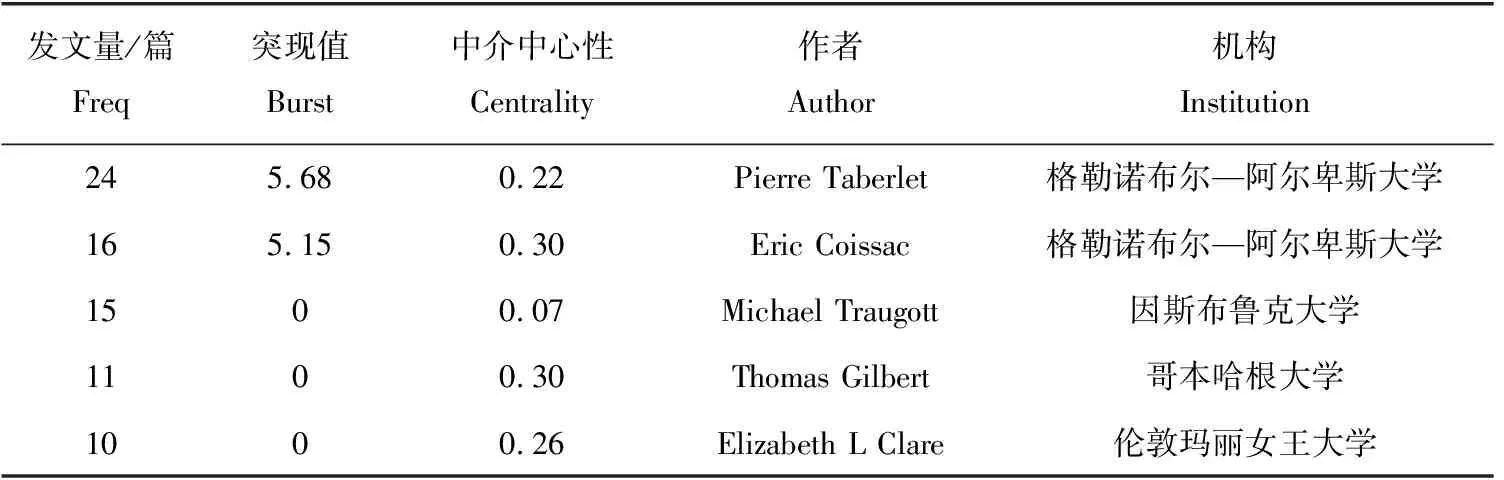
表7 合作发文量前5的作者Tab.7 Top five authors in the number of cooperative papers

图5 作者合作共现图谱Fig.5 The network of author cooperation
3 结语与展望
笔者利用WOS、HistCite和CiteSpace对基于分子生物学食性研究的相关文献进行了定性、定量和可视化分析,厘清了该领域的发展脉络、研究热点、研究趋势以及合作关系。通过发文量的年度波动分析发现,1996—2019年该领域历经了探索—发展—高速发展3个阶段,近13年来,主要是研究技术和研究概念的革新,研究主题聚焦于捕食者—猎物营养关系的确定,向其他领域拓展较少。1996—2004年探索阶段主要聚焦于基于DNA的猎物鉴定技术,PCR鉴定技术与肠道内容物分析技术的结合分析。研究重心为优化PCR扩增环节,如合适的目的基因、循环条件以及引物选择等;2005—2013年为发展阶段,King等[26]2008年为该领域提供了合理的实践指南,基于DNA分子生物学食性分析方法得到了规范,PCR鉴定技术依然为当时流行的食性分析方法。但自加拿大学者Hebert等[14]2003年提出DNA条形码理念后,基于测序鉴定的DNA条形码技术异军突起,DNA食性分析方法的两大主线PCR鉴定和测序分析开始齐驱并进发展;2014—2019年为高速发展阶段,由于宏条形码技术可以在分类学物种水平上识别猎物,且其允许对大量样本进行并行处理和测序。鉴于上述潜力,食性分析方法开始由PCR鉴定技术逐渐过渡到宏条形码技术。目前该领域发表论文的数量几乎呈指数增长[40,51],宏条形码技术成为当下无可争议的研究热点。
从上述3个阶段研究内容的变化可以看出,该领域的发展与分子生物学技术和测序技术的进步密切相关。随着基于DNA食性分析方法的规范化和体系化,研究方法不仅追求科学严谨的试验设计和严格规范的试验操作,还要求无偏见的、严谨的生物信息学数据处理流程。研究内容不再局限于食物组成,还涉及食性比较、摄食变化、食物选择性、营养生态位划分、食物网及其营养动力学等研究[52-55]。随着分子生物学技术的发展,研究人员开始用宏条形码技术解决基于营养关系的生态问题,如恢复生态学、关键种识别、生态系统脆弱性评估和入侵物种响应等[38,56-58]。研究尺度由个体扩展到种群、群落、生态系统等多个层次。
与各阶段相对应,学科基础知识分析的前3篇综述(表2)分别对应各阶段的研究热点和研究问题,为本领域影响力最高的3篇文章,均不同程度地推动并引领了下一阶段的发展,表现出紧密的传承性。通过发文期刊排名和LCS排名也可以看出,《Molecular Ecology》和《Molecular Ecology Resources》是推动该领域发展的核心期刊,这两个顶级期刊均隶属于美国Wiley出版社,美国的科研力量全方位推动和引领了该领域的发展。在国家合作关系图谱中也可以看出,分子生物学食性分析的研究力量主要分布于美国、法国和澳大利亚。但就研究机构而言,法国格勒诺布尔—阿尔卑斯大学的研究较为深入,合作较为频繁,有极高的学术影响力。在分子生物学食性研究领域贡献较高的作者有Pierre Taberlet(法国)、Eric coissac(法国)、Michael Traugott(澳大利亚)、Thomas Gilbert(丹麦)、Elizabeth Clare(英国)等。从合作图谱的3个层面来看,中国与美国、法国等国家在该领域仍存在着较大差距,其主要原因在于我国分子摄食生态学研究起步较晚,主要聚焦于传统的胃含物分析法,仍将分子生物学食性分析作为一种辅助手段,大规模实际应用欠缺[4]。因此使得我国该领域研究发文量较少,且多为定性分析或者与其他食性分析方法结合等[59-61],缺少高被引和具有影响力的高水平论文。
DNA食性分析方法凭借其高效性和特异性,广泛应用于生态学研究。但是每种DNA食性分析技术都有各自的优缺点,并且适用性不一。如PCR诊断需要事先了解捕食者潜在猎物,据此设计特异性引物,而且处理大量样品时耗时耗力[62],但在定量分析方面优于其他分析方法;基于初代测序的条形码技术只能针对单个样本,受限于工作量和测序成本,仅适用于猎物鉴定精确到种的小样品量分析[63];与前者相比,宏条形码技术在研究食物多样性和食谱范围时更具优势,且灵敏度高,但易受污染,数据处理方法不够完善[16]。除此之外,DNA食性分析方法还存在一些技术层面的问题,如引物选用组合、扩增效率差异、测序误差、数据库缺失等。但随着技术的发展和完善,鸟枪法测序[64]、数字PCR[65]、三代测序技术[66]等无偏见精确定量技术的出现,以及数据库的丰富[67]、分析方法的创新[68]、数据处理方法和软件的更新迭代[69],都为该领域研究提供了广阔的发展平台。从长远看,基于DNA食性分析方法有广阔的应用前景,可能会替代一些传统分析技术。但目前该领域研究尚未成熟,不同研究方法的结合应用能够取得更好的效果、解决更多复杂且富有研究意义的问题[2]。如DNA食性分析仅能反映短期摄食状况,稳定同位素能够反映长期摄食状况,两者相辅相成,适用于复杂食物网及营养动力学研究[42]。随着科学技术的发展和研究方法的交叉互补,DNA食性分析技术将得到更加宽泛、更加深入的应用。在未来,新定量方法开发、宏条形码技术数据优化、食物网营养动力学和生态系统功能研究将成为该领域的主流趋势[18]。基于高通量测序的食性分析技术是当下的研究热潮,鉴于其优势和潜力,很可能在未来数十年继续保持这一迅猛的发展势头[40]。在生态学研究越来越倾向于多方位合作交流的今天,我国应当加强国际间的交流合作,培养核心团队,加强实践,完善操作体系和操作流程,进一步补充我国生物物种的DNA条形码数据库,扩大分子生物学食性分析研究的规模,提高食物网营养动力学和生态系统功能研究的层次和水平。

