土地类芽胞杆菌(Paenibacillus terrae)NK3-4 EsxA结构与系统发育分析
于文清,闫凤超,刘文志,*,郑桂萍,肖俊杰
(1.上饶师范学院 生命科学院,江西 上饶334001;2.黑龙江省农垦科学院,哈尔滨 150036; 3.黑龙江八一农垦大学,黑龙江 大庆 163319)
EsxAs(早期分泌抗原靶6,Early secreting antigenic target-6,ESAT6)是一类小分子分泌蛋白,一般含有100左右个氨基酸,具有保守的WXG氨基酸基序[1],因此被称为WXG超级家族成员。该蛋白呈螺旋状结构,与EsxB(CFP-10)共同组成VII型分泌系统(T7SS),EsxA最先在动物病原菌结核分支杆菌(Mycobacteriumtuberculosis)中发现[2],与其致病力密切相关[3-5],后陆续在其它动物病原菌中也发现了该类蛋白[6]。EsxA除了与动物致病菌的致病性相关外[7-8],还能够诱导动物的免疫原活性[9-11],也有研究发现,在单核球增多性李斯特菌中(Listeriamonocytogenes),EsxA与宿主病原菌的致病性无关[12]。在非致病细菌中也存在EsxA编码基因,如在天蓝色链霉菌中,EsxA与EsxB形成同源二聚体,与宿主菌成孢相关[13]。
前期研究发现,植物根际促生菌土地类芽胞杆菌(Paenibacillusterrae)NK3-4基因组中携带着EsxA编码基因,但EsxA在类芽胞杆菌中发挥何种功能尚无相关报道,是否与类芽胞杆菌对植物的促生效应相关也尚未可知。本文采用生物信息学技术分析了该EsxA的基本性质、蛋白的3D结构及系统发育关系,以期为包括NK3-4菌株在内的类芽胞杆菌EsxA基因外源分泌表达、活性保持条件及功能的深入研究提供理论依据。
1 材料与方法
1.1 NK3-4菌株EsxA基本性质分析
将NK3-4基因组中注释为毒力因子(Virulence factor EsxA)的氨基酸序列提交ExPASy Proparam tool(https://web.expasy.org/cgi-bin/protparam/protparam)分析其基本性质,将氨基酸序列提交NCBI中的BLSTp进行序列相似性比对,并检索保守域。
1.2 NK3-4菌株EsxA3D结构预测
将NK3-4 菌株EsxA氨基酸序列提交在线蛋白质结构预测网站SWISS-MODEL(https://www.swissmodel.expasy.org/interactive/rgsxPX/models/),通过同源建模预测其3D结构[14],大致步骤如下:
(1)模板搜索选择及建模:将序列提交SWISS-MODEL后,会自动匹配相似的模板,对于每个识别的模板,都列出了预测模板-目标匹配质量,选择质量最高的模板建模。将模板与目标序列比对,模板与序列之间保守匹配的结构被从模板复制建模,用片段库插入或删除,再重新建模,而后重建侧链,最后利用最小作用力的几何学特性构建模型。
(2)建模质量评估及配基建模:利用QMEAM评分功能进行全局性和残基模型质量评估。配基建模时,只有满足以下全部条件后才代表存在配基:(i)配体在模板库中注释为生物学相关;(ii)配体与模型联结在一起;(iii)配体与模型之间不冲突;(iv)与配体结合的残基在模板与目标蛋白之间是特异保守的。
1.3 NK3-4菌株EsxA系统发育树构建
利用MEGA6软件对NK3-4菌株EsxA及与其同源性较高的蛋白序列进行聚类分析,这些蛋白的氨基酸序列从Uniport数据库(https://www.uniprot.org/)中获得。对比序列类型选择“Translated protein Sequenences”,统计方法采用邻接法(Neighbor-joining),模板采用最大似然法(Maximum Composite Likeliood method)。系统发生检测采用自举法(Bootstrap method),成对比对参数(Pairwise Alignment)为缺口开启罚分(Gap Opening Penalty):10,且缺口延伸罚分(Gap Extenson Penalty):0.1;多重比较参数(Multiple Alignment)为缺口开启罚分:10,缺口延伸罚分:0.2;蛋白权值矩阵(Protein Weight Matrix):Gonnet;特殊氨基酸罚分(Residue-specific Penalties):ON,亲水性罚分(Hydrophilic Penalties):ON;缺口分离距离(Gap Separation Distance):4;末端缺口分离(End Gap Separation):OFF。
2 结果与分析
2.1 NK3-4菌株EsxA的基本性质
2.1.1 NK3-4 菌株EsxA的理化性质
NK3-4菌株EsxA含有91个氨基酸,分子质量10 276.53 道尔顿,理论等电点 5.29,分子式为C445H711N125O146S4,原子数目1 431,消光系数(280 nm)5 500,Abs 0.1%(=1 g/L)0.535,估计半衰期为30 h(哺乳动物的网状细胞,体外),>20 h(酵母,体内)和>10 h(大肠杆菌,体内)。不稳定指数46.28,归为不稳定。脂肪族指数65.38,大致亲水性平均值-0.548。表明NK3-4菌株EsxA为弱酸性、亲水性蛋白质,三级结构不稳定。
2.1.2 NK3-4菌株EsxA的保守域
CDD分析表明,EsxA 含有WXG100 ESAT6保守域及WXG高度保守基序第44到46个氨基酸:W(甘氨酸)-E(谷氨酸)-G(色氨酸),是WXG超级家族成员,该EsxA氨基酸序列及其保守域(见图1)。

图1 土地类芽胞杆菌NK3-4 EsxA氨基酸序列及其保守域Fig.1 Amino acide sequence and conserved domain of EsxA in Paenibacillus terrae NK3-4
2.2 NK3-4菌株EsxA 3D结构预测结果
2.2.1 模板筛选及模型构建结果
将NK3-4菌株EsxA氨基酸序列提交SWISS-MODEL后,共查出417个模板,其中一个名为EsxA的蛋白与提交序列识别率30%,相似性36%,可以该蛋白为模板建模。蛋白模板是同源二聚体,没有配基,A、B链(见图2)分别覆盖NK3-4菌株EsxA 1~90及7~90个氨基酸,覆盖率99%。

图2 土地类芽胞杆菌NK3-4 EsxA结构预测模板Fig.2 Template for Paenibacillus terrae NK3-4 EsxA 3D structure prediction
2.2.2 建模质量评估结果
建模评估表明,分子概率统计表明,分子概率分值1.22,冲突分数4.42,拉氏亲和99.41%,拉氏离群值0.0%,旋转异构体异常值0.0%,C-Beta偏差为0。全局性模型质量评估显示,QMEAN为-1.06(见图3a);局部模板质量评估表明,预测的局部文库与目标序列预测相似性(见图3b);预测模型与同源蛋白质三级结构的相似性分析表明,与PDB结构的非冗余集比较,标准QMEAN4分值为0.74(见图3c),结果表明(散点图中以红色五角星标记),Z得分<1,显示该预测模型结构稳定,是最佳的理想结构。

图3 土地类芽胞杆菌NK3-4 EsxA结构预测模板质量评估结果Fig.3 Assessment results of template for Paenibacillus terrae NK3-4 EsxA structure prediction
拉氏构象分析表明,预测模型坏键0/1 394、坏角6/1 874,包括A66 Phe、(B8 Thr-B9 Pro)、A59 Ala、A82 Asn、B11 Gln、B49 Lys,两个亚基中,分别有97.8%和95.6%的氨基酸残基位于深绿色最佳允许区域,且全部残疾均位于额外允许区域(见图4)。表明预测模型中全部氨基酸残基均形成了合理的二面角,构成的蛋白质结构稳定。
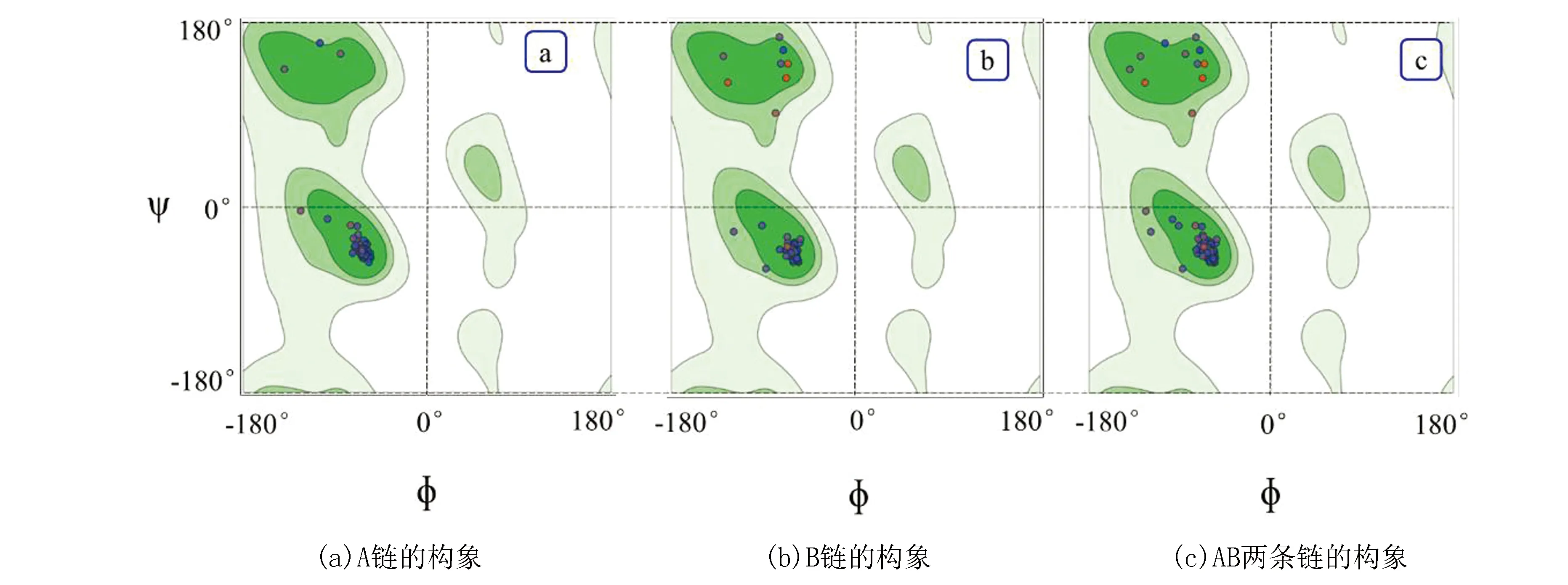
图4 土地类芽胞杆菌NK3-4 EsxA结构预测模板拉氏构象图Fig. 4 Ramachandran plots of template for Paenibacillus terrae NK3-4 EsxA注:图中深绿色区域代表键角稳定,绿色区域代表键角虽然不稳定,但可在自然界存在,淡绿色区域代表键角不稳定.
2.2.3 NK3-4菌株EsxA 3D结构预测结果
NK3-4菌株EsxA 结构分析显示,EsxA形成同源二聚体,其中每个亚基都由一个β折叠连接两个α螺旋组成,这两个α螺旋反向平行排列。二聚体中两个亚基的肽链反向排列,所有N末端和两个C末端均暴露在外。这个二聚体是不对称的结构,其中一个亚基(B链)自由的N端肽段暴露出来,形成3D结构(见图5)。

图5 预测的NK3-4菌株EsxA激发子结构Fig.5 Predicted structure of Paenibacillus terrae NK3-4 EsxA注:a,b为EsxA二聚体;c和d为计算出的二聚体表面电势图(蓝色-正电荷,橙色-负电荷,紫色-中性);b和d分别为a和c旋转180°后的图象;图中标注了每个肽链的C端和N端(标注1的为A链,标注2的为B链).
聚类分析表明,EsxA主要分成两个大簇(见图6a,6b),NK3-4菌株EsxA与其中一簇中的两个P.terrae及两株多粘类芽胞杆菌(Paenibacilluspolymyxa)聚为一个小支,该小支内的氨基酸序列是完全相同的,同时与其它类芽胞杆菌聚为一小簇(见图6c),这一小簇再依次与侧短芽胞杆菌(Brevibacilluslaterosporus),Paenibacillusyonginensis,高温放线菌(Thermoactinomycessp)及韩氏芽胞杆菌(Bacilluskoreens)聚为一个大簇(见图6a);在另一个大簇中,另一些类芽胞杆菌属菌株EsxAs聚为一小簇(见图6b),这两个大簇与红色梭菌(Clostridiumroseum)和毡毛梭菌(ClostridiumfelsineumDSM 794)最终聚到一起。可见,EsxAs在种间及属间进化关系是不保守的,它们被明显的分为两个进化关系较远的类簇,EsxAs做为一类分泌型小分子蛋白,广泛存在于包括类芽胞杆菌的微生物中,且包括NK3-4菌株在内的类芽胞杆菌的EsxAs与动物致病菌的毒力因子EsxAs亲缘关系甚远。亲缘关系较远可能是EsxA功能存在差异的原因。

图6 土地类芽胞杆菌NK3-4 EsxA的系统发育树Fig. 6 Phylogenetic tree of Paenibacillus terrae NK3-4 EsxA注:树上分类单元标注了各蛋白在Uniport中的登录号及来源菌种或菌株名称,a,b,c分别为不同亚分支。
3 讨 论
本文分析了类芽胞杆菌NK3-4 EsxA的基本性质、3D分子结构及系统发育关系,为其后续研究与应用提供了理论依据,主要表现在明确该蛋白的基本理化性质,可为EsxA基因外源分泌表达条件优化提供依据,也可为分泌表达至胞外的蛋白纯化条件优化提供参考,还可为优化蛋白活性保持条件提供依据。3D结构预测有助于了解EsxA的特殊结构及功能,有助于认识该蛋白可能存在的与其它分子之间的相互作用的位点,并指导进行功能确认的生物学实验设计。系统发育关系分析可为判断该EsxA在进化过程中的保守性与变异性提供依据,本文将NK3-4菌株EsxA与其相似性最高的34个蛋白质进行聚类分析,表明这些EsxA在种内与种间均是不保守的,说明其编码基因变异性强,是一类进化速度较快的蛋白,这也是我们认为其在类芽胞杆菌中与在病原细菌中具有不同功能的依据。在包括NK3-4菌株在内的类芽胞杆菌中,在类芽胞杆菌与植物进行互作时,其EsxA是否通过分泌到细胞外参与类芽胞杆菌对植物的促生效应,如参与宿主与植物的互作,其作用的分子机制如何,将是作者下一步研究将要揭示的答案。
4 结 论
采用生物信息学技术,明确了土地类芽胞杆菌NK3-4基因组携带的EsxA基因编码的小分子分泌型蛋白的理化性质,分析了保守域并定位了及保守基序WEG。采用建模的方式,通过对模板的一系列质量评估,预测了蛋白质的3D结构,并基于该EsxA氨基酸序列构建了系统发育树,系统发育分析表明,包括NK3-4菌株在内的类芽胞杆菌的EsxA与致病细菌的EsxA同源关系较远,这暗示非致病菌分泌的EsxA可能具有与病原菌分泌的EsxA具有截然不同的功能。结果为NK3-4菌株在内的非致病菌EsxA基因外源分泌表达条件、蛋白活性保持及功能的深入研究奠定了理论基础。

