海南槟榔黄化植原体分子检测及其系统发育关系研究
于少帅 宋薇薇 覃伟权
摘 要:由植原体引起的槟榔黄化病是海南特色经济作物槟榔种植上的一种毁灭性病害。本研究通过PCR扩增测序、序列多重比对和系统发育分析,对海南部分代表性地区槟榔致死性病原植原体的序列信息与系统发育关系进行检测分析。结果表明:本研究检测的保亭、屯昌、万宁等海南部分代表性地区的槟榔黄化植原体的16S rDNA序列一致;BLAST分析表明各株系16S rDNA与16SrI组植原体同源性为100%。序列多重比对分析表明,本研究各槟榔黄化植原体株系与海南苦棟丛枝、长春花绿变、马松子变叶、辣椒黄化皱缩、蛇婆子丛枝、细圆藤丛枝和日本洋葱黄化、美国翠菊黄化等植原体同源性为100%;与已报道的海南万宁、印度、海南三亚的槟榔黄化植原体16S rDNA序列同源性分别为99.9%、99.9%、99.8%。系统发育分析表明,本研究槟榔黄化植原體与海南苦棟丛枝、长春花绿变、马松子变叶、辣椒黄化皱缩、蛇婆子丛枝、细圆藤丛枝等株系聚于一个分枝,支持率为100%。此外,在发病槟榔叶片、花穗、心叶等组织部位均可检测到植原体。研究结果表明,海南槟榔黄化植原体与海南苦棟丛枝、长春花绿变、马松子变叶、辣椒黄化皱缩、蛇婆子丛枝、细圆藤丛枝等植原体株系的同源性极高,槟榔黄化病很可能会以这些寄主植物作为病原传播载体进行传播扩散。
关键词:植原体;槟榔黄化病;分子检测;亲缘关系;传播载体
中图分类号:S436.67 文献标识码:A
Abstract: Areca palm yellow leaf (AYL) disease caused by phytoplasma is a devastating disease in the cultivation of areca palm, an important economic crop in Hainan. Through PCR amplification and sequencing, multiple sequence alignment and phylogenetic analysis, the sequence information and phylogeny of the lethal pathogen infecting areca palm from part representative regions of Hainan were analyzed. The 16S rDNA sequence of the AYL phytoplasma in part representative areas of Hainan, such as Baoting, Tunchang, Wanning County, were identical. BLAST analysis based on 16S rDNA showed that the phytoplasm in the study was 100% homology with 16SrI group phytoplasma. Multiple sequence alignment indicated that the phytoplasma in the study was 100% homology with the phytoplasma strains of Chinaberry witches’-broom, Periwinkle virescence, Melochia corchorifolia phyllody, Pepper yellow crinkle, Waltheria indica virescence, Pericampylus glaucus witches’-broom in Hainan and Onion yellows in Japan, Aster yellow witches’-broom in USA. Homology with the AYL phytoplasma from Wanning, Sanya (Hainan, China) and India was 99.9%, 99.9% and 99.8%, respectively. Phylogenetic analysis indicated that AYL strains in the study were clustered into one clade with the phytoplasma strains infecting the plant hosts like chinaberry, periwinkle, Melochia corchorifolia, pepper, Waltheria indica, Pericampylus glaucus in Hainan with 100% bootstrap value. Furthermore, the phytoplasma could be detected in the leaves, flower spikes, and heart leaves from the diseased areca palm. The study indicated that the homology of the AYL phytoplasmas was significantly high with the strains of chinaberry witches’-broom, periwinkle virescence, Melochia corchorifolia phyllody, pepper yellow crinkle, Waltheria indica virescence, Pericampylus glaucus witches’-broom. AYL disease is very likely to spread using the host plants as pathogen transmission vectors.
Keywords: phytoplasma; areca palm yellow leaf disease; molecular detection; genetic relationship; transmission vector
DOI: 10.3969/j.issn.1000-2561.2021.11.003
植原体(phytoplasma)是一类寄生于植物韧皮部通过刺吸式口器昆虫传播的植物毁灭性病原菌,1967年由日本学者土居养二首次发现[1]。植原体很难从植物或昆虫寄主中分离纯化培养,对其遗传信息及表达代谢等了解相对不足,严重制约植原体研究[2-3]。植原体引起的病害寄主种类多、分布范围广、危害程度大,对社会经济、生态环境等影响严重,该病害目前尚无有效的治疗药剂,以防為主。海南省植原体病害种类十分丰富,约占我国已鉴定植原体病害的五分之一,海南植原体病害遍及整个海南岛且危害程度严重,给海南省农林业造成巨大损失[4]。海南省受植原体病害影响的作物有槟榔(Areca catechu L.)、花生(Arachis hypogaea)、苦楝(Melia azedarach)、长春花(Catharanthus roseus)、辣椒(Capsicum annuum)、细圆藤(Pericampylus glaucus)、山黄麻(Trema tomentosa)等海南特色经济作物或绿化植物,对海南的社会经济和生态环境等造成严重的影响[4-8],其中槟榔黄化病(areca palm yellow leaf disease, AYL)在海南发生普遍且严重,影响巨大。
槟榔是我国“四大南药”之首和海南省重要特色经济作物,具有很高的药用价值和经济价值。由植原体引起的槟榔黄化病是海南槟榔种植上的一种毁灭性病害,对海南的社会经济和生态环境造成严重影响。海南槟榔黄化病于1981年首次发现于屯昌县,目前已由发病初期的屯昌县蔓延至海口、文昌、琼海、万宁、陵水、琼中、定安、乐东、保亭、三亚、五指山等多个市(县)[4, 9]。金开璇等[10]通过超薄电镜切片在槟榔黄化病染病组织中观察到了植原体。车海彦[4]、罗大全等[11]、通过超薄电镜切片观察、四环素注射处理、PCR扩增检测等技术进一步证实植原体是海南槟榔黄化病的病原。近年来,在海南不同槟榔种植区均有不同程度的发生,且发病地区及面积仍在不断扩大,严重影响海南槟榔产业的健康可持续发展[4, 9]。目前,海南植原体病害的发生传播、扩散流行等问题尚不清楚,海南不同县(市)、不同生境、不同发病区植原体的来源、传播途径等问题尚不明确,这些问题严重制约海南相关植原体病害的防控管理。
本研究对海南不同代表性地区的槟榔黄化病进行调查,采集槟榔黄化病及疑似植原体引起的其他植物病害样品,通过PCR扩增检测、序列多重比对、系统发育分析等技术方法,从分子水平上对海南槟榔黄化植原体进行检测鉴定,丰富对槟榔黄化植原体分子水平上的认知。对本研究中槟榔黄化植原体与前期已报道的槟榔黄化植原体及亲缘关系或地理分布密切相关的植原体株系间的系统发育关系进行分析,揭示不同寄主植原体株系间遗传变异水平和系统发育关系。为槟榔黄化病在海南的传播扩散、流行监测和科学防控等研究提供科学参考,以促进海南该类病害防控理念的发展与提升。
1 材料与方法
1.1 材料
1.1.1 样品采集 实验材料于2019—2020年采自海南保亭、屯昌、万宁等地疑似槟榔黄化植原体引起的槟榔黄化病植株,每个地点采集3份叶片样品;针对同一株发病槟榔,采集叶片、花穗、心叶、茎皮、根等不同组织部位,每个组织部位采集3份样品,同时采集健康的槟榔样品作为对照,用作阳性对照的泡桐丛枝植原体样品取自中国林业科学研究院,所采样品低温保存备用。
1.1.2 试剂 植物基因组DNA提取试剂盒(离心柱型)、2×PCR Master Mix预混液、50×TAE、SYBR Green I染料、6×Loading Buffer、DNA分子量标准DL2000购自天根生化科技(北京)有限公司,其他试剂均为国产分析纯。PCR扩增引物由生工生物(上海)工程股份有限公司合成。
1.2 方法
1.2.1 总DNA提取 叶片、花穗、心叶、茎皮、根等样品取样量均为0.1 g,利用CTAB法提取染病槟榔组织中的总DNA,植原体总DNA提取方法参考天根植物基因组DNA提取试剂盒说明书方法。
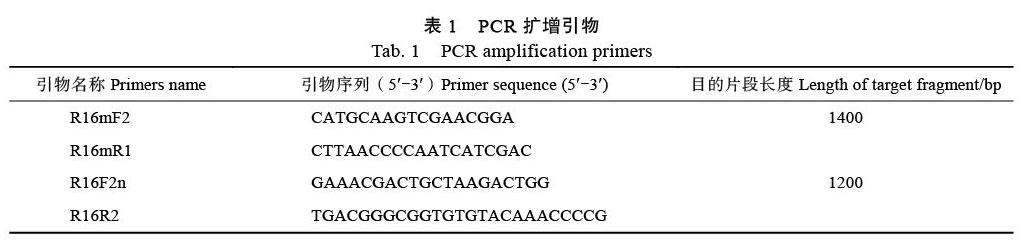
1.2.2 PCR扩增测序 槟榔黄化植原体通过植原体通用引物R16mF2/R16mR1、R16F2n/R16R2[2]进行扩增。用引物R16mF2/R16mR1扩增的PCR产物稀释50倍后用作引物R16F2n/R16R2进行巢式PCR扩增的模板,引物序列信息见表1。PCR反应体系和条件如下:PCR反应体系为25 μL,包括1.0 μL DNA模板,1.0 μL上游引物和下游引物(10 μmol/L),12.5 μL 2×PCR Master Mix(包含0.05 U/μL Taq DNA聚合酶,4 mmol/L MgCl2和0.4 mmol/L dNTPs),用ddH2O补至25 μL。反应条件为94.0 ℃,5 min;94.0 ℃,30 s,53.0 ℃,40 s,72.0 ℃,1 min 30 s(直接PCR)或1 min 20 s(巢式PCR),共35个循环;72.0 ℃,10 min。PCR扩增产物用SYBR Green I染色、经1.0%(w/V)琼脂糖凝胶、凝胶成像系统检测。相关PCR扩增引物由生工生物工程(上海)股份有限公司合成。PCR扩增产物用1.0%琼脂糖凝胶检测,有明显目的条带的扩增产物送生工生物工程(上海)股份有限公司测序。本研究所得核苷酸序列均上传至GenBank数据库。
1.2.3 序列比对分析 获得的核苷酸序列采用DNAMAN 6.0软件(Lynnon Corporation, Vau-dreuil-Dorion, Quebec, Canada)进行编辑,并进行多重序列比对,通过多重序列比对揭示不同植原体株系间的相似性及其变异位点。基于槟榔黄化植原体16S rDNA序列进行BLAST分析(https://blast.ncbi.nlm.nih.gov/Blast.cgi)。
1.2.4 系统发育分析 利用MEGA 7.0软件的邻接法(neighbor-joining, NJ)构建系统发育树[12]。自展值(bootstrap value)设为1000,用以评估软件生成的系统进化树的稳定性和支持率[13]。洋葱黄化植原体(onion yellows phytoplasma)OY-M(AP006628)、翠菊黄化植原体(aster yellow phytoplasma)AYWB(CP000061)、苹果簇生植原体(apple proliferation phytoplasma)CPM(CU469464)、澳大利亚葡萄黄化植原体(Australia grape yellow phytoplasma)CPA(AM422018)和草莓致死黄化植原体(strawberry lethal yellow phytoplasma)SLY(CP002548)等全基因组测序已完成的植原体株系作为构建系统发育树的外类群。
1.3 数据处理
本研究所得核苷酸序列均通过Submission Portal(https://submit.ncbi.nlm.nih.g-ov/subs/genb-ank/)上传至GenBank数据库并获得序列号。
2 结果与分析
2.1 槟榔黃化植原体分子检测
在海南保亭、屯昌、万宁等3个地点采集的9份槟榔黄化病样品中,均扩增到植原体目的条带,在健康的槟榔样品中未扩增到目的条带,槟榔黄化病症状及其扩增检测结果见图1。部分样品PCR扩增产物送测序,均获得植原体靶标基因序列。所得靶标基因序列经BLAST比对分析表明:所得序列为植原体16S rDNA序列,与16SrI组植原体的16S rDNA序列同源性极高甚至一致,如本研究所得植原体的16S rDNA序列与GenBank数据库中的16SrI组油菜变叶病植原体(rapeseed phyllody phytoplasma)(CP055264)的16S rDNA序列的同源性为100%,覆盖率为100%。所得序列已上传至GenBank数据库,采自海南省保亭县的槟榔黄化植原体株系AYL-hnbt的16S rDNA基因序列号为MZ427307,采自海南省屯昌县的槟榔黄化植原体株系AYL-hntc的16S rDNA基因序列号为MZ427308,采自海南省万宁市的2个槟榔黄化植原体株系AYL-hnwn1和AYL-hnwn2的16S rDNA基因序列号分别为MZ427309和MZ427310。
2.2 序列同源性分析
基于植原体16S rDNA序列多重比对分析表明:海南保亭、屯昌、万宁等3个地点9份样品中检测到的槟榔黄化植原体16S rDNA序列一致,无变异位点,序列同源性为100%(表2)。同源比对分析表明:本研究采自海南保亭、屯昌、万宁等3个地点的槟榔黄化植原体株系AYL-hnbt、AYL-hntc、AYL-hnwn1、AYL-hnwn2与前期已鉴定的来自海南万宁(FJ998269)、印度(KF728948)的槟榔黄化植原体株系的16S rDNA序列同源性为99.9%,与来自海南三亚(FJ694685)的槟榔黄化植原体株系的16S rDNA序列同源性为99.8%。本研究采自海南保亭、屯昌、万宁等3个地点的槟榔黄化植原体株系与前期已鉴定的来自海南的苦楝丛枝植原体(chinaberry witches’- broom phytoplasma)(KP662119、KP662120)、长春花绿变植原体(periwinkle virescence phytoplasma)(KP662136)、马松子变叶植原体(Melochia corchorifolia phyllody phytoplasma)(KX150461)、辣椒黄化皱缩植原体(pepper yellow crinkle phytoplasma)(MT760793)、细圆藤丛枝植原体(Pericampylus glaucus witches’- broom phytoplasma)(MT872515)、蛇婆子绿变植原体(Waltheria indica virescence phytoplasma)(MW353909)及来自日本的洋葱黄化植原体(AP006628)、来自美国的翠菊黄化丛枝植原体(CP000061)等各个植原体株系间的同源性为100%(表2)。
2.3 系统发育分析
采用MEGA 7.0软件基于邻接法构建系统发育树。本研究中采自海南保亭、屯昌、万宁等地的槟榔黄化植原体与车海彦[4]检测的海南三亚槟榔黄化植原体株系(FJ694685)、周亚奎等[9]检测的海南万宁槟榔黄化植原体株系(FJ998269)、Muddumadiah等[14]检测的印度槟榔黄化植原体株系(KF728948)聚于一个进化分枝,支持率为100%,均为16SrI组植原体(图2)。Ramaswamy等[15]通过16S rRNA基因序列分析发现印度Sullia地区引起槟榔黄化病的槟榔黄化植原体属于16SrXI组植原体株系(JN967909),与海南槟榔黄化植原体区别明显(图2,表2)。由此可见,槟榔黄化病可以由不同组或亚组的植原体导致,但目前未见2种以上植原体复合侵染引起槟榔黄化病的报道。此外,本研究鉴定的海南槟榔黄化植原体AYL-hnbt、AYL-hntc、AYL- hnwn1、AYL-hnwn2与海南引起苦楝丛枝(KP662119、KP662120)、长春黄花绿变(KP662136)、马松子变叶(KX150461)、辣椒黄化皱缩(MT760793)、细圆藤丛枝(MT872515)、蛇婆子绿变(MW353909)等相关病害的植原体株系聚于一个进化分枝,支持率为100%(图2)。
2.4 发病槟榔不同组织部位植原体检测
在海南保亭、屯昌、万宁采集表现黄化症状的同一株槟榔的不同组织部位进行检测,基于植原体16S rRNA基因序列扩增分析表明:在发病槟榔的叶片、花穗、心叶这3个组织部位中均检测到植原体16S rRNA基因序列目的条带,目的条带约为1200 bp;在发病槟榔的根、茎皮这2个组织部位均未检测到植原体16S rRNA基因序列目的条带;在健康无症状的槟榔叶片、花穗、心叶、茎皮、根等不同组织部位以及去离子水阴性对照中均未检测到植原体16S rRNA基因序列的目的条带(图3)。
3 讨论
本研究对我国海南保亭、屯昌、万宁等地的槟榔黄化植原体检测鉴定表明不同地区槟榔黄化植原体同源性很高甚至一致,本研究中不同槟榔黄化植原体株系间序列相似性为100%,均归类于16SrI组植原体。研究结论与海南前期已报道的槟榔黄化植原体株系为同一组,但存在一定程度的变异。车海彦[4]基于16S rRNA基因在海南屯昌、琼海、定安、万宁、三亚等地均检测到槟榔黄化植原体,通过序列比对和系统发育分析确定其鉴定的槟榔黄化植原体归属于16SrI组;周亚奎等[9]基于16S rRNA基因PCR扩增分析也表明海南槟榔黄化病是由16SrI组槟榔黄化植原体引起的。本研究中来自海南的槟榔黄化植原体与车海彦[4]、周亚奎等[9]检测的海南槟榔黄化植原体的16S rDNA序列同源性极高,分别为99.9%和99.8%。
与国外槟榔黄化植原体检测鉴定研究相比,本研究报道的槟榔黄化植原体与Muddumadiah等[14]报道的印度槟榔黄化植原体均属于16SrI组植原体,16S rDNA序列同源高达99.9%。Ramaswamy等[15]基于16S rRNA基因序列分析表明印度檳榔黄化病可以由16SrXI组槟榔黄化植原体引起;Silva等[16]基于16S rRNA基因序列分析表明斯里兰卡槟榔黄化病由16SrXIV组槟榔黄化植原体引起。目前,海南已报道的槟榔黄化植原体均为16SrI组植原体,未见其他16Sr组的植原体株系或2种以上的植原体复合侵染而引起槟榔黄化病的报道
海南属热带岛屿,具有独特的生物多样性与地理生态特征。已有报道表明海南的植原体及其相关病害的种类十分丰富,数量相对较多,地理分布几乎遍及整个海南,对海南一些主要的经济作物、生态植物危害十分严重[3-4]。本研究表明海南引起槟榔黄化病的槟榔黄化植原体株系与海南引起苦楝丛枝、长春黄花绿变、马松子丛枝、辣椒黄化皱缩、细圆藤丛枝、蛇婆子绿变等病害[5-7]的植原体株系同源性极高,亲缘关系很近,相关植原体株系间的16Sr DNA序列的同源性高达100%,极有可能是同一种植原体侵染不同的植物寄主引起相应的植物病害症状。
植原体主要通过寄主种苗、媒介昆虫、菟丝子(Cuscuta chinensis)等传播介体进行长距离或短距离传播。本研究发现海南槟榔黄化植原体与海南苦楝丛枝、长春黄花绿变、马松子丛枝、辣椒黄化皱缩、细圆藤丛枝等植原体[5-7]同源性高达100%,相关植原体株系亲缘关系极近,侵染槟榔引起槟榔黄化病的植原体可能侵染苦楝、长春花、马松子(Melochia corchorifolia)等寄主植物引起相关植物丛枝、绿变等症状,即槟榔黄化植原体的传播除了可以通过槟榔种苗、媒介昆虫、菟丝子等载体进行传播,也可能会通过苦楝、辣椒、长春花等植原体寄主植物的运输流通而进行传播扩散。因此,在槟榔黄化病的防控过程中,除了对槟榔种苗、媒介昆虫等进行防控管理外,也应对苦楝、辣椒、长春花等相关植原体寄主植物的运输流通加强检疫管理,从源头上切断病原传播途径,杜绝病害传播扩散。
参考文献
[1] Doi Y, Teranaka M, Yora K, et al. Mycoplasma or PLT group like microorganisms found in the phloem elements of pants infected with mulberry dwarf, potato witches’-broom, aster yellows or pauwlonia witches’-broom[J]. Annals of the Phytopathological Society of Japan, 1967, 33(4): 259-266.
[2] Dickinson M, Hodgetts J. Phytoplasma: Methods and Protocols[M]. Totowa: Humana Press, 2013.
[3] 于少帅, 徐启聪, 林彩丽, 等. 植原体遗传多样性研究现状与展望[J]. 生物多样性, 2016, 24(2): 205-215.
[4] 车海彦. 海南省植原体病害多样性调查及槟榔黄化病植原体的分子检测技术研究[D]. 杨凌: 西北农林科技大学, 2010.
[5] 于少帅, 李 永, 任争光, 等. 多位点序列分析揭示我国16SrI组植原体不同株系间遗传变异和系统发育关系[J]. 林业科学, 2017, 53(3): 105-118.
[6] Yu S S, Tang Q H, Fu D Q, et al. Molecular identification and characterization of a phytoplasma strain associated with pepper (Capsicum annuum L.) yellow crinkle disease in China[J]. Journal of General Plant Pathology, 2021, https://doi.org/10.1007/s10327-021-01012-x.
[7] Yu S S, Tang Q H, Wu Y, et al. First report of 16SrI-B subgroup related phytoplasma associated with witches’-broom symptoms in Pericampylus glaucus in China[J]. Plant Disease, 2021, 105(3): 695.
[8] Yu S S, Tang Q H, Wu Y, et al. First report of 16SrXXXII group related phytoplasma associated with Trema tomentosa witches’-broom disease in China[J]. Plant Disease, 2021, 105(4): 1191.
[9] 周亚奎, 甘炳春, 张 争, 等. 利用巢式PCR对海南槟榔(Areca catechu L.)黄化病的初步检测[J]. 中国农学通报, 2010, 26(22): 381-384.
[10] 金开璇, 孙福生, 陈慕容, 等. 槟榔黄化病的病原的研究初报[J]. 林业科学, 1995, 31(6): 556-558.
[11] 罗大全, 陈慕容, 叶沙冰, 等. 海南槟榔黄化病的病原鉴定研究[J]. 热带作物学报, 2001, 22(2): 43-46.
[12] Kumar S, Stecher G, Tamura K. MEGA7: Molecular evolu-tionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874.
[13] Felsenstein J. Confidence limits on phylogenies: An appro-ach using the bootstrap[J]. Evolution, 1985, 39(4): 783-791.
[14] Muddumadiah C, Madhupriya, Kumar S, et al. Detection and characterization of 16SrI-B phytoplasmas associated with yellow leaf disease of arecanut palm in India[J]. Phytopathogenic Mollicutes, 2014, 4(2): 77-82.
[15] Ramaswamy M, Nair S, Soumya VP, et al. Phylogenetic ana¬lysis identifies, a ‘Candidatus phytoplasma oryzae’-re-lat¬ed strain associated with yellow leaf disease of areca palm (Are¬ca catechu L.) in India[J]. International Journal of System¬atic and Evolutionary Microbiology, 2013, 63: 1376- 1382.
[16] Silva C K D, Dahanayake D, Silva R D, et al. Molecular and scanning electron microscopic proof of phytoplasma asso-ciated with areca palm yellow leaf disease in Sri Lanka[J]. Plant Disease, 2015, 99(11): 1641.
責任编辑:谢龙莲

