基于新遗传连锁图谱的豇豆抗豆象QTL定位
王彦,范保杰,曹志敏,张志肖,苏秋竹,王珅,王学清,彭秀国,梅丽,武玉华,刘少兴,田胜民,徐俊杰,蒋春志,王伟娟,刘长友,田静
基于新遗传连锁图谱的豇豆抗豆象QTL定位
王彦1,范保杰1,曹志敏1,张志肖1,苏秋竹1,王珅1,王学清2,彭秀国3,梅丽4,武玉华1,刘少兴1,田胜民1,徐俊杰1,蒋春志1,王伟娟5,刘长友1,田静1
1河北省农林科学院粮油作物研究所/河北省作物遗传育种实验室,石家庄 005035;2河北省农林科学院,石家庄 005031;3石家庄市植物园,石家庄 050000;4北京市农业技术推广站,北京 100029;5张家口市崇礼区植保站,河北张家口 076350
【目的】豆象是危害豇豆最主要的仓储害虫。发掘豇豆抗豆象基因为抗性品种的选育,以及减少豆象对豇豆生产的危害奠定基础。【方法】以中豇1号(感)和Pant-lobia-1(抗)为亲本构建的包含282个株系的RIL群体为研究材料,利用人工接种法分别对282个株系接种绿豆象和四纹豆象,进行抗豆象表型鉴定,并利用两亲本对3 992个来源于绿豆、小豆和豇豆的SSR标记进行多态性筛选,然后利用筛选到的多态性标记对282个株系进行基因型分析,最后结合RIL群体各株系抗豆象表型鉴定数据和基因型分型数据,采用完备区间作图法(ICIM-ADD)进行抗豆象QTL定位分析,在此基础上构建遗传连锁图谱,并定位豇豆抗豆象基因。【结果】中豇1号和F1籽粒的被害率均为100%,Pant-lobia-1籽粒的被害率分别为22.5%和42.5%。推测Pant-lobia-1对绿豆象和四纹豆象的抗性均为隐性遗传;筛选到182个多态性标记,利用这些多态性标记构建了一个包含11个连锁群的豇豆遗传连锁图谱,图谱总长1 065.23 cM,相邻标记间平均遗传距离为5.85 cM;经2种豆象处理,分别在连锁群1和连锁群5上检测到2个稳定的QTL位点,暂定名为和,其中位于标记XD11-44和HAAS_VR_2274之间,标记间的遗传距离为7.6 cM,在2种豆象处理中分别可以解释表型变异的7.16%和6.92%;位于标记XD1-14和CP185之间,标记间的遗传距离为2.90 cM,在2种豆象处理中分别可以解释表型变异的6.96%和6.37%。【结论】构建了一个包含11个连锁群、182个多态性标记的豇豆遗传连锁图谱,检测到2个与抗豆象相关的QTL位点。
豇豆;SSR;抗豆象;遗传图谱;QTL定位
0 引言
【研究意义】豆象是豇豆((Linn.) Walp.)等食用豆类作物的主要仓储害虫,以幼虫蛀入籽粒内进行危害[1],导致豇豆品质和质量下降,发生严重时可造成整仓库的豇豆籽粒全部受损,严重影响豇豆产业的发展[2-3]。在中国危害豇豆的豆象主要是绿豆象()和四纹豆象()[4-5]。其中,四纹豆象是中国进境检疫三类危险性害虫之一。目前,国内外主要通过化学防治和生物防治方法防治豆象危害[6-8]。化学防治方法容易导致农药残留和环境污染,而生物防治方法成本高、见效慢[9-10]。因此,利用作物本身的抗性培育抗豆象品种是防治豆象最为经济有效的方法。【前人研究进展】目前,国内外筛选到的豇豆抗豆象资源较少,1985年国际热带农业研究所(International Institute of Tropical Agriculture,IITA)筛选了8 000多份豇豆资源,仅得到3份抗性资源,分别为TVU11952、TVU11953和TVU20273抗源[11-12]。近年来,随着分子生物学技术的快速发展,国内外关于豇豆QTL定位的研究不断增多。2015年Huynh等[13]利用1 536个SNP标记定位了2个与蚜虫抗性相关的QTL位点,分别位于第1连锁群和第7连锁群。2018年Lo等[14]利用17 739个多态性标记构建了一个高密度遗传连锁图谱,并定位了9个与豇豆驯化相关的QTL位点。但是关于豇豆抗豆象性状遗传研究较少。1983年Redden[15]研究认为TVU2027的抗豆象特性是由1个主要隐性基因及其修饰基因起作用。2005年Singh[16]通过研究TVU11952、TVU11953和TVU2027的抗性遗传,认为它们的抗豆象基因相同,均由2个隐性基因控制,并命名为和。2018年Miesho等[17]利用41 948个SNP标记对217份豇豆核心种质进行全基因组关联分析(genome-wide association studies,GWAS),筛选到6个与抗豆象相关的候选基因,分别位于豇豆第1、2、6和8染色体。【本研究切入点】目前,国外关于豇豆抗豆象分子遗传基础研究结果不一,而国内关于豇豆抗豆象遗传研究鲜见报道。【拟解决的关键问题】本研究利用来自尼尔利亚的抗豆象豇豆品种Pant-lobia-1作为抗豆象基因来源,与中国豇豆品种中豇1号杂交构建重组自交系(recombinant inbred lines,RIL)群体,进行豇豆遗传连锁图谱构建和抗豆象QTL定位,为豇豆抗豆象分子标记辅助育种和抗豆象基因克隆提供依据。
1 材料与方法
1.1 供试材料
供试品种:抗豆象材料Pant-lobia-1由国际热带农业研究所(IITA)Bir Bahadur Singh博士惠赠,其籽粒白色黑脐环;感豆象材料中豇1号由中国农业科院作物科学研究所育成,其籽粒红色。以中豇1号作母本,Pant-lobia-1作父本进行杂交,获得F1籽粒,种植F1单株,F1植株上所收获种子的种皮全部为黑色,可由此判断是否为真杂交种(图1)。取1株F1单株上结的种子进一步加代繁殖,经过连续自交,采用单粒传法构建F8RIL群体(282个株系),用于遗传连锁图谱构建和抗豆象QTL定位。
供试豆象:绿豆象和四纹豆象,由河北省农林科学院粮油作物研究所食用豆研究室繁殖保存。

图1 亲本及F1植株籽粒
1.2 室内抗豆象鉴定
选取F1杂交籽粒及RIL群体每个株系正常的籽粒置于圆形小盒内(直径6 cm、高2 cm),将小盒并排放入大塑料箱内(66 cm×40 cm×15 cm),分别接种绿豆象(处理Ⅰ)和四纹豆象(处理Ⅱ)成虫(平均每份被鉴定材料3对),每个处理设3次重复,每个重复20粒种子,以感豆象亲本中豇1号和抗豆象亲本Pant-lobia-1作为对照,置于养虫室培养。养虫室温度为(30±1)℃,湿度为55%—65%。每个籽粒上着卵3—5粒时,移除成虫,防止豆象二次危害。当全部感豆象亲本籽粒被蛀食后开始调查,记录鉴定材料的被害粒数,计算平均被害率,并使用SPSS 19.0统计平均被害率频次分布及显著性分析。
1.3 DNA提取及PCR扩增
采集两亲本及RIL群体每个株系幼嫩的三出复叶置于1.5 mL离心管内,经液氮处理后-80℃保存。利用改良的CTAB法[18]提取基因组DNA,用0.8%琼脂糖凝胶电泳检测DNA质量,并将DNA稀释到10 ng·µL-1,用于PCR扩增。参照Liu等[19]的PCR反应体系及程序进行PCR扩增,其中,根据豇豆基因组设计的SSR标记退火温度为50℃,其他标记退火温度为55℃。
1.4 SSR标记开发及多态性标记筛选
利用已发布的豇豆基因组序列信息,使用SSR Locator软件查找SSR位点并设计引物,根据染色体长度,从每条染色体上每隔一定距离挑选一个标记,并命名为“JDX-N”,其中X为染色体号,N为挑选的第N个位点;另外从较长的congtig上设计开发SSR标记,命名为congtig-n,其中n为挑选的第n个位点,共设计合成基因组SSR标记620个;根据Cowpea Genomics Knowledge Base(CGKB)[20]上发布的豇豆SSR标记序列信息合成豇豆SSR引物300对;河北省农林科学院粮油作物研究所食用豆研究室前期开发设计的小豆基因组SSR标记1 100个、绿豆转录组SSR标记1 972个。共计筛选3 992个SSR标记,采用聚丙烯酰胺凝胶电泳技术进行亲本间多态性标记筛选,电泳方法同Liu等[19]。
1.5 豇豆遗传连锁图谱构建
利用亲本间多态性SSR标记对RIL群体每个株系进行基因分型分析。母本带型记作“0”,父本带型记作“2”,杂合带型记作“1”,空缺或其他带型记作“-1”,并利用QTL IciMapping 4.0作图软件构建遗传连锁图谱[21],以LOD值作为标记分组依据,用Grouping命令对所有标记进行分组,其他参数采用软件默认值。利用卡方检测分析,确定标记偏分离情况。
1.6 豇豆抗豆象QTL分析
利用QTL IciMaping 4.0软件的完备区间作图法(ICIM-ADD)分别对处理Ⅰ和处理Ⅱ的抗豆象表型鉴定结果进行QTL定位及效应分析[22]。LOD阈值采用10 000次“Permutation”获得,其他参数采用软件默认值。以豇豆拉丁名缩写+豆象英文名称前2个字母+所在连锁群编号+QTL位点序号对QTL位点进行命名,如代表该QTL位点是第1连锁群上的第1个QTL。
2 结果
2.1 RIL群体抗豆象鉴定
由抗豆象鉴定结果可知,两亲本的籽粒被害率差异较大,其中,感豆象亲本中豇1号在2个处理中的籽粒被害率均为100.0%,表现为极感豆象;抗豆象亲本Pant-lobia-1在处理Ⅰ中籽粒被害率为22.5%,处理Ⅱ中籽粒被害率为42.5%,结果表明,抗豆象亲本Pant-lobia-1籽粒被害率显著低于感豆象亲本,表现为抗豆象;F1籽粒被害率均为100.0%(表1)。处理Ⅰ中RIL群体各株系籽粒被害率范围为12.5%—100.0%,平均被害率为76.56%,处理Ⅱ中RIL群体各株系籽粒被害率范围为25.0%—100.0%,平均被害率为84.58%。根据抗豆象表型鉴定结果,推测Pant-lobia-1抗豆象性状为隐性遗传。
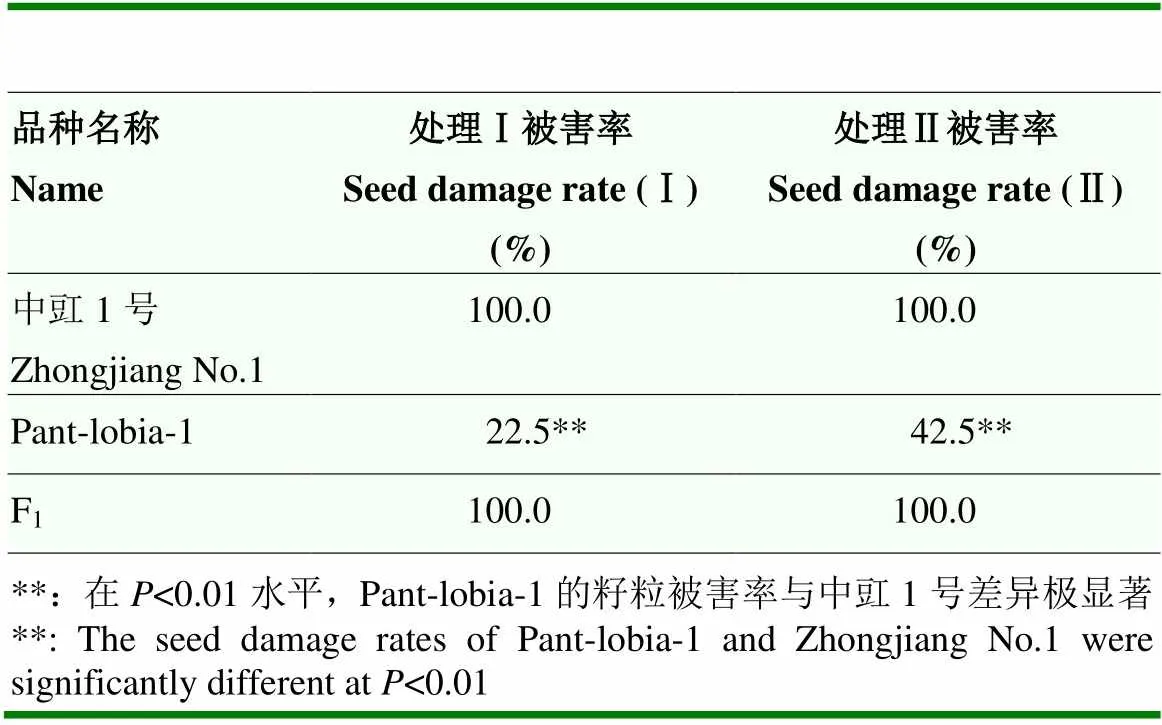
表1 亲本及F1籽粒被害率
分别对2个处理中282个株系的平均被害率进行频次统计分析。结果表明,2个处理中282个株系的平均被害率均呈连续性变异(图2),符合数量性状遗传的频率分布特点,说明豇豆的豆象抗性为数量性状。此外,豇豆RIL群体对绿豆象和四纹豆象的抗性均具有单向超亲分离现象。
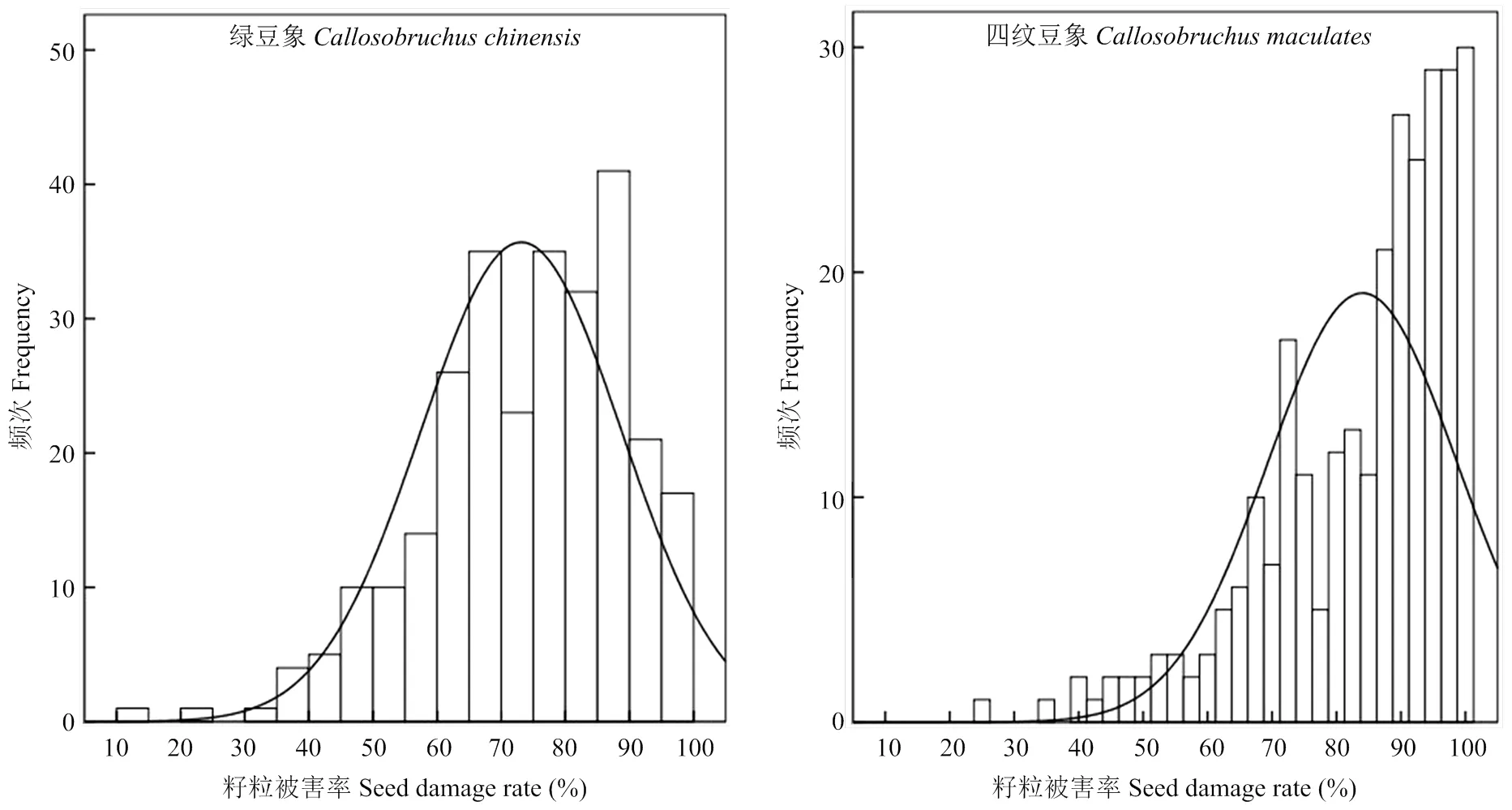
♀:中豇1号;♂:Pant-lobia-1。下同 ♀: Zhongjiang no.1; ♂: Pant-lobia-1. The same as below
2.2 多态性SSR标记分析
利用3 992个不同来源SSR标记筛选亲本间多态性标记(表2)。其中3 185个标记在豇豆中能有效扩增,有效扩增率为80.11%。豇豆SSR标记有效扩增率最高,为93.70%;小豆SSR标记有效扩增率最低,仅为65.18%。
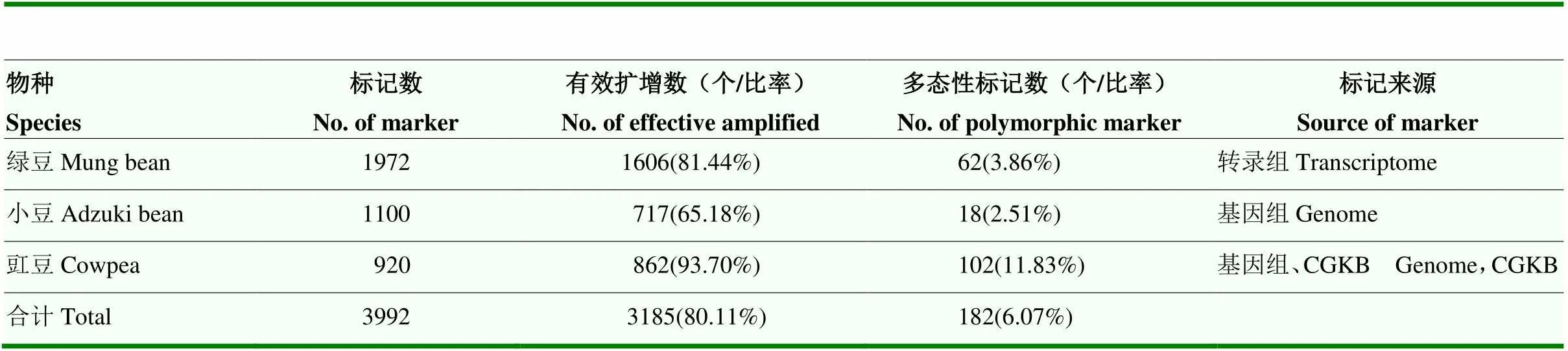
表2 多态性SSR标记分析
从3 185个有效扩增标记中筛选到182个在中豇1号与Pant-lobia-1间具有多态性的标记,多态性比率为6.07%。其中,豇豆SSR标记多态性比率最高,为11.83%;绿豆SSR标记多态性比率居中,为3.86%;小豆SSR标记的多态性比率最低,仅为2.51%。
2.3 遗传连锁图谱的构建
利用182个多态性标记对豇豆RIL群体282个株系进行基因型分析(图3),并利用QTL IciMapping 4.0进行遗传连锁图谱构建。以LOD值作为标记分组依据,在LOD=10.3时,182个多态性标记可分成11个连锁群,与豇豆的单倍体染色体数目(2n=22)相一致[23]。最终获得的遗传连锁图谱总长1 065.23 cM,相邻标记间平均遗传距离为5.85 cM。各连锁群的长度范围为(LG8)29.71—185.91(LG1)cM,平均长度为96.84 cM。各连锁群上的标记分布不均匀,标记数目范围为4(LG8)—28(LG2)个,标记间平均遗传距离范围为4.15—7.43 cM(表3和图4)。
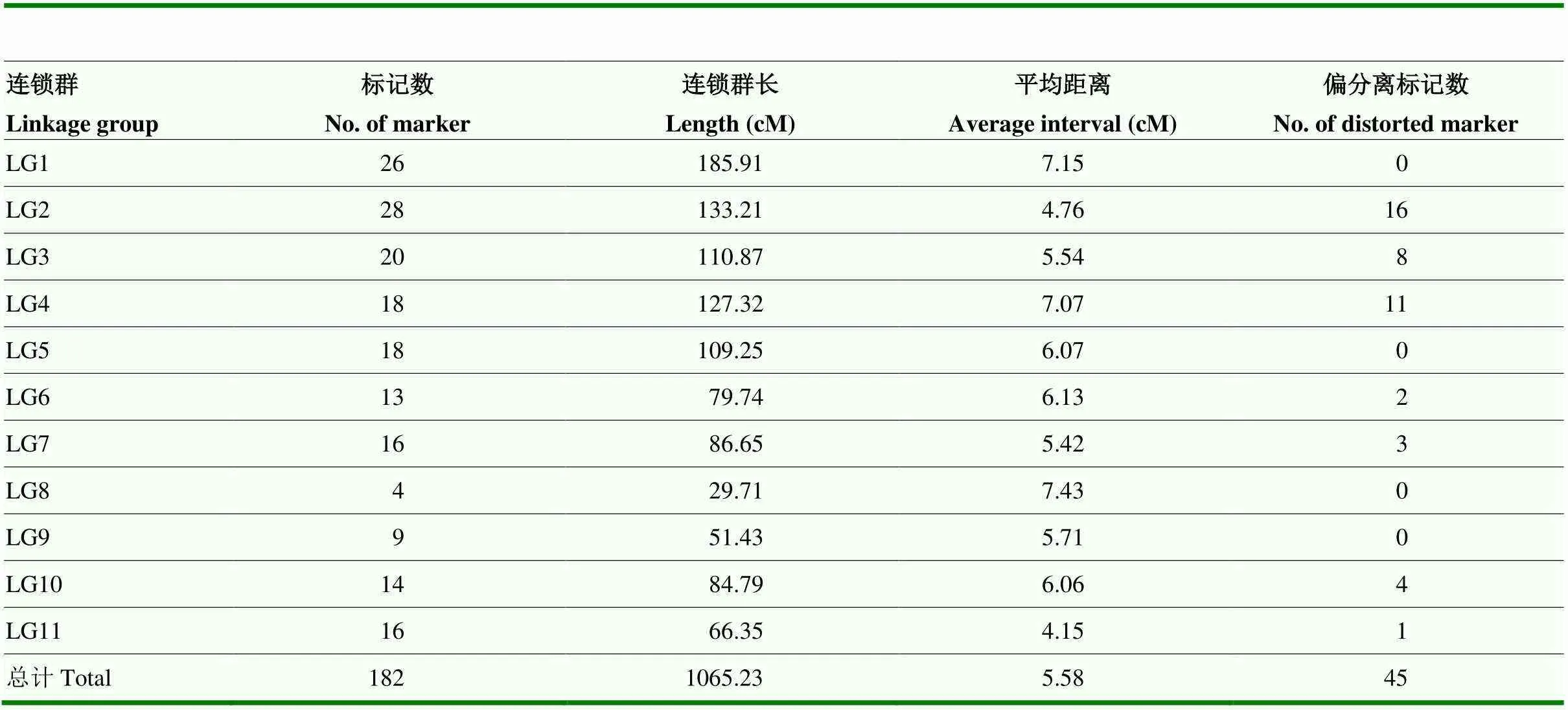
表3 图谱中连锁群标记及其分布
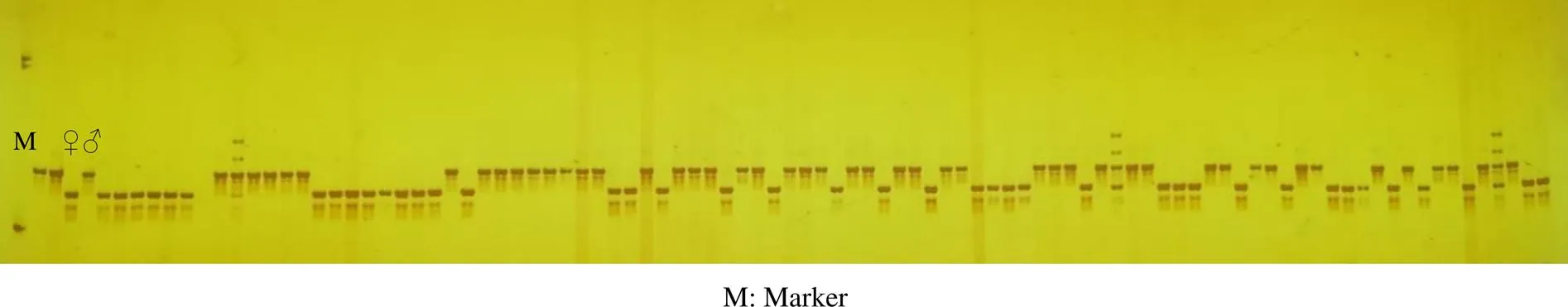
图3 SSR标记JD6-28在豇豆RIL群体部分株系中的扩增
182个标记中有45个标记表现偏分离(<0.05),占24.46%,主要分布于LG2和LG4上。其中,22个标记偏向母本中豇1号,占偏分离标记的48.89%;23个标记偏向父本Pant-lobia-1,占偏分离标记的51.11%。45个偏分离标记中有15个标记呈严重偏分离(<0.001),占偏分离标记的33.33%,主要位于LG2中部、LG3下部和LG4中下部(图4)。
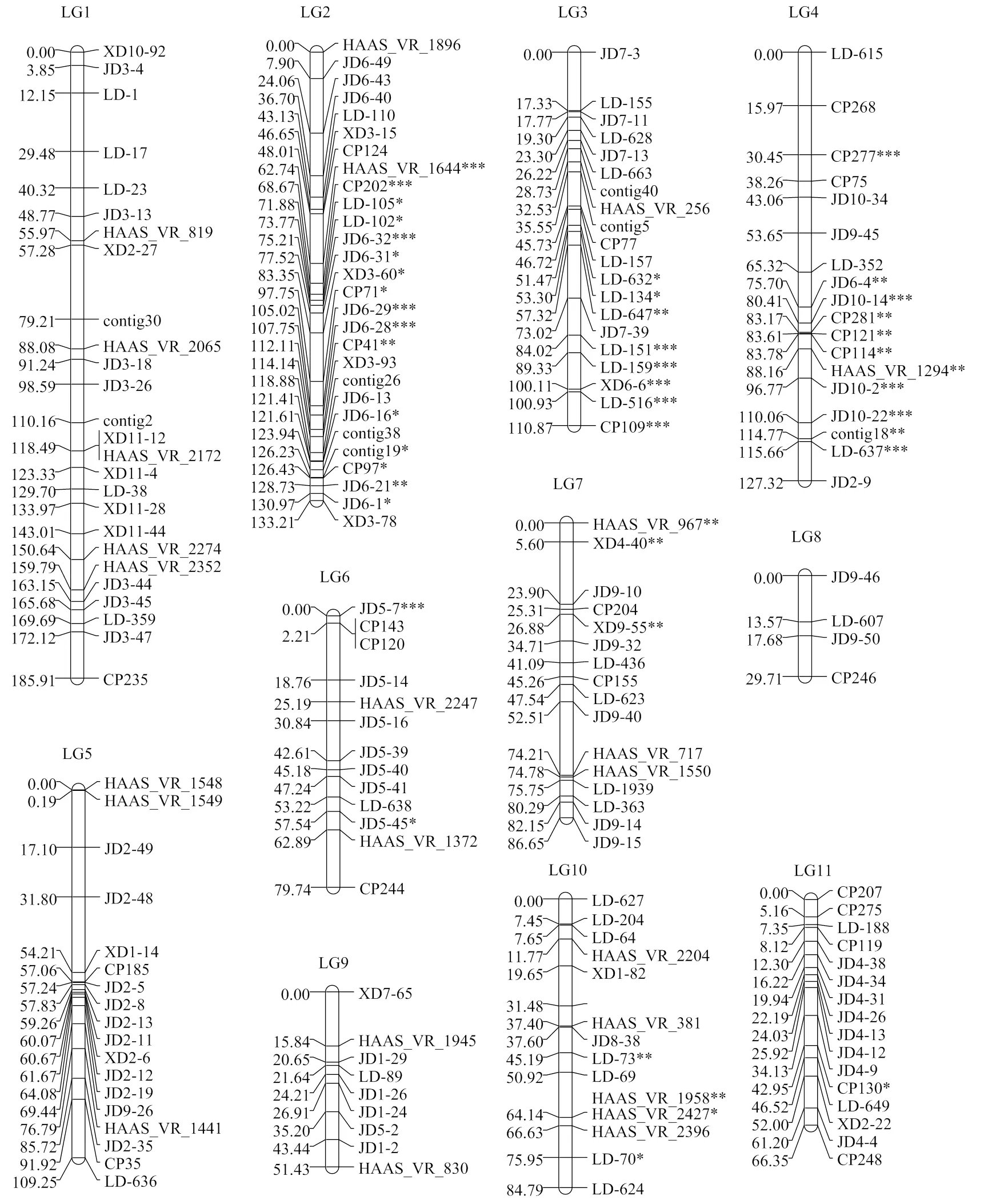
*、**和***分别表示该标记在0.05、0.01和0.001显著水平上表现偏分离
2.4 豇豆抗豆象QTL分析
整合RIL群体282个株系的抗豆象表型鉴定结果和基因型分型结果,利用QTL IciMapping 4.0完备区间作图法(ICIM-ADD)进行QTL定位分析(表4和图5)。处理Ⅰ共检测到2个与绿豆象抗性相关的QTL位点,一个位于第1连锁群标记XD11-44和HAAS_VR_2274区间内,另一个位于第5连锁群标记XD1-14和CP185区间内,标记之间的遗传距离分别为7.6和2.90 cM,LOD分别为3.86和4.58,表型贡献率分别为7.16%和6.96%,加性效应分别为-4.20和-5.30;处理Ⅱ同样检测到2个与四纹豆象抗性相关的QTL位点,这两个位点在连锁群上的位置与处理Ⅰ完全一致,LOD分别为3.91和4.44,表型贡献率分别为6.92%和6.37%,加性效应分别为-4.29和-3.70。综上所述,处理Ⅰ检测到的2个QTL位点和处理Ⅱ的相同,且稳定存在于2个处理中。另外,2个处理中检测到的QTL位点的加性效应均为负值,表明来自父本Pant-lobia-1的等位基因可以降低籽粒被害率,将2个QTL位点暂命名为和。利用2个位点两侧的标记序列信息,经过Blast序列比对,可知位于豇豆第3染色体,位于第2染色体。
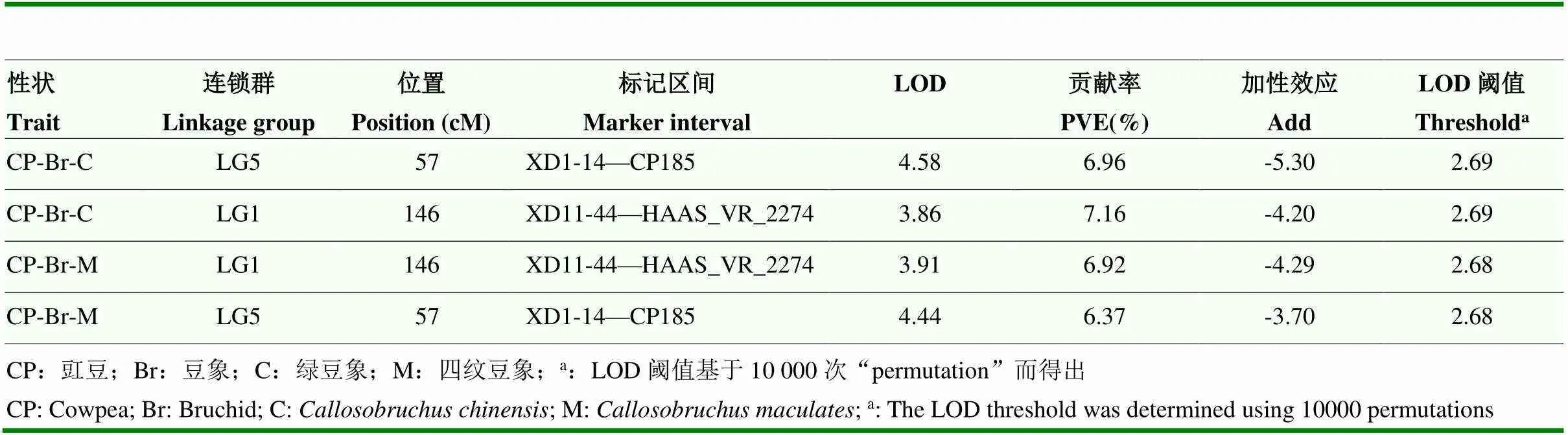
表4 豇豆RIL群体抗豆象QTL定位
3 讨论
3.1 SSR标记的选择
SSR分子标记以其多态性高、重复性好、操作简便等特点被广泛应用于不同作物遗传连锁图谱构建及QTL定位研究。另外,据报道SSR标记在相近的物种间具有一定的通用性,并且亲缘关系越近,通用性越高,利用近缘物种间SSR标记的通用性,可以减少引物开发成本,且有助于比较基因作图等方面的研究。王丽侠等[24]研究表明小豆SSR标记在绿豆基因组中通用性比率为75%,多态性比率为35%。钟敏等[25]研究表明绿豆基因组SSR标记在豇豆、小豆和饭豆中的通用性比率分别为50.0%、73.3%和81.6%;多态性比率分别为4.1%、1.7%和1.5%。本研究利用绿豆(1 972个)和小豆(1 100个)SSR标记筛选豇豆多态性标记,绿豆SSR标记在豇豆中的通用性为81.44%,显著高于钟敏等[25]的研究结果,可能原因是这些绿豆SSR标记多设计于绿豆基因组编码区[13];多态性比率为3.86%,低于钟敏等[25]的研究结果,但差异不大,原因可能是用于筛选的亲本材料及材料数目的差异。本研究中仅利用了2份豇豆亲本材料,随着材料数目的增加,多态性比率势必会随之升高。
3.2 豇豆遗传连锁图谱构建
构建遗传连锁图谱是QTL定位的前提,Fatokun等[26]以F2分离群体为作图群体,构建了第一个豇豆遗传连锁图谱,总长度684 cM。随着豇豆全基因组测序的完成,豇豆分子生物学研究取得突飞猛进的发展。Muñoz-Amatriaín等[27]基于豇豆高通量SNP平台(Cowpea iSelect Consortium Array)构建了一个包含37 372个SNP位点的遗传连锁图谱。Ohlson等[28]利用99个SNP标记构建了一个包含11个连锁群,总长763.4 cM的豇豆遗传连锁图谱,用于豇豆栽培种KN1抗褐斑病QTL定位研究。Ndeve等[29]利用17 208个SNP标记构建了一个包含11个连锁群、总长985.89 cM的豇豆遗传连锁图谱,用于豇豆抗线虫QTL定位研究。本研究利用中豇1号×Pant-lobia-1杂交形成的RIL作图群体,构建了一个包含182个多态性标记、11个连锁群、总长1 065.23 cM的遗传连锁图谱用于豇豆抗豆象QTL定位研究,该遗传连锁图谱与Muñoz-Amatriaín等[27]、Ohlson等[28]、Ndeve等[29]构建的遗传连锁图谱的连锁群数目一致,且图谱密度适中。但该图标记分布不均匀,有的连锁群标记数目较少,尤其是LG8连锁群上仅有4个标记,且通过该图标记分析发现LG7和LG8连锁群均对应于豇豆第9染色体。在后续研究中可以考虑增加标记类型和标记数目,如SNP和Indel标记等,进一步加密该遗传连锁图谱,使该图谱可更准确的应用于其他农艺性状的定位研究。
本研究的182个多态性标记中有45个标记为偏分离标记,偏分离比例为24.46%。偏分离标记主要分布于连锁群2、连锁群3和连锁群4,且成簇分布,这与赵向前等[30]报道的偏分离标记有成簇分布的特点相符。导致标记偏分离现象的原因可能是RIL群体连续自交选择过程中群体发生偏离。
3.3 豇豆抗豆象基因定位
近年来,国内外关于食用豆抗豆象QTL定位的研究主要集中在绿豆上。相关研究表明绿豆抗豆象是由主效的显性基因控制[31],但还可能存在其他微效基因的修饰作用[32]。吴传书[33]、孙蕾等[34]、刘长友等[35]分别以绿豆抗豆象资源ACC41、V2709、V1128为试验材料,定位了绿豆抗豆象QTL位点、和。关于豇豆抗豆象基因定位研究较少。Redden[15]研究表明豇豆抗豆象性状由1个主要隐性基因及其修饰基因起作用,而Singh[16]则认为该性状由2个隐性基因控制。Miesho等[17]利用GWAS对豆象的平均产卵数、平均出虫数、平均虫洞数等5个性状进行了分析,检测到11个与抗性相关的SNP,并通过在Phytozome网站进行比对分析,筛选到6个与豇豆抗豆象相关的候选基因,分布于豇豆第1、2、6和8染色体。本研究中F1籽粒表现完全感豆象,推测豇豆抗豆象性状为隐性遗传。在第1连锁群和第5连锁群分别检测到2个绿豆象和四纹豆象处理下稳定存在的抗豆象QTL位点和,由此推断豇豆抗豆象性状可能由2个隐性基因控制,该结果与Singh[16]的研究结果相吻合。通过Blast序列比对,位于第3染色体,位于第2染色体。与Miesho等[17]报道的豇豆抗豆象候选基因均位于第2染色体,但在染色体上位置存在差异。位于物理位置2 612 671—3 850 060 bp,而位于物理位置28 196 653—28 200 516 bp,这可能是由检测到的不是同一个位点或者用于抗豆象研究的材料不同造成的。
4 结论
利用中豇1号与Pant-lobia-1杂交构建的RIL群体和182个亲本间多态性SSR标记,构建了一个包含11个连锁群、总长1 065.23 cM的豇豆遗传连锁图谱,定位了2个绿豆象和四纹豆象处理下稳定存在的豇豆抗豆象QTL位点和,分别位于豇豆第3染色体和第2染色体,两侧的SSR标记分别为XD11-44—HAAS_VR_2274和XD1-14—CP185。
[1] BITTNER J A, SUSAN B, PITTENDRIGH B R, POPOVICS J S. Monitoring of the cowpea bruchid,(Coleoptera: Bruchidae), feeding activity in cowpea seeds: Advances in sensing technologies reveals new insights. Journal of Economic Entomology, 2018, 111(3): 1469-1475.
[2] SANCHEZ L J, FARBER C, LEI J, ZHU-SALZMAN K, KUROUSKI D. Non-invasive and non-destructive detection of cowpea bruchid within cowpea seeds with a hand-held raman spectrometer. Analytical Chemistry, 2019, 91(3): 1733-1737.
[3] 金文林, 谭瑞娟, 王进忠, 张志勇, 刘长安, 濮绍京, 赵波. 田间小豆绿豆象卵空间分布型初探. 植物保护, 2004, 30(6): 34-36.
JIN W L, TAN R J, WANG J Z, ZHANG Z Y, LIU C A, PU S J, ZHAO B. Preliminary analysis on spatial distribution patterns ofeggs in adzuki bean fields. Plant Protection, 2004, 30(6): 34-36. (in Chinese)
[4] 刘长友, 田静, 范保杰, 曹志敏, 苏秋竹, 张志肖, 王素华. 豇豆属3种主要食用豆类的抗豆象育种研究进展. 中国农业科学, 2010, 43(12): 2410-2417.
LIU C Y, TIAN J, FAN B J, CAO Z M, SU Q Z, ZHANG Z X, WANG S H. Advances in breeding research on bruchid-resistant cultivars of three mainfood legumes. Scientia Agricultura Sinica, 2010, 43(12): 2410-2417. (in Chinese)
[5] GUJAR G T, YADAV T D. Feeding of(Fab.) and(Linn.) in green gram. Indian journal of entomology, 1978, 40(2): 108-112.
[6] BABARINDE S A, PITAN O, AJALA M O, OLATUNDE G O. Insectifugal and insecticidal potentials of two tropical botanical essential oils against cowpea seed bruchid. Environmental Science and Pollution Research, 2017, 24(24): 19785-19794.
[7] BABARINDE S A, PITAN O, OLATUNDE G O, AJALA M O. Chemical composition of the essential oil of nigeria grown hoslundia opposita Vahl (Lamiaceae) dried leaves and its bioactivity against cowpea seed bruchid. Chemistry & Biodiversity, 2017, 14(6): e1600418.
[8] SANG W, SPEAKMON M, ZHOU L, WANG Y, LEI C, PILLAI S D, ZHU-SALZMAN K. Detrimental effects of electron beam irradiation on the cowpea bruchid. Pest Management Science, 2016, 72(4): 787-795.
[9] AMEVOIN K, SANON A, APOSSABA M, GLITHO I A. Biological control of bruchids infesting cowpea by the introduction of dinarmus basalis (Rondani) (Hymenoptera: Pteromalidae) adults into farmers' stores in West Africa. Journal of Stored Products Research, 2007, 43(3): 240-247.
[10] KOLAWOLE A O, OKONJI R E, AJELE J O. Tithonia diversifolia, cyperus rotundus and hyptis suaveloensis ethanol extracts combinatorially and competitively inhibit affinity purified cowpea storage bruchid () glutathione S-transferase. Arthropod-Plant Interactions, 2011, 5(3): 175-184.
[11] SINGH B B, SINGH S R, ADJADI O. Bruchid resistance in cowpea. Crop Science, 1985, 25(5): 736-739.
[12] AMUSA O D, OGUNKANMI L A, ADETUNBI J A, AKINYOSOYE S T, OGUNDIPE O T. Assessment of bruchid () tolerance of some elite cowpea () varieties. Journal of Agriculture & Sustainability, 2014, 6(2): 164-178.
[13] HUYNH B L, EHLERS J D, NDEVE A, WANAMAKER S, LUCAS M R, CLOSE T J, ROBERTS P A. Genetic mapping and legume synteny of aphid resistance in African cowpea (L. Walp.) grown in California. Molecular Breeding, 2015, 35(1): 36.
[14] LO S, Muñoz-Amatriaín M, BOUKAR O, HERNITER I, CLOSE T J. Identification of QTL controlling domestication-related traits in cowpea (L. Walp). Scientific Reports, 2018, 8(1): 6261.
[15] REDDEN R. The inheritance of seed resistance toF. in cowpea (L. Walp.): II. Analyses of percentage emergence and emergence periods of bruchids in F4seed generation of two reciprocal crosses. Australian Journal of Agricultural Research, 1983, 34(6): 697-705.
[16] SINGH B B. Cowpea chrimosome engineering and crop improvement: Grain legumes. Genetic Resources, 2005: 117-161.
[17] MIESHO B, HAILAY M, MSISKA U, BRUNO A, KYAMANYWA S. Identification of candidate genes associated with resistance to bruchid () in cowpea. Plant Breeding, 2019, 138(5): 1-9.
[18] AFANADOR L K, HALEY S D, KELLY J D. Adoption of a mini-prep DNA extraction method for RAPD marker analysis in common bean. Bean Improvement Cooperative, 1993, 36: 10-11.
[19] LIU C, FAN B, CAO Z, SU Q, WANG Y, ZHANG Z, WU J, TIAN J. A deep sequencing analysis of transcriptomes and the development of EST-SSR markers in mungbean (). Journal of Genetics, 2016, 95(3): 1-9.
[20] CHEN X, LAUDEMAN T W, RUSHTON P J, SPRAGGINS T A, TIMKO M P. CGKB: an annotation knowledge base for cowpea (L.) methylation filtered genomic genespace sequences. BMC Bioinformatics, 2007, 8(1): 129.
[21] MENG L, LI H, ZHANG L, WANG J. QTL IciMapping: Integrated software for genetic linkage map construction and quantitative trait locus mapping in biparental populations. The Crop Journal, 2015, 3(3): 269-283.
[22] LI H, RIBAUT J M, LI Z, WANG J. Inclusive composite interval mapping (ICIM) for digenic epistasis of quantitative traits in biparental populations. Theoretical & Applied Genetics, 2008, 116(2): 243-260.
[23] IWATA A, GREENLAND C M, JACKSON S A. Cytogenetics of legumes in the phaseoloid clade. Plant Genome, 2013, 6(3): 1-8.
[24] 王丽侠, 程须珍, 王素华, 刘长友, 梁辉. 小豆SSR引物在绿豆基因组中的通用性分析. 作物学报, 2009, 35(5): 816-820.
WANG L X, CHENG X Z, WANG S H, LIU C Y, LIANG H. Transferability of SSR from adzuki bean to mung bean. Acta Agronomica Sinica, 2009, 35(5): 816-820. (in Chinese)
[25] 钟敏, 程须珍, 王丽侠, 王素华, 王小宝. 绿豆基因组SSR引物在豇豆属作物中的通用性. 作物学报, 2012, 38(2): 223-230.
ZHONG M, CHENG X Z, WANG L X, WANG S H, WANG X B. Transferability of mungbean genomic-SSR markers in otherspecies. Acta Agronomica Sinica, 2012, 38(2): 223-230. (in Chinese)
[26] FATOKUN C A, MENANCIO-HAUTEA D I, DANESH D, YOUNG N D. Evidence for orthologous seed weight genes in cowpea and mung bean based on RFLP mapping. Genetics, 1992, 132(3): 841-846.
[27] MUÑOZ-AMATRIAÍN M, MIREBRAHIM H, XU P, WANAMAKER S I, LUO M C, ALHAKAMI H, ALPERT M, ATOKPLE I, BATIENO B J, BOUKAR O. Genome resources for climate-resilient cowpea, an essential crop for food security.The Plant Journal, 2017, 89(5): 1042-1054.
[28] OHLSON E W, THIO G I, MAHAMADOU S, PACO S, TIMKO M P. Quantitative trait loci analysis of brown blotch resistance in cowpea variety KN1. Molecular Breeding, 2018, 38(9): 110.
[29] NDEVE A D, SANTOS J, MATTHEWS W C, HUYNH B L, GUO Y N, LO S, MU OZ-AMATRIA N M, ROBERTS P A. A novel root-knot nematode resistance QTL on chromosome Vu01 in cowpea. Genes Genomes Genetics, 2018, 9: 1199-1209.
[30] 赵向前, 吴为人. 水稻ILP标记遗传图谱的构建. 遗传, 2008, 30(2): 225-225.
ZHAO X Q, WU W R. Construction of a genetic map based on ILP markers in rice. Hereditas, 2008, 30(2): 225-230. (in Chinese)
[31] KITAMURA K, ISHIMOTO M, SAWA M. Inheritance of resistance to infestation with adzuki bean weevil inand vuccessful incorporation to. Japanese Journal of Breeding, 2008, 38(4): 459-464.
[32] CHEN H M, KU H M, SCHAFLEITNER R, BAINS T S, KUO C G, LIU C A, NAIR R M. The major quantitative trait locus for mungbean yellow mosaic Indian virus resistance is tightly linked in repulsion phase to the major bruchid resistance locus in a cross between mungbean [(L.) Wilczek] and its wild relativess. Euphytica, 2013, 192(2): 205-216.
[33] 吴传书. 绿豆SSR标记的开发及高密度分子遗传连锁图谱的构建[D]. 兰州: 甘肃农业大学, 2014.
WU C S. Development of SSR markers and construction of a genetic linkage map in mungbean (L.) [D]. Lanzhou: Gansu Agricultural University, 2014. (in Chinese)
[34] 孙蕾, 程须珍, 王素华, 王丽侠, 刘长友, 梅丽, 徐宁. 栽培绿豆V2709抗豆象特性遗传及基因初步定位. 中国农业科学, 2008, 41(5): 1291-1296.
SUN L, CHENG X Z, WANG S H, WANG L X, LIU C Y, MEI L, XU N. Heredity analysis and gene mapping of bruchid resistance of a mungbean cultivar V2709. Scientia Agricultura Sinica, 2008, 41(5): 1291-1296. (in Chinese)
[35] 刘长友, 苏秋竹, 范保杰, 曹志敏, 张志肖, 武晶, 程须珍, 田静. 栽培绿豆V1128抗豆象基因定位. 作物学报, 2018, 44(12): 137-143.
LIU C Y, SU Q Z, FAN B J, CAO Z M, ZHANG Z X, WU J, CHENG X Z, TIAN J. Genetic mapping of bruchid resistance gene in mungbean V1128. Acta Agronomica Sinica, 2018, 44(12): 1875-1881. (in Chinese)
Quantitative trait locus mapping of bruchids resistance based on anovel genetic linkage map in cowpea ()
WANG Yan1, FAN BaoJie1, CAO ZhiMin1, ZHANG ZhiXiao1, SU QiuZhu1, WANG Shen1, WANG XueQing2, PENG XiuGuo3, MEI Li4, WU YuHua1, LIU ShaoXing1, TIAN ShengMin1, XU JunJie1, JIANG ChunZhi1, WANG WeiJuan5, LIU ChangYou1, TIAN Jing1
1Institute of Cereal and Oil Crops, Hebei Academy of Agricultural and Forestry Sciences/Hebei Laboratory of Crop Genetic and Breeding, Shijiazhuang 050035;2Hebei Academy of Agricultural and Forestry Sciences, Shijiazhuang 050031;3Shijiazhuang Botanical Garden, Shijiazhuang 050000;4Beijing Agricultural Technology Extension Centre, Beijing 100029;5Plant Protection Department of Chongli District, Zhangjiakou 076350, Hebei
【Objective】Bruchids are the main storage pests of cowpea. The discovery of bruchids resistance genes is helpful for breeding resistant varieties and reducing the harm of bruchids to cowpea production.【Method】In this study, a RIL population of 282 lines derived from the cross between Zhongjiang No.1 (a bruchid-susceptible cultivar) and Pant-lobia-1 (a bruchid-resistant cultivar) was used for phenotype identification of bruchids resistance by artificial inoculation ofand. The two parents were used to screen polymorphic markers from 3 992 SSR markers of mung bean, adzuki bean and cowpea. Genotypes of 282 lines were analyzed using the polymorphic SSR markers. Based on the phenotype identification and genotype analysis, a genetic linkage map was constructed and the bruchids resistance gene(s)of cowpea was located using Inclusive Composite Interval Mapping (ICIM-ADD). 【Result】The results showed that Zhongjiang No. 1 and F1seeds were 100% susceptible toand, and the damage rates of Pant-lobia-1 were 22.5% and 42.5%, respectively. It was speculated that the resistance of Pant-lobia-1 to bruchids was recessive inheritance. 182 polymorphic markers were obtained from 3 992 SSR markers of mung bean, adzuki bean and cowpea. Using those polymorphic SSR markers, a genetic linkage map with 11 linkage groups was constructed. The map covered a total length of 1 065.23 cM with an average interval of 5.85 cM between adjacent markers. Twobruchid-resistant QTLs from linkage groups 1 and 5were discovered, which were temporarily named asand. QTLwas located between markers XD11-44 and HAAS_VR_2274, which genetic distance was 7.6 cM, explaining7.16% and 6.92% of the phenotypic variation in the two bruchid-resistanttests respectively. QTLwas located between markers XD1-14 and CP185, which genetic distance was 2.9 cM, explaining6.96% and 6.37% of the phenotypic variation in the two bruchid-resistant tests respectively. 【Conclusion】A genetic linkage mapcontaining 11 linkage groups and 182 polymorphic markers was constructed. Two QTLs linked with bruchids resistance were identified on linkage groups 1 and 5.
; SSR; bruchid resistance; genetic mapping; quantitative trait locus
2021-05-06;
2021-07-02
国家重点研发计划(2019YFD1000700/2019YFD1000703)、财政部和农业农村部:国家现代农业产业技术体系(CARS-08)、河北省重点研发计划(19226353D)、河北省现代农业产业技术体系(HBCT2018070203)、河北省青年拔尖人才(2018)、河北省农林科学院创新工程项目(2019-4-02-08)
王彦,Tel:0311-87670607;E-mail:411678486@qq.com。通信作者田静,Tel:0311-87670655;E-mail:nkytianjing@163.com。通信作者刘长友,Tel:0311-87670607;E-mail:35931915@qq.com
(责任编辑 李莉)

