虫生真菌非核糖体多肽活性产物生物合成潜力预测
张礼文,István MOLNÁR,徐玉泉
(1 中国农业科学院生物技术研究所,北京 100081;2 美国亚利桑那大学西南天然产物研究中心,亚利桑那州 图森85706,美国)

通过测序和注释,发现真菌基因组中存在大量参与非核糖体多肽天然产物生物合成的基因簇[1],具有合成结构多样化合物的巨大潜力[2]。大多数真菌非核糖体多肽合成酶(nonribosomal polypeptide synthetase,NRPS)包括多个由腺苷酰化(adenylation,A)、巯基化(thiolation,T)和缩合(condensation,C)等结构域组成的模块。在生物合成的过程中,腺苷酰化结构域先将氨基酸或羟基酸前体单元加载到巯基化结构域上,然后缩合结构域缩合前体单元和中间产物合成非核糖体多肽[3]。除了这3个核心结构域,非核糖体多肽合成酶还可能包含修饰前体单元或中间产物甲基化、差向异构化的结构域,以及终止延伸和释放产物、末端缩合等功能的结构域,从而使合成的产物结构更加复杂多样。
肉座菌目虫生真菌能够产生大量具有抗菌、杀虫、抗癌、调节免疫等活性的天然产物,如环孢霉素和白僵菌素等,具有重要药用和农用价值[3-8]。通过基因缺失和互补、异源表达、化合物结构解析等方法,阐明了部分肉座菌目虫生真菌天然产物生物合成基因簇的功能,如合成破坏素(destruxins)、白僵菌素(beauvericin)、白僵菌环四肽(bassianolide)和serinocyclins 等[9-13],然而,很多基因簇在常规实验室条件下不表达,因此绝大多数基因簇的产物未知[14-15]。在过去数年中,使用特定方法激活沉默基因簇的表达在发现新型真菌天然产物方面虽然取得了重大进展[1,16-17],然而,面对数以千万计的生物合成基因簇,通过实验的手段鉴定其功能十分费时费力。如何高效选取最具合成新颖天然产物潜力的基因簇成为亟待解决的问题。因此,本研究基于隐马尔可夫模型从虫生真菌基因组中预测可能合成非核糖体多肽类天然产物的非核糖体多肽合成酶,通过构建序列相似性网络将其分类,并与已知产物的标签结构域进行比较,从而推测出可能合成新颖产物的生物合成基因簇。准确有效预测基因乃至基因簇的功能,不仅可以指导大规模实验鉴定和筛选,而且也为后续利用组合生物合成改造天然途径提供了候选的基因元件。
1 材料与方法
1.1 数据集和标签结构域的收集
本研究使用来自SwissProt(2019年发行版)审核和人工注释的全部真菌蛋白作为标签蛋白。40种肉座菌目虫生真菌的基因组序列和预测的蛋白序列来自于NCBⅠ数据库。使用基于隐马尔可夫模型(hidden Markov model,HMM)的 HMMSearch 方法预测腺苷酰化(PFAM00501.21) 和缩合(PFAM00668.13) 等结构域 (表 1),并使用Python 提取相应的序列。来自于SwissProt 已知功能的结构域被定义为“标签”结构域。

表1 非核糖体多肽合成酶结构域注释Tab.1 Annotion for NRPS domains in hypocrealean entomopathotenic fungi(HEF)
1.2 序列相似网络的建立
为了建立序列相似性网络,使用BLASTp程序计算数据集中每一对腺苷酰化结构域的氨基酸序列一致性(identity)、覆盖率(coverage)和e-value。经统计分析得出,一致性数据在20%~60%区间符合正态分布的规律,高于60%则逐渐偏离正态分布,离群值的下边界为62.06%,这表明当两个腺苷酰化结构域的氨基酸序列一致性高于62.06%时为“真”相似的概率较高。因此在构建网络和聚类时,将一致性0 参数的阈值设定为62.06%。类似地,覆盖率值通常在91%~99%之间,低于75%为异常值,因此覆盖率的阈值设定为75%。图形可视化使用Cytoscape ver 3.7.2。利用马尔可夫聚类算法(MCL),根据序列相似性矩阵来识别相关节点的分支。再根据NRPS的结构域组成,并结合MultiGeneBlast 评估的特定分支中基因簇的相似性,来进一步分析特定分支的特点并预测可能的代谢产物。
2 肉座菌目虫生真菌基因组测序及次生代谢产物合成基因簇概况
2014—2019年期间,已发表的肉座菌目虫生真菌基因组序列数量大幅增加:2014 年以前为8 个,2014年以后增至31个,这为分析天然产物的合成潜力、比较基因簇之间的异同提供了丰富的数据。本研究选取的40种真菌来自于白僵菌属(Beauveria)、冬虫夏草属(Cordyceps和Ophiocordyceps)、被毛孢属(Hirsutella)、座壳孢属(Hypocrella或Aschersonia)、棒束孢属(Isaria)、轮枝孢属(Lecanicillium)、莫勒菌属(Moelleriella)、弯颈霉属(Tolypocladium)等,为农业上应用广泛的生防真菌,也是常用的药用真菌(菌种目录详见http://www.researchgate.net/publication/354887574_Table S1)。
通过HMMsearch检索所得的腺苷酰化结构域、缩合结构域和非核糖体多肽合成酶数量见表1、表2。腺苷酰化结构域的氨基酸序列一致性的25%分位数为27.70%,75%分位数为41.30%,离群值的下边界为62.06%。覆盖率值通常在91%~99%之间,异常值低于75%。因此,在后续分析中,分别选择62.06%和75%作为一致性和覆盖率的阈值,从而得到了182 个腺苷酰化结构域的分支(clade)。
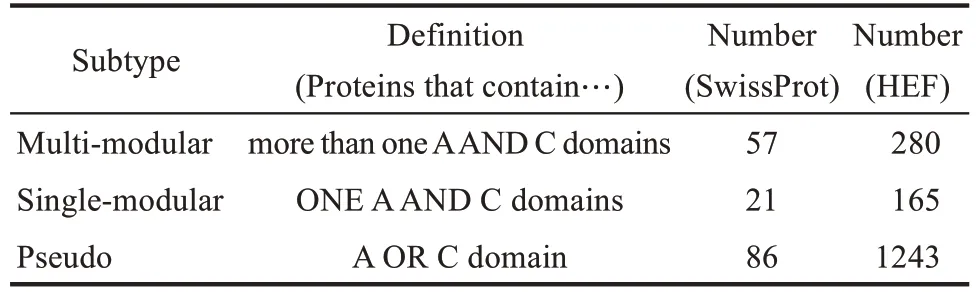
表2 非核糖体多肽合成酶统计信息Tab.2 Statistic summary of NRPSs in hypocrealean entomopathotenic fungi(HEF)
3 非核糖体多肽合成酶序列相似性网络和马尔可夫聚类分析
序列相似性网络(sequence similarity network,SSN)能够通过同源蛋白之间成对的序列相似关系,自动化评估蛋白的分类,并从家族/亚家族之间的关系来研究特定蛋白的功能。在SSN 中,每个蛋白用一个符号(“节点”)表示;如果两个节点的序列相似性超过指定阈值,则通过一条线(“边”)连接。已有多篇文章综述了SSN 在蛋白质序列与功能研究中的成功应用[18-20]。
腺苷酰化和缩合结构域均已成功用于非核糖体多肽合成酶系统发育研究[4,21-26]。本研究在网络构建和聚类分析腺苷酰化结构域的基础上,分析了部分非核糖体多肽合成酶的模块和结构域组成,以及所在的基因簇特征。研究结果显示肉座菌目虫生真菌的基因组中广泛存在功能未知的非核糖体多肽合成酶家族,即使在亲缘关系十分接近的种属之间,它们在基因或基因簇水平上也存在高度变异性,表明肉座菌目虫生真菌是合成新型非核糖体多肽类天然产物的潜在资源。
大多数非核糖体多肽合成酶具有多个腺苷酰化结构域,因此,利用对腺苷酰化结构域进行聚类分析来阐明合成酶的功能并预测产物结构变得十分复杂。在真菌聚酮合酶中,酮脂酰合酶(ketoacyl synthase,KS)结构域往往可以代表整个酶的特征;与此相对的,非核糖体多肽合成酶被视为具有不同系统发育特征的模块复合体,而腺苷酰化结构域的聚类则代表其所在模块的特征。收集到的2676 个腺苷酰化结构域(包括302 个已知功能的标签结构域)的氨基酸序列平均一致性为36.30%,其中2358 个被成功地分为174 个分支(不含聚酮合酶-非核糖体多肽合成酶杂合酶中的腺苷酰化结构域)。由于数目较多,本文图中仅列出具有5 个以上节点的分支簇,且排除了参与短链脂肪酸合成的酰基辅酶A 连接酶(http://www.researchgate.net/publication/354887574_Table S1)。由此产生的网络可将腺苷酰化结构域分为多模块、双模块、单模块和杂合的非核糖体多肽合成酶。这与先前报道的系统发育分析基本一致,即真菌非核糖体多肽合成酶分为两大分支:远古起源的单/双模块和新近起源的多/双模块[24,27-28]。
3.1 具有新型非核糖体多肽合成潜力的多模块非核糖体多肽合成酶
肉座菌目虫生真菌基因组预测的2440 个腺苷酰化结构域中,有1056 个来自于多模块的非核糖体多肽合成酶(图 1)[9,11-14,29-30],这类合成酶仅发现于真菌基因组,其腺苷酰化结构域可能由垂直遗传、基因重排(如复制、插入和丢失)或水平转移事件进化而来[4,24],是发现新型活性产物的宝贵资源。
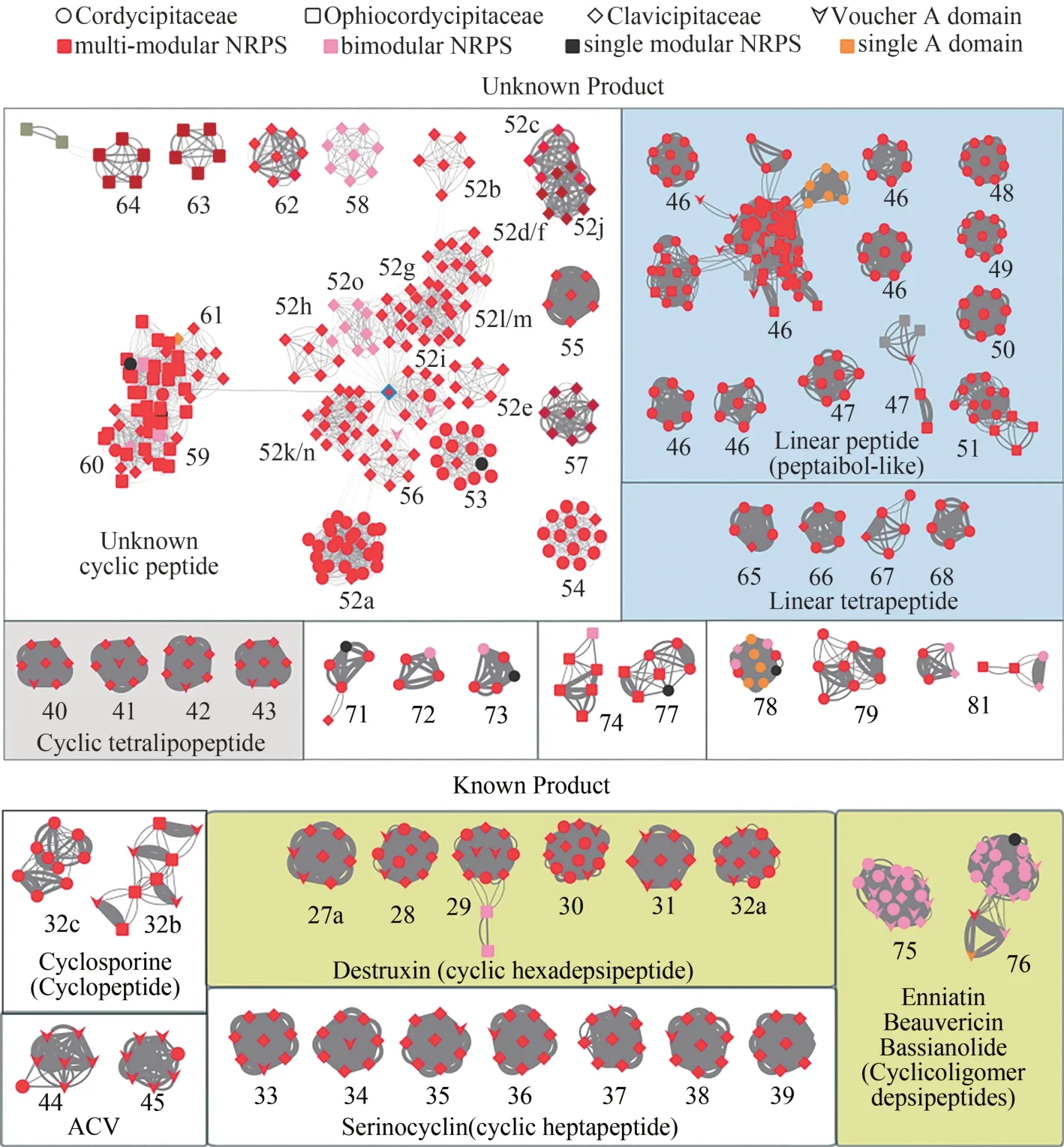
图1 肉座菌目虫生真菌多模块非核糖体多肽合成酶的相似性网络图Fig.1 Network for the subgroups of multi-modular NRPSs in Hypocrealean Entomopathogenic fungi
这些多模块非核糖体多肽合成酶中,绝大多数的产物是未知的。如图1 所示,第52~64 分支的非核糖体多肽合成酶形成了一个较大的超级分支(super clade)。根据氨基酸序列相似性,这些合成酶的最后一个结构域属于环肽类的终端缩合结构域,可以通过分子内环化释放延伸的肽链,因此推测其产物是环肽类化合物。其中,52~60分支可以进一步划分为3 种含有4 个以上模块的非核糖体多肽合成酶,其代表为B. bassiana的BBA_06727、M. robertsii的 MAA_09953 和 MAA_09772(图 1)。通过系统发育分析,这些非核糖体多肽合成酶曾被归为“昆虫病原菌特有”的进化分支[3-4]。与BBA_06727 相似的蛋白有13 个,结构域组成均为(AT)-(CAT)-(CAT)-(CAT)-(CT),它们的第1 个腺苷酰化结构域归于第53 个分支,第3 个腺苷酰化结构域归于第54分支,第2个和第4个腺苷酰化结构域均归于分支52a。同一个非核糖体多肽合成酶中的两个腺苷酰化结构域聚类在一起通常表明它们来源于进化上的复制事件。这一类非核糖体多肽合成酶所在的基因簇中还含有编码细胞色素P450 单加氧酶和转运蛋白的基因[图2(a)]。BBA_06727 的转录在昆虫感染期间被激活[27],表明可能在昆虫致病过程中起作用。52b~52h 亚分支和第55 分支的非核糖体多肽合成酶(如MAA_09953)存在于6 种绿僵菌属真菌基因组中,其结构域组成均为(AT)-(CAT)-(CATE)-(CAT)-(CAT)-(CAT)-(CAT)-(CT),所在基因簇中还包含编码细胞色素P450 单加氧酶、β-内酰胺酶和转运蛋白的基因。以MAA_09772 为代表的非核糖体多肽合成酶在8 种绿僵菌属真菌中十分保守,具有(AT)-(CAT)-(CATE)-(CAT)-(CAT)-(CAT)-(CTT)的结构域组成,其腺苷酰化结构域归于分支59、亚分支52i~52n、分支60,相应的合成基因簇包含细胞色素P450 单加氧酶、Ser/Thr 蛋白磷酸酶和两个转运蛋白的基因。亚分支52o 和分支58 的腺苷酰化结构域也来源于相同的非核糖体多肽合成酶,其结构域的组成为(AT)-(CATE)-(CT)-(CT),基因簇含有编码细胞色素P450 单加氧酶、两种氨基转移酶、脱羧酶、双加氧酶和含有腺苷酰化结构域的连接酶/合成酶的基因。值得注意的是,超级分支52中几乎所有的分支都是通过52i中的一个节点连接的,这个节点对应于M.rileyi基因组中NOR_07696 的第2 个腺苷酰化结构域(介数betweenness为0.826),表明这个腺苷酰化结构域与共同的祖先最为相似。分支59~61中来自于线虫草科(Ophiocordycipitaceae)和麦角菌科(Clavicipitaceae)的非核糖体多肽合成酶也通过NOR_07696相连。
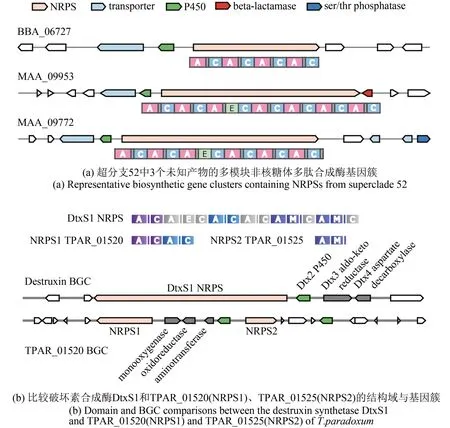
图2 肉座菌目虫生真菌基因簇分析Fig.2 Analysis of representative biosynthetic gene clusters in hypocrealean entomopathogenic fungi
分支46~51 对应的非核糖体多肽合成酶可能产生peptaibols 类线性多肽[30],其前体单元除氨基酸外,还包括由基因簇中其他酶合成的非常规前体。这些多模块非核糖体多肽合成酶大多来自Trichotheciam和Tolypocladium属真 菌[30],由 8 个以上CAT 模块组成,有的还具有特殊模块或结构域,如T. inflatum的ctg24_orf003 具有一个酮酯酰合成酶和丙二酰/酰基转移酶结构域组成的加载模块。O.australis的CDD81_5223 的终端结构域为硫酯酶结构域,而不是CT。这些非核糖体多肽合成酶所在的基因簇并不保守,普遍存在转氨酶基因,可能用于合成非常规氨基酸前体。
当非核糖体多肽合成酶两两比较时,经常发现它们的部分腺苷酰化结构域能够聚类在一起,而其余腺苷酰化结构域则属于不同的分支。例如,来自T.paradoxum的TPAR_05120 对应于绿僵菌属破坏素合成酶DtxS1 的第1 和第3 个结构域,而TPAR_01525 则与 DtxS1 的第 5 或第 6 模块相似[图2(b)]。这种嵌套关系展示了非核糖体多肽合成酶复杂的进化过程,使其产物有不同的结构和生物学功能。
除此之外,一些分支包含产物已知的非核糖体多肽合成酶,具有相同的结构域组成,所在的基因簇也高度相似,可能产生与已知化合物结构类似的产物。其中,分支75~76 包含白僵菌素/白僵菌环四肽合成酶的腺苷酰化结构域[11,31],分支27~32 和 33~39 则 分 别 包 含 破 坏 素[9-10]和serinocyclin[13]合成酶的腺苷酰化结构域(图1),说明这些分支中的菌株具有合成杀虫化合物的潜力。
3.2 较为保守的单模块和多模块非核糖体多肽合成酶
如图3 所示,单/双模块的非核糖体多肽合成酶包括赖氨酸生物合成途径中还原α-氨基己二酸的L-氨基己二酸半醛脱氢酶(分支23)[24],参与形态发育但产物未知的ChNPS10 合成酶(分支15)[40],产生聚硫二酮哌嗪的 ChNPS11/ETP 毒素合成酶(分支12 和13)等[41],其合成的代谢物参与了真菌重要的生物过程,如性发育、生殖、分生孢子发育等,因此在基因组中也较为保守[24,32-39]。其中,部分基因的生理功能已有研究,但其合成产物仍然未知。
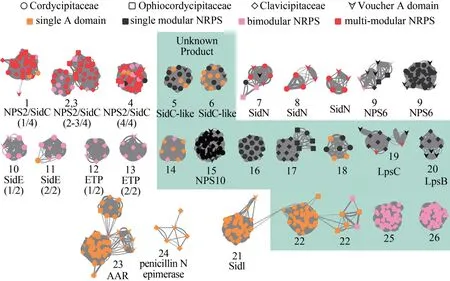
图3 肉座菌目虫生真菌单模块和双模块非核糖体多肽合成酶的相似性网络图Fig.3 Overview of the A domain distance network for monomodular,bimodular or siderophore-like NRPSs in Hypocrealean entomopathogenic fungal species
铁载体是一种低分子量的铁螯合物,可分为胞外和胞内两种类型,因与病原真菌的致病性相关而受到广泛关注[37]。铁载体合成酶具有保守的结构域,合成产物的结构和生物学功能相似,在各种营养模式的真菌中广泛分布,包括动植物病原菌和腐生菌。铁载体合成酶是多模块的非核糖体多肽合成酶,在系统发育中属于较新的分支,然而由于其序列和功能的保守性,将它们与单/双模块的非核糖体多肽合成酶一起分析。大部分肉座菌目虫生真菌的基因组中都有铁载体合成酶NPS2/SidC(分支 1~4)[36]、NPS6(分支 9)[39]、SidN(分支7 和8)[38]和/或 SidⅠ(分支21)[37]的编码基因。值得注意的是,分支5 和6 的腺苷酰化结构域与NPS2/SidC 的第1 个腺苷酰化结构域具有很近的亲缘关系,但其所属的非核糖体多肽合成酶与NPS2/SidC截然不同:仅有一个模块,几乎只存在于白僵菌/冬虫夏草属真菌中,与醇脱氢酶、细胞色素P450 单加氧酶、聚酮合酶或聚酮合酶-非核糖体多肽合成酶编码基因成簇存在。这表明该分支的类SidC 非核糖体多肽合成酶可能是通过基因复制从铁载体合成酶进化而来,并获得了不同的功能。分支10~11 的双模块非核糖体多肽合成酶与人类病原菌A.fumigatus的SidE相似[42]。SidE尽管与NPS2/SidC铁载体合成酶有着密切的系统发育关系,但其产物不是铁载体,而是反丁烯二酰丙氨酸,其结构与已知具有免疫调节活性的药物十分相似[42]。在球孢白僵菌中,类SidE 编码基因BBA_07589 在感染小菜蛾期间上调[27]。因此,分支10~11 的非核糖体多肽合成酶可能产生类似反丁烯二酰丙氨酸的产物,用于抵抗宿主免疫系统的攻击。
分支16~20 的腺苷酰化结构域来自于单模块非核糖体多肽合成酶。其中,分支16 主要存在于白僵菌/冬虫夏草属真菌的基因组中,它们的编码基因与羧肽酶、双加氧酶、铁(Ⅱ)加氧酶、酰胺水解酶、磷酸酯酶和转运体蛋白等基因共同组成基因簇,其中一些基因簇还编码聚酮合酶-非核糖体多肽合成酶杂合酶、酮基还原酶、烯基还原酶、甲基转移酶和细胞色素P450 蛋白。分支17 中的腺苷酰化结构域主要存在于绿僵菌中,它们的基因簇含有一个寡肽转运蛋白基因,但没有修饰酶基因。分支19~20 中的腺苷酰化结构域存在于大多数已测序的绿僵菌属真菌中,与LpsC 或LpsB型麦角生物碱非核糖体多肽合成酶十分相似[32],其基因簇中包含萜烯合成酶,可能产生麦角酸α-羟乙基酰胺型或麦角碱类产物[43]。
4 结论与讨论
肉座菌目虫生真菌是一个极有特色的真菌类群,它们在不同的情况下从植物病原菌和腐生真菌进化成为昆虫病原菌,并发展出多种侵染昆虫、抑制其免疫系统、利用昆虫作为自身营养源的机制。同时,许多肉座菌目虫生真菌还保持着与植物共生以及营腐生生活的能力,研究它们的生物合成基因簇不仅有助于理解天然产物在生物和非生物相互作用中的功能,也有助于挖掘临床、兽用或农用药物分子。
近年来基因组测序成本持续降低,基因组分析工具不断完善,公共数据库中高质量真菌基因组序列数量激增,这些极大改变了基因和基因簇功能研究的模式。利用简并引物PCR、黏粒基因文库和类似的传统方法迅速被基于组学方法的基因功能研究所取代,如产生活性代谢产物的真菌菌株的基因组草图测序与分析、基于目标产物结构的逆生物合成分析、针对产生类似产物菌株的比较基因组分析以及在产生和不产生目标代谢产物的条件下的转录组分析等。类似的基因组学方法已经成为挖掘活性代谢产物及其合成基因簇的主要手段。
越来越多的基因组序列也使得人们发现了大量产物未知的生物合成基因簇。利用异源表达方法,将基因簇转入较为成熟的底盘细胞中激活并表达,逐渐成为天然产物挖掘以及生物合成途径研究的主流方法之一。然而,这些方法仍然面临着重复发现已知产物的问题。因此,尽可能准确地预测这些未知基因簇的功能、筛选最具潜力的研究对象是十分有必要的。对未知基因和基因簇进行聚类分析可以:①发现与合成已知活性产物的基因簇相似的基因簇,从而得到活性产物的类似物,例如分支75~76、分支27~32 的合成酶产生具有杀虫、抗菌、细胞毒性等活性的白僵菌素/白僵菌环四肽、破坏素类化合物,分支10~11 的合成酶产生可能具有免疫调节活性的类似反丁烯二酰丙氨酸的化合物;②挖掘全新的生物合成基因簇,得到新结构、新功能的化合物,例如分支52~64 的合成酶可合成4~8 个氨基酸单体组成的环肽,这些合成酶与已知功能的非核糖体多肽合成酶的序列差异较大,且仅发现于肉座菌目虫生真菌基因组中。由于大部分基因在实验室常规培养条件下不表达,因此,研究其产物需要进行异源表达、调控因子激活/抑制或其他转录激活方法。此外,在利用合成生物学方法创制新结构人工产物方面,可根据聚类分析的结果,选取特定功能的基因或基因簇,异源表达部分基因簇、或来源于不同基因簇的基因(即组合生物合成)从而改造已知化合物。
通过对腺苷酰化结构域的聚类来预测真菌非核糖体多肽合成酶的功能也存在一定的局限性。即使在系统发育上接近且整体氨基酸序列一致性很高的腺苷酰化结构域之间,其底物范围也有可能显著不同,如在酶的活性部位的关键氨基酸位点发生改变,就可能改变被激活的氨基酸单体的范围。与原核生物相比,真菌腺苷酰化结构域的底物选择性预测还不够精确,这是因为现有的预测算法往往是根据细菌来源的非核糖体多肽合成酶构建和训练的。因此,为了更好地建立蛋白序列与产物结构之间的映射关系、从而实现更加准确的功能预测,需要更丰富完善的数据库和更加先进的分析方法。

