基因组挖掘在天然产物发现中的应用和前景
杨谦,程伯涛,汤志军,刘文,2
(1 中国科学院上海有机化学研究所,生命有机化学国家重点实验室,上海 200032;2 中国科学院上海有机化学研究所,湖州生物制造中心,浙江 湖州 313000)
自然界作为活性天然产物的重要来源孕育了数以万计的生命有机体。在过去的几十年里,绝大多数抗癌、抗感染和抗菌药物都源于生命体所产生的天然产物及其衍生物,如青霉素、洛伐他汀、紫杉醇[1-2]。其中,许多来源于土壤、海洋及特殊环境的微生物由于能够产生一系列活性显著且具有成药潜力的天然产物而备受关注,这些化合物的发现也为新药创制开辟了新的思路。然而,人类疾病谱的变化以及多药耐药等一系列问题的出现,使得开发新的药物成为人类健康的迫切需求。基于活性导向药物发现的方法虽然可以从植物、动物及微生物中分离获得具有生物活性的小分子,但是这些筛选方法不仅耗时耗力,而且不能避免重复性、盲目性以及低效率等弊端。
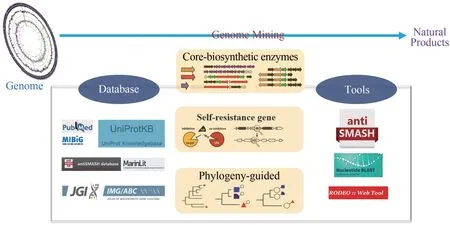
基因组时代的到来为我们提供了来源于不同有机体数以万计的DNA(deoxyribo nucleic acid,脱氧核糖核酸)序列,这些数据不仅能够为生物学各个分支学科提供丰富的物质基础,同时也为天然药物的发现带来了新的曙光。基因组数据不仅可以用来识别潜在的药物靶标[3],还能用于寻找先前被忽视的次级代谢产物的生物合成途径[4],定向发现新的天然产物药物。每一个旨在预测生理或代谢特性的生物信息学研究都可以被认为是基因组挖掘(genome mining)。然而,在与天然产物相关的文献中,“genome mining”经常被赋予更特殊的意义,它不再局限于通过计算模拟的方法检测生物合成基因,还涉及到它们的功能研究,最终阐明相关的化学机制。随着基因组数据的丰富,次级代谢产物的基因簇不再匮乏,更大的挑战转向了如何高效快速地锁定具有挖掘潜力的生物合成基因簇(biosynthetic gene clusters,BGCs),从而快速地获得药物实体分子。在生物信息学发展的同时,许多专业的网络工具也被研究人员运用到基因组数据的挖掘过程中,目前已有许多综述进行了报道[5-13]。本文综述了基因组挖掘在天然产物发现中的运用,包括最新的生物信息学工具、常用的各类数据库以及主要的挖掘方法,并对基因组挖掘在药物发现和多个学科领域中的影响和发展进行了展望。
1 基因组挖掘过程中的数据库和网络工具
数字革命正在改变人们储存、传播和使用信息的方式。随着关联数据、机器学习和大规模网络推理等新技术的出现,天然产物研究领域开始实现数字化实验数据的实时共享和大规模分析。数据库在这方面发挥了关键作用,因为它们允许对基本和高级应用程序的数据进行系统的注释和存储[14]。
1.1 天然产物数据库
2020 年,Maria Sorokina 等整理了 2000 年以来所有的天然产物数据库,到目前正在使用的数据库超过了120个,其中只有50个可以免费访问[15]。在这些数据库中,有3个在微生物领域运用最为广泛,即NPASS、StreptomeDB 和The Natural Products Atlas。其余常用的数据库还包括Dictionary of Natural Products(DNP)[16]、PubMed[17]、MarinLit、AntiBase、KNApSAcK[18]、Norine[19](非核糖体肽专门的数据库)和MacrolactoneDB[20]等,这些数据库包含了来源于植物、海洋微生物、真菌及细菌等生命有机体产生的天然产物。
NPASS 是 2018 年发展起来的一个数据库[21],旨在提供天然产物的来源及其生物活性。它包含了部分来自植物、无脊椎动物和微生物天然产物的化学结构,共含有超过35 000 种化合物,其中大约9000种来源于微生物。StreptomeDB是一个链霉菌属天然产物的专门数据库[22]。在2020 年的更新信息中,该数据库包含了7100 多个化合物的来源、生物活性及其光谱信息。The Natural Products Atlas[23]是2019年建立的一个新的数据库,它提供了所有微生物产生的天然产物衍生物的化学结构。当前该数据库包含了超过25 500 个化合物,它具有一个特殊的检索链接,能够双向连接到另外两个天然产物资源库:一是生物合成基因簇的MⅠBiG (Minimum Ⅰnformation about a Biosynthetic Gene cluster)[24]数据库;另一个是天然产物质谱数 据 的 GNPS (Global Natural Products Social Molecular Networking)[25]数 据 库 。 Dictionary of Natural Products是一个非开源数据库,主要收录天然产物的来源、物理特性及其生物学数据,目前已经收录了超过29 万个条目。MarinLit 是一个海洋天然产物的文献数据库,包含超过35 000 个化合物的化学结构、分类及其全合成数据。它是目前海洋天然产物最新和最全面的数据库。值得一提的是,目前世界上最大的两个非开放的天然产物数据库:Scifinder和Reaxys。这两个平台包括了来自天然产物文献的大多数天然产物及其衍生物、合成中间体。
1.2 生物合成基因簇数据库
自2000 年以来,越来越多的基因组数据被报道,而在NCBⅠGenBank[26]中能够找到“基因-结构”相对应的数据屈指可数。为了解决这一问题,研究者开发了一系列专门的生物合成基因簇数据库,例如ClusterMine360[27]、DoBⅠSCUⅠT(Database of BⅠoSynthesis clusters CUrated and ⅠnTegrated)[28]、MⅠBiG 2.0[24]、ⅠMG-ABC[29]、antiSMASH Database[30]和Recombinant ClustScan Database[31]。ClusterMine360作为早期的生物合成基因簇分析平台,将那些通过实验证实的生物合成基因簇与其对应的化合物进行了链接,主要聚焦于非核糖体肽(non-ribosomal peptide,NRP)和聚酮(polyketide,PK)类化合物,它包含了超过300个天然产物生物合成基因簇的信息。2015年,通过150多名天然产物科学家的共同努力发布了“生物合成基因簇的最小信息库”(Minimum Ⅰnformation about a Biosynthetic Gene Cluster,MⅠBiG),对已被实验证实天然产物的生物合成基因簇进行了人工注释[32]。利用联合基因研究所(Joint Genome Ⅰnstitute,JGⅠ)的细菌基因组平台发布的 ⅠMG/M[29]和 ⅠMG-ABC[29],旨在发展成为一个最全面的细菌基因组数据库,它包含已知天然产物的生物合成基因簇(有些信息间接地来源于MⅠBiG)的信息,并且能够通过计算模拟预测未知生物合成基因簇的功能。到目前为止,该数据库包含了来源于antiSMASH 和ClusterFinder算法模拟获得的超过100万个生物合成基因簇。由于JGⅠ的数据使用限制,部分数据目前处于未公开状态。同样由JGⅠ开发的真菌基因组门户MycoCosm[33],包含超过1000 个真菌基因组信息,它通过提供交互式网络工具,支持真菌基因组序列和其他“组学”(omics)数据的整合、分析和共享。2016 年, antiSMASH 的开发团队发布了antiSMASH database[30-34](antiSMASH-DB),作为antiSMASH 运行的中央存储库。与ⅠMG-ABC 不同的是,antiSMASH-DB 旨在提供一个有限的、复杂的假定生物合成基因簇列表,这些生物合成基因簇来自最高质量的细菌基因组。在2018 年发布的第二版中,antiSMASH-DB 包含了来源于24 000 多个细菌基因组的152 000 多个生物合成基因簇[35]。
1.3 蛋白家族数据库
蛋白质通常由一个或多个功能区域组成,识别这些功能区域有助于预测未知蛋白的功能。UniProtKB[36]是目前世界上最大的蛋白数据库,囊括了超过2 亿个非重复的条目。它分为两个部分 : UniProtKB/SwissProt 和 UniProtKB/TrEMBL。其中,UniProtKB/SwissProt 带有功能性的注释,这些注释来源于各类文献中已经被人工核实的数据。截至 2020 年 6 月,SwissProt 收录了 563 972 个条目,由于人工注释并不能做到面面俱到,因此它收录的功能并不是全面的,但是SwissProt 能够接受使用者输入注释数据,从而达到数据库的实时更新。在UniProtKB/TrEMBL 中,基于序列同源性的分析会进行自动分配注释,系统会自动将满足条件的注释从储存序列转移到假定的同源序列中[37]。
目前,常用的蛋白家族数据库包括Pfam[38]和ⅠnterPro[39]。Pfam[38-40]是注释信息最为全面的蛋白家族数据库,每个家族都由多序列比对和隐马尔可夫模型(HMMs)表示。在最新发布的版本Pfam 33.1 中,定义了18 259 个同源家族,有70%的条目与UniProtKB 密切相关。ⅠnterPro 是一个更大的蛋白家族数据库。截至2020 年11 月,该数据库定义了38 345个条目,包括3326个同源超家族、22 618 个家族、11 162 个功能域。但是,并不是所有的序列都能被Pfam和ⅠnterPro所包含,因此,在蛋白质领域存在着大部分尚未注释的基因组“暗物质”,它们可能具有某些特殊的功能[5]。为此,生物学家开发出了一系列能够有效挖掘蛋白和基因组数据的工具,称为“基因组酶学计算机模拟工具”[5]。
1.4 识别生物合成基因簇的网络工具
自从第一个链霉菌基因组被测序[41],基因组挖掘迅速成为发现天然产物的一种重要方式,人们发现神秘的生物合成基因簇为新颖天然产物的发现开启了新的篇章。基因组挖掘利用遗传/基因组信息来评估微生物产生新化合物的遗传潜力,通过运用多种生物信息学工具在众多的基因序列中识别具有潜在价值的生物合成基因簇,并根据基因簇中的信息来预测其产物,最终阐明生物合成过程。
序列相似性搜索是一项非常重要的生物信息学任务。基于局部比对搜索工具BLAST(Basic Local Alignment Search Tool)[42]和利用隐马尔可夫模型(hidden Markov model)进行蛋白序列分析的网络工具HMMer[43]是目前最简单的序列比对工具,它们使用人工构建的基因列表作为查询序列,对未知蛋白进行序列比对从而初步预测其功能。此外,DⅠAMOND[44]是一种基于双索引的开源算法,速度比BLASTx快20 000倍,但灵敏度与其不相上下。如今,这些分析方法已经变得越来越复杂,常用的分析工具包括:ClustScan(Cluster Scanner)[45]、 CLUSEAN (CLUster SEquence ANalyzer)[46]、np.searcher[47]、SMURF[48]和 anti SMASH[49-50]。ClustScan 是一个用于模块化生物合成基因簇的半自动注释和新型化学结构的计算机模拟预测的集成程序包。该程序包用于快速、半自动地对编码模块化生物合成酶的DNA 序列进行注释,包括聚酮合酶(PKS)、非核糖体肽合成酶(NRPS)和聚酮-非核糖体杂合酶(PKS/NRPS)。但由于版权限制目前只能提供用户30 天的试用期,属于半开放的程序包。CLUSEAN 是一个开放式的自动分析细菌次级代谢产物生物合成基因簇的计算机框架程序。它集成了标准的分析工具,如BLAST 和HMMer,以及能够识别非核糖体肽合成酶(NRPS)/Ⅰ型聚酮合酶(TⅠPKS)功能域和基序的特定工具,并且能够预测NRPS 的特异性。
为了促进真菌基因组中次级代谢产物生物合成基因簇的系统定位,Natalie D. Fedorova 等开发了一个基于网络的软件工具——Secondary Metabolite Unique Regions Finder (SMURF)[48],它基于真菌次级代谢产物生物合成途径的三个特征:①存在骨架基因;②成簇出现;③含有特征蛋白的结构域,对真菌基因组中的生物合成基因簇进行预测和归类。2011 年,Eriko Takano 等开发一个强大的网络工具,用于细菌和真菌基因组序列中次级代谢物生物合成基因簇的快速识别、注释和分析,并命名为antiSMASH (antibiotics &Secondary Metabolite Analysis Shell)[49]。直至2019年,antiSMASH 5.0[50]增加了编码酰基氨基酸、β-内酯、真菌RiPPs等多种类型基因簇的检测规则,尤其是对于编码Ⅱ型PKS 生物合成基因簇提供了更多详细的预测,同时在网页运行方面也缩短了一些时间。
近几年一些新颖的分析工具相继被开发使用,它们能够解决上述算法中的缺陷:在检测已知基因簇方面具有高特异性,但是并不会识别未知的生物合成基因簇。从基因组中预测未知类别基因簇应该是最具优先级的,因为这些基因簇可能编码了全新骨架的分子[51]。根据文献报道,目前实验室可培养的微生物只占总数的1%[52],而这1%的微生物就含有超过200 万株细菌或真菌(http://www.wfcc.info/ccinfo/),这意味着含有数量级的未被识别的生物合成基因簇有待开发和挖掘。这些基因簇被人们统称为“微生物的暗物质”,它们可能存在于未被开发的菌株中,也可能存在于像大肠杆菌这样被研究成熟的微生物中。这就需要运用更为复杂的算法提供强有力的检索能力来识别“暗物质”,从而成功地获取全新的天然产物分子。
目前开发了3 个研究策略:①ClusterFinder[53]算法,它首先识别基因组序列中可能的基因编码区域,利用Pfam 数据库对编码区域进行蛋白功能域注释,然后依据Pfam 数据库中的蛋白功能域在训练集生物合成基因簇中出现的频率,利用HMM 将其设定为 BGC 或者 non-BGC 状态。ClusterFinder 能够识别出富含 BGC 状态 Pfam 数据库功能域的基因组区域。这种策略能够发现新类型的基因簇,因为不同分子的生物合成途径往往利用相同家族的酶,如氧化还原酶、甲基转移酶、CoA 连接酶和 P450 氧化酶[53]。②基于所有次级代谢酶都是初级代谢酶同源物这个观点发展了EvoMining[54]方法,通过检测基因组中“额外”的代谢酶,使用系统发育分析来识别进化上具有明显差异的序列,并对其上下游基因进行功能分析,从而发现新的生物合成基因簇。③使用大规模的基因组序列比对。首先利用BLASTp 寻找不同基因组中的同源基因,从同源基因出发通过局部比对识别种子区域(seed regions),对种子区域进行扩张,锁定基因簇边界并进行共线性分析(synteny analysis),最终寻找到可能编码新颖次级代谢产物的基因簇[55]。这三种策略的综合运用可能成为未来识别生物合成基因簇最有效的方法[7]。
核糖体合成和翻译后修饰肽(RiPPs)是从基因编码的前体肽衍生而来的一类天然产物,由于不同类别前体肽缺乏共同的序列特征,因此通过计算识别其生物合成基因簇一直是极具挑战的任务。最近开发了几种新的算法,专门进行RiPPs的生物合成基因簇的挖掘。Andrew W. Truman 等开发了一种用于识别不同家族RiPP 前体肽工具RiPPER,运用该方法在放线菌中找到了新的含有硫酰胺结构的RiPPs[56]。许多RiPPs 后修饰的发生依赖于一个称为RiPP 识别元件(RRE)的蛋白结构区域。RRE 与前导肽(leader peptide)特异性结合,并引导翻译后修饰酶作用于核心肽(core peptide)。Douglas A.Mitchell 等开发了一种基因组挖掘的工具 RRE-Finder[57],它从 UniProtKB 蛋白数据库中调取25 000 条高可信度的RRE 蛋白序列作为样本数据库,进一步识别基因组中可能包含RRE 序列的生物合成基因簇。此外,还有一些新的挖掘工具也被开发出来,例如DeepRiPP[58]和RODEO (Rapid ORF Description and Evaluation Online)[59]。基因组挖掘过程中常用的数据库及网络工具见表1。

表1 基因组挖掘的数据库及网络工具Tab.1 Database and web tools of genome mining
2 基因组挖掘在天然产物发现中的应用
“基因组挖掘”,几乎与每一个生物信息学研究相关联,它可以用于检测生物活性天然产物的生物合成途径。对天然产物研究领域而言,基因组挖掘就是在没有化学结构的前提下,基于遗传信息来预测和分离活性天然产物。根据挖掘对象的不同,可以大致分为基于核心骨架酶的挖掘、基于抗性基因的挖掘以及基于系统进化的挖掘。
2.1 基于编码核心骨架的酶进行挖掘
以编码合成核心骨架的酶出发,挖掘具有特定结构片段的天然产物,是一种经典的基因组挖掘方法。尽管次级代谢产物的结构多种多样,但是同一类型代谢产物的生源途径往往是非常保守的,这是由于许多核心骨架生物合成的酶在序列上具有高度的相似性。如聚酮类(polyketides)、非核糖体肽类(non-ribosomal peptides)以及氨基糖苷类(aminoglycosides)。利用天然产物结构与其对应的生物合成基因一一对应的关系,在基因层面发现含有特定结构片段的天然产物,指导新化合物的发现。
烯二炔类抗生素是迄今为止发现的抗肿瘤活性最高的天然化合物[64](图1),其活性中心是双键偶联两个炔键构成的烯二炔核心结构,目前已有20 余例烯二炔天然产物陆续被报道,虽然它们的核心环不同(九元环或者十元环),但是核心的烯二炔单元却由相同的生物合成逻辑合成,由包含编码特殊Ⅰ型聚酮合成酶PKSE、硫酯水解酶TE和3 个未知功能蛋白在内的5 个连续基因组成的基因盒催化完成[65-66]。聚酮合成酶PKSE 重复使用7 次,完成结构独特的九元(C-1027)或十元(Calicheamicin)烯二炔核心结构的不饱和聚酮前体的合成,再由3 个未知功能的酶以及TE 催化完成核心烯二炔单元的合成。为了挖掘更多的烯二炔类天然产物,Shen Ben 研究组以PksE核心基因为探针,对4889个已经测序的微生物基因组分析,又找到51 个基因组中含有合成烯二炔结构特征的基因盒[67];此外,他们还基于实时PCR 技术,开发了基于核心基因盒快速分析菌株是否含有合成烯二炔的基因簇的高通量方法,从3000 株菌株中找到81 株具有烯二炔生物合成基因簇[68]。以上结果表明,虽然目前发现的烯二炔类天然产物很少,但是大自然有巨大的潜力合成更多这类高活性化合物。随着沉默基因激活技术的成熟、异源表达体系的完善、发酵分离技术的提高,有望利用基因组挖掘的方法,分离得到更多、活性更优的烯二炔类天然产物。
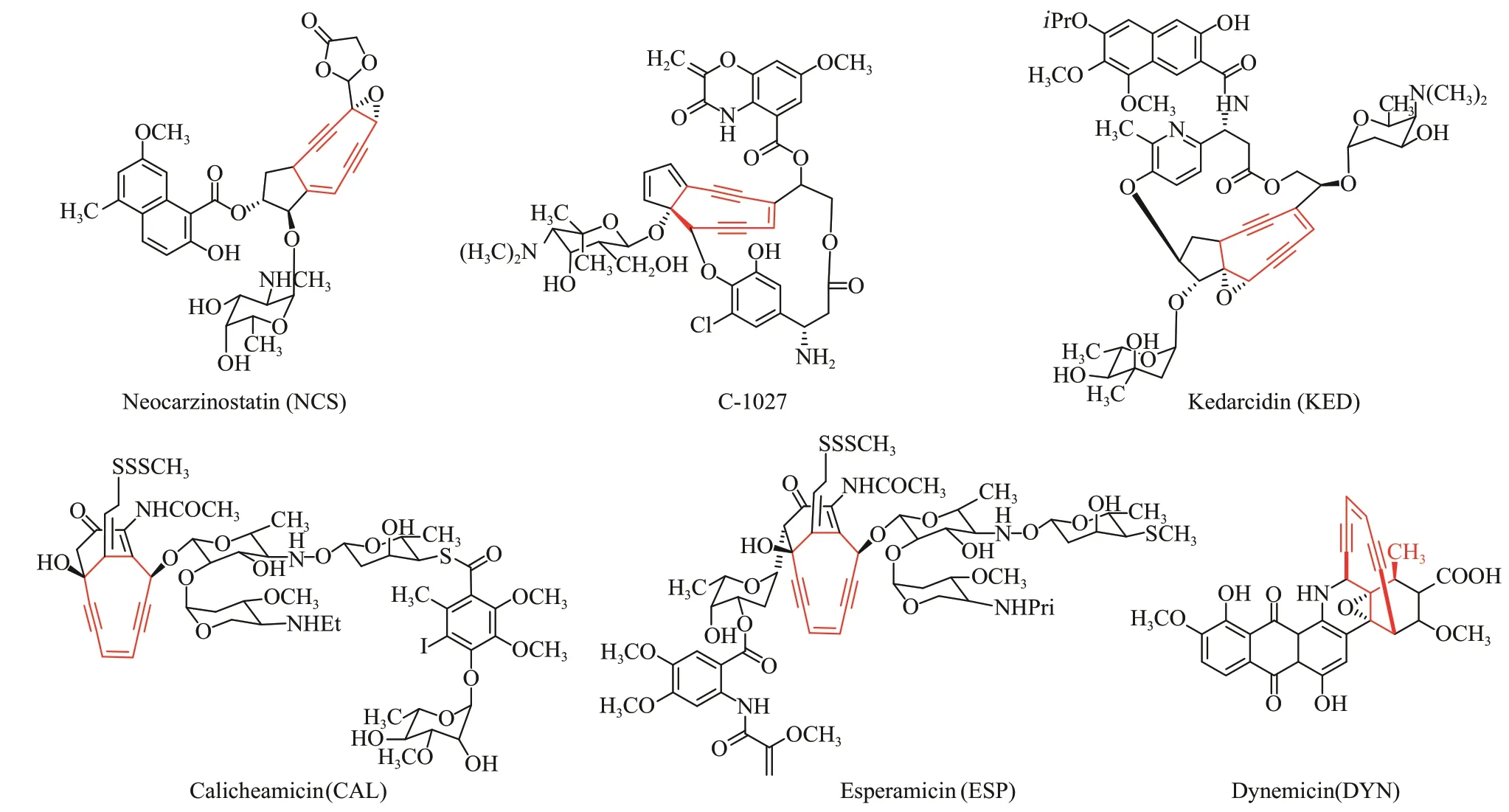
图1 代表性的烯二炔类化合物Fig.1 Representative compounds of Enediyne
脱水氨基酸是羊毛硫肽和硫肽类天然产物共同具备的特征结构片段。Ⅰ型羊毛硫肽和硫肽的脱水氨基酸通过相同的化学机制引入[69-70]。LanB蛋白的谷氨酰化结构域催化前体肽中丝氨酸/苏氨酸侧链羟基的谷氨酰活化,谷氨酸消除结构域催化谷氨酸离去形成脱水氨基酸(图2)。Van der Donk 研究组对超过100 000 个细菌基因组进行LanB 蛋白搜索,寻找到超过600 个与LanB 谷氨酰化结构域同源的基因,包含这些基因的基因簇或者基因组不包括LanB 谷氨酸消除结构域同源基因[71]。他们利用来源于Pseudomonas syringaepv.maculicolaES4326 的tgl基因簇为研究对象,揭示了一类新的核糖体肽分子pearlin 的生物合成过程。tgl簇中包含LanB 同源蛋白TglB,其催化前体肽TglA 的羧基端半胱氨酰化,在前体肽上实现一分子半胱氨酸的延伸。在整个生物合成过程中,不涉及对前体肽TglA 的额外修饰,前体肽仅作为骨架,接受后修饰酶识别,承载修饰对象半胱氨酸。最终,半胱氨酸被转化为thia-Glu成熟分子。

图2 LanB蛋白的催化机制及pearlin的生物合成过程Fig.2 Catalytic mechanism of LanB and the biosynthesis of pearlin
polytheonamide(图3)是一种具备高细胞毒性的核糖体肽类天然产物,成熟分子中含有DL-交替的氨基酸序列和AsmX5Asm 的天冬酰胺N-甲基化基序,保证成熟分子形成可插入细胞膜的稳定β-螺旋构象[72]。D构型非天然氨基酸的引入由依赖于S-腺苷甲硫氨酸的PoyD 蛋白负责[73],天冬酰胺的侧链N-甲基化则由N-甲基化酶PoyE 催化形成[74]。Jörn Piel 研究组以 polytheonamide 合成途径的前体肽基因poyA、异构酶基因poyD、N-甲基化酶poyE作为样本序列,分别对非冗余蛋白序列数据库进行BLASTp搜索,集合同时含有三者同源序列的基因组,挖掘到aer基因簇[75]。该簇导向发现了polytheonamide 类似结构终产物aeronamide A(图3),其同样具备高细胞活性,针对HeLa 细胞的ⅠC50值为1.48 nmol/L。
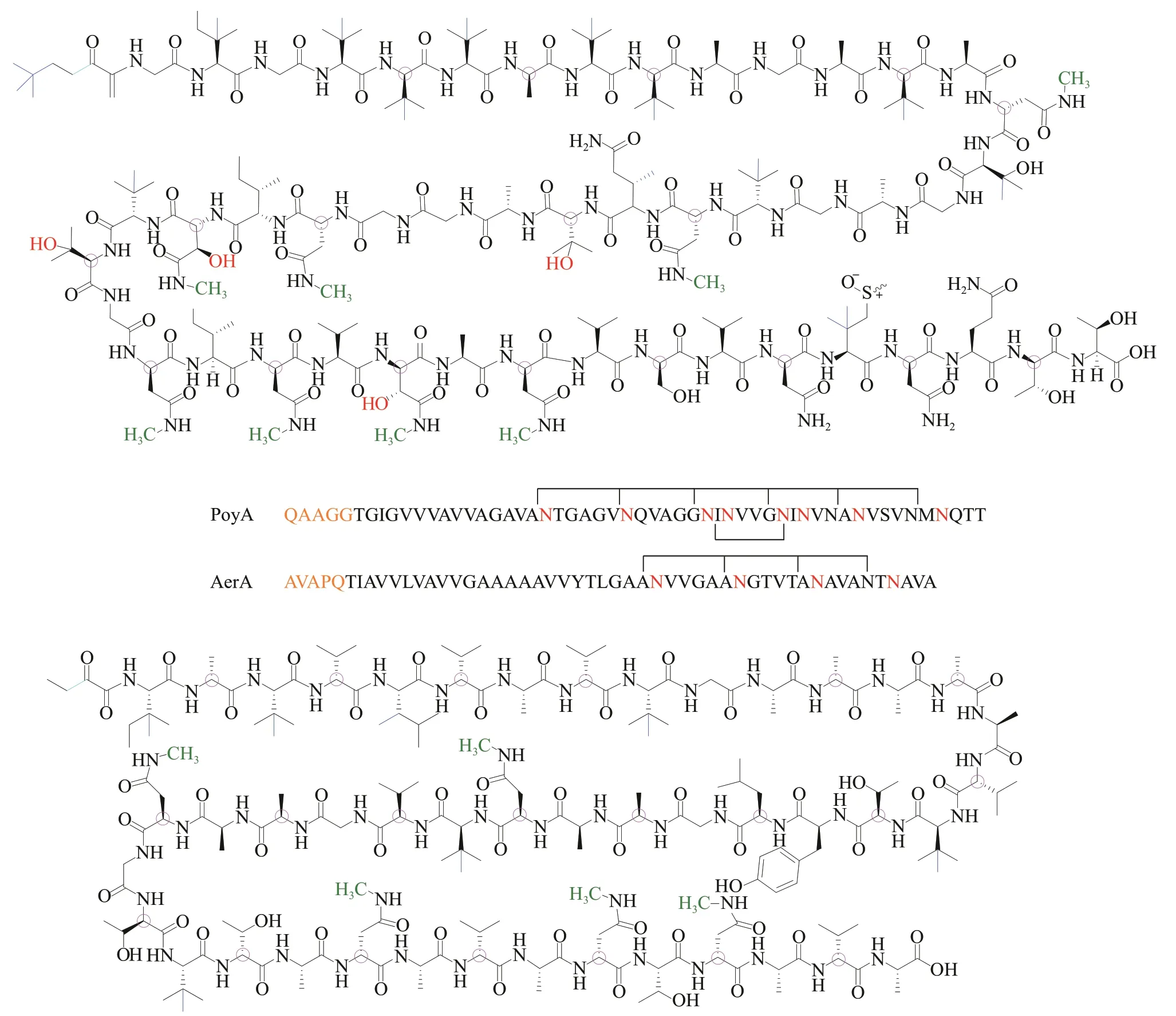
图3 polytheonamide和aeronamide A的化学结构Fig.3 Chemical structures of polytheonamide and aeronamide A
蛋白功能总是处在不断进化的过程中,尽管来源于相同的祖先序列,在经历如基因复制、水平基因转移等生理过程后,基因的功能趋向差异化。因此,具备一定序列同源性的蛋白可能存在催化功能上的差异性。蛋白功能的差异导致天然产物结构的差异。从催化特定反应的蛋白出发,建立其与同源蛋白之间的进化关系,可能寻找到催化差异反应的同源蛋白,定位到具备新结构的天然产物,表现出相似或者差异的生理功能。
自由基SAM 酶(rSAM,图4)普遍存在于核糖体肽合成途径中,其利用[4Fe-4S]簇还原性裂解S-腺苷甲硫氨酸生成5'-脱氧腺苷自由基[76]。该自由基进一步从底物中提取氢原子,从而引发不同的反应。Sactipeptide 分子中存在特征性的S—Cα硫醚键,该键由具有额外的C端[4Fe-4S]簇结合基序(SPASM)的自由基SAM 酶催化形成[77]。从6 个已知的催化前体肽S—Cα键形成的rSAM 酶出发,Douglas A. Mitchell 研究组利用 PSⅠ-BLAST 建立蛋白序列文库,并利用RODEO 注释备选蛋白本地基因组序列,对潜在的前体肽序列进行打分,通过该流程,他们极大地扩展了Sactipeptides 类化合物的序列多样性[78]。
不仅如此,通过对获得的蛋白文库进行序列相似性网络分析(sequence similarity networks,SSN),他们发现与QhpD 蛋白同源性较高的rSAM酶催化S—Cβ和S—Cγ的形成,不同于已知的S—Cα硫醚键。这一发现拓宽了rSAM 酶催化形成的硫醚结构多样性。Streptide是一类由链球菌产生的长度为9个氨基酸的核糖体肽,具有特征的赖氨酸Cβ和色氨酸吲哚C7 位碳碳键偶联结构,该结构由rSAM 蛋白 StrB 催化形成[79]。Streptide 的产生受到簇内shp/rgg群集感应系统的调控[80]。为了在链球菌中获得更多类似的受到群集感应系统调控的核糖体肽类天然产物,Mohammad R.Seyedsayamdost研究组对集合微生物基因组和微生物组[Ⅰntegrated Microbial Genomes and Microbiomes(ⅠMG/M) System]上的2875 个链球菌基因组进行了分析[81]。利用系统中的 Function Profile tool 进行搜索,列出所有包含Pfam04055(rSAM 蛋白)或者TⅠGR01716(Rgg/GadR/MutR family)的基因组。最终获得592 个同时包含rSAM 和Rgg 蛋白的潜在基因簇。对所有的rSAM 蛋白建立序列相似性网络,并对相应的前体肽生成序列标识图(sequence logo),以确定前体肽的保守序列区域。利用这种方法,他们获得了一系列在shp/rgg群集感应系统调控下可能产生的核糖体肽类产物。它们同样由rSAM 蛋白修饰,产生多种类型的化学键,包括碳碳键[81-82]、碳氧键[83]、碳硫键[84-85]。这一发现极大地拓展了rSAM 在核糖体肽后修饰中催化形成的结构类型及其酶学功能。
2.2 基于抗性基因的挖掘
活性导向天然产物的发现一直是高通量筛选活性化合物的重要方法。近年来,迅速发展的基因组测序技术使得天然产物的发现发生了革命性的变化。这种以基因组扫描为基础发现天然产物的策略已经成功地发现了许多新颖的代谢产物,并通过大量实验证实了这些天然产物能够极大地增加其化学结构的多样性[86]。尽管这些挖掘方法能够发现独特的生物合成酶和特异的化学物种,但在生物活性方面却没有一个明确的目标,如何利用基因组数据来预测天然产物生物活性成为基因组挖掘的一个热点。为了避免被代谢产物所误伤,微生物在产生活性天然产物的同时进化出了能够抵抗其毒性的基因,使其能够在产生防御机制的同时完整地保存自己。因此,基于抗性基因的挖掘,不仅能够发现结构多样的天然产物,而且能够预测其潜在的生物活性及其作用靶点,为新颖药物的发现提供强有力的研究基础。宿主的抗性或者自我保护机制主要包括以下几种(图5):其一,外排泵(主动运输代谢产物到细胞外);其二,对天然产物本身进行修饰从而防御其带来的伤害; 其三, 修饰宿主内部的管家酶(housekeeping enzyme)来避免天然产物的抑制作用[87-89]。
自然界用于自我保护的另一种策略是编码一个功能等价的自抗性酶(self-resistance enzyme,SRE),它是管家酶的变体。自抗性酶在序列上与管家酶高度相似,它不仅拥有管家酶的功能,同时还能抵御代谢物对宿主的伤害。SRE 往往与天然产物生物合成基因成簇存在,也与天然产物生物合成基因同时转录。因此,利用SRE 的序列相似性挖掘策略能够快速地定位细菌和真菌天然产物的生物合成基因簇[90-91]。DNA 的复制是一个基本的生命过程。然而,这种生命过程在细菌和真菌中却不尽相同。由于这一过程在原核生物中是高度保守的,因此抑制细菌中DNA 的复制就成为抗生素筛选的一个理想靶点。G.M.Savage 等从葡萄球菌中首次发现了能够抑制DNA 复制的抗生素novobiocin,研究表明该化合物的作用靶点是一个DNA 旋回酶(DNA gyrase),它属于Ⅱ型拓扑异构酶的一个亚型[92]。其生物合成研究显示,该化合物的生物合成基因簇中存在的gyrB基因编码一种对其不敏感的管家DNA 旋回酶的变种[93-94]。由于DNA 的复制在原核生物与真核生物之间的差异,寻找共同的、具有普适性的抗性基因挖掘策略成为微生物抗生素发现的关键。回溯到生物合成基因簇,参与蛋白质生物合成的酶是开发抗生素的经典靶点。在蛋白质生物合成过程中,转运RNA(tRNA)优先被20 个氨基酰化-tRNA 合成酶(aminoacyl-tRNA synthetases,aaRSs)编码的同源氨基酸进行酰化。有几个重要的天然产物以此为靶点被挖掘,如 mupirocin[95]、thiomarinol A[96]和borrelidin[97]( 图 6)。 在 这 些 天 然 抑 制 剂 中 ,mupirocin 被FDA 批准用于治疗皮肤感染性疾病脓疱疮。
许多参与脂类合成和降解的酶都是有机体所必需的,大部分天然产物的生物合成基因簇以脂肪酸生物合成路径编码的SRE 为靶标来实现自抗。来源于真菌最为著名的天然产物洛伐他汀(lovastatin),是一种被FDA 批准治疗高胆固醇的药物,它针对的是甲羟戊酸途径限速步骤中的3-羟基-3-甲基戊二酰辅酶 A 还原酶(HMGR)[98]。在土曲霉中,lovastatin 由lov生物合成基因簇编码合成,推测该化合物可能是为了对抗真菌中其他的甾醇生物合成途径而产生。在其基因簇中出现一个双拷贝的HMGR,通过实验证实该基因确实具有自抗能力[99-100]。
活性天然产物不仅是人类治疗药物的重要来源,也是许多农业药物的主要来源。支链氨基酸生物合成途径(branched-chain amino acid,BCAA)是植物生长的重要途径,它不存在于动物中,因此是高度特异性除草剂的有效靶点[101]。植物中的BCAA生物合成途径是由三种酶完成的:乙酰乳酸合成酶(acetolactate synthase,ALS)、乙酰羟基异构还原酶(acetohydroxy acid isomeroreductase,KARⅠ) 以及二羟基酸脱水酶(dihydroxy-acid dehydratase,DHAD)。DHAD 是一种重要且高度保守的植物催化酶,它催化β-脱水反应生成α-酮酸前体,进一步生成异亮氨酸、缬氨酸和亮氨酸,发展DHAD 的抑制剂成为制备除草剂的重要工业手段。为了鉴定可能编码DHAD 抑制剂的天然产物生物合成基因簇,Tang Yi 等[102]利用SRE 策略,假定其目标生物合成基因簇中包含一个对抑制剂不敏感的DHAD 拷贝,从DHAD 出发进行真菌基因组扫描,结合进化树分析等方法从土曲霉(Aspergillus terreus)中挖掘到一个与其高度同源的基因astD,对其所在的基因簇进行异源表达获得了新颖的天然产物分子,从而发现了一种天然除草剂aspterric acid(图7),并确定了其作用机制。
随着天然产物生物合成基因簇的进化,与其共簇的SRE 也会随之而进化,SRE 不仅能够为抗生素的耐药性提供新的见解,同时也为抗生素的靶点提供新的切入点。然而,从SRE 出发利用现有的知识和信息获得的天然产物,有时并不是我们期待的目标产物[103],因此,准确地预测SRE 还是目前天然产物发现过程中一个极具挑战性的工作。
2.3 基于系统进化进行基因组挖掘
天然产物的结构多样性是生物合成基因簇不断进化的结果。分子系统发育是一种常用的跟踪特定基因序列的进化足迹,并确定其与同源序列的进化关系的技术。以系统发育为导向发现新天然产物的基本思想是根据一个生物合成基因与其各自的生物合成基因簇共同进化,可以作为系统发育标志,代表其整个生物合成基因簇的进化路径,通过进化关系的远近判断天然产物的新颖程度[104](图8)。
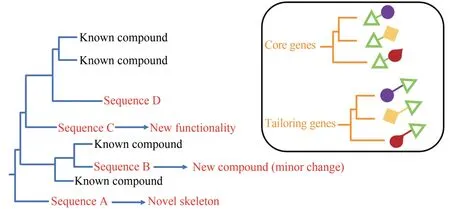
图8 利用标记基因序列建立系统发育树来指导新天然产物的发现[104]Fig.8 Phylogenetic tree built with marker gene sequences for guiding the discovery of novel natural products
利用系统进化分析挖掘天然产物最成功的案例是芳香聚酮类化合物[105-108]。芳香族聚酮是由Ⅱ型聚酮合酶(PKS)基因簇编码合成的,在Ⅱ型PKS 中最小的PKS 模块包括酮基合酶α(KSα)、酮基合酶β(KSβ)和酰基载体蛋白(ACP)[106]。这三个基因参与了芳香族聚酮生物合成过程的第一步,通过催化丙二酰辅酶A(malonyl-CoA)单元的重复缩合产生不同长度的线性聚酮链(图9)。这些最小的PKS 基因可能与它们各自的生物合成基因簇共同进化,因此可以作为系统发育标记。
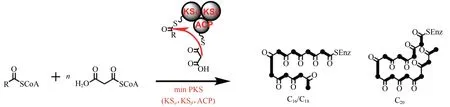
图9 最小化的PKS参与不同长度的线性聚酮链的合成Fig.9 minPKS involved in the synthesis of diverse linear polyketide chains
蒽醌类化合物(anthracyclines)是一类具有抗肿瘤活性的天然产物[109],其中具有代表性的多柔比星(doxorubicin)已用于临床抗癌化疗超过30 年[110]。在系统发育分析中,宏基因组 DNA 衍生的扩增子序列AZ129 与已知的蒽醌类化合物斯特菲霉素(steffimycin)生物合成基因簇的序列形成一个紧密的分支,Brady 等[111]利用 AZ129 扩增子序列作为探针从宏基因组中重新获得的AZ129基因簇的信息学分析表明,与斯特菲霉素生物合成基因簇相比,还存在一组额外的糖生物合成基因。在白色链霉菌(Streptomyces albus)中对AZ129 基因簇进行异源表达获得一个全新的天然产物arimetamycin A(图10),在体外肿瘤细胞抗增殖实验中,arimetamycin A 表现出比多柔比星更强的活性,并且对多柔比星耐药的癌细胞也表现出中等的抗肿瘤活性[111]。
系统进化分析除了利用上述编码聚酮合成酶这类骨架形成相关的基因作为标签,还可以利用前体供应基因、编码后修饰蛋白的基因、抗性基因等特征基因作为标签,通过进化关系将其与相应的代谢产物关联,指导新结构、新活性天然产物的发现。
3 展 望
大自然从数十亿年前就开始以“自然实验师”的身份进行生物工程实验。为了探究自然界神秘的面纱,人们开发了许多基于基因组与宏基因组的策略来剖析生物进化过程中涉及的途径,并发现了许多新的药物和高效的生物催化剂(酶),同时解析了这些新的反应机制。天然产物及其衍生物一直都是药物先导化合物的重要来源。在天然产物的获取途径中,传统的分离分析方法无法避免重复性、滞后性等问题,这不仅耗时耗力而且无法突破代谢产物“黑箱子”的魔咒。随着基因组测序技术的快速发展,以基因组学为导向的天然产物发现已经成为药物研究领域的重要组成部分。尽管持续更新的基因组数据为天然产物的研究和开发提供了源源不断的资源,然而,如何利用现有的实验条件和技术进行天然产物的挖掘还是一项极具挑战的任务。就微生物领域而言,目前所报道的微生物种群和基因组数据远远超过已知代谢产物的数量,换言之,还有数以万计的“沉默”基因簇等待着开发和利用。以数据为研究中心的方法正在从根本上改变自然科学的许多领域[112],多组学技术、系统生物学方法与合成生物学理论的联合使用推动着基因组、转录组和代谢组数据的自动化高通量分析,从而更有效地将基因与有机分子连接起来。基于这些理论的结合使用以及网络工具的更新与发展,许多新颖的挖掘技术被开发出来,研究方法已经从传统上基于活性导向天然产物的发现,转向基于核心骨架基因、基于抗性基因以及基于系统进化的基因组挖掘,通过对化学结构、基因组和代谢组学等数据的集成为我们提供了数据的优先排序。这样,基因组的挖掘不仅能发现“终点”药物分子,而且对多个研究领域的发展也起到了非常关键的承接作用。同时,参与次级代谢的酶催化各种各样的反应,这些反应可以在合成生物学中进化和利用。天然产物本身在介导微生物-微生物相互作用、宿主-微生物相互作用以及影响疾病、生长发育等方面都发挥着重要作用。许多合成化学家通过合成结构复杂、活性显著的有机小分子从而开发了许多高效、绿色环保的合成路线,加快了药物合成的步伐。生物学家通过研究生命体内包括转录、调控以及相应的酶学机制,阐明了生命传承过程中许多重要的途径。天然产物研究改革与创新正在创建一种多领域多学科交叉的研究模式,这种方式汇聚了多种学习方法、理论基础以及实时更新的网络信息学技术。如今,随着科技的快速发展,人工智能(artificial intelligence)在各个领域都开始崭露头角,在科技时代如何把握技术的更新和运用将成为基因组挖掘研究领域发展的一大挑战。

