豇豆品种资源的InDel分子标记分析
康研,陈珍珠,赵媛,何思晓,马雪洁,刘青青,陈禅友,潘磊
(江汉大学生命科学学院;湖北省豆类(蔬菜)植物工程技术研究中心;湖北省食用豆类植物自然科技资源中心,湖北武汉 430056)
0 引言
豇豆[Vigna unguiculata(L.)Walp.]为豆科豇豆属草本植物。作为一种重要豆类作物,豇豆耐高温干旱,在世界范围内种植广泛,包括非洲、亚洲、欧洲、美国南部地区等。豇豆是我国的优势豆类蔬菜作物之一,嫩荚和种子均可食用。因其营养丰富,既可鲜食,又适合加工,深受消费者青睐。由于播种季节长,生产周期短,对于我国8-9月夏秋蔬菜淡季市场的蔬菜供应具有重要作用。
采用DNA分子标记技术研究豇豆种质资源的遗传变异是当前关注的研究热点之一。利用显性分子标记技术如RAPD(random amplified polymorphic DNA)、ISSR(inter-simple sequence repeat)等在豇豆的研究中,已经筛选获得一批重复性好、稳定可靠的分子标记引物[1-2]。而在豇豆的共显性分子标记研究方面,如SSR(simple sequence repeat)和SNP(single nucleotide polymor⁃phism)等分子标记的发掘则报道愈来愈多。从长豇豆基因组数据库HarvEST和CGKB(http:∥cowpeagenomics.med.virginia.edu/CGKB)中发掘出了172个多态性豇豆SSR分子标记,包括45个EST-SSR标记和127个SSR标记[3-4]。有研究表明采用SNP标记可定位一批与豇豆驯化相关的基因,包括荚长、花色、果柄长度、种皮颜色等[5-6]。目前研究所揭示的豇豆种质资源的遗传变异有限,豇豆的分子生物学研究还比较滞后,尤其是在长豇豆的DNA分子标记研究方面还比较薄弱,亟待加强。
插入与缺失(InDel,insertion/deletion)标记是一种共显性分子标记,在基因组上分布广泛,密度大,适宜于进行全基因组分子标记的发掘。InDel标记可以通过凝胶电泳中的条带大小差异来确定不同的基因型。随着测序技术的发展以及各个物种参考基因组的公布,其InDel标记开发的成本和难度也随之降低,因此InDel标记是近年来一种较为热门的DNA分子标记技术,广泛应用于种质资源分析与分子辅助遗传育种、群体遗传分析、图位克隆、基因定位及遗传图谱的构建等研究[7-8]。李斯更等[9]基于黄瓜基因组重测序结果设计了全基因组分布的代表性InDel引物134对,其中具有多态性的引物116对,占引物总数的86.6%,116对引物充分揭示出16份种质的多样性和特异性。吉康娜等[10]通过茄子的全基因重测序数据,开发出具有多态性的62个InDel标记,基于这62个InDel标记聚类分析结果揭示出24份紫红长茄自交系的遗传多样性,并对茄子品种纯度进行鉴定,其结果与田间鉴定结果相吻合。
豇豆中InDel分子标记的研究鲜见报道。本研究拟采用InDel分子标记技术,检测豇豆种质资源的遗传变异,聚类分析其亲缘关系,尝试构建豇豆InDel分子指纹图谱,以期为豇豆的分子评价、鉴定、保存和新品种选育等提供分子生物学证据。
1 材料与方法
1.1 实验材料
本研究中采用来自国内外的24份豇豆品种材料,其中来自中国的有21份,美国的有2份和尼日利亚1份(表1)。

表1 24份豇豆品种材料的信息Tab.1 Information of 24 cowpea varieties
1.2 实验方法
1.2.1 豇豆基因组总DNA的提取与检测选取鲜嫩的豇豆叶片,加液氮将叶片迅速冷冻,并研磨成粉末,采用改良的CTAB法[11],提取植物基因组总DNA。采用1%的琼脂糖凝胶电泳,以LambdaDNA/HindIIIMarker为参照,检测基因组总DNA的质量和浓度。之后,将不同样品材料的总DNA稀释到30~50 ng/μL,置于-20℃保存备用。
1.2.2 引物的筛选和PCR扩增本研究所用的3个InDel分子标记引物序列由本实验室自主设计。由生物工程(上海)股份有限公司合成引物,引物信息见表2。

表2 3个InDel分子标记信息Tab.2 Information of 3 InDel molecular markers
1.2.3 PCR扩增体系InDel-PCR扩增中,PCR反应总体系为20μL:正反引物各1μL(10 ng/μL),Mix Buffer 10μL(含TaqDNA聚合酶),ddH2O 7μL,模板DNA 1μL(30~50 ng/μL)。
PCR反应在Eppendorf扩增仪上进行,扩增条件:95℃预变性5 min,然后95℃变性30 s,60℃退火30 s,72℃延伸30 s,35个循环;最后72℃延伸10 min。扩增完毕,取出放入冰箱,保存于4℃备用。
1.2.4 InDel-PCR扩增及检测PCR扩增产物用35%的琼脂糖凝胶电泳检测。在100 V恒压电泳条件下,电泳30~45 min,置于全自动凝胶成像分析系统上拍照保存。
1.3 数据处理
拍照记录凝胶电泳的DNA图谱。对于不同豇豆DNA样品在琼脂糖凝胶电泳统计时按照有条带为1,无条带为0的形式记录,录入Excel表格中生成0/1矩阵形式。用NTSYS pc2.1软件对豇豆InDel分子标记的0/1矩阵数据进行分子指纹图谱构建和UPGMA聚类分析。
2 结果与分析
2.1 引物扩增条带多态性结果分析
本研究利用3对InDel引物(VuFC30、VuFC33和VuFC34),共检测出6条多态性条带,多态性位点比率为100%,平均每个引物扩增2条多态性条带(见图1)。

图1 InDel引物VuFC30(A)、VuFC33(B)和VuFC34(C)检测24份豇豆材料的多态性电泳图谱Fig.1 Polymorphism electrophoresis patterns of 24 cowpea cultivars detected by the InDel primers VuFC30(A)、VuFC33(B)and VuFC34(C)
InDel引物VuFC30检测到2条多态性条带,大小分别为170 bp和300 bp。其中条带大小为170 bp的材料有20份,分别为X1、X2、X3、X4、X5、X6、X7、X8、X9、X10、X12、X13、X14、X17、X18、X19、X20、X21、X22和X23;条带大小为300 bp的材料有2份(X15和X24);X11和X16无条带。
InDel引物VuFC33检测到2条多态性条带,大小分别为250 bp和320 bp。其中条带大小为250 bp的 材料有7份,分 别为X1、X7、X10、X11、X12、X15和X19;条带大小 为320 bp的材料 有17份,分别为X2、X3、X4、X5、X6、X8、X9、X13、X14、X16、X17、X18、X20、X21、X22、X23和X24。
InDel引物VuFC34检测到2条多态性条带,大小分别为210 bp和250 bp。其中条带大小为210 bp的材料有17份,分别为X2、X3、X4、X5、X6、X8、X9、X10、X11、X13、X14、X17、X18、X19、X21、X22和X23;条带大小为250 bp的材料有6份,分别为X1、X7、X12、X15、X20和X24;X16无条带。
2.2 24个豇豆品种材料的聚类分析
遗传相似性系数反映群体间的亲缘关系,遗传距离常用来表示样本间遗传分化的程度。供试的24份豇豆品种材料的Jaccard相似性系数变化范围为0.20~1.00之间(图2),说明其遗传背景存在较大差异。
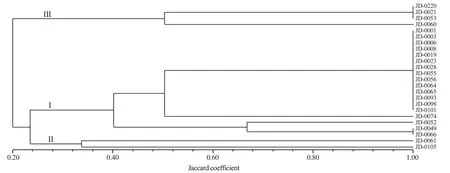
图2 24份豇豆品种材料的UPGMA聚类图Fig.2 UPGMA cluster of the 24 cowpea cultivars
利用聚类分析可以明确材料间的遗传差异,有助于合理选择亲本,充分利用种质资源。聚类分析将24份材料大致分为3个亚群:亚群Ⅰ:共18份,豇豆样品来源于湖北7份、辽宁3份、江苏2份、北京1份、江西1份、山东1份、湖南1份和美国2份;亚群Ⅱ:共2份,豇豆样品来源于辽宁1份和宁夏1份;亚群Ⅲ:共4份,豇豆样品来源于湖北1份、辽宁2份和尼日利亚1份。从UPGMA聚类结果来看,聚类结果与其来源地之间没有明显的相关性,但是豇豆资源之间还是存在一定的遗传分化。3份来自国外的豇豆品种材料之间能被区分开,说明供试的3份国外豇豆资源之间存在遗传分化。3份国外豇豆材料与部分国内豇豆品种无法区分。比如,两份外国材料(JD-0049和JD-0053)中,JD-0053与亚群Ⅰ中其余2份国内豇豆材料无法区分,与我国的部分豇豆材料尚未能完全区分;JD-0049与亚群Ⅱ中的JD-0066尚无法区分。在21份国内豇豆材料中,有14份豇豆资源聚类在一个分支中无法区分,其原因可能是这些豇豆品种的遗传背景相似,由于环境或人为因素使国内不同地区的豇豆种质间存在一定的基因流,也可能是3个InDel标记检测的位点太少所致。
2.3 豇豆品种材料InDel分子指纹图谱构建
供试的24份豇豆品种材料中,3个InDel分子标记共检测到8种类型的InDel分子指纹图谱(表3)。InDel指纹类型1为010110,属于此类型的有3份豇豆材料JD-0220、JD-0021和JD-0053。InDel指纹类型2为100110,有1份(JD-0060)豇豆材料属于此类型。InDel指纹类型3为011001,有14份 豇 豆 材 料 属 于 此 类 型,包 括JD-0001、JD-0003、JD-0006、JD-0008、JD-0019、JD-0023、JD-0028、JD-0055、JD-0056、JD-0064、JD-0065、JD-0093、JD-0098和JD-0101。InDel指 纹 类 型4为011010,包 含1份(JD-0074)豇 豆 材 料。InDel指 纹 类 型5为000101,包含1份(JD-0052)豇豆材料。InDel指纹类型6为010101,包含2份豇豆材料JD-0049和JD-0066。InDel指纹类型7为001000,包含1份(JD-0061)豇豆材料。InDel指纹类型8为101010,包含1份(JD-0105)豇豆材料。值得注意的是,在这8种InDel指纹类型中,有5份豇豆材料(JD-0052、JD-0060、JD-0061、JD-0074、JD-0105)检测到其独有的InDel指纹类型,可以与其他19份豇豆材料区分开。因此,这3个InDel分子标记能用于豇豆品种资源的DNA分子指纹鉴定。
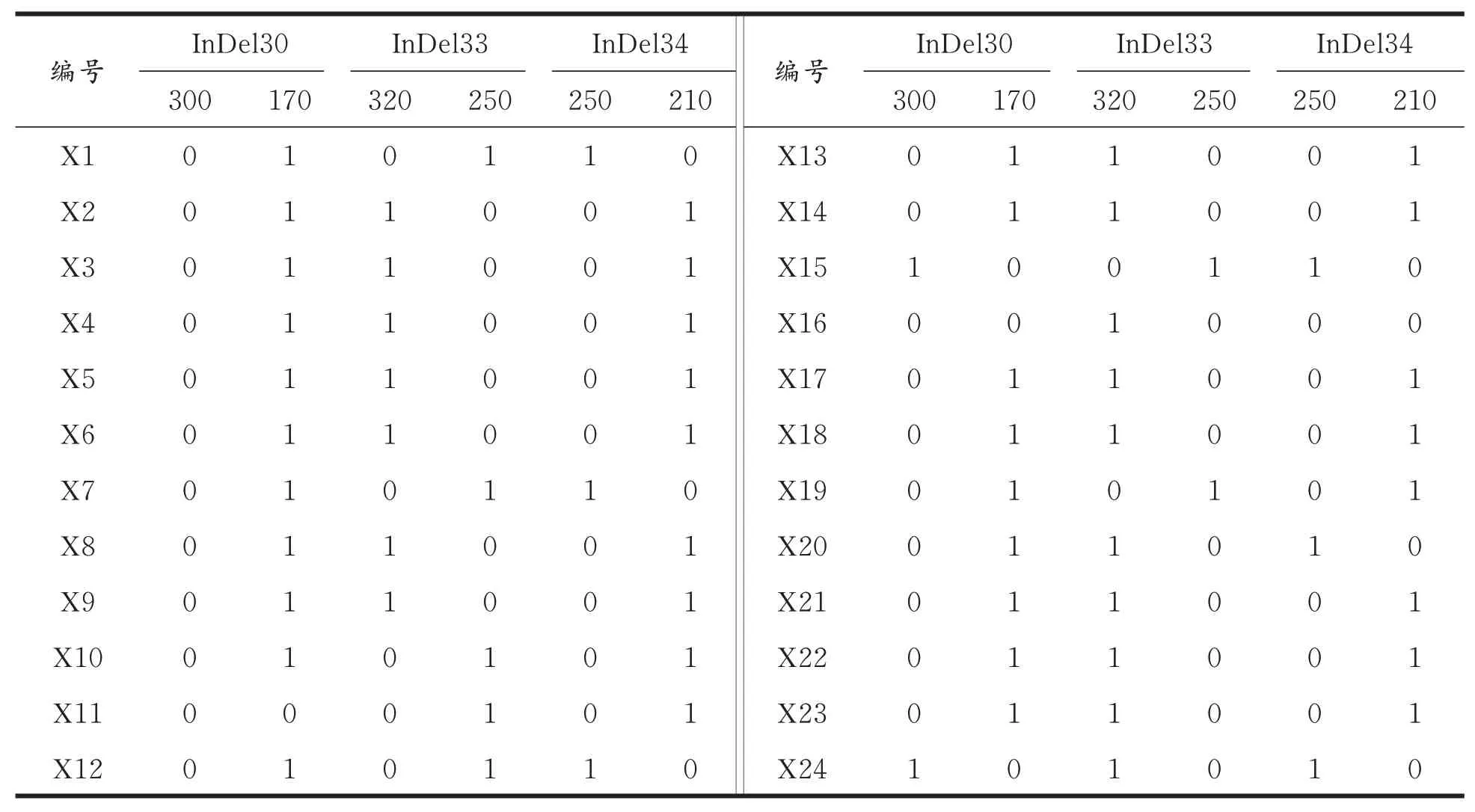
表3 24份豇豆品种材料的InDel分子指纹图谱类型Tab.3 The fingerprint types of the InDel markers of the 24 cowpea cultivars /bp
3 讨论
InDel标记为共显性标记,在整个基因组中,其分布频率高,且变异稳定性强[13]。随着DNA测序技术的进步,测序的时间成本和经济成本逐渐降低,基于高通量重测序技术的InDel标记开发及应用发展迅速,已经在作物研究中大量报道,包括大豆[14]、水稻[15]、番茄[16]、黄瓜[17]等。Adedze等[18]基于黄瓜的重测序数据,利用GATK等软件从10 470个InDel标记筛选出插入缺失片段>30 bp的InDel标记385个,使用琼脂糖凝胶电泳检测出211个具有多态性的InDel标记,利用这些标记成功地将48个黄瓜选育品系聚类为水果和普通黄瓜两大类。但是,豇豆中的InDel研究鲜见报道。
本研究以从国内外收集的24份豇豆种质资源为试验材料,采用InDel分子标记技术,进行了豇豆种质资源的亲缘关系和分子指纹图谱研究。结果表明:相似性系数在0.20~1.00之间,揭示其遗传变异较大,研究结果有利于豇豆的遗传多样性分析和遗传图谱构建等应用研究;UPGMA聚类分析可将24份豇豆材料大致分为3个亚群,结果表明豇豆品种资源与其地理来源之间没有明显的相关性,但是能反映出部分豇豆资源之间的遗传分化;对于本次供试的24个材料,使用3个InDel标记检测出8种类型的指纹图谱,由于使用的InDel标记较少无法将全部品种区分,但可为以后使用琼脂糖凝胶电泳大规模开发豇豆InDel标记提供基础。由于传统的InDel标记检测技术使用聚丙烯酰胺凝胶电泳,其效率较低、实验过程复杂、试剂安全性差,导致其检测成本升高。本研究中的InDel标记带型易读简单,琼脂糖凝胶电泳操作更为简单快捷,更适合品种遗传多样性分析及指纹图谱构建。对于构建指纹图谱,由于InDel标记的条带清晰且数量少,即可使用引物组合法去鉴别相应的品种,引物组合法通过不同引物的有限组合,可以大大提高引物的鉴别能力。
综上,本研究结果可为今后豇豆杂交选育及杂交种间遗传多样性研究提供分子证据,可为后续开展优良品种提供有效依据,对豇豆种质资源的开发与利用有着重要意义。

