转录组测序分析氯化钠对普鲁兰生物合成的影响
张高川,何超永,王崇龙,王大慧*,卫功元*
(苏州大学基础医学与生物科学学院,江苏 苏州 215123)
普鲁兰是一种由出芽短梗霉(Aureobasidium pullulans)合成的水溶性直链高分子物质[1]。作为一种微生物胞外多糖,普鲁兰本身安全无毒,同时具有良好的可塑性、成膜性和稳定性,以及耐热、耐酸碱、耐盐等特性,因而可以广泛地应用于食品加工、临床医药、环境保护和轻工化工等诸多领域[2-4]。美国食品药品监督管理局已经将普鲁兰认证为GRAS(Generally Recognized As Safe)微生物多糖,我国也于2006年将普鲁兰列为新增的4 种食品添加剂之一[5]。此后,普鲁兰在市场上的需求量日益增长。
在普鲁兰的发酵生产中,普鲁兰产量和分子质量是两个重要的过程参数,前者决定普鲁兰生产的得率和效率,而后者关系到普鲁兰的应用范围[6]。影响普鲁兰产量和分子质量的因素有很多,既包括培养基中各种营养成分及其配比,又涉及生产菌株培养的环境条件[7-8]。其中,培养基中无机盐及其含量在普鲁兰的生产中往往具有重要的作用。Badr-Eldin等[9]发现发酵培养基中磷酸盐水平偏低不利于菌体生长,偏高则抑制普鲁兰合成。Reeslev等[10]发现当培养基中Zn2+浓度高于0.45 μmol/L时,出芽短梗霉的形态即从酵母状细胞向菌丝状转变,普鲁兰的合成随之受到不利影响[6]。鞠宝等[11]分析了二价阳离子在普鲁兰和黑色素形成中的作用,结果表明合适的金属离子种类和浓度抑制了黑色素的形成,促进了普鲁兰的合成。Gao Wa等[12]在单因素试验的基础上,采用正交试验优化了培养基中无机盐含量,提高了普鲁兰产量。与此同时,还发现影响出芽短梗霉细胞生长的最显著因素是磷酸氢二钾,而氯化钠(NaCl)则是影响普鲁兰合成的最显著因素[13]。以上结果均是基于发酵过程优化而得到的,这些研究并没有对无机盐影响普鲁兰生物合成的内在机制进行深入分析。
在前期优化普鲁兰发酵培养基的研究中,发现培养基中NaCl的存在有利于提高普鲁兰产量,但却显著降低了普鲁兰的分子质量[6]。通过对普鲁兰合成关键酶(α-磷酸葡萄糖异构酶、尿苷二磷酸葡萄糖焦磷酸化酶、葡萄糖基转移酶)的活性、物质和能量代谢进行测定,发现NaCl同时提高了与普鲁兰生物合成和降解相关酶的活性,增加了能量物质ATP的供给,结果部分揭示了NaCl在提高普鲁兰产量的同时却降低了普鲁兰分子质量的生理机制[14]。尽管如此,NaCl如何在分子水平影响出芽短梗霉胞内相关基因的表达,并体现在普鲁兰的产量和分子质量上,这些问题至今仍然不得而知。
转录组测序(RNA-Seq)技术是通过对细胞转录产物进行测序,统计测得的每条序列,获得每个特定转录本的表达量,可更加精确地评估细胞表型,加深对细胞代谢的理解,也有助于对目标基因进行改良[15]。近年来,RNA-Seq因检测动态范围宽、数据重复性好等优势,逐渐成为研究基因表达差异、新基因挖掘与功能注释的重要手段[16]。RNA-Seq技术和生物信息学分析手段已经广泛运用于微生物过程分析中,为深入理解微生物现象中蕴含的分子机制提供了可信的证据[17-20]。为此,本实验研究NaCl对出芽短梗霉细胞转录组的影响,采用生物信息学方法筛选与分析差异表达基因(differentially expressed genes,DEGs),并对这些DEGs进行注释与富集分析。
1 材料与方法
1.1 材料与试剂
出芽短梗霉CCTCC M 2012259,由苏州大学微生物生理与代谢调控研究室保藏。
种子培养基:马铃薯200 g/L,葡萄糖20 g/L,自然pH值;发酵培养基:葡萄糖50 g/L,酵母粉3 g/L,(NH4)2SO40.6 g/L,K2HPO45 g/L,MgSO4·7H2O 0.2 g/L,pH 3.8,NaCl质量浓度根据具体实验设计而定。
所有试剂均为国产分析纯,购自生工生物工程(上海)股份有限公司。
1.2 仪器与设备
Minifors实验室台式5 L发酵罐 瑞士Infors公司;HZ-2010K恒温摇瓶柜 太仓市华利达实验仪器设备有限公司;J6-MI型冷冻离心机 美国Beckman公司;T6新世纪紫外-可见分光光度计 北京普析通用仪器有限责任公司;LDZX-50KBS型立式压力蒸汽灭菌器 上海申安医疗器械厂;1836稀释型乌式毛细管黏度计 浙江台州市椒江玻璃仪器厂;2100生物分析仪 美国安捷伦科技公司;Qubit 2.0荧光分光光度计 美国Invitrogen公司。
1.3 方法
1.3.1 种子培养
将在-70 ℃超低温冰箱中保藏的菌种(1 mL)接种至50 mL种子培养基中,于30 ℃、200 r/min摇床中培养24 h。
1.3.2 发酵培养
摇瓶发酵培养:以10%(V/V)的接种量,将种子液接种至装有50 mL发酵培养基的500 mL三角瓶中,在200 r/min摇床中培养72 h,温度30 ℃。
分批发酵培养:以10%(V/V)的接种量,将种子液接种至装有3 L发酵培养基的5 L发酵罐中,温度30 ℃,搅拌转速400 r/min,通气量3 L/min。pH值采用梅特勒电极在位监测,通过自动流加3 mol/L H2SO4或3 mol/L NaOH溶液控制pH值为3.80±0.02。
1.3.3 指标测定
细胞干质量和普鲁兰产量的测定方法参见文献[5];普鲁兰分子质量的测定采用黏度法[6];葡萄糖质量浓度的测定采用3,5-二硝基水杨酸法[21];分批发酵过程参数的计算方法参见文献[8]。
1.3.4 出芽短梗霉RNA-Seq
1.3.4.1 RNA提取
将分批发酵培养至36 h的出芽短梗霉细胞8 000 r/min、4 ℃离心10 min,液氮猝灭2 min;利用TRIzol试剂盒提取总RNA。采用Agilent 2100生物分析仪、NanoDrop和1%琼脂糖凝胶对总RNA进行品质和定量分析。选择1 μg RNA完整值(RNA integrity number,RIN)大于7的总RNA用于后续文库制备。
1.3.4.2 cDNA文库构建
根据NEBNext®UltraTMRNA文库制备试剂盒的操作步骤进行测序文库制备。首先使用NEBNext Poly(A) mRNA Magnetic Isolation Module从总RNA中分离Poly(A) mRNA,然后利用NEBNext First Strand Synthesis Reaction Buffer和NEBNext Random Primers对mRNA进行破碎(约300 bp)和引物配对。以此为模板,利用ProtoScript II逆转录酶合成第1条cDNA链,利用Second Strand Synthesis Enzyme Mix合成第2条cDNA链。之后用AxyPrep Mag PCR Clean-up纯化双链cDNA产物,使用Agilent 2100生物分析仪评估其品质,使用Qubit 2.0荧光分光光度计对其进行定量。最后使用End Prep Enzyme Mix修复纯化后的双链cDNA产物,添加dA尾巴,并通过T-A连接在其末端添加接头。
1.3.4.3 RNA-Seq数据分析
利用Illumina HiSeqTM2000对上述构建的cDNA文库进行150 bp读长的双末端测序,获得的RNA-Seq数据经FASTQC(http://www.bioinformatics.babraham.ac.uk/projects/fastqc/)质控分析后,使用Trimmomatic(v0.30)软件删除测序质量偏低的末端序列片段[22],处理后的干净读段(clean reads)用于后续分析。将RNA-Seq数据上传至NCBI数据库,登记号为PRJNA350822(https://dataview.ncbi.nlm.nih.gov/object/PRJNA350822)。
虽然已经对出芽短梗霉全基因组进行测序[23],但缺少完善的注释信息,所以RNA-Seq数据分析采用基于Trinity(v2.6.6)从头组装的无参分析流程[24]。利用Trinity软件对RNA-Seq数据进行从头组装,利用BLASTX(2.7.1+)程序[25],将Trinity组装获得的所有转录本与来自UniprotKB数据库(https://www.uniprot.org/)的34 084 个真菌已知蛋白进行比对(截止日期2019-05-14),鉴别这些组装转录本可能编码的蛋白(统计期望阈值为1×10-5)。
使用RSEM(v1.3.1)软件的RSEM-prepare-reference程序,根据Trinity组装结果创建参考转录组;使用RSEM-calculate-expression工具计算Trinity组装转录本在每个样本中的表达水平;利用RSEM-generate-datamatrix程序,将所有样本的基因表达数据合并为一个数据矩阵[26],用于DEGs分析。
1.3.4.4 DEGs的鉴别
利用R语言包edgeR(v3.20.9)对上述基因表达水平数据矩阵进行计算分析[27],鉴别因培养基中添加NaCl而产生的DEGs。将基因差异表达倍数(fold change,FC)阈值设为2 倍,统计显著性阈值设为0.05,作为DEGs的筛选标准[28]。对筛选结果进行可视化分析,绘制火山图。利用R语言包pheatmap(v1.0)对DEGs进行聚类和可视化分析,用来评估平行样本的一致性。
1.3.4.5 DEGs的功能注释与富集分析
利用UniProt Retrieve/ID mapping tool(http://www.uniprot.org/uploadlists)在线工具,从Uniprot数据库中获取所有DEGs对应蛋白质的基因本体论(Gene Ontology,GO)注释信息和序列信息。利用WEGO(http://wego.genomics.org.cn/)对其进行GO注释的富集分析[29],使用R语言包ggplot2对富集结果进行可视化。利用京都基因和基因组百科全书(Kyoto Encyclopedia of Genes and Genome,KEGG)数据库的Blast-KOALA工具(https://www.kegg.jp/blastkoala),分别对上调和下调的DEGs进行通路富集分析[30],并利用KEGG mapper工具对其中的通路模块进行深入挖掘。
1.4 数据统计
RNA-Seq设置2 个平行样品,其他所有实验数据均来自于3 组平行样品,应用SPSS 17.0软件对数据进行t检验分析,P<0.05,差异显著。
2 结果与分析
2.1 NaCl对普鲁兰生物合成的影响
在摇瓶发酵条件下,考察发酵培养基中NaCl质量浓度对出芽短梗霉细胞生长和普鲁兰合成的影响,结果见图1。可以看出,与对照(0 g/L NaCl)相比,NaCl的添加提高了普鲁兰产量,但降低了细胞干质量和普鲁兰分子质量。当NaCl质量浓度为3 g/L时,普鲁兰产量达到最大值23.17 g/L,比对照提高了22.9%;而细胞干质量和普鲁兰分子质量在此质量浓度下均处于最低水平,分别比对照降低了12.7%和44.9%。以上结果表明,培养基中NaCl的存在有利于普鲁兰产量的提高,但不利于将普鲁兰分子质量维持在较高的水平。
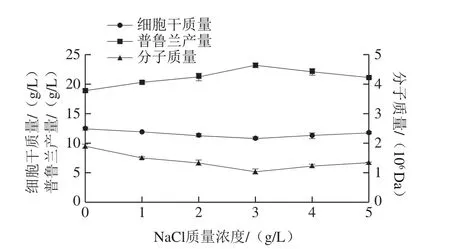
图1 NaCl质量浓度对普鲁兰生物合成的影响Fig.1 Effect of NaCl at different concentrations on pullulan biosynthesis
2.2 普鲁兰分批发酵过程参数比较
在5 L发酵罐中对出芽短梗霉CCTCC M 2012259进行分批培养,检测了3 g/L NaCl和对照条件下的葡萄糖消耗、细胞生长和普鲁兰合成情况,结果见图2和表1。通过对各个发酵过程参数进行比较可以发现,NaCl加快了葡萄糖的消耗速率,但却降低了细胞生长的速率。因此,当培养基中含有NaCl时,细胞并没有将多消耗的葡萄糖用于细胞生长,而是用于普鲁兰的合成,最终细胞干质量比对照降低了16.5%,普鲁兰产量却比对照提高17.6%。需要特别指出的是,在NaCl存在的情况下,普鲁兰最终分子质量显著下降55.8%。综合表1中分批发酵其他过程参数,可以得出结论,与对照相比,3 g/L NaCl显著提高了普鲁兰产量,却同时显著降低了细胞干质量和普鲁兰分子质量。以下将采用RNA-Seq技术和生物信息学分析手段对NaCl影响普鲁兰生物合成的分子机制进行解析。
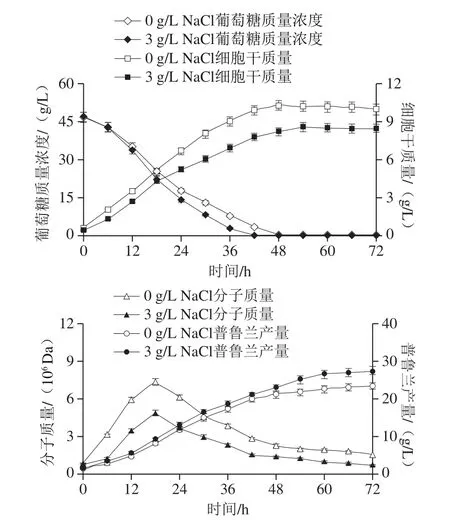
图2 不同NaCl质量浓度下的普鲁兰分批发酵过程Fig.2 Time-course curve of pullulan production in batch fermentation at different concentrations of NaCl

表1 普鲁兰分批发酵过程参数比较Table 1 Comparison of cell growth and pullulan production in batch fermentation in the presence and absence of NaCl
2.3 RNA-Seq分析
2.3.1 RNA-Seq数据分析
对来自0 g/L和3 g/L NaCl培养条件下的出芽短梗霉细胞样本进行RNA-Seq,共获得近9 240万 条读段(reads),总长度约27.72 G碱基。4 个转录组样本的Q30(0.1%碱基识别错误率)均在93%左右(表2),表明这些RNA-Seq数据具有较高的质量,能够满足后续生物信息学分析的要求。通过FASTQC质控分析和trimmomatic处理后,使用Trinity(v2.6.6)对这些测序结果进行从头组装,共获得16 204 个基因和29 523 条转录本。通过与真菌已知蛋白进行比对,BLASTX程序成功鉴别出其中的16 130 条转录本。
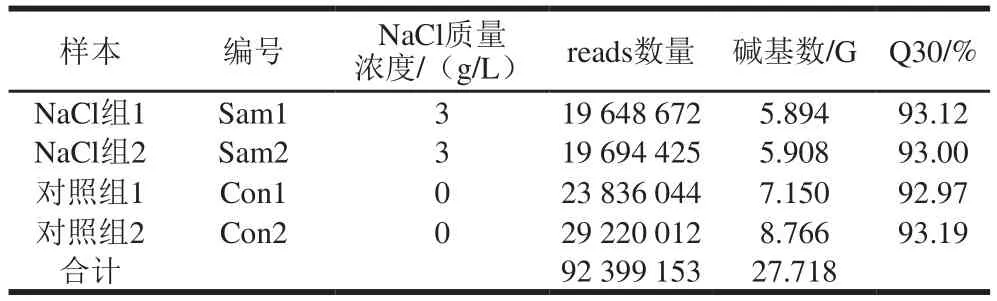
表2 RNA-Seq数据Table 2 Overview of RNA-Seq data for A.pullulans
2.3.2 DEGs分析
利用edgeR对RSEM软件计算获得的Trinity组装转录本表达水平数据矩阵进行筛选,除去低表达基因后,在剩下的14 098 个转录本中鉴别DEGs。将统计显著性阈值(P值)设为0.05,则有659 个DEGs被鉴别出,DEGs分析的火山图见图3A,其中227 个DEGs表达水平在NaCl存在时显著上调,432 个DEGs表达水平显著下调,表明培养基中的NaCl对出芽短梗霉的基因转录水平产生了显著影响。利用pheatmap对这些DEGs进行聚类分析(图3B),结果显示,该RNA-Seq数据分析结果可用于后续的基因功能注释和代谢通路分析。
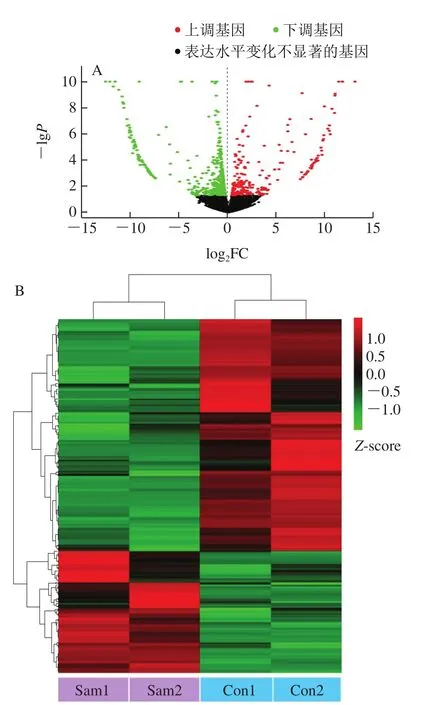
图3 NaCl存在时DEGs分析可视化图谱Fig.3 Volcano map and heatmap of DEGs in the presence of NaCl
2.3.3 DEGs功能注释和富集分析
利用WEGO对在NaCl组中表达水平显著上调的227 个基因和显著下调的432 个基因分别进行GO功能注释及富集分析,共有169 个上调基因和360 个下调基因获得GO功能注释信息。利用R语言包ggplot2对富集结果进行可视化分析,结果见图4。可以看出,在NaCl组中显著上调的基因,主要富集在初级代谢、氮化合物代谢、生物合成等生物学过程,具有离子绑定、水解酶活性、小分子绑定等分子功能,且大多在胞内细胞器尤其是具有膜结构的细胞器中发挥作用;在NaCl组中显著下调的基因,其富集的生物学过程、分子功能和细胞组分均与上调基因相似。由此可见,培养基中NaCl的存在,影响了细胞中的一些水解酶,以及具有离子和小分子绑定功能的蛋白编码基因的表达水平,这些均可能与普鲁兰产量和分子质量的改变有关。

图4 NaCl存在时DEGs的GO功能注释及富集分析Fig.4 GO functional annotation and enrichment analysis of DEGs in the presence of NaCl
2.3.4 DEGs的KEGG代谢通路分析
使用KEGG的Blast-KOALA工具对NaCl组中表达水平显著上调的227 个基因和下调的432 个基因进行代谢通路分析,分别有109 个上调基因和224 个下调基因被KEGG注释(表3)。结果表明,上调基因主要富集于遗传信息过程、碳水化合物代谢、氨基酸代谢等相关通路,而下调基因则主要富集于碳水化合物代谢、遗传信息过程、信号与细胞过程等相关通路。在此基础上,利用KEGG mapper工具对其中的通路模块进行深入挖掘,发现分别有58 个上调基因和112 个下调基因编码各类酶(表4),其中,上调基因编码的酶参与的代谢通路主要包括次级代谢物生物合成、抗生素生物合成、不同环境下的微生物代谢、淀粉和蔗糖代谢、糖酵解/糖异生等代谢过程,而下调基因编码的酶参与的代谢过程除了上述通路以外,还参与了碳代谢、氨基酸生物合成、丙酮酸代谢、氧化磷酸化等代谢过程。

表3 DEGs的KEGG通路富集分析Table 3 KEGG pathway enrichment analysis of DEGs

表4 DEGs的KEGG通路功能挖掘Table 4 KEGG pathway functional annotation of DEGs
需要特别说明的是,直接参与普鲁兰生物合成的关键酶基因(pgm1、pgm2、ugp、fks)和降解酶基因(amy1)在本研究中均能被注释到,其表达水平与前期研究[14]中real-time PCR所得到的结果一致。虽然这些基因的转录水平在NaCl存在时均有所上调,但基因FC均小于2,因此不在鉴别出的659 个DEGs范围之内。尽管如此,针对RNA-Seq数据的分析结果,仍然可以为今后出芽短梗霉的遗传改造提供可信的理论依据。
3 结 论
本研究考察NaCl在普鲁兰生物合成中的作用,发现3 g/L NaCl最有利于普鲁兰的过量合成,但培养基中NaCl的存在显著降低了普鲁兰的分子质量。在分批发酵的基础上,采用生物信息学方法对出芽短梗霉RNA-Seq数据进行分析,共鉴别出659 个DEGs,其中NaCl组中有227 个基因表达上调,432 个基因表达下调。通过对这些DEGs进行GO功能注释和KEGG通路富集分析,发现NaCl影响了参与碳水化合物代谢、淀粉和蔗糖代谢、糖酵解/糖异生等代谢过程的基因的表达水平,最终在提高了普鲁兰产量的同时降低了普鲁兰分子质量。研究结果有助于深入理解NaCl在普鲁兰生物合成中的作用机制,同时也为出芽短梗霉遗传改造以实现高分子质量普鲁兰的高效合成提供理论依据。

