广东省蛭态轮虫物种多样性及其系统发育研究
王 庆,李 莹,汪文博,杨宇峰
广东省蛭态轮虫物种多样性及其系统发育研究
王 庆,李 莹,汪文博,杨宇峰*
(暨南大学生态学系,广东 广州 510632)
为了解广东省蛭态轮虫物种多样性,采集广东省不同水陆生境样品98份,记录蛭态轮虫3目4科12属66种(含亚种).陆生环境中鉴定出蛭态轮虫56种,其中苔藓生境42种、落叶生境40种、地衣生境12种,水生生境蛭态轮虫15种,陆生生境的蛭态轮虫种类明显多于水生生境蛭态轮虫.为验证形态鉴定的准确性,获取蛭态轮虫COⅠ基因序列4属16种37条,种间遗传距离8.59%~23.17%,其中楔足盘网轮虫()和迅捷旋轮虫()的COⅠ基因序列为全球首次报道.COI基因序列中腺嘌呤(A)与胸腺嘧啶(T)的总含量(71.2%)远大于鸟嘌呤(G)与胞嘧啶(C) (28.8%)的含量.系统发育树的聚类结果与形态学物种鉴定结果一致;同属形态相近的物种,具有较高的同源性.研究结果表明,形态学鉴定与DNA 条形码技术相结合,有助于提高蛭态轮虫种类鉴定的准确性和探究蛭态轮虫各属的发育地位,促进蛭态轮虫物种多样性和生态学研究.
蛭态轮虫;生物多样性;DNA条形码;COI基因;生物地理
蛭态轮虫是水生无脊椎微型动物,属于轮虫动物门真轮纲蛭态亚纲,是目前为止有形态学、细胞发生学和基因学证据的最大、最古老以及最多样化的无性进化多细胞动物类群[1-2].蛭态轮虫具有专性孤雌生殖和隐生两大特性,是研究分类学、生态学以及遗传进化的理想模型[3-6].蛭态轮虫广泛分布于淡水水体中,并栖息于苔藓、地衣、落叶等半水生环境[7].蛭态轮虫鉴定较为困难,只有在活体时才能观察到关键结构特征,并极易收缩.由于蛭态轮虫物种鉴定需要活体状态,不同于常见水体轮虫,一些研究只将蛭态轮虫统一作为一大类别进行分类和讨论,导致蛭态轮虫物种多样性和生态学研究进展较慢.随着DNA条形码技术在物种鉴定方面的发展,该技术与传统形态学鉴定相结合,使得蛭态轮虫种类鉴定结果更准确可靠,并可探究蛭态轮虫物种间的系统发育关系.系统学分类分析还可帮助了解蛭态轮虫的进化历史过程,推断物种之间的亲缘关系.
Hebert等首次提出了DNA条形码概念,并表明线粒体COI基因具有变异速度适中、可用通用引物扩增的特点[8].因此利用线粒体COI基因作为分子标记已被广泛应用于研究物种进化.目前在轮虫种群遗传结构及系统地理学研究中采用的分子遗传标记主要是线粒体COI 基因序列和少量的核 DNA标记,如 ITS 和微卫星[9].由于轮虫个体小、基因组DNA含量少以及微卫星引物筛选难度大等原因,核DNA标记目前在轮虫种群遗传结构及系统地理学研究中应用并不普遍[10].随着基因标记技术的使用,蛭态轮虫潜在的物种多样性和分子隐种逐渐被发掘[11-12].
广东省位于南岭以南,南海之滨,是我国陆地最南部的省份,地域辽阔,地形复杂,生态类型多样.全境位于北纬20°13¢~25°31¢和东经109°39¢~117°19¢之间,北回归线横贯广东.因此,广东省从北向南分别为中亚热带、南亚热带和热带气候.广东省为东亚季风区,年平均气温高,且降水充沛,非常适合常绿植物的生长.对于广东省河流、城市湖泊和大型水库等典型淡水栖息环境,单巢纲轮虫物种多样性和群落结构研究都较为全面[13-15],蛭态轮虫研究较少,仅以蛭态轮虫提及,鲜有种类多样性记载.本研究基于形态学鉴定和DNA条形码技术,对广东省蛭态轮虫物种多样性进行了较为全面的调查,研究结果对于丰富我国的蛭态轮虫物种多样性和广东省生物多样性具有重要意义.
1 材料与方法
1.1 采样点设置与样品信息
自2017年9月至2019年12月,选择广东省城市公园、山脉、岛屿等不同水陆区域站点98个,包括白云山、帽峰山、火炉山、珠海万山群岛、汕头南澳岛等区域,采集水体、苔藓、地衣和落叶等不同生境样品,其中水库、湖泊、河流、溪流、水坑、咸淡水池塘等不同类型水体样品13个,土壤、石壁和树干等生境苔藓样品47个,地衣样品6个,树木和竹林落叶样品30个,花朵露水样品2个.
1.2 样品采集与鉴定
对土壤、苔藓、地衣和落叶等陆生环境样品的采集:用取土环刀或工具铲采集样品放入特制的牛皮纸袋中,实验室敞口自然风干后置入封口袋中密封保存.对于水生环境样品:采集激流中底栖样品时,用500mL瓶装容器刮取收集水底基质;采集水生植物附着样品时,摘取植物根茎进行冲洗后收集冲洗液;带回实验室后立即进行活体观察和鉴定.
陆生样品蛭态轮虫的提取:取样品放置于锥形瓶内,加入纯水浸泡,2~12h后,待蛭态轮虫与样品分离,经20μm筛网过滤3~4次,将滤液转移至5mL计数框中镜检;或将样品放入50mL规格的离心管中,加入50%的蔗糖溶液,离心机3000转离心5分钟,取上清液稀释后观察.
使用显微镜(OLYMPUS BX51)观察蛭态轮虫的形态学特征,录制其运动和摄食状态下的视频,随后仔细分析并记录视频中获取的轮盘、唇片(lip)、咀嚼器齿式等结构特征,参考分类书籍和文献[16-18]进行物种鉴定.为跟踪物种分类系统的最新进展,同时参考轮虫专业分类网站[19].
1.3 蛭态轮虫单克隆培养和DNA提取
使用生菜汁进行蛭态轮虫的单克隆培养.将生菜磨碎后过滤,滤液灭菌后冷冻保存待用.挑取蛭态轮虫置于24孔板中,每孔1只并加入1mL超纯水和10μL生菜汁,每3d更换1次培养液并观察记录蛭态轮虫的生长繁殖状况.
挑取单克隆培养轮虫,置于载玻片上,使用超纯水清洗数次,镜检确保虫体周围和表面均无杂质颗粒,将轮虫转移至0.2mL PCR管中,吸出多余水份,在解剖镜下将轮虫破碎,确保DNA充分提取.
样品DNA提取使用HotSHOT方法,配制碱性裂解液(NaOH 25mmol/L, EDTA 0.2mmol/L, pH 8.0)和中和缓冲液(Tris-HCl 40mmol/L, pH 5.0),按1:1比例混合.向每个PCR管加入裂解液10 μL,涡旋振荡后放入PCR仪95℃裂解30min;-20℃冷冻5~10min后加入10μL中和液,提取的DNA在-80℃中保存.
1.4 PCR扩增
PCR反应体系共25μL,设置3个平行.PCR反应体系: 引物(LCO1490: 5’-GGTCAACAAATCATA- AAGATATTGG-3’), 0.5μL;引物(HCO2198: 5’-TA- AACTTCAGGGTGACCAAAAAATCA-3’), 0.5μL; DNA模板: 5μL; ExTaq酶: 0.125μL; dNTP(Mg+): 2μL; Buffer: 2.5μL; ddH2O补足至25μL.PCR反应循环参数为:95℃预变性1min;95℃变性40s,48℃退火30s,72℃延伸45s,共30个循环;72℃延伸5min后4℃保存.PCR产物经1%琼脂糖凝胶电泳检测合格后利用Sanger法进行双向测序.
1.5 数据分析
所获原始序列经Bioedit软件检查校正测序结果峰图,利用SeqMan软件对序列进行拼接,人工校正剪去两端不可靠碱基序列;在NCBI进行Blast比对,查找相似度高的物种序列,从GenBank上下载其他蛭态轮虫COⅠ序列.用MAFFT进行多重序列比对,删除两端不对齐序列,使所有片段长度一致,比对结果使用MEGA7.0软件分析.使用软件DAMBE 6.0[20]进行碱基替换饱和度分析,COⅠ序列的碱基转换数和颠换数随种间遗传距离的增大呈明显上升趋势,可以进行系统发育树的构建.采用Kimura 2-parameter模型计算不同物种间的遗传距离,通过最大似然法(Maximum Likelihood method,ML)使用BEAST v1.8.4构建蛭态轮虫贝叶斯系统发育树.
2 结果
2.1 蛭态轮虫种类组成
共鉴定出蛭态亚纲轮虫3目4科12属66种(含亚种)(表1),包括旋轮属15种,粗颈轮属14种,盘网轮属10种,宿轮属10种,轮虫属7种,敖突轮属2种,原盘轮属2种,突盘轮属2种,盔甲轮属1种,水蛭轮属1种,围轮属1种和双趾轮属1种.其中,污轮虫()出现频次最高(23.5%),其次为贝氏盘网轮虫() (14.3%),栖居粗颈轮虫() (13.3%),游荡盘网轮虫() (11.2%)和褶皱旋轮虫() (10.2%).
2.2 不同生境中蛭态轮虫物种多样性比较

图1 四类生境中蛭态轮虫物种多样性的比较
陆生环境中鉴定出蛭态轮虫56种,其中苔藓生境42种、落叶生境40种、地衣生境12种,水体15种(图1).陆生生境的蛭态轮虫种类明显多于水体;28种蛭态轮虫既可在苔藓中生存,也可栖息在落叶中;苔藓生境和水体中有5种共有物种,水体独有种类10种.不同生境蛭态轮虫物种多样性差异较大.
2.3 COI基因序列组成分析与种间遗传距离
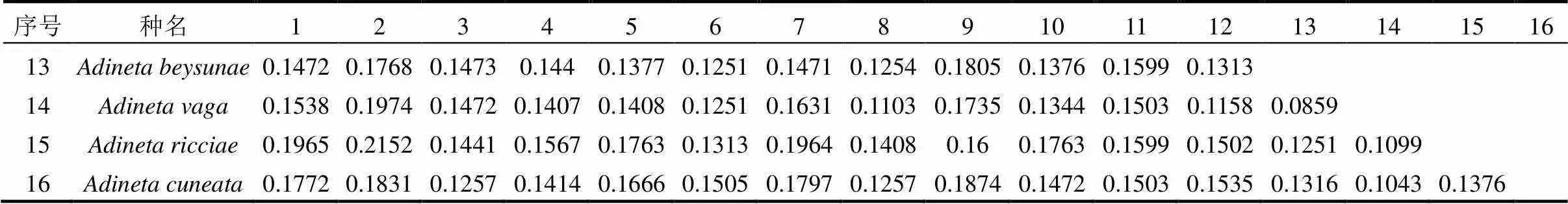
共获得37条COⅠ序列,通过BLAST比对分析,鉴定出16个形态学物种的分子信息(表2).对蛭态轮虫测序获得的COⅠ基因序列进行核苷酸组成分析,序列中A、C、G、T的平均含量分别为25%、10%、18.8%、46.2%,其中A+T的总含量(71.2%)远大于G+C的总含量(28.8%),高AT特性是明显的碱基偏倚现象,符合COⅠ基因的碱基组成特点.
根据Kimura 2-parameter模型,所有的种间遗传距离结果均大于3%,物种间最大的遗传距离为0.2317,为轮虫属的转轮虫()和旋轮属的巨环旋轮虫(),它们的栖息环境均为淡水水体,但形态、大小等特征具有明显差异.种间距离最小的是同属于盘网轮科盘网轮属的游荡盘网轮虫()和贝氏盘网轮虫(),为0.0859.
2.4 各属系统发育树分析
2.4.1 粗颈轮属 获得4个粗颈轮属()物种的COⅠ基因,共9条序列,分别为宽阔粗颈轮虫()、四角粗颈轮虫()、乳突粗颈轮虫()和粗多刺粗颈轮虫().经Blast比对,与该4种相似性最高的序列均为同种轮虫序列.其中,宽阔粗颈轮虫与基因库中宽阔粗颈轮虫EU751246.1的相似度为99%;四角粗颈轮虫与基因库中四角粗颈轮虫EF650532.1、FJ426410.1、EU499789.1的相似度分别为100%、99%和99%;乳突粗颈轮虫序列1和2与乳突粗颈轮虫 KM043196.1的相似度为100%;粗多刺粗颈轮虫序列1、2、3与基因库中多刺粗颈轮虫EF650498.1的相似度为99%.形态学物种鉴定与DNA分子鉴定的结果一致.以同属于旋轮科的间盘轮属作为分析时的外类群,MH251791.1- MH251792.1单独聚为一支,粗颈轮属所有物种序列聚为另一支(图2).
根据Kimura 2-parameter模型计算粗颈轮属各物种的种间距离,发现宽阔粗颈轮虫和的遗传距离最小为0.0995,系统发育树中这两个物种聚为最相近的一支.粗多刺粗颈轮虫和的遗传距离最大,为0.1924,系统发育树中这两个物种的亲缘关系最远.粗多刺粗颈轮虫为多刺粗颈轮虫()的亚种之一,与该属其他物种的亲缘关系较远,遗传距离也较高,该物种与其亚种单独聚为一支.宽阔粗颈轮虫、四角粗颈轮虫这两个物种的序列除了基因库中的相同种类外,与粗颈轮属的、泡状粗颈轮虫()和同源性较高,亲缘关系较近.

表1 广东省蛭态亚纲轮虫调查物种

续表1

表2 基于COI基因序列通过K2P模型计算的蛭态轮虫遗传距离
续表2
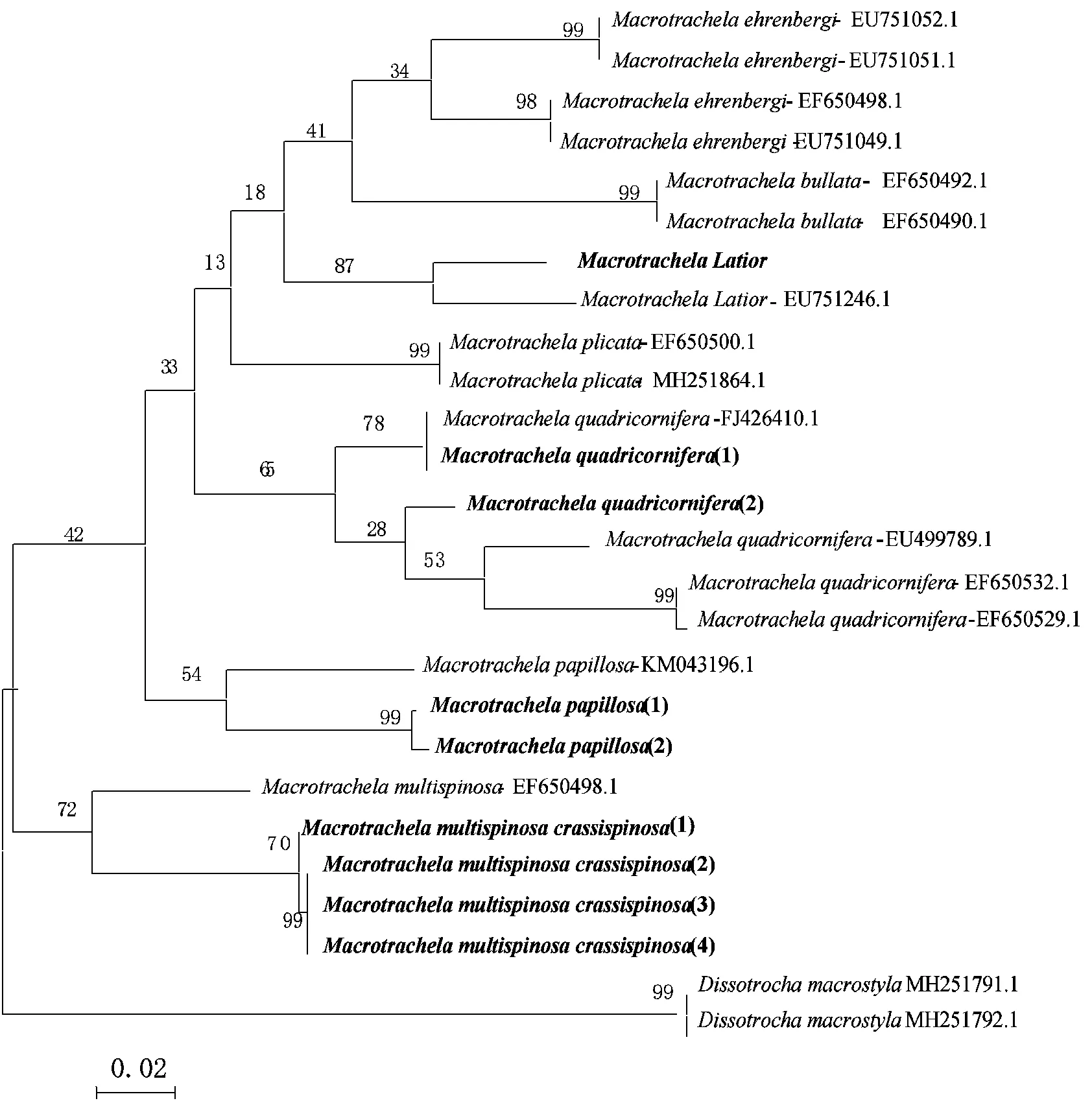
图2 基于COI基因构建的粗颈轮属系统发育树
2.4.2 旋轮属 获得6个旋轮属()物种的COⅠ基因序列共12条,分别是宽体旋轮虫()、微戟旋轮虫()、尖角旋轮虫()、巨环旋轮虫()、褶皱旋轮虫()和迅捷旋轮虫().经BLAST比对,前5个物种在GenBank中均找到了相似度高的同一物种序列,相似度分别为98%、99%、99%、97%和100%,均不低于97%,形态学鉴定的物种结果与基因序列比对结果一致.该6种轮虫中5种来自陆生生境(苔藓和落叶),只有巨环旋轮虫生活在稳定水体中,且分布广泛.微戟旋轮虫与种间遗传距离最小为0.1249;巨环旋轮虫与褶皱旋轮虫和双戟旋轮虫()遗传距离最大,同为0.1896.旋轮属轮虫种间遗传距离数值比较集中,基因之间重组率较高.经比对,GenBank中无迅捷旋轮虫的基因序列,本研究首次获得了该轮虫的COⅠ基因,其与同属的遗传距离最小为0.1278,在发育树中聚为一支,两个物种的同源性较高(图3).本研究获得的序列除了与同种物种的序列聚为一支以外,进化树的其他分支联系紧密,聚类并无较明显的分歧.
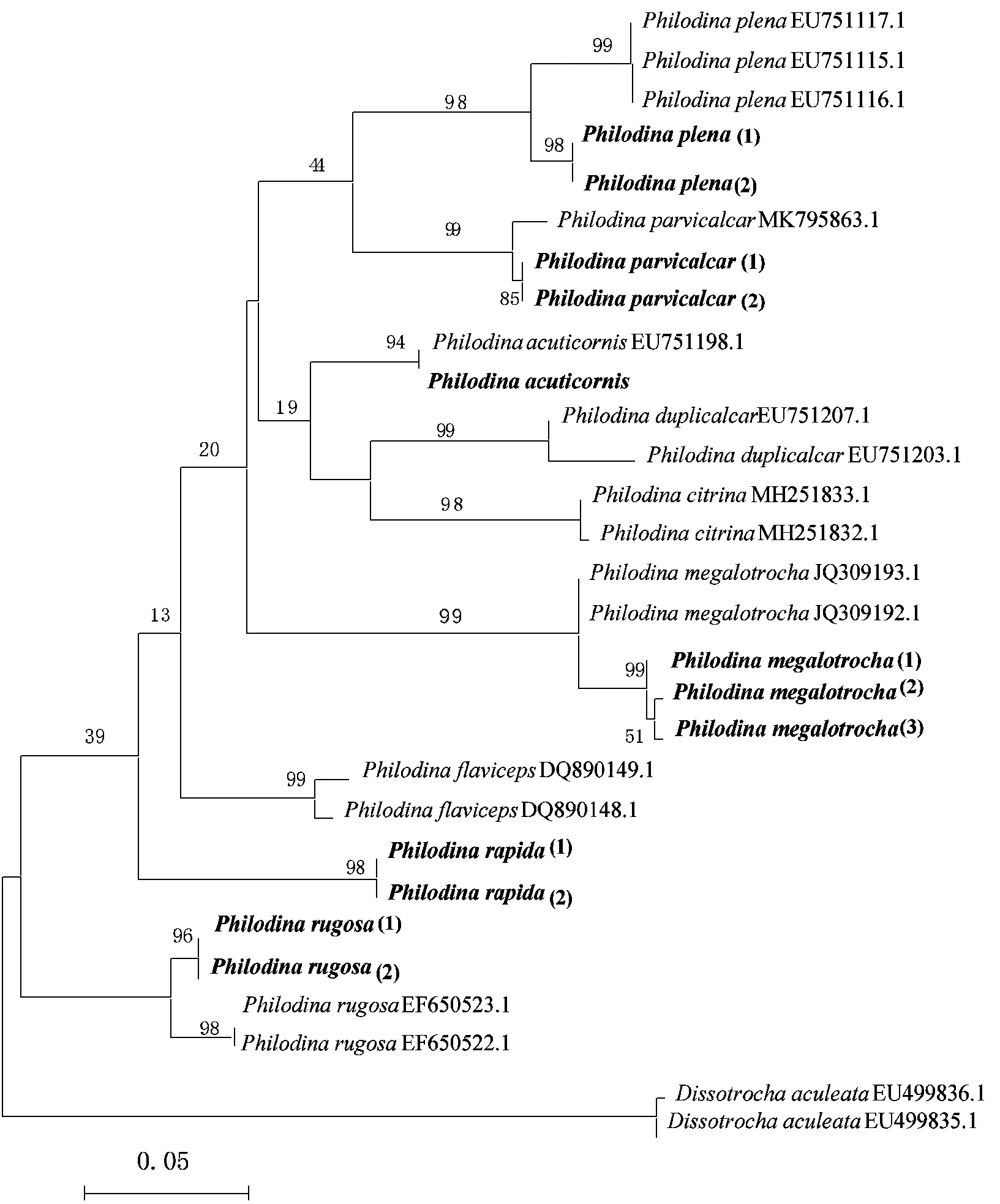
图3 基于COI基因构建的旋轮属系统发育树
2.4.3 轮虫属 发现两种轮虫属()种类,污轮虫()和转轮虫(),获得8条COⅠ基因序列.经BLAST比对,污轮虫与EU751161.1-751162.1的相似度为99%,转轮虫与JQ309463.1-309467.1的相似度为98%,种群间差异度小于3%.根据Kimura 2-parameter模型计算轮虫属的种间遗传距离,采用最大似然法构建系统发育树,以蛭态轮虫两条序列作为外类群,轮虫属所有物种序列聚为一支(图4).轮虫属最小种间距离0.1188,为和懒轮虫()两个物种,系统发育树结果显示这两个物种亲缘关系最近,聚为同一支(图4).污轮虫和转轮虫遗传距离最大,为0.1977,其亲缘关系相对最远.轮虫属的聚类结果分为两大类群,第一类群的物种包括污轮虫、长足轮虫()、懒轮虫和;第二类群包括转轮虫和,这两个物种的栖息生境均为池塘的水生植物区.长足轮虫和生活在稳定水体中,与陆生环境生存的其他2个物种聚为一支,表明物种的同源性可能与地理距离或虫体形态特征相关,不仅仅取决于生境.污轮虫的聚类情况较为复杂,同一生境地点获得的基因序列分开,与基因库中已有序列分别聚类,显示同域物种分化现象.
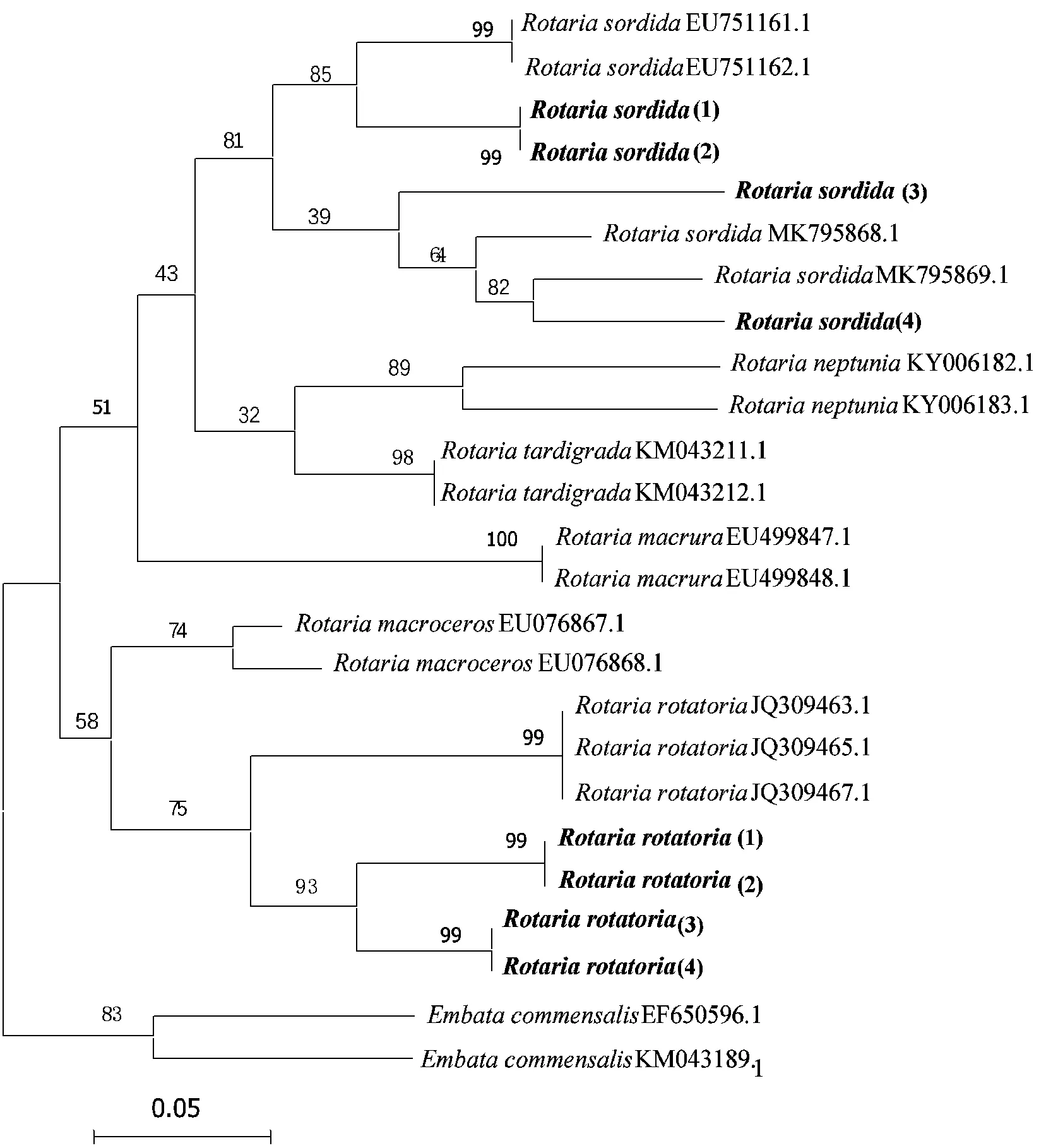
图4 基于COI基因构建的轮虫属系统发育树
2.4.4 盘网轮属 获得4种盘网轮属()物种的COⅠ基因序列8条.经BLAST序列比对,游荡盘网轮虫()、瑞氏盘网轮虫)、贝氏盘网轮虫()与其聚为一支对应的物种相似度分别为99%、98%和99%.以蛭态轮虫和作为外类群,盘网轮属物种全部聚为一支(图5).基因库中未发现楔足盘网轮虫()基因序列,本研究首次获得该轮虫的COI基因.该属中,与楔足盘网轮虫种间遗传距离最大,为0.1478;贝氏盘网轮虫与南方盘网轮虫()种间遗传距离最小,为0.1032,亲缘关系最近(图5),2个物种的形态结构特征相似,其喙部比较发达且都具有左右各一的坚硬纤毛,身体各部分形态和长度也无较大差别,唯一较明显的区别是贝氏盘网轮虫的足部有3个圆环状结构,南方盘网轮虫无此结构,这也是贝氏盘网轮虫的重要分类特征.楔足盘网轮虫和游荡盘网轮虫在形态上仅有足部和趾存在较大差异,其他部位类似,种间遗传距离为0.1091,相对于楔足盘网轮虫与其他物种的遗传距离较小,具有较高的同源性.
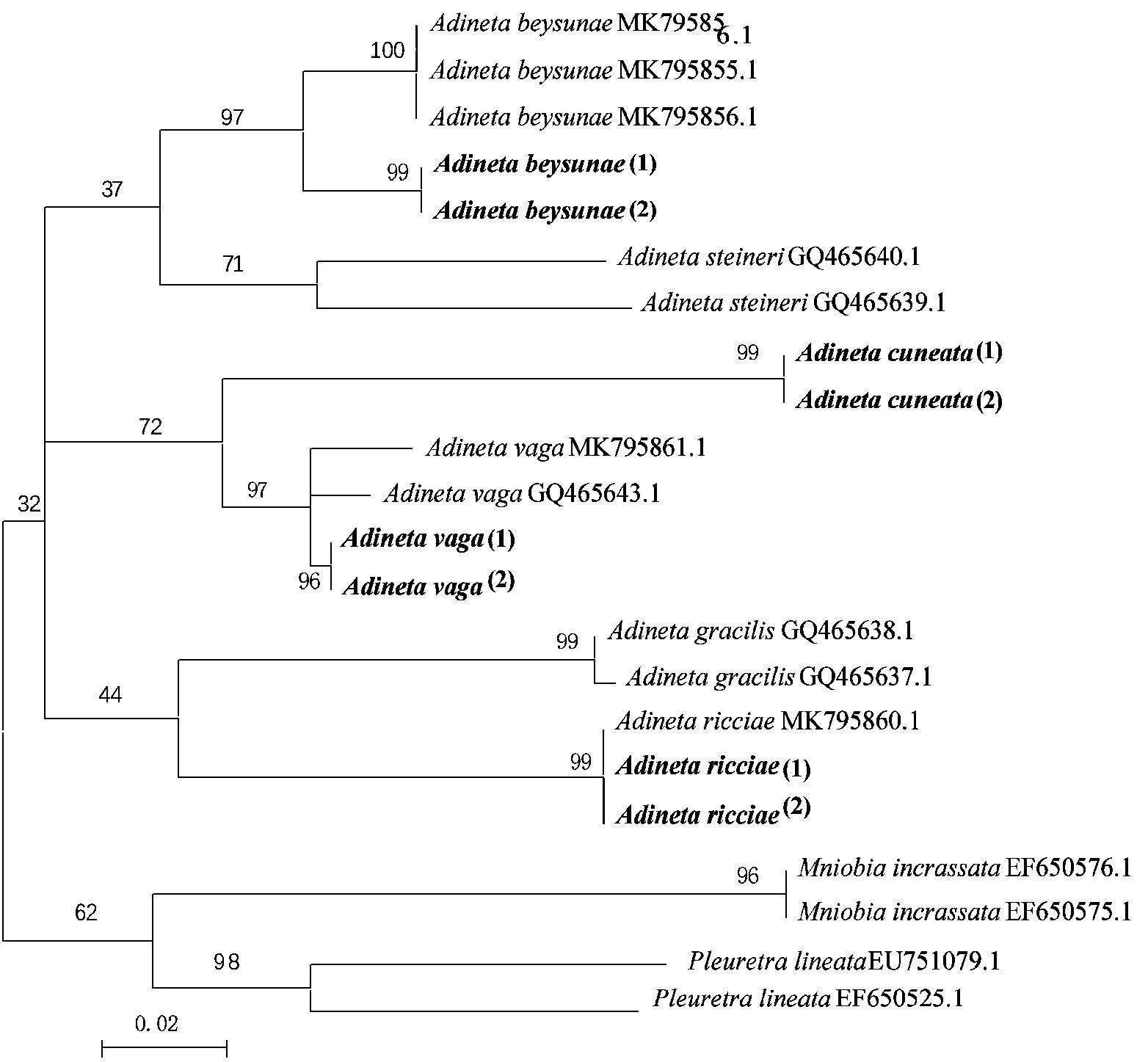
图5 基于COI基因构建的盘网轮属系统发育树
3 讨论
3.1 蛭态轮虫的物种多样性
目前全球共发现蛭态轮虫3目4科19属574种,其中原轮目(Philodinavida)3属9种、盘网轮目(Adinetida)2属28种、旋轮目(Philodinida)14属537种[19].我国的蛭态轮虫记录有4科13属89种[21-22].目前为止,蛭态亚纲轮虫的4科在中国已全部记录,仍有6属(、、、、、)的物种有待发现探究,这些属的物种相对于常见种,形态上都有其自身的特殊性,可能是为适应特殊生境而发生的适应性进化.由于蛭态轮虫分类学研究力度较弱以及高度的形态变异性,潜在的物种丰富度和分子隐种被严重低估[23-25].目前已记载的蛭态轮虫数仅能代表真实物种多样性的一部分.在今后的研究中,寻找特殊的新生境和研究方法的更新可能会发现更多蛭态轮虫物种.
蛭态轮虫分布广泛,常见种类出现在多个不同类型的生境中[7].调查发现,苔藓和落叶中的蛭态轮虫物种丰富度较高,很多常见种在2种生境中均可生存(图2).本次采集的23个苔藓、地衣、落叶和水体样品中都发现了污轮虫,涵盖了多种水陆栖息地生境,数量多、分布广,为物种地理区域格局的研究奠定了基础.研究发现,落叶中的蛭态轮虫体表常有附属结构,如短刺、环状凸起物等.这些结构增加了虫体本身的摩擦力,可以牢固地附着在基质表面,更好地适应多风环境.苔藓地衣的组织结构密集,保水抗风性较好,苔藓生境稳定,适合蛭态轮虫生长繁殖,物种较丰富,多样性高[22-24].水生生境中的蛭态轮虫一般聚集在水生植物生长的地方,水生植物为其提供了附着基质,有利于蛭态轮虫固着和摄食[11,22], 并可根据蛭态轮虫评价水体状况[26].蛭态轮虫物种丰富度在稳定水体中最低,原因可能是蛭态轮虫可通过低湿休眠度过不定期干燥等多变环境,以此成为其竞争和生存的重要策略.有研究显示,蛭态轮虫经历了低湿休眠后繁殖力反而增强,且有助于保持适合度的稳定[27].蛭态轮虫为无性生殖方式,能在环境适宜时迅速拓殖栖息地.因此周期性干燥等环境压力可能对其并非胁迫,而是生活史策略中不可或缺的因素,利于其与同生境的其他生物竞争,从而有助于蛭态轮虫物种的生存与延续[28].
蛭态轮虫体型微小无背甲,易收缩为无法辨认的球状,因此种类鉴定时不能对其进行有效固定或刺激性干扰.除了盘网轮科以外,其他3科不仅需要获取虫体的爬行体态特征,还须获取摄食状态下的轮盘特征才能进行形态学鉴定;其次,虫体运动剧烈难以在高倍显微镜下捕捉关键分类特征和进行及时有效的追踪观察;再者,目前最为全面的鉴定资料依然为早期发表的德语书籍,由于语言障碍使得鉴定和检索资料利用困难[2].因此,该类群动物的分类学研究一直进展较为缓慢,尤其是在非德语或非英语的亚非国家[22, 29-30].在现有技术手段下,借助分子生物学手段获取传统形态学物种的遗传信息进行比对分析,有助于揭示该类群潜在的物种多样性和生物地理分布格局.
3.2 蛭态轮虫的分子系统发育研究
本研究共获取蛭态轮虫COⅠ基因序列37条,包括4属16种,通过DNA条形码技术验证了形态学分类的准确性.蛭态轮虫体型微小,同属不同物种间形态差异小,特别是旋轮属的物种,数量多、特征相近,在鉴定过程中易混淆,为避免这种错误的出现,将分子生物学技术与传统分类学方法相结合,可以确保物种鉴定的准确性.蛭态轮虫COI序列中A+T的平均含量(71.2%)高于4种淡水臂尾轮虫A+T的平均含量(65.6%)[31],与8种淡水臂尾轮虫4和5基因序列中A+T含量相当(71.7%)[32],高AT特性是明显的碱基偏倚现象,符合COⅠ基因的碱基组成特点,在其他无脊椎动物线粒体基因中广泛存在.
目前对蛭态轮虫盘网轮属的分类学研究相对最多,也最易通过形态识别[33].本研究共采集了盘网轮属的4个形态学物种,其中游荡盘网轮虫分布非常广泛,几乎所有地区水陆生境都有其存在,变异度高,已有研究显示其形态学物种之下有36个分子隐种[11];瑞氏盘网轮虫易于识别与培养,该种具有较为广泛的地理分布.由于易采集和培养,在获得游荡盘网轮虫和瑞氏盘网轮虫全基因组信息后,其已成为研究基因组特征和遗传进化的重要模式生物[6,34].
对于部分稀有物种,本研究未能获取相关的COⅠ基因数据,因为DNA提取和扩增成功率较低,且多数稀有物种的样品数量较少,并且不容易培养,实验失败后很难再获取轮虫样品.为了提高基因提取和扩增的成功率,获取更多物种的基因序列,可做如下改进:(1) DNA提取时尽可能地将蛭态轮虫个体破碎完全,释放更多的内容物质,提高基因提取和扩增效率;(2) 利用蛭态轮虫孤雌生殖特点,进行单克隆培养,提取DNA时向PCR管中加入多只单克隆培养轮虫,提高DNA含量,更易扩增成功.
4 展望
蛭态轮虫体型微小,在环境不适时进入低湿休眠利于传播,往往被认为具有全球性分布特征,而不具备地域性特征[7].近年来研究显示南极地区存在极高比例的地域性物种,有高达95%的独立进化单元,已描述12个新种[27].对热带地区泰国水体蛭态轮虫调查也发现蛭态轮虫既有全球性广布种,也存在地域性物种[35].对不同栖息地的探索有助于发现更多占据不同生态位的蛭态轮虫.形态学分类工作未记录到大量地域性新物种的存在,可能是因为很多分子隐种未被发现,以及很多非常细微的形态学变异未被有效记录,因此后续研究不仅应利用更多样的显微镜技术,也应利用DNA分类技术对不同生境和不同地理区域的物种进行研究.我国具有多样化的生境,应尝试运用多种研究方法扩展蛭态轮虫调查的广度和深度,增加对青藏高原和热带地区湿地环境中轮虫群落的调查,将有助于全面了解蛭态轮虫多样性及其分布模式,丰富我国蛭态轮虫物种多样性,进一步探究蛭态轮虫的生态学作用.
5 结论
利用形态学和DNA分类手段,调查广东省不同水陆生境蛭态轮虫物种多样性,鉴定出蛭态轮虫3目4科12属66种,并获取4属16种共37条COⅠ序列.调查结果表明:(1)广东省蛭态轮虫物种多样性高,不同生境间物种组成差异较大,陆生生境物种更为丰富;(2)系统发育分析揭示了相应物种的分类地位和亲缘关系,证实了形态学观察配以DNA分类手段鉴定的有效性,为我国蛭态轮虫物种多样性和生物地理学研究提供了基础资料.
[1] Fontaneto D, Barraclough T G. Do species exist in asexuals? Theory and evidence from bdelloid rotifers [J]. Integrative and Comparative Biology, 2015,55(2):253-263.
[2] Ricci C. Bdelloid rotifers: ‘sleeping beauties’ and ‘evolutionary scandals’, but not only [J]. Hydrobiologia, 2017,796(1):277-285.
[3] Welch D B M, Meselson M. Evidence for the evolution of bdelloid rotifers without sexual reproduction or genetic exchange [J]. Science, 2000,288(5469):1211-1215.
[4] Gladyshev E A, Meselson M, Arkhipova I R. Massive horizontal gene transfer in bdelloid rotifers [J]. Science, 2008,320(5880):1210-1213.
[5] Wilson C G, Sherman P W. Anciently asexual bdelloid rotifers escape lethal fungal parasites by drying up and blowing away [J]. Science, 2010,327(5965):574-576.
[6] Flot J F, Hespeels B, Li X, et al. Genomic evidence for ameiotic evolution in the bdelloid rotifer Adineta vaga [J]. Nature, 2013,500 (7463):453-457.
[7] Fontaneto D. Long-distance passive dispersal in microscopic aquatic animals [J]. Movement Ecology, 2019,7(1):10.
[8] Hebert P D N, Cywinska A, Ball S L, et al. Biological identifications through DNA barcodes [J]. Proceeding of Royal society of London. Biological Science, 2003,270(1512):313-321.
[9] Schenk J, Fontaneto D. Biodiversity analyses in freshwater meiofauna through DNA sequence data [J]. Hydrobiologia, 2020,847:2597–2611.
[10] 项贤领,陶媛媛,席贻龙.轮虫种群遗传结构及系统地理格局研究进展[J]. 生态学杂志, 2015,34(7):2048-2057.
Xiang X L, Tao Y Y, Xi Y L. Review on phylogeographical pattern and population genetic structure of rotifer [J]. Chinese Journal of Ecology, 2015,34(7):2048-2057.
[11] Fontaneto D, Iakovenko N S, Eyres I, et al. Cryptic diversity in the genus Adineta Hudson & gosse, 1886 (Rotifera: Bdelloidea: Adinetidae): a DNA taxonomy approach [J]. Hydrobiologia, 2011,662:27-33.
[12] Iakovenko N S, Smykla J, Convey P, et al. Antarctic bdelloid rotifers: diversity, endemism and evolution [J]. Hydrobiologia, 2015,761:5-43.
[13] Wang Q, Yang Y F, Chen J F. Impact of environment on the spatio- temporal distribution of rotifers in the tidal Guangzhou segment of the Pearl River Estuary, China [J]. International Review of Hydrobiology, 2009,94(6):688-705.
[14] 梁迪文,王 庆,魏 南,等.广州市不同类型水体轮虫群落结构的时空变动及与理化因子间的关系[J]. 湖泊科学, 2017,29(6):1433-1443.
Liang D W, Wang Q, Wei N, et al. Spatial and temporal variation in rotifer community structure and the response to environmental factors among different water bodies in Guangzhou City [J]. Journal of Lake Sciences, 2017,29(6):1433-1443.
[15] 黄 珂,李慧明,徐少林,等.不同水力调度模式的两座调水型水库轮虫群落结构的比较[J]. 湖泊科学, 2019,31(3):853-861.
Huang K, Li H M, Xu S L, et al. Comparative analysis of rotifer community structures in two pumped-storage reservoirs with different retention times [J]. Journal of Lake Sciences, 2019,31(3):853-861.
[16] 王家楫.中国淡水轮虫志[M]. 北京:科学出版社, 1961.
Wang J J. Freshwater rotifers of China [M]. Beijing: Science Press, 1961.
[17] Donner J. Ordnung Bdelloidea (Rotatoria, Radertiere) Bestimmungsbü cher zur Bodenfauna Europas 6 [M]. Berlin: Akademie-Verl, 1965.
[18] Ricci C, Melone G. Key to the identification of the genera of bdelloid rotifers [J]. Hydrobiologia, 2000,418:73-80.
[19] Jersabek C D, Leitner M F. The rotifer world catalog [EB/OL]. http://www.rotifera.hausdernatur. at/, 2021-01-05.
[20] Xia X. DAMBE6: New tools for microbial genomics, phylogenetics and molecular evolution [J]. Journal of Heredity, 2017,108:431-437.
[21] 李 莹,王 庆,魏 南,等.蛭态轮虫中国新记录种及其研究展望[J]. 水生生物学报, 2020,44(2):423-428.
Li Y, Wang Q, Wei N, et al. Newly recorded species of bdelloid rotifer in China and research prospects [J]. Acta Hydrobiologica Sinica, 2020,44(2):423-428.
[22] Zeng Y, Wei N, Wang Q, et al. Bdelloid rotifers (Rotifera, Bdelloidea) of China: diversity and new records [J].ZooKeys, 2020,941:1-23.
[23] Kaya M, Herniou E A, Barraclough T G, et al. Inconsistent estimates of diversity between traditional and DNA taxonomy in bdelloid rotifers [J]. Organisms Diversity and Evolution, 2009,9(1):3-12.
[24] Fontaneto D, Kaya M, Herniou E A, et al. Extreme levels of hidden diversity in microscopic animals (Rotifera) revealed by DNA taxonomy [J]. Molecular Phylogenetics and Evolution, 2009,53(1):182-189.
[25] Robeson M S, King A J, Freeman K R, et al. Soil rotifer communities are extremely diverse globally but spatially autocorrelated locally [J]. Proceedings of the National Academy of Sciences, 2011,108(11):4406-4410.
[26] Kuczyńska-Kippen N. The use of bdelloids in reference to rotifer biocoenotic indices as an indicator of the ecological state of small field water bodies: The effect of macrophytes, shading and trophic state of water [J]. Ecological Indicators, 2018,89:576-583.
[27] Hinz C, Ahlrichs W H, Bininda-Emonds O R P. Immediate and heritable costs of desiccation on the life history of the bdelloid rotifer Philodina roseola [J]. Organisms Diversity & Evolution, 2018,18(4):399-406.
[28] Sommer S, Fontaneto D, Ozgul A. Demographic processes underlying fitness restoration in bdelloid rotifers emerging from dehydration [J]. Freshwater Biology, 2019,64(7):1295-1302.
[29] Song M O, Lee C H. A new and five rare bdelloids from Korea [J]. Zootaxa, 2017,4242(3):529-547.
[30] Song M O, Min G S. A new species and ten new records of bdelloid rotifers from Korea [J]. Zootaxa, 2015,3964(2):211-227.
[31] 鲍 蕾,牛翠娟,马 蕊,等.四种臂尾轮虫线粒体COI基因部分序列及系统发育关系[J]. 动物学研究, 2003,24(3):200-204.
Bao L, Niu C J, Ma R, et al. Sequences and phylogeny of partial mitochondrial COⅠ gene from four brachionus species [J]. Zoological Research, 2003,24(3):200-204.
[32] 程双怀,魏世祥,张 逸,等.基于ND4和ND5基因序列研究八种臂尾轮虫的系统关系和分类地位[J]. 水产学报, 40(12):1823-1829.
Cheng S H, Wei S X, Zhang Y, et al. Phylogenetic relationship and taxonomic status of eightspecies (rotifera) inferred from ND4 and ND5 genes [J]. Journal of Fisheries of China, 2016,40(12):1823-1829.
[33] Örstan A. Taxonomic morphology of the genus Adineta (Rotifera: Bdelloidea: Adinetidae) with a new species from a suburban garden [J]. Zootaxa, 2018,4524(2):187-199.
[34] Nowell R W, Almeida P, Wilson C G, et al. Comparative genomics of bdelloid rotifers: Insights from desiccating and nondesiccating species [J]. PLoS Biology, 2018,16(4):e2004830.
[35] Jaturapruek R, Fontaneto D, Meksuwan P, et al. Planktonic and periphytic bdelloid rotifers from Thailand reveal a species assemblage with a combination of cosmopolitan and tropical species [J]. Systematics and Biodiversity, 2018,16(2):128-141.
Species diversity and phylogenetic analysis of bdelloid rotifers in Guangdong Province.
WANG Qing, LI Ying, WANG Wen-bo, YANG Yu-feng*
(Department of Ecology, Jinan University, Guangzhou 510632, China)., 2021,41(9):4367~4377
In order to investigate species diversity of bdelloid rotifer in Guangdong Province, 98 samples from different habitats in Guangdong Province were collected, and 66 species (including subspecies) of bdelloid rotifers were recorded belonged to 3 orders, 4 families, 12 genera. Fifty-six species of bdelloid rotifer were identified in terrestrial environment, including 42 species in bryophyte habitats, 40 species in deciduous habitats, 12 species in lichen habitats, and 15species in aquatic habitats. The species number of bdelloid rotifer in terrestrial habitats was significantly more than that in aquatic habitats. To verify the accuracy of morphological identification, 37 COI gene sequences of 16 species belonged to 4genera were obtained, and the genetic distance among them ranged from 8.59% to 23.17%. The COI gene sequences ofandwere first reported in the world. The total content (71.2%) of adenine (A) and thymine (T) was higher than that (28.8%) of guanine (G) and cytosine(C). The cluster results of phylogenetic tree were consistent with those of morphological species identification; the species with similar morphology belonged to the same genus have higher homology. The results showed that the combination of morphological identification and DNA barcoding technology could improve the accuracy of species identification for bdelloid rotifers, explore their phylogenetic position at molecular level, and promote the species diversity and ecological research of bdelloid rotifers.
bdelloid rotifer;biodiversity;DNA barcoding;COI gene;biogeography
X17
A
1000-6923(2021)09-4367-11
王 庆(1981-),男,安徽安庆人,副研究员,博士,主要从事轮虫生态学研究.发表论文60余篇.
2021-02-18
广东省自然科学基金面上项目(2021A1515010814)
* 责任作者, 教授, tyyf@jnu.edu.cn

