水稻响应热胁迫核心基因的筛选与鉴定
张斌 杨昕霞 袁志辉


摘要: 基于热胁迫条件下的水稻转录组数据,通过HTSeq和DESeq软件筛选差异表达基因;对其进行功能富集和构建蛋白质互作网络,鉴定最重要模块中的核心基因。结果显示:水稻热胁迫0 h、1 h、6 h和12 h条件下,共有278个差异表达基因,生物学过程主要富集在刺激反应和蛋白质折叠上;鉴定出含有12个基因的重要模块.基因表达模式显示重要模块中7个核心基因受热胁迫诱导上调,推测这7个核心基因在水稻响应热胁迫过程中发挥关键作用。
关键词: 水稻;高温胁迫;核心基因;转录组
中图分类号: S511.01 文献标识码: A 文章编号: 1000-4440(2021)04-0817-06
Screening and identification of core genes responding to heat stress in rice
ZHANG Bin, YANG Xin-xia, YUAN Zhi-hui
(Hunan University of Science and Engineering, Yongzhou 425199, China)
Abstract: In this study, rice transcriptome data under heat stress were used to screen differentially expressed genes by HTSeq and DESeq software, then functional enrichment analysis was carried out and protein interaction network was constructed to identify the core genes in most important module. The results showed that there were 278 differentially expressed genes under heat stress for 0 h, 1 h, 6 h and 12 h, and the biological process was mainly concentrated in stimulation response and protein folding. The important module containing 12 genes was identified. The gene expression patterns showed that seven core genes in important modules were up-regulated under heat stress, suggesting that these seven core genes played a key role in the response of rice to heat stress.
Key words: rice;high temperature stress;core gene;transcriptome
水稻作为全球重要的粮食作物,其生长过程中极易受到各种生物胁迫和非生物胁迫的影响,其中高温胁迫严重影响水稻的产量和稻米的品质。因此,研究者在水稻热胁迫的危害及其应答热胁迫的生理和分子机制方面做了大量的工作。在成熟期,高温导致水稻籽粒灌浆提前结束[1];高温还可以引起稻米品质下降[2]。植物可以启动热激响应基因表达从而抵御胁迫伤害[3]。拟南芥钙调蛋白AtCaM3激活钙离子/钙调蛋白结合蛋白激酶,钙离子内流激活钙离子依赖的蛋白激酶,进而磷酸化热胁迫反应的关键调控因子,从而启动下游基因表达[4]。热激蛋白在植物响应热胁迫过程中发挥重要作用[5-7]。随着研究的深入,人们发现植物响应热胁迫的分子机制极其复杂。Takehara等发现OsSUS3基因导入日本晴后提高了该基因的表达水平,增强了抗热性[8]。Moon等报道异源表达OsHSP1基因可提高拟南芥的耐热性[9]。但是,到目前为止,水稻响应热胁迫的生理和分子调控机理仍未被阐述清楚。因此,本研究利用转录组学手段探究水稻响应热胁迫过程中的关键基因,并进一步阐释其功能途径。
1 材料与方法
1.1 材料
热胁迫处理的水稻转录组原始数据从NCBI数据库(https://www.ncbi.nlm.nih.gov/bioproject/PRJNA530826/)下载。本研究进行试验的水稻品种为日本晴 (Nipponbare)。
1.2 差异表达基因筛选
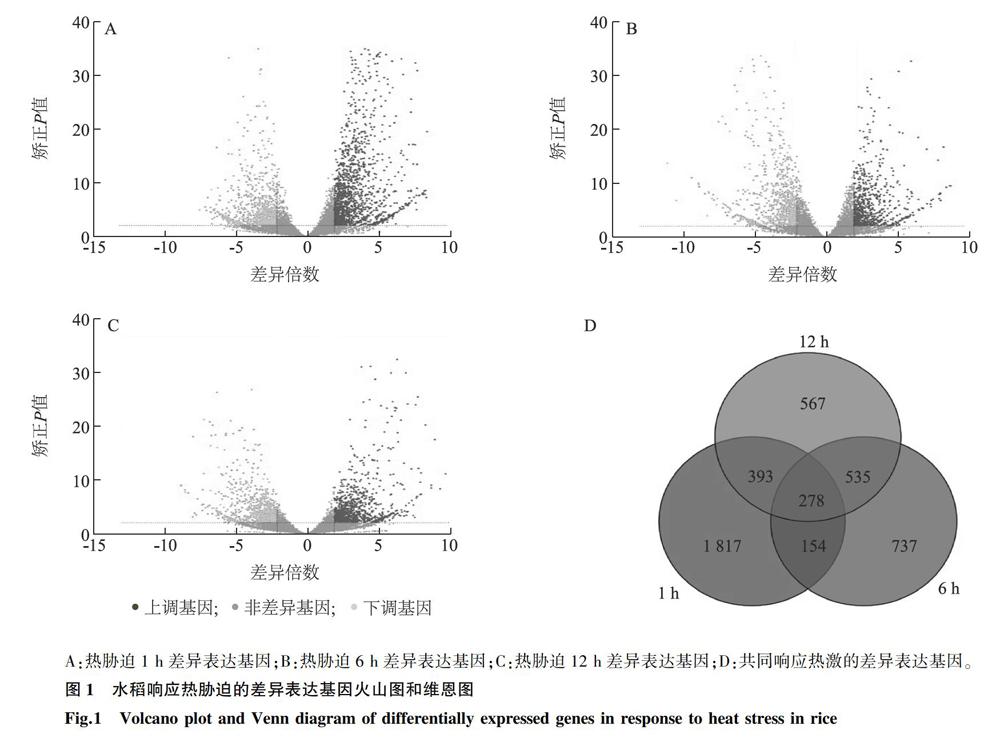
利用Hisat2软件建立索引以及比对到参考基因组,通过Stringtie软件进行转录本组装、合并和定量,基因差异分析则通过DESeq2软件进行。以|log2foldChange|>2且P值<0.01为条件分别筛选高温胁迫1 h、6 h和12 h时间点的差异表达基因,再通过Ggplot2绘制所有差异基因的火山图和韦恩图。
1.3 蛋白质互作网络构建及重要模块分析
利用在线数据库(http://string-db.org),以参数combined score>0.4,构建蛋白质互作网络, 并通过Cytoscape进行可视化。参数设置:scores>5,degree cut-off=2,node scorecut-off=0.2,Max depth=100和k-score=2。此外,利用MCODE(分子复合物检测)软件筛选最重要模块。
1.4 核心基因筛选及表达分析
设置马修斯相关系数(MCC)算法,利用Cytoscape软件的cytoHubba插件鉴定相互作用最紧密的10个基因,与最重要模块基因取交集,篩选核心基因。取log2TPM值(TPM为每千碱基记录本),分析水稻在高温胁迫0 h、1 h、6 h、12 h、24 h和48 h时间点的基因表达模式。
1.5 差异表达基因富集分析
差异表达基因使用在线软件(http://systemsbiology.cau.edu.cn/agriGOv2/)进行GO富集分析,取FDR(错误发现率)值≤0.05的通路,利用Ggplot2插件进行绘图。差异表达基因使用在线软件(http://kobas.cbi.pku.edu.cn/kobas3/genelist/)进行KEGG富集分析,取P值≤0.05的通路,利用Ggplot2插件绘图。
1.6 qRT-PCR验证
种子发芽后培养14 d(28 ℃,14 h白天/10 h黑夜),45 ℃热激1 h取样。利用TRIzol法提取幼苗总RNA,添加脱氧核糖核酸酶I(Dnase I)去除DNA污染。使用反转录试剂盒SuperScript III First-Strand Synthesis SuperMix(Thermo)合成cDNA。利用ABI7500定量PCR仪进行qRT-PCR检测,利用2-△△Ct法计算基因相对表达水平。反应程序:94 ℃预变性32 min;94 ℃变性10 s,60 ℃退火10 s,72 ℃延伸10 s,40个循环。总体系20 μl:10.0 μl SYBR Green Mix(Thermo),8.0 μl RNAase-free水,1.0 μl引物,1.0 μl模板。Os10g0510000基因为内参,引物序列见表1。
2 结果与分析
2.1 水稻热胁迫下差异表达基因筛选
热胁迫1 h,检测到2 642个差异表达基因,1 624个上调,1 018个下调(图1A);热胁迫6 h,检测到1 704个差异表达基因,812个上调,892个下调(图1B);热胁迫12 h,检测到1 773个差异表达基因,899个上调,874个下调(图1C)。共有278个重叠基因(图1D)。
2.2 水稻热胁迫下差异表达基因富集分析
278个基因富集到GO项上,生物学过程主要富集在刺激反应和蛋白质折叠;分子功能主要富集在结合功能;细胞组分无显著富集(图2A)。KEGG分析结果显示278个差异基因主要富集在内质网蛋白质加工和次生代谢产物合成通路上(图2B)。
2.3 蛋白质互作网络构建与重要模块中关键核心基因筛选
通过蛋白质互作网络(图3A)获得了一个含有12个基因表达全部上调的最重要模块(图3B)。进一步通过Cytoscape软件的cytoHubba插件鉴定出作用最紧密的10个基因(图3C),其中7个基因存在最重要模块上,确定为核心基因(图3D)。
2.4 水稻响应热胁迫核心基因表达模式分析
分析核心基因的表达模式,发现在热胁迫条件下不同时间点(1 h、6 h、12 h、24 h和48 h)基因表达都表现上调(图4)。随热胁迫时间的延长,基因表达水平有一定波动。Os01g0840100、Os07g0641700、Os12g0277500、Os03g0366000和Os02g0612000 5個基因的表达在热胁迫1 h时达到最大值,呈现先上升,后下降的趋势;Os08g0338700和Os09g0491772响应热胁迫时间比较长,在48 h内基因表达表现出上升的趋势。
2.5 qRT-PCR方法验证转录组测序结果
利用qRT-PCR方法对转录组数据进行验证。结果(图5)显示,野生型水稻日本晴幼苗经热胁迫处理1 h后,筛选出的7个核心基因Os12g0277500、Os08g0338700、Os07g0641700、Os09g0491772、 Os01g0840100、Os02g0612000和Os03g0366000的表达水平都上调,结果与转录组测序数据基本一致,表明转录组测序结果可靠,也进一步证明了通过转录组数据筛选鉴定水稻响应热胁迫过程中核心基因的可靠性。
3 讨论
生物具有感知温度变化的系统[10]。植物响应热胁迫涉及受体激活、活性氧产生、热激蛋白和热激转录因子等多种信号途径[11],过程复杂是其分子机制没有研究清楚的原因之一。因此,迫切要求判断植物响应热胁迫的潜在核心基因。转录组技术能够分析热胁迫中植物基因表达的改变,是一种确定核心基因有效方法。
本研究分析了水稻热胁迫下4个转录组数据集,共鉴定出278个差异表达基因,GO分析结果显示这些基因主要富集在对刺激的反应和蛋白质折叠的生物过程上,KEGG分析结果显示这些基因主要富集在内质网蛋白质加工的代谢途径上。蛋白质折叠在生物适应高温胁迫的过程中起着重要的作用[12]。热激蛋白(HSP)分为HSP100、HSP90、HSP70、HSP60和小HSP家族,具有阻止变性蛋白质聚集和协助变性蛋白质重新折叠的功能[13]。HSP101基因功能缺失的拟南芥变得对热敏感[14]。因此, HSP家族成员在植物响应热胁迫过程中发挥重要作用。叶绿体和线粒体HSP70s作为转运子的一部分可以帮助蛋白质前体转移[15-17]。研究结果表明拟南芥通过细胞周期转录因子介导叶绿体AtHSP70-4基因的表达适应温度变化[18]。HSP70-4和E3泛素连接酶通过泛素-26S蛋白酶系统介导质体靶向蛋白质前体的降解,减轻细胞损伤[19]。线粒体mtHsc70-1基因在拟南芥细胞色素c氧化酶(COX)依赖性呼吸系统中发挥作用,基因敲除后表现出严重的生长缺陷[20]。Os01g0840100和Os09g0491772同属HSP70家族,在水稻热胁迫响应过程中的具体功能未见报道,本研究中其受热胁迫诱导,可能发挥调控作用。质体Cpn60是线粒体Hsp60的同源物,参与核酮糖-1,5-二磷酸羧化酶的组装,在此过程中需要蛋白质GroES/Cpn10进行底物封装[21-23]。Cpn60家族在拟南芥生长发育过程中发挥着非常重要的作用,短日照条件下,拟南芥AtCpn60β1基因缺失导致幼苗死亡,突变体对热胁迫敏感[24] 。OsCpn60α1(Os12g0277500)对核酮糖-1,5-二磷酸羧化酶大亚基的折叠至关重要,突变体表现出淡绿色和幼苗致死的表型,高温对OsCpn60α1的转录有较强的诱导作用[25]。同源基因OsCpn60β1对水稻叶绿体发育至关重要[26]。这与我们的分析结果一致,说明OsCpn60在热应激过程中发挥了重要的作用。拟南芥AT Cpn10基因与大肠杆菌groES基因功能互补,在不同器官中mRNA都有表达,受热胁迫诱导[27]。Os07g0641700和Os03g0366000同属Cpn10基因,表达水平在0~24 h持续上调,说明在热胁迫过程中发挥了一定的作用。DnaK-DnaJ-GrpE系统是蛋白质稳态的重要组成部分[28]。拟南芥GrpE表达受热胁迫诱导,与大肠杆菌grpE基因功能互补[29]。Os08g0338700和Os02g0612000同属GrpE基因,此基因的功能亦未见报道,本研究中受热胁迫的诱导,推测其在热胁迫响应中发挥调控作用。水稻响应热胁迫与蛋白质折叠息息相关,7个核心基因同属HSP家族,蛋白质互作网络分析结果表明它们联系非常紧密,同源蛋白质参与蛋白质的运输、组装和折叠,因此,我们认为这些基因在热胁迫过程中协同发挥关键性作用。
总之,本研究旨在确定水稻热胁迫响应过程中的核心基因。共鉴定出278个差异基因和7个核心基因,7个基因可能在水稻响应热胁迫过程中发挥重要作用。但是,需要进一步研究来阐明这些基因在水稻响应热胁迫过程中的生物学功能。
参考文献:
[1] KIM J, SHON J, LEE C K, et al. Relation-ship between grain filling duration and leaf senescence of temperate rice under high temperature[J]. Field Crops Research, 2011, 122: 207-213.
[2] ZHONG L J, CHENG F M, WEN X, et al. The deterioration of eating and cooking quality caused by high temperature during grain filling in early-season indica rice cultivars[J]. Journal of Agronomy & Crop Science, 2005, 191: 218-225.
[3] LIM C J, YANG K A, HONG J K, et al. Gene expression profiles during heat acclimation in Arabidopsis thaliana suspension-culture cells[J]. J Plant Res, 2006, 119: 373-383.
[4] SUZUKI N, SEJIMA H, TAM R, et al. Identification of the MBF1 heat-response regulon of Arabidopsis thaliana[J]. Plant J, 2011, 66(5): 844-851.
[5] GUAN J C, YEH C H, LIN Y P, et al. A 9 bp cis-element in the promoters of class I small heat shock protein genes on chromosome 3 in rice mediates L-azetidine-2-carboxylic acid and heat shock responses[J]. Journal of Experimental Botany, 2010, 61(15): 4249-4261.
[6] JAGADISH S V, MUTHURAJAN R, OANE R, et al. Physiological and proteomic approaches to address heat tolerance during anthesis in rice(Oryza sativa L.)[J]. Journal of Expermental Botany, 2010, 61: 143-156.
[7] JUNG K H, GHO H J, NGUYEN M X, et al. Genome-wide expression analysis of HSP70 family genes in rice and identification of a cytosolic HSP70 gene highly induced under heat stress[J]. Functional & Integrative Genomics, 2013, 13(3): 391-402.
[8] TAKEHARA K, MURATA K, YAMAGUCHI T, et al. Thermo-responsive allele of sucrose synthase 3 (Sus3) provides high-temperature tolerance during the ripening stage in rice (Oryza sativa L.)[J]. Breeding Science, 2018, 68(3): 336-342.
[9] MOON J C, HAM D J, HWANG S G, et al. Molecular characterization of a heat inducible rice gene, OsHSP1, and implications for rice thermotolerance[J]. Genes Genom, 2014, 36: 151-161.
[10]MITTLER R, FINKA A, GOLOUBINOFF P. How do plants feel the heat?[J]. Trends Biochem Sci, 2012, 37(3): 118-125.
[11]陳思婷,郭房庆.植物耐热性及热激信号转导机制研究进展[J].中国科学:生命科学,2013,43(12): 1072-1081.
[12]MORO F, MUGA A. Thermal adaptation of the yeast mitochondrial Hsp70 system is regulated by the reversible unfolding of its nucleotide exchange factor[J]. J Mol Biol,2006, 358(5): 1367-1377.
[13]WANG W, VINOCUR B, SHOSEYOV O, et al. Role of plant heat-shock proteins and molecular chaperones in the abiotic stress response[J]. Trends Plant Sci, 2004, 9(5): 244-252.
[14]HONG S W, VIERLING E. Mutants of Arabidopsis thaliana defective in the acquisition of tolerance to high temperature stress[J]. Proc Natl Acad Sci USA, 2000, 97(8): 4392-4397.
[15]JACKSON C D, KEEGSTRA K. Arabidopsis genes encoding components of the chloroplastic protein import apparatus[J]. Plant Physiol, 2001, 125:1567-1576.
[16]SU P H, LI H M. Stromal Hsp70 is important for protein translocation into pea and Arabidopsis chloroplasts[J].Plant Cell, 2010, 22:1516-1531.
[17]ZHANG X P, GLASER E. Interaction of plant mitochondrial and chloroplast signal peptides with the Hsp70 molecular chaperone[J]. Trends Plant Sci, 2002, 7:14-21.
[18]ZHOU S, SUN H, ZHENG B, et al. Cell cycle transcription factor E2F2 mediates non-stress temperature response of AtHSP70-4 in Arabidopsis[J]. Biochem Biophys Res Commun, 2014, 455(3-4): 139-146.
[19]LEE S, LEE D W, LEE Y, et al. Heat shock protein cognate 70-4 and an E3 ubiquitin ligase, CHIP, mediate plastid-destined precursor degradation through the ubiquitin-26S proteasome system in Arabidopsis[J]. Plant Cell, 2009, 21(12): 3984-4001.
[20]WEI S S, NIU W T, ZHAI X T, et al. Arabidopsis mtHSC70-1 plays important roles in the establishment of COX-dependent respiration and redox homeostasis[J]. J Exp Bot, 2019, 70(20): 5575-5590.
[21]BARRACLOUGH R, ELLIS R J. Protein synthesis in chloroplasts IX assembly of newly-synthesized large subunits into ribulose bisphosphate carboxylase in isolated intact pea chloroplasts[J]. Biochim Biophys Acta, 1980, 608(1):19-31.
[22]HEMMINGSEN S M, ELLIS R J. Purification and properties of ribulosebisphosphate carboxylase large subunit binding protein[J]. Plant Physiology, 1986, 80(1):269-276.
[23]HEMMINGSEN S M, WOOLFORD C, VAN D, et al. Homologous plant and bacterial proteins chaperone oligomeric protein assembly[J]. Nature, 1988, 333(6171):330-334.
[24]ATSUSHI I, HIDEAKI T, MASATO N, et al. Deletion of a chaperonin 60 beta gene leads to cell death in the Arabidopsis lesion initiation 1 mutant.[J]. Plant and Cell Physiology, 2003(3):255-261.
[25]KIM S R, YANG J I, AN G. OsCpn60α1, encoding the plastid chaperonin 60α subunit, is essential for folding of rbcL[J]. Moleculer Cells, 2013, 35(5) :402-409.
[26]WU Q, ZHANG C, CHEN Y, et al. OsCpn60β1 is essential for chloroplast development in rice (Oryza sativa L.)[J]. International Journal of Molecular Sciences, 2020, 21(11) :4023.
[27]KOUMOTO Y, TSUGEKI R, SHIMADA T, et al. Isolation and characterization of a cDNA encoding mitochondrial chaperonin 10 from Arabidopsis thaliana by functional complementation of an Escherichia coli groES mutant[J]. Plant J,1996, 10(6): 1119-1125.
[28]BEN-ZVI A P, GOLOUBINOFF P. Review: mechanisms of disaggregation and refolding of stable protein aggregates by molecular chaperones[J]. J Struct Biol, 2001, 135(2): 84-93.
[29]HU C, LIN S Y, CHARNG C, et al. Recent gene duplication and subfunctionalization produced a mitochondrial GrpE, the nucleotide exchange factor of the Hsp70 complex, specialized in thermotolerance to chronic heat stress in Arabidopsis[J]. Plant Physiology, 2012, 158(2):747-758.
(責任编辑:张震林)
收稿日期:2020-01-25
基金项目:湖南省自然科学基金项目(2020JJ4030);湖南省创新平台与人才计划项目(2020NK4222)
作者简介:张 斌(1981-),男,湖南永州人,博士,讲师,主要从事植物发育生物学研究,(E-mail)zhangbin27104@163.com
通讯作者:袁志辉,(E-mail)zhh_yuan@126.com

