罗氏沼虾淀粉酶基因克隆及其表达规律分析
姜建萍 杨学明 袁翔 邱庆庆 杨秀荣 蒋钦杨 黄光华 蒋和生


摘要:【目的】克隆羅氏沼虾(Macrobrachium rosenbergii)淀粉酶基因(AMY),并进行生物学信息分析及其表达规律研究,为揭示AMY基因的生物学功能及其对罗氏沼虾生长发育的调控作用机理提供理论依据。【方法】PCR克隆罗氏沼虾AMY基因编码区(CDS)序列,利用ExPASy ProtParam、ExPASy ProtScale、SignalP-5.0、MegAlign及Lasergene v8.0等在线软件进行生物信息学分析,应用实时荧光定量PCR检测AMY基因在罗氏沼虾不同组织(腹神经节、胃、心脏、鳃组织、性腺、肝胰腺、肌肉和肠道)中的表达情况,并明确其在生长快速家系(FG)和生长慢速家系(SG)个体肌肉中的表达模式。【结果】罗氏沼虾AMY基因CDS序列全长2121 bp,共编码706个氨基酸残基;其编码蛋白分子量为76.87 kD,理论等电点(pI)为4.63,属于不稳定的亲水性蛋白,包含2个典型的淀粉酶结构域;在罗氏沼虾AMY蛋白二级结构中,α-螺旋占13.88%,延伸链占21.39%,无规则卷曲占64.73%。罗氏沼虾AMY氨基酸序列与克氏原螯虾AMY氨基酸序列的相似性最高(62.6%),与大珠母贝AMY氨基酸序列的相似性较低(48.9%);基于AMY氨基酸序列相似性构建的系统发育进化树显示,罗氏沼虾与克氏原螯虾的亲缘关系最近,而与中华绒螯蟹的亲缘关系较远。AMY基因在罗氏沼虾不同组织中广泛表达,但存在性别差异性和组织特异性,具体表现为:雌性个体肠道中的相对表达量极显著高于雄性个体(P<0.01,下同),鳃组织中的相对表达量显著高于雄性个体(P<0.05),而肝胰腺中的相对表达量极显著低于雄性个体。罗氏沼虾AMY基因在FG个体肌肉中的相对表达量极显著高于SG个体。【结论】AMY基因在罗氏沼虾的生长发育过程中发挥重要作用,广泛表达于罗氏沼虾的不同组织中,但存在性别差异性和组织特异性。
关键词: 罗氏沼虾;淀粉酶基因(AMY);表达规律;生物信息学分析;生长性状
中图分类号: S917.4 文献标志码: A 文章编号:2095-1191(2021)05-1362-08
Abstract:【Objective】Macrobrachium rosenbergii amylase gene(AMY) was cloned, biological information analysis and expression regulation were studied, to provide theoretical foundation for further understanding biological function of AMY gene in the growth and development of M. rosenbergii. 【Method】The coding area sequence (CDS) of AMY gene in M. rosenbergii was cloned by PCR. Bioinformatic analysis of AMY sequencing was performed using ExPASy ProtParam, ExPASy ProtScale, SignalP-5.0, MegAlign and Lasergene v8.0 softwares. Real-time fluorescence quantitative PCR (RT-PCR) was used to detect the expression of AMY gene in different tissues(including abdominal ganglion,stomach,heart,gill,gonad,hepatopancreas,muscle and intestine), and further to explore its expression in the muscle of individuals from fast-growing families(FG) and slow-growing families(SG). 【Result】The length of CDS of AMY gene was 2121 bp, encoding 706 amino acids residues. The molecular weight of AMY protein was 76.87 kD,and the theoretical isoelectric point(pI) was 4.63,belonging to an unstable hydrophilic protein, which contained two typical amylase domains. The secondary structure of AMY protein was consisted of α-helix,extended strand and random coil,accounting for 13.88%, 21.39% and 64.73%, respectively. The homology of amino acid sequence of AMY gene in M. rosenbergii was the highest(62.6%) and lower(48.9%) compared with the amino acid sequences of Procambarus clarkii and Pinctada maxima,respectively. Phylogenetic tree constructed based on the amino acid sequence similarity of AMY genes howed that M. rosenbergii had the closest genetic relationship with P. clarki, but had the far genetic relationship with Eriocheir sinensis. AMY gene was widely expressed in the different tissues, but with differences between sexes and tissue specificity. The details were as follows:it had the extremely higher expression in the intestines and gills of female individuals than that of male individuals(P<0.01, the same below),it had the significantly higher expression in gill of female individuals than that of male indivi-duals(P<0.05),but the expression in the hepatopancreas of female individuals was lower than that of male individuals. In addition, the expression level of AMY gene in muscle of in FG was extremely higher than that in SG. 【Conclusion】AMY gene plays an important role in growth and development of M. rosenbergii. It is widely expressed in different tissues of M. rosenbergii, but with sex specificity and tissue specificity.
Key words:Macrobrachium rosenbergii; amylase gene(AMY); expression regulation; bioinformatic analysis; growth traits
Foundation item:Guangxi Natural Science Foundation(2018GXNSFBA281209); Science and Technology Major Project of Guangxi(Guike AA17204080-6); Guangxi Postdoctoral Fundamental Research Fund(T3340097968)
0 引言
【研究意义】罗氏沼虾(Macrobrachium rosenbergii)原产于东南亚国家,因具有生长快、食性广、肉质鲜美及养殖周期短等优点,现已发展成为我国重要的淡水养殖品种之一(姜建萍等,2019)。截至2018年,我国罗氏沼虾产量达13.3万t(农业农村部渔政渔业管理局,2019)。随着集约化养殖规模的不断扩大,近亲繁殖等原因致使罗氏沼虾种质不断退化,“老头虾”“蝎子虾”现象频繁出现,严重制约了罗氏沼虾养殖业的持续健康发展,亟待进一步加强罗氏沼虾遗传改良以提高罗氏沼虾养殖效益和健康养殖水平。淀粉酶(Amylase,AMY)是机体内重要的消化酶类,在甲壳类动物碳水化合代谢及营养物质消化吸收方面扮演着重要角色(Simon,2009;Karasov and Douglas,2013;Rodríguez-Viera et al.,2017),因此开展罗氏沼虾AMY基因克隆及其表达规律分析,对深入探究AMY基因生物学功能和提高罗氏沼虾遗传改良及分子辅助育种均具有重要意义。【前人研究进展】至今,已有众多学者针对AMY基因克隆及其生物信息学分析展开了一系列研究,从鳜鱼(陈亮等,2009)、斜带石斑鱼(胡永乐等,2010)、大珠母贝(潘俐玲等,2013)、日本囊对虾(宋晓红,2014)、九孔鲍(栗志民等,2017)及斑节对虾(杨其彬等,2017)等多种水产动物中克隆获得AMY基因编码区(CDS)序列,针对AMY基因的表达模式及其规律也有较多研究报道。辛静静等(2011)研究证实,AMY基因多态性对凡纳滨对虾的生长性状有显著影响,可作为影响凡纳滨对虾生长性状的候选基因。宋晓红(2014)研究表明,AMY基因在不同变异类型日本囊对虾的不同组织中均有表达,以肝胰腺中的相对表达量最高,故推测AMY基因与日本囊对虾的消化代谢相关。彭涛(2015)通过免疫印迹法和实时荧光定量PCR分析克氏原螯虾AMY基因的组织表达差异,结果显示AMY基因在各组织中的表达量差异显著,以在肝胰腺中的表达量最高,其次是胃组织;经蜕皮激素刺激48 h后其表達量明显下降,说明AMY基因能应答克氏原螯虾蜕皮激素诱导信号。唐小红等(2015)研究表明,肝胰脏并不是草鱼生成淀粉酶的唯一场所,在其肠道组织中也有AMY基因表达;自出膜72 h仔鱼开口摄食之后,AMY基因的表达量明显增加,故推测摄食能促进草鱼AMY基因的表达。栗志民等(2017)研究发现,AMY基因在九孔鲍右侧壳肌、肠道、肝脏、胃、外套膜、吻和腹足等7种组织中均有不同程度的表达,经ANOVA分析显示消化组织与非消化组织间的AMY基因表达量存在显著差异,即AMY基因表达量与九孔鲍的生长性状呈显著正相关。杨其彬等(2017)研究表明,AMY基因在斑节对虾的不同组织及整个生长阶段均有表达,且以糠虾时期的表达量最高,说明AMY基因可能与斑节对虾的幼体发育相关。以上研究证明,AMY基因是水产类动物重要的生长性状候选基因。【本研究切入点】至今,关于罗氏沼虾AMY基因的克隆、生物信息学分析及其表达规律鲜见研究报道。【拟解决的关键问题】基于NCBI已公布的AMY基因EST序列,克隆罗氏沼虾AMY基因CDS序列,并进行生物学信息分析及其表达规律研究,为揭示AMY基因的生物学功能及其对罗氏沼虾生长发育的调控作用机理提供理论依据。
1 材料与方法
1. 1 试验材料
5月龄罗氏沼虾由广西南宁国家级罗氏沼虾良种场提供。以肝胰腺为素材,进行AMY基因克隆;分别选取7尾雌/雄罗氏沼虾采集样品,包括腹神经节、胃、心脏、鳃组织、性腺、肝胰腺、肌肉和肠道共8个组织,用于AMY基因表达分析;同时采集生长快速家系(FG)和生长慢速家系(SG)中体重和体长极端性状的雌性罗氏沼虾肌肉组织(邱庆庆,2019),分别置于液氮中保存备用。总RNA提取试剂TRIzol、反转录试剂盒、凝胶回收试剂盒、pMD18-T载体和Premix Ex TaqTM Ⅱ均购自TaKaRa公司;DL2000 DNA Marker购自广州东盛生物科技有限公司;Taq PCR Master Mix购自Vazyme公司;大肠杆菌Trans5α感受态细胞购自北京全式金生物技术有限公司。
1. 2 总RNA提取及cDNA合成
采用TRIzol提取罗氏沼虾样品总RNA,利用NanoDrop 2000和1.0%琼脂糖凝胶电泳检测RNA浓度及其完整性,再以PrimeScriptTM RT Reagent Kit with gDNA Eraser试剂合成cDNA。
1. 3 AMY基因克隆及生物信息学分析
根据GenBank已公布的罗氏沼虾AMY基因EST序列(KM886337.1),利用AMY-1引物扩增AMY基因CDS序列,PCR反应体系15.0 μL:2×Taq Master Mix 7.5 μL,上、下游引物各0.5 μL,cDNA模板1.0 μL,RNase-free ddH2O 5.5 μL。扩增程序:95.0 ℃预变性5 min;95.0 ℃ 30 s,57.6 ℃ 30 s,72.0 ℃ 1 min,进行35个循环;72.0 ℃延伸5 min,4 ℃保存。PCR扩增产物经1.5%琼脂糖凝胶电泳检测后,利用胶回收试剂盒进行目的条带回收及纯化,过夜连接至pMD18-T载体后转化Trans5α感受态细胞,挑取阳性克隆进行菌液PCR鉴定,并送至生工生物工程(上海)股份有限公司进行测序。
1. 4 AMY基因生物信息学分析
利用ExPASy ProtParam(https://web.expasy.org/protparam/)分析罗氏沼虾AMY基因编码蛋白理化性质;采用ExPASy ProtScale(https://web.expasy.org/protscale/)进行亲/疏水性预测;运用SignalP-5.0(http://www.cbs.dtu.dk/services/SignalP/)预测其信号肽;使用SMART(http://smart.embl.de/)进行结构域预测;利用MLRC(https://npsa-prabi.ibcp.fr/NPSA/npsa_mlrc.html)和SWISS-MODEL(http://swissmodel.expasy.org/)分别进行编码蛋白的二、三级结构预测;并以DNASTAR中的MegAlign和Lasergene v8.0分别进行同源比对及系统发育进化树分析。
1. 5 AMY基因表达分析
基于克隆获得的AMY基因CDS序列,以18S rRNA序列(DQ642856.1)为内参基因(俞炎琴,2013),利用Primer 3(http://primer3.ut.ee/)和Oligo 7.0设计扩增引物(表1),通过实时荧光定量PCR检测罗氏沼虾AMY基因的表达情况。PCR反应体系20.0 μL:Premix Ex Taq? II 10.0 μL,cDNA模板5.0 μL,正、反向引物各0.5 μL,RNase free ddH2O 4.0 μL。扩增程序:95.0 ℃预变性30 s;95.0 ℃ 5 s,60.0 ℃ 30 s,进行40个循环;72.0 ℃延伸5 min,4.0 ℃保存。每个样品进行3次生物学重复,采用2?ΔΔCt法计算AMY基因的相对表达量(Livak and Schmittgen,2001)。
2 结果与分析
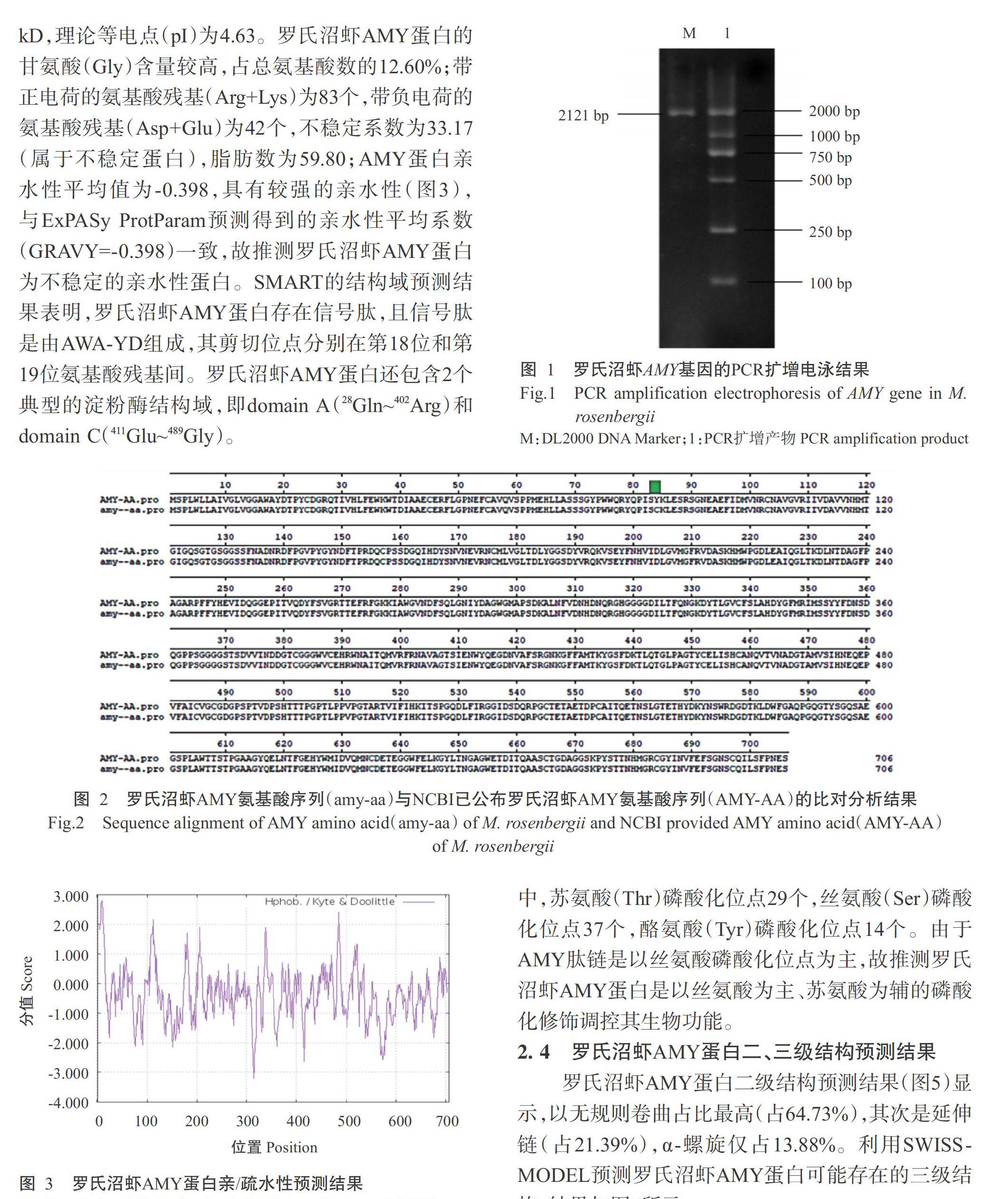
2. 1 罗氏沼虾AMY基因CDS序列及测序分析结果
PCR扩增产物经1.5%琼脂糖凝胶电泳检测,结果获得1条明亮清晰、大小约2000 bp的特异性条带(图1),与预期结果相符。测序结果显示,罗氏沼虾AMY基因CDS序列全长2121 bp,共编码706个氨基酸残基;氨基酸序列比对分析发现其与NCBI已发布罗氏沼虾(GenBank登录号KM886337.1)相应氨基酸序列的相似性达99.9%,仅是第251位碱基由A→G,导致第84位酪氨酸(Tyr)突变为半胱氨酸(Cys)(图2)。
2. 2 罗氏沼虾AMY蛋白理化性质预测分析结果
罗氏沼虾AMY基因编码蛋白分子量为76.87 kD,理论等电点(pI)为4.63。罗氏沼虾AMY蛋白的甘氨酸(Gly)含量较高,占总氨基酸数的12.60%;带正电荷的氨基酸残基(Arg+Lys)为83个,带负电荷的氨基酸残基(Asp+Glu)为42个,不稳定系数为33.17(属于不稳定蛋白),脂肪数为59.80;AMY蛋白亲水性平均值为-0.398,具有较强的亲水性(图3),与ExPASy ProtParam预测得到的亲水性平均系数(GRAVY=-0.398)一致,故推测罗氏沼虾AMY蛋白为不稳定的亲水性蛋白。SMART的结构域预测结果表明,罗氏沼虾AMY蛋白存在信号肽,且信号肽是由AWA-YD组成,其剪切位点分别在第18位和第19位氨基酸残基间。罗氏沼虾AMY蛋白还包含2个典型的淀粉酶结构域,即domain A(28Gln~402Arg)和domain C(411Glu~489Gly)。
2. 3 罗氏沼虾AMY蛋白糖基化位点和磷酸化位点预测结果
罗氏沼虾AMY蛋白不存在糖基化位点;其磷酸化位点预测结果(图4)显示,在AMY肽链中可能发生磷酸化且分值在0.5以上的氨基酸位点有80个,其中,苏氨酸(Thr)磷酸化位点29个,丝氨酸(Ser)磷酸化位点37个,酪氨酸(Tyr)磷酸化位点14个。由于AMY肽链是以丝氨酸磷酸化位点为主,故推测罗氏沼虾AMY蛋白是以丝氨酸为主、苏氨酸为辅的磷酸化修饰调控其生物功能。
2. 4 罗氏沼虾AMY蛋白二、三级结构预测结果
罗氏沼虾AMY蛋白二级结构预测结果(图5)显示,以无规则卷曲占比最高(占64.73%),其次是延伸链(占21.39%),α-螺旋仅占13.88%。利用SWISS-MODEL预测罗氏沼虾AMY蛋白可能存在的三级结构,结果如图6所示。
2. 5 罗氏沼虾AMY基因同源性比对分析及系统发育进化树构建
根据NCBI已公布的AMY基因EST序列,可知罗氏沼虾AMY基因CDS序列编码706个氨基酸残基。针对罗氏沼虾(AKL71614.1)、斑节对虾(AME17649.1)、凡纳滨对虾(AIJ02079.1)、太平洋牡蛎(CAA69658.1)、褐虾(AWU67110)、合浦珠母贝(AGN55419.1)、克氏原螯虾(AXC43909.1)、大珠母贝(AEI58897.1)及中華绒螯蟹(ANG56301.1)的AMY氨基酸序列进行同源比对分析,结果(图7)显示,罗氏沼虾与克氏原螯虾的相似性最高(62.6%),与大珠母贝的相似性较低(48.9%)。基于AMY氨基酸序列相似性构建的系统发育进化树(图8)显示,罗氏沼虾与克氏原螯虾的亲缘关系最近,而与中华绒螯蟹的亲缘关系较远。
2. 6 罗氏沼虾AMY基因的组织表达差异
如图9所示,AMY基因在雄性罗氏沼虾不同组织中的相对表达量以肝胰腺最高,其次是胃,鳃组织的相对表达量最低;在雌性罗氏沼虾中以肝胰腺的相对表达量最高,其次是胃,而肌肉的相对表达量最低。对比相同组织不同性别个体的AMY基因相对表达量发现,罗氏沼虾AMY基因在雌性个体肠道中的相对表达量极显著高于雄性个体(P<0.01,下同),鳃组织中的相对表达量显著高于雄性个体(P<0.05),肝胰腺中的相对表达量极显著低于雄性个体,而腹神经节、胃、心脏、性腺和肌肉中的相对表达量差异不显著(P>0.05)。可见,AMY基因在罗氏沼虾不同组织中广泛表达,但存在性别差异性和组织特异性。
2. 7 罗氏沼虾AMY基因在不同生长速度家系个体中的表达差异
为探究罗氏沼虾AMY基因在不同生长速度家系个体中的表达规律,以体重和体长等生长性状表型值差异极显著的FG和SG个体为供试材料,实时荧光定量PCR检测不同生长速度家系个体肌肉中的AMY基因表达情况。由图10可知,罗氏沼虾AMY基因在FG个体肌肉中的相对表达量极显著高于SG个体的相对表达量。
3 讨论
AMY是甲壳类动物体内主要的消化酶类,对其新陈代谢及生长发育等活动发挥着极其重要的作用(陈亮等,2009)。AMY活性对营养物质的消化和吸收效率有显著影响,进而影响机体的生长发育(Hidalgo et al.,1999;Huvet et al.,2008;李浩,2018;朱书礼等,2020)。为此,本研究以罗氏沼虾为研究对象,开展AMY基因克隆并进行生物信息学分析,结果显示,罗氏沼虾AMY基因CDS序列全长2121 bp,共编码706个氨基酸残基,其编码蛋白分子量为76.87 kD,理论等电点(pI)为4.63,属于不稳定的亲水性蛋白,包含2个典型的淀粉酶结构域,即domain A(28Gln~402Arg)和domain C(411Glu~489Gly);羅氏沼虾AMY氨基酸序列与克氏原螯虾AMY氨基酸序列的相似性最高,与大珠母贝AMY氨基酸序列的相似性较低;基于AMY氨基酸序列相似性构建的系统发育进化树显示,罗氏沼虾与克氏原螯虾的亲缘关系最近,而与中华绒螯蟹的亲缘关系较远。
本研究对罗氏沼虾AMY基因的组织表达差异进行分析,发现AMY基因在罗氏沼虾不同组织中广泛表达,但存在性别差异性和组织特异性,具体表现为:雌性个体肠道中的相对表达量极显著高于雄性个体,鳃组织中的相对表达量显著高于雄性个体,而肝胰腺中的相对表达量极显著低于雄性个体。AMY基因在雄性罗氏沼虾不同组织中的相对表达量排序为肝胰腺>胃>腹部神经>肠道>精巢>心脏>肌肉>鳃组织,在雌性罗氏沼虾中则表现为肝胰腺>胃>肠道>卵巢>腹神经节>心脏>鳃组织>肌肉,与唐小红等(2015)、栗志民等(2017)、杨其彬等(2017)的研究结果相似。罗氏沼虾AMY基因表达的性别差异性和组织特异性,与胰蛋白酶原基因(TRY)在黄羽肉鸡中的表达规律类似,TRY基因在雄性黄羽肉鸡肺脏中的相对表达量显著高于雌性黄羽肉鸡,而在肌肉中的相对表达量显著低于雌性黄羽肉鸡(许丽惠等,2018)。
本研究结果显示,罗氏沼虾AMY基因在生长快速家系个体肌肉中的相对表达量极显著高于生长慢速家系个体,与斑节对虾AMY基因的整个生长阶段均有表达,且在幼体发育过程中糠虾期的表达量达峰值(杨其彬等,2017),文蛤AMY基因在不同发育时期的壳顶幼虫期表达量最高(高晓艳,2015),以及草鱼出膜72 h后AMY基因表达量明显升高(唐小红等,2015)的研究结果相似。此外,AMY基因与甲壳类动物的生长性状显著相关(Prudence et al.,2006;Huvet et al.,2008;栗志民等,2017)。综合前人的相关研究结果可知,AMY基因在罗氏沼虾的生长发育过程中发挥重要作用,AMY基因表达量高的生长快速家系个体,其AMY活性相应也较高,因而生长速率快,与在中华绒螯蟹(田华梅等,2003)、华贵栉孔扇贝(邓岳文等,2008)、美洲龙虾(Perera et al.,2008)及马氏珠母贝(王庆恒等,2010)中研究发现AMY活性与生长速率紧密相关的结论相似,进一步提示AMY基因可能介导罗氏沼虾的生长发育。
4 结论
AMY基因在罗氏沼虾的生长发育过程中发挥重要作用,广泛表达于罗氏沼虾的不同组织中,但存在性别差异性和组织特异性。
参考文献:
陈亮,梁旭方,王琳,李观贵,林群,刘秀霞. 2009. 鳜鱼胰蛋白酶和淀粉酶与胃蛋白酶原基因的克隆与序列分析[J]. 中国生物化学与分子生物学报,25(12):1115-1123. doi:10.13865/j.cnki.cjbmb.2009.12.005. [Chen L,Liang X F,Wang L,Li G G,Lin Q,Liu X X. 2009. Molecular clo-ning and sequences analysis of trypsin,amylase and pepsinogen genes from Chinese perch(Siniperca chuatsi)[J]. Chinese Journal of Biochemistry and Molecular Biology,25(12):1115-1123.]
邓岳文,张凌飞,杜晓东,王庆恒,黄荣莲. 2008. 华贵栉孔扇贝两种壳色群体生长和消化酶活力比较[J]. 广东海洋大学学报,28(3):20-23. doi:10.3969/j.issn.1673-9159. 2008.03.005. [Deng Y W,Zhang L F,Du X D,Wang Q H,Huang R L. 2008. A comparison of growth and digestive enzymes between two shell color stocks of Huagui scallop Chlamys nobilis[J]. Journal of Guangdong Ocean University,28(3):20-23.]
高晓艳. 2015. 文蛤4个生长相关基因克隆、时空表达及与生长的相关性分析[D]. 宁波:浙江万里学院. [Gao X Y. 2015. Cloning,spatiotemporal expression and association analysis of 4 genes with growth traits in Hard clam Meretrix meretrix[D]. Ningbo:Zhejiang Wanli University.]
胡永乐,梁旭方,王琳,李观贵,刘秀霞,王云新,张海发. 2010. 斜带石斑鱼胰蛋白酶原和淀粉酶全长cDNA的克隆与序列分析[J]. 热带海洋学报,29(5):125-131. doi:10.3969/j.issn.1009-5470.2010.05.019. [Hu Y L,Liang X F,Wang L,Li G G,Liu X X,Wang Y X,Zhang H F. 2010. Mole-cular cloning and sequence analysis of pancrea-tic trypsinogen and amylase from orange-spotted grouper (Epinephelus coioides)[J]. Journal of Tropical Oceanography,29(5):125-131.]
姜建萍,袁翔,邱庆庆,黄光华,蒋钦杨,杨秀荣,蒋和生. 2019. 罗氏沼虾性别相关基因研究进展及其单性化养殖现状[J]. 南方农业学报,50(9):2111-2118. doi:10.3969/j.issn.2095-1191.2019.09.30. [Jiang J P,Yuan X,Qiu Q Q,Huang G H,Jiang Q Y,Yang X R,Jiang H S. 2019. Research progress of sex-related genes and research status of monosex culture in Macrobrachium rosenbergii:A review[J]. Journal of Southern Agriculture,50(9):2111-2118.]
李浩. 2018. 缢蛏选育系生长参数估计和α-淀粉酶基因外显子区域SNPs筛选及生长关联分析[D]. 上海:上海海洋大学. [Li H. 2018. Heritability estimates and SNPs detection of alpha amylase exon region and its association with growth traits about selected line of Sinonovacula constricta[D]. Shanghai:Shanghai Ocean University.]
栗志民,钱佳慧,刘建勇,艾加林,檀克勤. 2017. 九孔鲍α-淀粉酶基因克隆、表达分析及与生长性状相关性[J]. 南方水产科学,13(6):14-21. doi:10.3969/j.issn.2095-0780. 2017.06.002. [Li Z M,Qian J H,Liu J Y,Ai J L,Tan K Q. 2017. Molecular cloning,expression of alpha-amylase gene of Haliotis diversicolor supertexta and correlation with growth traits[J]. South China Fisheries Science,13(6):14-21.]
农业农村部渔政渔业管理局. 2019. 中国渔业统计年鉴[M]. 北京:中国农业出版社. [Bureau of Fisheries,Ministry of Agriculture and Rural Affairs. 2019. China fisheries statistics yearbook[M]. Beijing:China Agriculture Press.]
潘俐玲,黄桂菊,成书营,王晓宁,喻达辉. 2013. 大珠母贝(Pinctada maxima)α-淀粉酶基因cDNA及内含子克隆分析[J]. 热带海洋学报,32(3):52-58. doi:10.3969/j.issn. 1009-5470.2013.03.008. [Pan L L,Huang G J,Cheng S Y,Wang X N,Yu D H. 2013. Cloning and characterization of alpha amylase cDNA and its introns in the pearl oyster Pinctada maxima[J]. Journal of Tropical Oceanography,32(3):52-58.]
彭涛. 2015. 克氏原螯虾α-淀粉酶基因的克隆与功能分析[D]. 合肥:安徽农业大学. [Peng T. 2015. Cloning and functional analysis of α-amylase gene in Procambarus clarkii[D]. Hefui:Anhui Agricultural University.]
邱庆庆. 2019. 罗氏沼虾生长性状候选基因的克隆及mRNA表达规律研究[D]. 南宁:广西大学. [Qiu Q Q. 2019. Cloning and mRNA expression of candidate genes for growth performance of Macrobrachium rosenbergii[D]. Nanning:Guangxi University.]
宋晓红. 2014. 两种形态变异类型日本囊对虾α-淀粉酶基因的克隆、表达与比較[D]. 厦门:厦门大学. [Song X H. 2014. The cloning,expression and comparison of α-amylase genes for the two morphologically similar varieties of Marsuprnaeus japonicus[D]. Xiamen:Xiamen University.]
唐小紅,樊佳佳,白俊杰. 2015. 草鱼α-淀粉酶基因组织表达特征和早期发育的表达谱[J]. 海洋渔业,37(1):31-37. doi:10.13233/j.cnki.mar.fish.2015.01.005. [Tang X H,Fan J J,Bai J J. 2015. Expression analysis of α-amylase gene in various tissues and early development of Ctenopharyngodon idellus[J]. Marine Fisheries,37(1):31-37.]
田华梅,王群,赵云龙,罗文,樊玉杰. 2003. 中华绒螯蟹胚胎发育过程中的消化酶活力及氨基酸组成[J]. 中国水产科学,10(5):404-408. doi:10.3321/j.issn:1005-8737.2003. 05.010. [Tian H M,Wang Q,Zhao Y L,Luo W,Fan Y J. 2003. Digestive enzyme activities and amino acids composition during embryonic development of Eriocheir sinensis[J]. Journal of Fishery Sciences of China,10(5):404-408.]
王庆恒,张善发,杜晓东,邓岳文,黄荣莲. 2010. 生长适温下马氏珠母贝黄壳色选系F1与养殖群体消化酶活力的比较[J]. 中国水产科学,17(2):252-257. [Wang Q H,Zhang S F,Du X D,Deng Y W,Huang R L. 2010. A comparative analysis of digestive enzyme activities of first gene-ration yellow shell color and cultivated stocks of Pinctada martensii at suitable growth temperatures[J]. Journal of Fishery Sciences of China,17(2):252-257.]
辛静静,刘小林,李喜莲,王宪总,黄皓,相建海. 2011. 凡纳滨对虾α-淀粉酶基因PCR-RFLP多态性与生长性状的相关分析[J]. 海洋学报,33(3):124-130. [Xin J J,Liu X L,Li X L,Wang X Z,Huang H,Xiang J H. 2011. PCR-RFLP polymorphism of alpha-amylase gene and its association with growth traits of Litopenaeus vannamei[J]. Acta Oceanologica Sinica,33(3):124-130.]
许丽惠,胡世忠,吴卓秋,詹湉湉,王全溪,王长康. 2018. 黄羽肉鸡胰蛋白酶原的原核表达及其在体内分布的研究[J]. 中国兽医科学,48(9):1176-1186. doi:10.16656/j.issn. 1673-4696.2018.0170. [Xu L H,Hu S Z,Wu Z Q,Zhan T T,Wang Q X,Wang C L. 2018. Studies on prokaryotic expression and distribution in vivo of trypsinogen in ye-llow broiler[J]. Chinese Veterinary Science,48(9):1176-1186.]
杨其彬,李运东,江世贵,黄建华,姜松,邱丽华,朱彩艳,周发林. 2017. 斑节对虾α-淀粉酶基因的克隆及其表达分析[J]. 水生生物学报,41(6):1186-1192. doi:10.7541/2017. 147. [Yang Q B,Li Y D,Jiang S G,Huang J H,Jiang S,Qiu L H,Zhu C Y,Zhou F L. 2017. Cloning and expression analysis of alpha amylase cDNA of Penaeus Monodon[J]. Acta Oceanologica Sinica,41(6):1186-1192.]
俞炎琴. 2013. 罗氏沼虾中性别发育相关基因Sxl和Dmrt基因的分子特征和功能研究[D]. 杭州:浙江大学. [Yu Y Q. 2013. The molecular characterization and functional ana-lysis of sexual development related genes Sxl and Dmrt in the prawn,Macrobrachium rosenbergii[D]. Hangzhou:Zhejiang University.]
朱书礼,张迎秋,陈蔚涛,杨计平,李捷,武智,李新辉. 2020. 胰α-淀粉酶基因5'端调控序列与鱼类食性的关系[J]. 中国水产科学,27(3):277-285. doi:10.3724/SP.J.1118.2020. 19202. [Zhu S L,Zhang Y Q,Chen W T,Yang J P,Li J,Wu Z,Li X H. 2020. Analysis of the relationship between the pancreatic alpha amylase gene 5? flanking sequence and the feeding habits of fish[J]. Journal of Fi-shery Sciences of China,27(3):277-285.]
Hidalgo M C,Urea E,Sanz A. 1999. Comparative study of digestive enzymes in fish with different nutritional habits. Proteolytic and amylase activities[J]. Aquaculture,170(3-4):267-283. doi:10.1016/S0044-8486(98)00413-X.
Huvet A,Jeffroy F,Fabioux C,Daniel J Y,Quillien V,van Wormhoudt A,Moal J,Samain J F,Boudry P,Pouvreau S. 2008. Association among growth,food consumption-related traits and amylase gene polymorphism in the Paci-fic oyster Crassostrea gigas[J]. Animal Genetics,39(6):662-665. doi:10.1111/j.1365-2052.2008.01776.x.
Karasov W H,Douglas A E. 2013. Comparative digestive physiology[J]. Comprehensive Physiology,3(2):741-783. doi:10.1002/cphy.c110054.
Livak K J,Schmittgen T D. 2001. Analysis of relative gene expression data using real-time quantitative PCR and the 2?ΔΔCT method[J]. Methods,25(4):402-408. doi:10.1006/meth.2001.1262.
Perera E,Moyano F J,Díaz M,Perdomo-Morales R,Montero-Alejo V,Rodríguez-Viera L,Alonso E,Carrillo O,Galich G S. 2008. Changes in digestive enzymes through deve-lopmental and molt stages in the spiny lobster,Panulirus argus[J]. Comparative Biochemistry and Physiology. Part B:Biochemistry and Molecular Biology,151(3):250-256. doi:10.1016/j.cbpb.2008.07.005.
Prudence M,Moal J,Boudry P,Daniel J Y,Quéré C,Jeffroy F,Mingant C,Ropert M,Bédier E,van Wormhoudt A,Samain J F,Huvet A. 2006. An amylase gene polymorphism is associated with growth differences in the Pacific cupped oyster Crassostrea gigas[J]. Animal Genetics,37(4):348-351. doi:10.1111/j.1365-2052.2006.01481.x.
Rodríguez-Viera L,Perera E,Montero-Alejo V,Perdomo-Morales R,García-Galano T,Martínez-Rodríguez G,Mancera J M. 2017. Carbohydrates digestion and metabolism in the spiny lobster(Panulirus argus):Biochemical indication for limited carbohydrate utilization[J]. PeerJ,5:e3975. doi:10.7717/peerj.3975.
Simon C J. 2009. Digestive enzyme response to natural and formulated diets in cultured juvenile spiny lobster,Jasus edwardsii[J]. Aquaculture,294(3-4):271-281. doi:10. 1016/j.aquaculture.2009.06.023.
(責任编辑 兰宗宝)

