栽培番茄与野生番茄NBS-LRR类抗病基因家族的全基因组鉴定及表达分析
史建磊 熊自立 苏世闻 张海利 宰文珊


摘要:【目的】對栽培番茄和野生番茄NBS-LRR类抗病基因家族进行全基因组鉴定及表达分析,为NBS-LRR类抗病基因功能研究及其在植物抗病育种中的应用提供理论参考。【方法】应用生物信息学和比较基因组学方法,从栽培番茄和野生番茄[潘那利番茄(Solanum pennellii)和醋栗番茄(Solanum pimpinellifolium)]基因组中鉴定出NBS-LRR类抗病基因家族成员,明确其类型,并进行序列特征、染色体定位、系统进化及生物胁迫下表达模式分析。【结果】从栽培番茄、潘那利番茄和醋栗番茄基因组中分别鉴定获得238、202和217个NBS-LRR类抗病基因家族成员,根据这些抗病基因编码的结构域差异,将其分为TNL(TIR-NBS-LRR)、CNL(CC-NBS-LRR)、RNL(RPW8-NBS-LRR)、TN(TIR-NBS)、CN(CC-NBS)、RN(RPW8-NBS)、NL(NBS-LRR)和N(NBS)8种类型,其中TNL、CNL和RNL型基因分别为58、87和13个。番茄NBS-LRR类抗病蛋白整体呈弱酸性,具有不稳定性和亲水性,以丝氨酸(Ser)磷酸化为主,二级结构以α-螺旋和无规则卷曲为主,定位于细胞核、细胞质和叶绿体。栽培番茄和野生潘那利番茄的NBS-LRR类抗病基因不均匀地散布在13条染色体(0号虚拟染色体~12号染色体)上,分别有58.47%和52.48%的基因形成多个基因簇,有35.59%和22.77%的基因形成串联重复。栽培番茄和2个野生番茄共477个NBS-LRR类抗病蛋白可聚为十大类群,其中N端为TIR的蛋白基本聚在一起,但N端为CC和RPW8结构域的蛋白未能很好分开,聚在多个类群中;NL和N型基因散布于各类群中。在477个番茄NBS-LRR类抗病基因中共发现115对直系同源基因和8对旁系同源基因,主要分布在4号和5号染色体上,其中51.57%的NBS-LRR类抗病基因以同源基因形式存在,大多数Ka/Ks均小于1,说明其进化中主要受到纯化选择。基于转录组测序数据(RNA-Seq)分析结果表明,多数NBS-LRR类抗病基因能响应不同细菌胁迫。【结论】番茄NBS-LRR类抗病基因具有成簇存在的特点,在进化过程中经重复扩张,已形成大量同源基因,且能响应多种细菌胁迫。
关键词: 番茄;NBS-LRR类抗病基因;序列特征;染色体定位;系统进化;生物胁迫;基因簇;基因重复
中图分类号: S641.203.6 文献标志码: A 文章编号:2095-1191(2021)05-1158-09
Abstract:【Objective】Genome-wide identification and expression analysis of NBS-LRR disease-resistantgenes in cultivated tomatoand its wild relatives were performed,to provide a theoretical basis for the research on the function of NBS-LRR disease-resistant genes and the application in plant disease resistance breeding. 【Method】NBS-LRR disease-resistant gene family members were identified and analysed on classification, sequence characteristics, chromosome location, phylogeny, and expression patterns under biotic stresses from cultivated(Solanum lycopersicum) and two wild(S. pennellii and S. pimpinellifolium) tomato species genomes using bioinformatics and comparative genomics methods. 【Result】In total,238,202 and 217 NBS-LRR disease-resistant genes were identified in cultivated tomato and S. pennellii and S. pim-pinellifolium genomes,respectively. Based on the differences in the domains encoded by these disease-resistant genes, they were divided into eight types,which were TNL(TIR-NBS-LRR), CNL(CC-NBS-LRR), RNL(RPW8-NBS-LRR), TN(TIR-NBS), CN(CC-NBS), RN(RPW8-NBS), NL(NBS-LRR) and N(NBS),with 58 TNL,87 CNL and 13 RNL genes,respectively. NBS-LRR disease-resistant genes were weakly acidic in general, and were unstable and hydrophilic,with serine(Ser) phosphorylation and secondary structure of alpha-helix and random coil,which were mainly located in the nucleus,cytoplasm and chloroplast. Chromosome localization revealed that the NBS-LRR disease-resistant genes in cultiva-ted tomato and S. pennellii genomes were unevenly distributed on 13 different chromosomes(virtual chromosome 0-chromosome12),with 58.47% and 52.48% genes forming multiple gene clusters,35.59% and 22.77% genes forming tandem duplicates,respectively. Systematic clustering divided 477 NBS-LRR disease-resistant genes in the three genomes into ten groups,the N-terminal TIR-type genes were basically clustered together,but CC-and RPW8-type genes were not well separated and clustered in multiple groups; NL- and N-type genes were scattered in various groups. Gene homology analysis revealed 115 pairs of orthologous genes and 8 pairs of paralogous genes in 477 NBS-LRR disease-resistantgenes, equivalent to 51.57% of NBS-LRR genes had a homologous gene,and that they were mainly distributed on chromosomes 4 and 5, with most Ka/Ks values being less than 1,indicating that purification selection had occurred in evolution. It revealed that the most NBS-LRR disease-resistant genes could respond to different bacterial stresses by transcriptome sequencing(RNA-seq)data analysis. 【Conclusion】The tomato NBS-LRR disease-resistant genes exist in cluster. During the evolution,a large number of homologous genes have appeared through gene expansion. Also,they can respond to a variety of bacterial stresses.
Key words:tomato; NBS-LRR disease-resistant gene;sequence characteristics; chromosomal localization;phyletic evolution;biotic stress; gene cluster;gene duplication
Foundation item: Major Science and Technology Project for Breeding New Agricultural(Vegetable) Varieties in Zhejiang(2016C02051-1-2); Wenzhou Basic Scientific Research Project(N20180003); General Scientific Research Pro-ject of Education Department in Zhejiang(Y201840413)
0 引言
【研究意义】植物在自然进化过程中面对病原微生物的侵染已形成一整套识别与防御机制,在此过程中抗病基因发挥着非常重要的作用。NBS-LRR是植物中最大抗病基因家族之一,目前已克隆的抗病基因多为NBS-LRR类抗病基因家族成员(Dangl and Jones,2001)。NBS-LRR类抗病蛋白具有核苷酸结合位点(Nucleotide-binding site,NBS)和富亮氨酸重复(Leucine-rich repeat,LRR)典型特征结构域(刘云飞等,2014a,2014b),通过与病原效应子结合,触发ETI(Effector-triggered immunity)免疫反应,从而抑制病原在植物细胞中的定殖与扩展。番茄(Solanum lycopersicum)是世界主要蔬菜作物和科研模式植物之一,同时其近缘野生种中含丰富的抗病虫和抗逆资源,鉴定与评价番茄NBS-LRR类抗病基因,对研究其数目特征、遗传进化和生物功能具有重要理论意义;同时在相关抗病基因深入挖掘的基础上,对番茄栽培品种性状改良和优良新品种选育也具有积极实践意义。【前人研究进展】随着越来越多植物基因组测序的完成,水稻(Oryza sativa)(Bai et al.,2002)、拟南芥(Arabidopsis thaliana)(Meyers et al.,2003)、苜蓿(Medicago sativa)(Ameline-Torregrosa et al.,2008)、马铃薯(S. tuberosum)(Lozano et al.,2012)、玉米(Zea mays)(Cheng et al.,2012)、番茄(刘云飞等,2014b)、猕猴桃(Actinidia chinensis)(文欢和刘永立,2015)、大麦(Hordeum vulgare)(Habachi-Houimli et al.,2018)、蝴蝶兰(Phalaenopsis aphrodite)(蒋卉等,2018)、桑树(Morus alba)(刘潮等,2019)、谷子(李任建等,2020)等多种植物NBS-LRR类抗病基因已被鉴定。研究结果显示,NBS-LRR类抗病蛋白主要包含由氨基端(N端)到羧基端(C端)的TIR/CC、NBS和LRR 3个结构域(Meyers et al.,2003),有的还含有RPW8等结构域(Xiao et al.,2001)。其中,NBS又称NB-ARC,是最保守的结构域,由8个保守基序组成(Meyers et al.,2003),通过结合水解ATP和GTP并进行信号转导,调节随后的病原物识别(Fenyk et al,2012);LRR由多个亮氨酸或脯氨酸和天冬氨酸组成(Lozano et al.,2012),是最主要的病原物特异识别区,通过蛋白互作识别病原物并决定抗病基因的专化性和抗病谱(Bai et al.,2002)。根据N端是否存在TIR(Toll/interleukin-1 receptor like)结构域,可将NBS-LRR类抗病基因分为TNL(TIR-NBS-LRR)和nTNL(non-TIR-NBS-LRR)两大类,后者通常会编码卷曲螺旋(Coiled-coil)结构,也称为CNL(CC-NBS-LRR)(Meyers et al.,2003)。但并非所有NBS-LRR类抗病蛋白均具有3个结构域,通常会缺少N端或C端结构域。【本研究切入点】目前番茄全基因组测序已完成(Sato et al.,2012),为在全基因组水平上鑒定NBS-LRR类抗病基因提供了基础条件。目前尽管已有番茄NBS-LRR类抗病基因家族的全基因组鉴定,但鲜见有关野生番茄NBS-LRR类抗病基因家族的研究报道。【拟解决的关键问题】应用生物信息学和比较基因组学方法,从栽培番茄和野生番茄[潘那利番茄(S. pennellii)和醋栗番茄(S. pimpinellifolium)]基因组鉴定出NBS-LRR类抗病基因家族成员,明确其类型,并进行序列特征、染色体定位、系统进化及生物胁迫下表达模式分析,为番茄相关基因功能验证及其在抗病育种中的应用提供理论依据。
1 材料与方法
1. 1 番茄NBS-LRR类抗病基因家族成员鉴定
从茄科基因组网站SGN(http://solgenomics.net/)下载番茄基因组序列及其注释信息,并利用BioEdit 7.2.5构建本地数据库。在Pfam(http://pfam.xfam.org/)中下载NB-ARC(PF00931)氨基酸序列,并以此为靶序列进行BLASTp(E-value<1)比对检索,获得候选基因。然后通过Pfam、SMART和COILS Server鉴定TIR、CC、RPW8、NBS和LRR等结构域,将不含NBS结构域编码序列的候选基因排除。
1. 2 番茄NBS-LRR类抗病基因序列特征分析
利用ExPASy在线网站的Compute pI/MW工具计算蛋白分子量和理论等电点(pI);以ProtParam在线网站进行蛋白理化性质预测;利用ProtScale分析蛋白质亲/疏水性;运用NetPhos 3.1 Server在线程序分析蛋白磷酸化位点;采用ExPASy在线网站的SOPMA预测蛋白二级结构;利用WoLF PSORT在线工具进行蛋白亚细胞定位预测;并以WebLogo 3在线工具绘制保守序列Logo图。
1. 3 番茄NBS-LRR类抗病基因定位
结合SGN网站中的基因注释信息,利用MapDraw 2.1进行基因染色体定位并绘制其分布图。随后对番茄NBS-LRR类抗病基因进行基因簇和串联重复分析,划分基因簇的原则:(1)2个相邻基因间距小于200 kb;(2)2个相邻基因间其他基因不多于8个(Meyers et al.,2003;Yang et al.,2008);判断串联重复的原则:(1)相邻基因距离小于100 kb;(2)基因间相似度大于70%(Huang et al.,2012)。采用在线程序PAL2NAL(http://www.bork.embl.de/pal2nal/index.cgi?example=Yes#RunP2N)计算基因间的选择压力。
1. 4 番茄NBS-LRR类抗病基因系统进化分析
首先提取出NBS结构域的氨基酸序列,利用ClustalX 1.83进行多序列比对,再以MEGA 7.0的邻接法(Neighbor-joining,NJ)(参数设置:Bootstrap method 1000,Poisson model,and Pairwise deletion)构建系统发育进化树,去除Bootstrap支持率低于50%的节点。采用DNAMAN 7.0计算序列间的相似性。
1. 5 番茄NBS-LRR类抗病基因表达分析
从番茄功能基因组数据库TFGD(http://ted.bti.cornell.edu/cgi-bin/TFGD/digital/home.cgi)下载番茄转录组测序(RNA-Seq)数据(D007),利用MeV 4.9.0绘制基因的表达热图,并分析基因的表达模式。
2 结果与分析
2. 1 番茄NBS-LRR类抗病基因家族成员鉴定结果
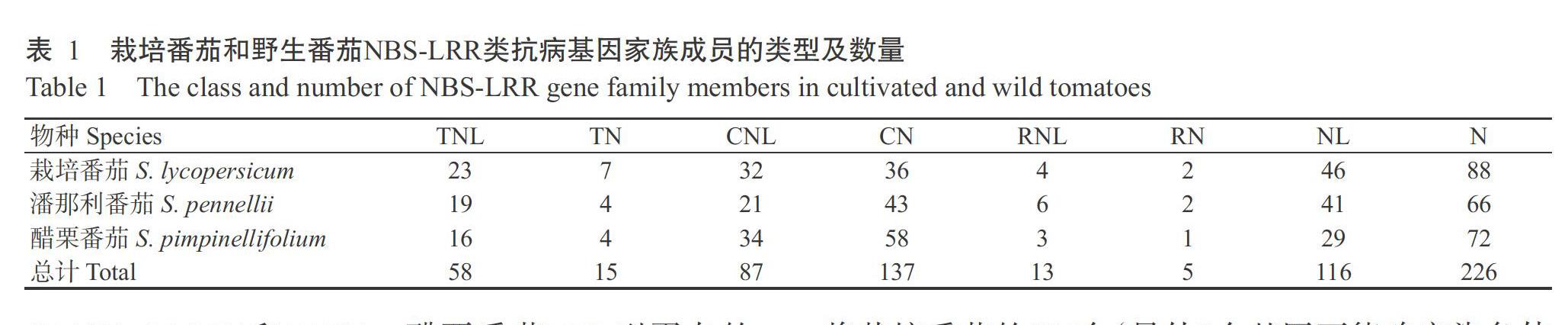
从栽培番茄及野生潘那利和醋栗番茄的基因组序列中分别检索获得238、202和217个NBS-LRR类抗病基因。根据这些抗病基因编码蛋白的结构域差异,将其分为TNL(TIR-NBS-LRR)、CNL(CC-NBS-LRR)、RNL(RPW8-NBS-LRR)、TN(TIR-NBS)、CN(CC-NBS)、RN(RPW8-NBS)、NL(NBS-LRR)和N(NBS)8种类型(表1)。其中,N型基因最多,为226个,占NBS-LRR类抗病基因总数(657个)的34.4%;TNL、CNL和RNL型基因分别为58、87和13个,共计158个。
2. 2 番茄NBS-LRR类抗病蛋白理化性质、二级结构及亚细胞定位结果
158个番茄TNL、CNL和RNL型NBS-LRR类抗病蛋白的理化性质、二级结构及亚细胞定位如表2所示。栽培番茄TNL型蛋白的氨基酸数为856~2871个,分子量为98.40~325.87 kD,pI为5.43~8.77,不稳定系数为37.88~51.45,磷酸化氨基酸平均为136个,其中丝氨酸(Ser)、苏氨酸(Thr)和酪氨酸(Tyr)分别占比67.11%、22.58%和10.30%;nTNL型蛋白的氨基酸数为696~2613个,分子量为79.05~298.53 kD,pI为4.85~9.03,不稳定系数为37.93~50.34,磷酸化氨基酸数平均为100个,其中Ser、Thr和Tyr分别占比64.69%、27.07%和8.24%。潘那利番茄TNL型蛋白的氨基酸数为673~2871个,分子量为76.33~326.23 kD,pI为5.48~8.96,不稳定系数为37.25~52.73,磷酸化氨基酸数平均为118个,其中Ser、Thr和Tyr分别占比66.87%、22.78%和10.34%;nTNL型蛋白的氨基酸数为617~1687个,分子量为69.79~194.21 kD,pI为5.34~8.62,不稳定系数为39.82~52.41,磷酸化氨基酸数平均为101个,其中Ser、Thr和Tyr分别占比67.42%、24.80%和7.78%。醋栗番茄TNL型蛋白的氨基酸數为973~2871个,分子量为111.40~325.79 kD,pI为5.42~9.23,不稳定系数为40.45~51.45,磷酸化氨基酸数平均为128个,其中Ser、Thr和Tyr分别占比67.02%、22.86%和10.11%;nTNL型蛋白的氨基酸数为645~2595个,分子量为73.57~296.37 kD,pI为4.85~8.91,不稳定系数为38.96~49.79,磷酸化氨基酸数平均为95个,其中Ser、Thr和Tyr分别占比64.29%、27.14%和8.56%。番茄NBS-LRR类抗病蛋白整体上呈弱酸性,且具有不稳定性和亲水性,以Ser磷酸化为主,其二级结构以α-螺旋和无规则卷曲为主,主要定位于细胞核、细胞质和叶绿体。
对番茄TNL、CNL和RNL型NBS-LRR类抗病蛋白的保守结构域进行分析,绘制得到相应氨基酸序列的Logo图(图1-A、图1-B和图1-C)。保守氨基酸组成的NBS结构域P-loop、Kinase2、RNBS-B和GLPL,TNL和nTNL型NBS-LRR类抗病基因不完全一致,如TNL型基因NBS保守结构域的Kinase2一般以天冬氨酸(Asp)结尾,而nTNL型一般以色氨酸(Trp)结尾。说明番茄NBS-LRR类抗病基因在进化过程中具有较高的保守性。
2. 3 番茄NBS-LRR类抗病基因染色体定位结果
通过获取番茄NBS-LRR类抗病基因的注释信息(醋栗番茄基因组注释不完整,故未作分析),分别将栽培番茄的236个(另外2个基因不能确定染色体位置)和野生潘那利番茄的202个NBS-LRR类抗病基因分别不均匀地散布于12条染色体和0号虚拟染色体上(图2-A和图2-B)。番茄NBS-LRR类抗病基因集中在染色体端部,且具有成簇分布的特点。栽培番茄各染色体的基因数为1(0号虚拟染色体)~45(11号染色体)个,平均为18.15个,其中138个(58.47%)基因形成47个基因簇,平均每簇有2.94个基因,4号染色体10簇37个基因,最大簇7个基因,6号和4号染色体成簇基因均超过80.00%,其他染色体成簇基因数为0~16个;同时,84个(35.59%)基因为串联重复基因,以4号染色体的串联重复基因最多,为29个,6号染色体上串联重复基因比例最大,为76.92%。潘那利番茄各染色体的基因数为3(0号虚拟染色体)~31(11號染色体)个,平均为15.54个,其中,106个(52.48%)基因形成39个基因簇,平均每簇2.72个基因,11号染色体11簇24个基因,4号染色体7簇23个基因,6号染色体最大簇8个基因,11号、4号和6号染色体成簇基因均超过70.00%,其他染色体成簇基因数为0~14个;同时,46个(22.77%)基因为串联重复基因,以4号染色体最多,为17个,6号和4号染色体上串联重复基因均超过50.00%。整体来看,番茄4号、6号和11号染色体NBS-LRR类抗病基因成簇和串联重复特点最明显。
2. 4 番茄NBS-LRR类抗病蛋白系统进化分析结果
提取NBS-LRR类抗病蛋白NBS结构域的氨基酸序列,去掉氨基酸序列过短的180个蛋白,对477个NBS-LRR类抗病蛋白进行多重序列比对分析,结果发现这些抗病蛋白含有多个相对保守的氨基酸位点,如24-I、26/29/31/127/188-G、32-K、33/34/133/134-T、36/192-A、47/166-F、102/105/152/160/189/191/200/234-L、106/107-D、128/230-S、135-R、190-P和208-W等。由系统发育进化树(图3)可知,栽培番茄和2个野生番茄共477个NBS-LRR类抗病蛋白可聚为十大类群,其中,N端为TIR结构域的蛋白中仅有1个蛋白聚在Ⅰ类群,其他65个蛋白聚在Ⅴ类群;N端为CC结构域的蛋白聚在Ⅱ、Ⅲ、Ⅳ、Ⅶ、Ⅷ和Ⅹ等6个类群;N端为RPW8结构域的蛋白聚在Ⅱ、Ⅲ、Ⅵ和Ⅸ等4个类群。因此,N端为TIR的蛋白基本聚在一起,但N端为CC和RPW8结构域的蛋白未能很好分开,聚在多个类群中;NL和N型基因散布于各类群中。
结合477个番茄NBS-LRR抗病基因的聚类分析结果及其核苷酸序列相似性,结果共发现115对直系同源基因和8对旁系同源基因,占比51.57%,主要分布在4号和5号染色体上。其中,栽培番茄分别与野生醋栗番茄和潘那利番茄形成97(84.35%)和11(9.57%)对直源基因,2个野生番茄间形成7(6.09%)对直源基因,且同源基因对所在染色体对应,4号和5号染色体分别为27和16对,其他染色体为5~9对。野生潘那利番茄和栽培番茄各自分别形成7和1对旁源基因。计算同源基因间非同义(Ka)和同义(Ks)替换率及Ka/Ks值,结果发现有4对直源基因(Solyc04 g009120.2和Sopim04g009120、Solyc04g009130.3和Sopim04g009130、Solyc11g069020.2和Sopim11g06 9020、Solyc12g044190.2和Sopim12g044190)及2对旁源基因(Sopen10g029640和Sopen10g029660、Sopen11g028600和Sopen11g028610)的Ka/Ks值大于1,说明这6对同源基因受到自然正选择作用,其他同源基因对Ka/Ks值均小于1,为纯化选择。[Ⅰ][Ⅹ][Ⅸ][Ⅶ][Ⅵ][Ⅴ][Ⅳ][Ⅲ][Ⅱ][Ⅷ]
2. 5 番茄NBS-LRR类抗病基因在细菌胁迫下的表达情况
从54个栽培番茄TNL、CNL和RNL型NBS-LRR类抗病基因(其他5个基因数据缺失,2个野生番茄对应注释不可用,故未作分析)的表达聚类热图(图4)可看出,不同基因可明显分为两大类群,其中,Ⅰ类群的16个基因表达量较低,Ⅱ类群的38个基因表达量较高。其中,31个基因在4或5种生物胁迫下有较高表达量,10个基因在所有生物胁迫下表达量均较低。Solyc02g082050.3和Solyc01g087200.3在2种假单胞菌和根癌农杆菌胁迫下有较高表达量,Solyc08g0055 10.2在根癌农杆菌胁迫下表达量最高,Agrobacterium tumefaciens和DC3000hrcQ-UfliC胁迫下基因表达量整体偏低。[Ⅰ][Ⅱ]
3 讨论
植物NBS-LRR类抗病基因的数量和结构,在某种程度上代表了其识别病原物的能力。部分基因NBS结构域不完整,可能与基因进化过程中的丢失、假基因化或功能异化有关。本研究从栽培番茄及野生潘那利番茄和醋栗番茄中分别鉴定获得238、202和217个NBS-LRR类抗病基因,共计657个,根据这些基因结构的差异进一步将其划分为8种类型(TNL、CNL、RNL、TN、CN、RN、NL和N),各类型中的分布数目各不相同,其中,TNL、CNL和RNL型基因数分别为58、87和13个。前人在与番茄同属的马铃薯中发现419个NBS-LRR类抗病基因,其中TNL型基因37个、CNL型基因65个,二者比值为0.57(Lozano et al.,2012),与本研究中TNL和CNL型基因数比值(0.67)基本一致,但明显高于拟南芥的TNL和CNL型基因数比值1.80(Meyers et al.,2003)。这种不均匀分布可能是基因进化和植物环境适应的自然选择结果。RNL型蛋白包含RPW8结构域,与植物广谱抗性密切相关(Xiao et al.,2003)。磷酸化作为重要的蛋白表观修饰,参与基因的转录调控(唐文武等,2017)。而本研究发现番茄NBS-LRR类抗病蛋白以Ser磷酸化为主,主要是通过改变蛋白结构激活酶活性。此外,本研究对番茄NBS-LRR类抗病基因的NBS结构域进行保守性分析,发现P-loop、Kinase2、RNBS-B和GLPL序列具有较高的相似性,与拟南芥(Meyers et al.,2003)和蝴蝶兰(蒋卉等,2018)相关研究结果一致,表明不同物种NBS-LRR类抗病基因在进化过程中具有较高的保守性。进一步研究发现,NBS保守结构域的Kinase2末端的氨基酸(Asp或Trp)是区分CNL和TNL型的关键因素(Lozano et al.,2015)。本研究的染色体定位结果显示,栽培番茄和野生潘那利番茄的NBS-LRR类抗病基因不均匀地散布在12条染色体和0号虚拟染色体上,其中以4号、5号和11号染色体较多,且主要位于染色体两端,因此这些基因在进化过程中可能更易变异;栽培番茄和野生潘那利番茄均有超过50.00%的基因成簇分布,且分别有35.59%和22.77%的基因为串联重复基因。可见,番茄NBS-LRR类抗病基因具有在染色体上成簇分布的特征,以4号、6号和11号染色体最突出,与拟南芥(Meyers et al.,2003)和马铃薯(Lozano et al.,2012)研究结果相似。基因簇形式有利于抗病基因间的序列交换,且串联重复通常出现在基因簇中,表明串联重复在NBS-LRR抗病基因家族的扩张中具有重要作用,也是基因簇形成的主要原因(Liu et al.,2007;刘云飞等,2014a)。
本研究通过构建番茄NBS-LRR类抗病蛋白的系统发育进化树发现,大部分N端为TIR结构域的NBS-LRR类抗病蛋白聚类在一起,但N端为CC和RPW8结构域的蛋白未能很好分开,其中以N端为CC结构域的蛋白扩张程度最大,散布于6个类群,其原因可能是含TIR结构域的编码基因在进化过程中发生丢失现象,或含CC结构域的编码基因起源更早(Tarr and Alexander,2009)。一般而言,同一进化分支上的基因具有较高相似性,系统发育进化树包含不同染色体上基因组成的混合进化分支,反映出这些基因在染色体上的扩张情况,可能是基因产生新功能的源泉;同一进化分支包含不同物种基因,说明NBS-LRR类抗病基因可能按照物种特异性的方式进行扩张。本研究对番茄NBS-LRR类抗病基因与已克隆的NBS-LRR类抗病基因(Liu et al.,2007)进行比对分析,结果显示,NBS-LRR类抗病基因Solyc04g008130.2与Hero、Solyc06g008450.3与Mi-1、Solyc09g098130.2与Sw-5、Solyc09g018220.2与Tm-2、Solyc05g007850.2与Bs4、Solyc11g071995.1与I-2的相似性均在80%以上,说明其具有类似抗病虫害功能。115对直系同源基因主要源自栽培番茄与野生醋栗番茄之间,且主要分布在4号和5号染色体上,证明其具有较高的遗传相似性;8对旁系同源基因中有5对是串联重复形成,其中1对来自栽培番茄、4对来自野生潘那利番茄。串联重复基因中同源性偏低,可能与筛选条件过严有关,也可能是基因重复后在长期演化中发生了功能异化。除了6对同源基因外,其他同源基因(95.12%)的Ka/Ks均小于1,表明其受纯化选择作用,基因进化动力可能是遗传漂变(Edwards,2000)。
基于RNA-Seq的番茄NBS-LRR类抗病基因表达分析结果显示,大多数基因在4~5种生物胁迫下表达量较高。虽然抗病基因表达促使植株产生抗性,但大量抗病基因的高表达会导致植物细胞死亡,因此为减少植物负担,抗病基因表达量通常较其他基因低(Zhai et al.,2011)。同时,抗病基因的转录可能通过小RNA调控(Fei et al.,2013),或进化为假基因甚至在基因组中丢失。可见,植物在与环境长期互作中形成了完善的自我调节机制,以保证个体生存与群体繁衍。对于番茄NBS-LRR类抗病基因家族仍需进行针对性地生物学功能鉴定及挖掘,以推动相关抗病基因的农业生产应用。
4 结论
番茄NBS-LRR类抗病基因具有成簇存在的特点,在进化过程中经重复扩张,已形成大量同源基因,且能响应多种生物胁迫。
参考文献:
蒋卉,张晶,符真珠,董晓宇,王慧娟,李艳敏,高杰,王利民,张和臣. 2018. 蝴蝶兰NBS-LRR家族基因挖掘和生物信息学分析[J]. 分子植物育种,16(9):2786-2794. doi:10. 13271/j.mpb.016.002786. [Jiang H,Zhang J,Fu Z Z,Dong X Y,Wang H J,Li Y M,Gao J,Wang L M,Zhang H C. 2018. Mining and bioinformatics analysis of NBS-LRR gene family in Phalaenopsis[J]. Molecular Plant Breeding,16(9):2786-2794.]
李任建,申哲源,李旭凱,韩渊怀,张宝俊. 2020. 谷子NBS-LRR类基因家族全基因组鉴定及表达分析[J]. 河南农业科学,49(2):34-43. doi:10.15933/j.cnki.1004-3268.2020. 02.005. [Li R J,Shen Z Y,Li X K,Han Y H,Zhang B J. 2020. Genome-wide identification and expression analysis of NBS-LRR gene family in Setaria italica[J]. Journal of Henan Agricultural Sciences,49(2):34-43.]
刘潮,褚洪龙,韩利红,杨云锦,高永,唐利洲. 2019. 桑树NBS-LRR类基因家族的全基因组鉴定及其调控micro-RNAs分析[J]. 江苏农业学报,35(3):544-553. doi:10.3969/ j.issn.1000-4440.2019.03.007. [Liu C,Chu H L,Han L H,Yang Y J,Gao Y,Tang L Z. 2019. Genome-wide identification of NBS-LRR genes and regulation analysis by microRNAs in mulberry[J]. Jiangsu Journal of Agricultural Sciences,35(3):544-553.]
刘云飞,万红建,李志邈,叶青静,王荣青,阮美颖,姚祝平,周国治,韦艳萍,杨悦俭. 2014a. 植物NBS-LRR抗病基因的结构、功能、进化起源及其应用[J]. 分子植物育种,12(2):377-389. doi:10.13271/j.mpb.012.000377. [Liu Y F,Wan H J,Li Z M,Ye Q J,Wang R Q,Ruan M Y,Yao Z P,Zhou G Z,Wei Y P,Yang Y J. 2014. Analysis of plant NBS-LRR resistance gene:structure,function,origin,evolution and their application[J]. Molecular Plant Breeding,12(2):377-389.]
劉云飞,万红建,韦艳萍,李志邈,叶青静,王荣青,阮美颖,姚祝平,周国治,杨悦俭. 2014b. 番茄NBS-LRR抗病基因家族全基因组分析[J]. 核农学报,28(5):790-799. [Liu Y F,Wan H J,Wei Y P,Li Z M,Ye Q J,Wang R Q,Ruan M Y,Yao Z P,Zhou G Z,Yang Y J. 2014. Genome-wide analysis of NBS-LRR resistance genes in tomato[J]. Journal of Nuclear Agricultural Sciences,28(5):790-799.]
唐文武,吴秀兰,李桂花. 2017. 芸薹属作物花粉发育相关基因MS1的生物信息学分析[J]. 南方农业学报,48(12):2122-2128. doi:10.3969/j.issn.2095-1191.2017.12.02. [Tang W W,Wu X L,Li G H. 2017. Bioinformatics analysis for pollen development related gene MS1 of Brassica crops[J]. Journal of Southern Agriculture,48(12):2122-2128.]
文欢,刘永立. 2015. ‘红阳猕猴桃全基因组NBS-LRR类基因家族的生物信息学分析[J]. 果树学报,32(2):413-419. doi:10.13925/j.cnki.gsxb.20140408. [Wen H,Liu Y L. 2015. Bioinformatic analysis of the NBS-LRR gene family in kiwifruit ‘Hongyang[J]. Journal of Fruit Science,32(2):413-419.]
Ameline-Torregrosa C,Wang B B,O'Bleness M S,Deshpande S,Zhu H,Roe B,Young N D,Cannon S B. 2008. Identification and characterization of nucleotide-binding site-leucine-rich repeat genes in the model plant Medicago truncatula[J]. Plant Physiology,146(1):5-21. doi:10. 1104/pp.107.104588.
Bai J F,Pennill L A,Ning J C,Lee S W,Ramalingam J,Webb C A,Zhao B Y,Sun Q,Nelson J C,Leach J E,Hulbert S H. 2002. Diversity in nucleotide binding site-leucine-rich repeat genes in cereals[J]. Genome Research,12(12):1871-1884. doi:10.1101/gr.454902.
Cheng Y,Li X,Jiang H,Ma W,Miao W,Yamada T,Zhang M. 2012. Systematic analysis and comparison of nucleotide-binding site disease resistance genes in maize[J]. The FEBS Journal,279(13):2431-2443. doi:10.1111/j.1742-4658. 2012.08621.x.
Dangl J L,Jones J D. 2001. Plant pathogens and integrated defence responses to infection[J]. Nature,411(6839):826-833. doi:10.1038/35081161.
Edwards A W F. 2000. The genetical theory of natural selection[J]. Genetics,154:1419-1426. doi:10.1038/126595a0.
Fei Q L,Xia R,Meyers B C. 2013. Phased,secondary,small interfering RNAs in posttranscriptional regulatory networks[J]. The Plant Cell,25(7):2400-2415. doi:10.1105/ tpc.113.114652.
Fenyk S,Campillo A D S E,Pohl E,Hussey P J,Cann M J. 2012. A nucleotide phosphatase activity in the nucleotide binding domain of an orphan resistance protein from rice[J]. Journal of Biological Chemistry,287(6):402-4032. doi:10.1074/jbc.M111.314450.
Habachi-Houimli Y,Khalfallah Y,Mezghani-Khemakhem M,Makni H,Makni M,Bouktila D. 2018. Genome-wide identification,characterization,and evolutionary analysis of NBS-encoding resistance genes in barley[J]. 3 Biotech,8(11):453. doi:10.1007/s13205-018-1478-6.
Huang S,Gao Y,Liu J,Peng X,Niu X,Fei Z,Cao S,Liu Y. 2012. Genome-wide analysis of WRKY transcription factors in Solanum lycopersicum[J]. Molecular Genetics and Genomics,287(6):495-513. doi:10.1007/s00438-012-0696-6.
Liu J L,Liu X L,Dai L Y,Wang G L. 2007. Recent progress in elucidating the structure,function and evolution of di-sease resistance genes in plants[J]. Journal of Genetics and Genomics,34(9):765-776. doi:10.1016/S1673-8527(07)60087-3.
Lozano R,Ponce O,Ramirez M,Mostajo N,Orjeda G. 2012. Genomewide identification and mapping of NBS-enco-ding resistance genes in Solanum tuberosum group phureja[J]. PLoS One,7(4):e34775. doi:10.1186/s12864-015-1554-9.
Lozano R,Hamblin M T,Prochnik S,Jannink J L. 2015. Identification and distribution of the NBS-LRR gene family in the cassava genome[J]. BMC Genomics,16:360. doi:10.1371/journal.pone.0034775.
Meyers B C,Kozik A,Griego A,Kuang H,Michelmore R W. 2003. Genome-wide analysis of NBS-LRR-encoding genes in Arabidopsis[J]. The Plant Cell,15(4):809-834. doi:10.1105/tpc.009308.
Sato S,Tabata S,Hirakawa H,Asamizu E,Shirasawa K,Isobe S,Kaneko T,Nakamura Y,Shibata D,Aoki K. 2012. The tomato genome sequence provides insights into fleshy fruit evolution[J]. Nature,485(7400):635-641. doi:10. 1038/nature11119.
Tarr D E K,Alexander H M. 2009. TIR-NBS-LRR genes are rare in monocots:Evidence from diverse monocot orders[J]. BMC Research Notes,2(197):1-10. doi:10.1186/1756-0500-2-197.
Xiao S Y,Brown S,Patrick E. 2003. Enhanced transcription of the Arabidopsis disease resistance genes RPW8.1 and RPW8.2 via a salicylic acid-dependent amplification circuit is required for hypersensitive cell death[J]. The Plant Cell,15(1):33-45. doi:10.1105/tpc.006940.
Xiao S,Ellwood S,Calis O,Patrick E,Li T,Coleman M,Turner J G. 2001. Broad-spectrum mildew resistance in Arabidopsis thaliana mediated by RPW8[J]. Science,291(5501):118-120. doi:10.1126/science.291.5501.118.
Yang S,Zhang X,Yue J X,Tian D,Chen J Q. 2008. Recent duplications dominate NBS-encoding gene expansion in two woody species[J]. Molecular Genetics and Genomics,280(3):187-198. doi:10.1007/s00438-008-0355-0.
Zhai J X,Jeong D H,De Paoli E,Park S,Rosen B D,Li Y P,González A J,Yan Z,Kitto S L,Grusak M A,Jackson S A,Stacey G,Cook D R,Green P J,Sherrier D J,Meyers B C. 2011. Micrornas as master regulators of the plant NBS-LRR defense gene family via the production of phased,transacting siRNAs[J]. Genes and Development,25(23):2540-2553. doi:10.1101/gad.177527.111.
(責任编辑 陈 燕)

