基于eDNA技术的白洋淀微型生物群落监测
陈家琪,董丽,麻晓梅,田凯,白洁,赵彦伟
(北京师范大学环境学院,水环境模拟国家重点实验室,北京 100875)
白洋淀是华北地区最大的内陆浅水湖泊湿地,被誉为“华北之肾”,其对维持区域生态平衡和保护生物多样性发挥着生态安全屏障作用[1]。长期以来,受人为和自然双重胁迫的影响,白洋淀面临水质下降与水体富营养化[2]、湿地萎缩[3]、生物多样性下降[4]等生态环境问题,因此亟待加强生态管理与综合修复。生物与生态监测可以用来评估生态现状,查明存在的生态问题,识别退化的驱动因子,为管理与修复提供重要的基础信息与依据,开展白洋淀生物监测具有重要意义。
微型生物群落是生物监测的重要类群之一,其主要由细菌、真菌、藻类、原生动物以及小型后生动物等在显微镜下才能观测到的微小生物组成。微型生物群落是湿地生态系统中生物要素的重要组成,对外界环境条件的变化十分敏感,在物质循环和能量流动中发挥着重要作用,其个体或群落变化可以客观地反映水体变化[5-8],在生态监测实践中应用广泛。传统的基于形态学与生理生化特征的生物分类鉴定虽然能基本满足自然生态系统中常规物种的监测,但该方法在检测数量和种类上有先天不足,加之受鉴定人员的主观影响,很难对体积微小的微型生物实现精准识别。为实现对微型生物的全面评估,需要突破传统生物监测方法的瓶颈,探索高灵敏度的生物监测新技术。
环境DNA(Environmental DNA,eDNA)技术为微型生物群落监测提供了新手段。该技术是DNA 条形码与高通量测序方法的结合,又称eDNA 宏条形码(Metabarcoding)技术,其主要通过对目标DNA 片段进行高通量测序,将所得序列与标准数据库进行比对,获取环境中的物种组成及丰度等信息[9-10]。eDNA技术是近年生态学领域最重要的技术之一,其快速发展,推动了物种识别方法从传统形态学鉴定向分子生物学领域转变,极大提高了人类从生态系统中获取数据的能力。因具有通量高、易发现新物种,在探究生物群落组成和检测物种多样性方面更准确快捷等优势[11],eDNA 技术已被广泛应用在鱼类[12]、哺乳动物[13]、藻类[14-15]等多种生物群落的监测中,并逐渐被应用到入侵物种[16]和濒危物种[17]的研究中。eDNA 宏条形码技术在白洋淀的浮游动物监测[18]、沉积物细菌多样性研究[19]等方面有过初步应用,但尚未对白洋淀的微型生物群落开展过全面的监测和研究。
本研究采用eDNA 宏条形码技术,对白洋淀微型生物群落进行监测和分析,明确了白洋淀微型生物的分布和组成,比较论证了eDNA 方法在白洋淀生物监测中的灵敏度和高效性,揭示了白洋淀微型生物群落的多样性变化,探究了季节变化和空间变化对白洋淀微型生物群落的影响。
1 材料与方法
1.1 样品采集及形态学鉴定
选取白洋淀9 个点位采集水样(图1),采集时间分别为2020年4月(春季)、6月(夏季)、9月(秋季)和11 月(冬季)。每个采样点用水质采样器(JC-8000B)采集2 L 地表水,其中1 L 用于eDNA 宏条形码分析,将水样立即转移到冰中,并在6 h内进行抽滤,滤膜孔径为0.45 µm,直径为50 mm,抽滤体积为250~300 mL。抽滤后立即将滤膜取下,放置在2 mL 无菌离心管内,并在-20~-80 ℃范围内保存直至DNA提取。
为便于分析对比,采用形态学鉴定方法进行藻类鉴定。用于形态学鉴定的1 L水样立即加入10 mL 鲁哥试剂固定,静置48 h 后,抽取上层清液并将下层沉淀浓缩至30 mL 左右,用光学显微镜对浓缩后的样品进行鉴定。
1.2 DNA提取、PCR扩增和高通量测序
采用Magen Hipure 土壤DNA 试剂盒从样本中提取DNA,使用Qubit®dsDNA HS 分析试剂盒检测提取到DNA的浓度。
16S rRNA 测序被广泛应用在原核生物的分类鉴定中,真核生物则主要应用18S rRNA 测序,因微型生物既有原核生物又有真核生物,本研究同时进行了16S rRNA 和18S rRNA 测序。以20~30 ng DNA 为模板,采用包含序列“CCTACGGRRBGCASCAGKVRVGAAT”的正向引物和包含序列“GGACTACNVGGGTWTCTAATCC”的反向引物扩增原核生物16S rRNA 的V3 和V4 区。对于真核生物,扩增的是18S rRNA 的V7 和V8 区,其正反向引物分别包含序列“CGWTAACGAACGAG”和“AICCATTCAATCGG”。PCR 扩增体系为25 µL,其中包含TransStart 缓冲液2.5µL,dNTPs 2µL,正向引物和反向引物各1µL,模板DNA 20 ng。原核生物和真核生物的扩增方案不同,原核生物:94 ℃初始变性3 min,94 ℃变性5 s,57 ℃退火90 s,72 ℃延伸10 s,72 ℃最终延伸5 min,共循环24 次。真核生物:94 ℃初始变性5 min,94 ℃变性30 s,57 ℃退火30 s,72 ℃延伸30 s,72 ℃最终延伸5 min,共循环25次。扩增后的产物经1.5%琼脂糖凝胶电泳检测。
通过Qubit3.0荧光仪(Invitrogen,Carlsbad,CA)检测DNA文库浓度。将文库定量到10 nmol·L-1,按Illumina MiSeq(Illumina,San Diego,CA,USA)仪器使用说明书进行PE250/FE300 双端测序,由MiSeq 自带的MiSeq控制软件(MCS)读取序列信息。
1.3 生物信息学分析
双端测序得到的正反向Reads 首先进行两两组装连接,过滤拼接结果中含有N 的序列,保留序列长度大于200 bp的序列。经过质量过滤、去除嵌合体序列后,最终得到的序列用于可操作分类单元(Operational Taxonomic Units,OTU,等同于传统分类学中的“种”)分析,使用VSEARCH(1.9.6)进行序列聚类,序列的相似性设为97%。之后用RDP Classifier(Ribosomal Database Program)贝叶斯算法对OTU 的代表性序列进行物种分类学分析,并在不同物种分类水平下统计每个样本的群落组成,最后综合多篇文献中的微型生物名录,对检测到的结果进行筛选分类[20-22]。其中16S rRNA 的参考数据库为Silva 132[23],18S rRNA的参考数据库为Protist Ribosomal Reference(PR2)[24]。
本文所有的数据分析均是基于高通量测序获得的OTUs序列。为了直观地展示所检测到的微型生物群落的序列和分类信息,本研究将门水平的序列数和OTU 数进行了对数(lg)转换。需特别说明的是,真核生物的注释数据库存在分类信息和正常分类级别无法对应的情况(真菌类群和无脊椎后生动物类群),为进行统一,本研究在统计各分类级别数目时遵循了数据库的规则,但在后续的分析中仍然按照正常的分类处理。
基于上述结果,主要对藻类优势度、季节间差异及微型生物类群的多样性指数进行了计算,同时也比较分析了检出的白洋淀微型生物群落的组成和丰度以及序列数的四季变化。
1.3.1 藻类优势度
物种优势度(Y)表示原核藻类或真核藻类类群某一物种所占的优势程度,计算公式为:
式中:ni为第i种藻类的个数;N为所有藻类总个数;fi为第i种藻类在各采样点出现的频率[25]。为便于比较,本文对原核藻类和真核藻类优势度分别进行计算,其中传统形态学计算的是种水平的优势度,而eDNA 宏条形码计算的是OTU 的优势度。本文将优势度Y>0.02的藻类确定为优势种。
1.3.2 ANOSIM分析
ANOSIM 用于计算季节间差异,它是一种非参数检验方法,用于评估两组实验数据整体的相似性及相似的显著性。该方法主要有两个数值结果:R用于评估不同组间是否存在差异,P用于说明组间差异是否显著。
R值计算公式为:
式中:rb表示组间距离排名的平均值;rw表示组内距离排名的平均值;n表示样品总数[26]。R的范围为[-1,1],R>0说明组间差异大于组内差异,R<0说明组间差异小于组内差异。
P值通过置换检验(Permutation test)获得。当P<0.01时,差异显著。
本研究在R 语言中用vegan 包的anosim()函数分别对检测到的原核生物和真核生物进行了ANOSIM分析。
1.3.3 α多样性指数
选用香农-威纳指数(H)反映样本中不同微型生物群落的生物多样性,在R 语言中用vegan 包的diversity()函数进行计算,计算公式为:
式中:S为群落物种丰富度指数,即OTU 数;pi为物种i的相对丰度;x通常为2或e[27],本研究中选取2 为底数。
2 结果与分析
对36 个水样进行高通量测序,获得2 730 899 条原核生物序列和2 801 272 条真核生物序列。为保证数据可比性,按照每个样本36 897条原核生物序列和33 484条真核生物序列的标准对测序结果进行随机抽平处理,36个样本最终共得到1 328 292条原核生物序列(占抽平前原核生物序列的48.64%)和1 205 424 条真核生物序列(占抽平前真核生物序列的43.03%)。对抽平后的序列按照97%的相似性进行OTU 聚类,获得4 629个OTU(原核生物1 517个、真核生物3 112个),其中4 580 个OTU 能被数据库注释到门水平以上(占总OTU的98.94%)。
2.1 白洋淀微型生物群落多样性
2.1.1 微型生物群落组成
依据eDNA 宏条形码测序中的OTU 注释结果进行分类,检测到的白洋淀微型生物群落分为7 个类群,包括原核与真核生物,原核生物包括古细菌、细菌(除蓝藻门)、原核藻类,真核生物包括真核藻类、真菌、原生动物、无脊椎后生动物。其中,细菌(除蓝藻门)的序列数和OTU 数在原核生物中最多,原核藻类次之;真菌的序列数和OTU 数在真核生物中最多,原生动物次之,无脊椎后生动物最少,这些微型生物类群的序列数和OTU 数详见表1。按照这些OTU 所能注释到的最高分类水平进行统计,它们分属于50 个门、119个纲、228个目、386个科、614个属、567个种。
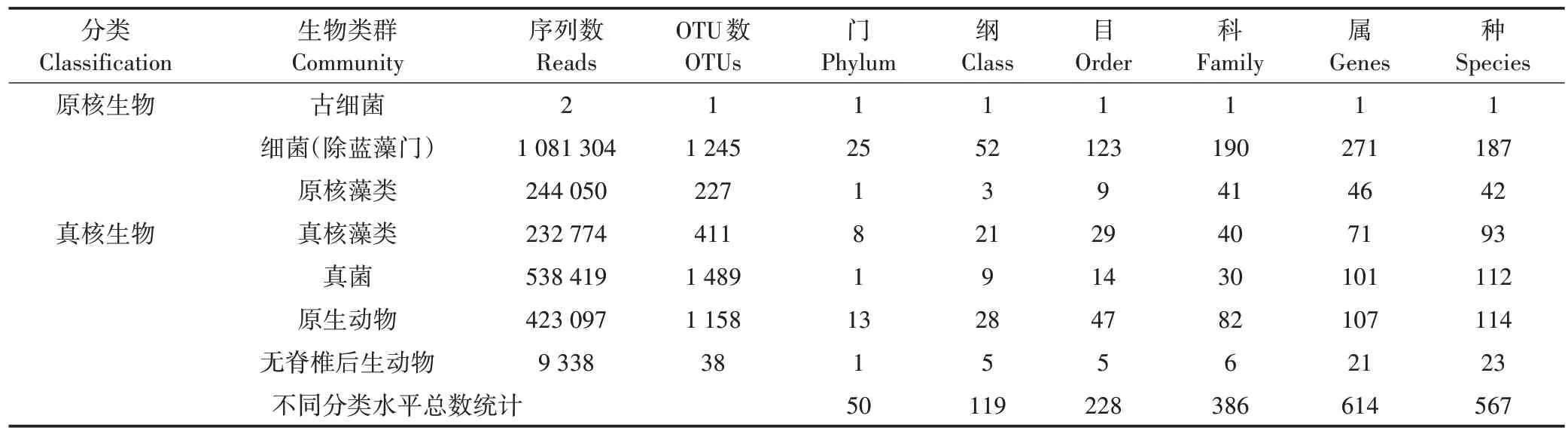
表1 微型生物群落注释结果Table1 The annotation results of microbial community
7个微型生物群落的组成见图2。由图2可见,变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria)三者序列数和OTU 数在细菌(除蓝藻门)中占主要优势;隐藻门(Cryptophyta)在真核藻类中最丰富,其序列数占真核藻类总序列数的82.65%,OTU 数占总OTU 数的54.74%;真菌中最丰富的门类是壶菌门(Chytridiomycota)和隐真菌门(Cryptomycota),二者序列数分别占真菌总序列数的67.16%和22.15%,OTU 数占总OTU 数的56.15%和20.89%;丝足虫类(Cercozoa)占原生动物总序列数的53.15%,领鞭毛虫门(Choanoflagellida)占序列数的25.81%,二者在原生动物中占主要优势,它们的OTU数分别占原生动物总OTU 数的39.90%和27.98%。腔肠动物门(Cnidaria)和轮虫类(Rotifera)在无脊椎后生动物中最丰富。原核藻类中仅检测到蓝藻门(Cyanobacteria),其序列数和OTU数也较高。
2.1.2 藻类监测结果
基于eDNA 宏条形码技术共注释到196个原核藻类OTU 和287 个真核藻类OTU,分属于9 门21 纲34目76 科100 属112 种,其中蓝藻门(196 个OTU)和隐藻门(163 个OTU)的种类最丰富,Ochrophyta(55 个OTU)和Katablepharidophyta(31 个OTU)次 之(图3A)。共有14 个OTU 被确定为优势种(表2),蓝藻门9 个,隐藻门5 个。OTU3 的优势度最大(0.290 4),OTU134 的优势度最小(0.021 7),这两个物种均属于隐藻门。
为验证eDNA 宏条形码技术用于生物监测的可靠性,同步对秋季样品进行藻类的形态学鉴定。共鉴定出73种藻类,基本被eDNA宏条形码技术的监测结果所覆盖,分属于7 门9 纲18 目31 科46 属,其中绿藻门(Chlorophyta,35 种)和硅藻门(Bacillariophyta,17种)种类最丰富,分别占鉴定出所有藻类的47.95%和23.29%(图3B)。鉴定出的73 种藻类中,15 种被确定为优势种(表2),其中绿藻门种类最多,为6 种,蓝藻门(Cyanophyta)4 种,硅藻门3 种,隐藻门1 种,甲藻门(Dinophyta)1 种。伪鱼腥藻(Pseudanabaenasp.)为绝对优势种,优势度为0.750 2,四角十字藻(Crucigenia lauterbornii)为次优势种,优势度为0.195 3。
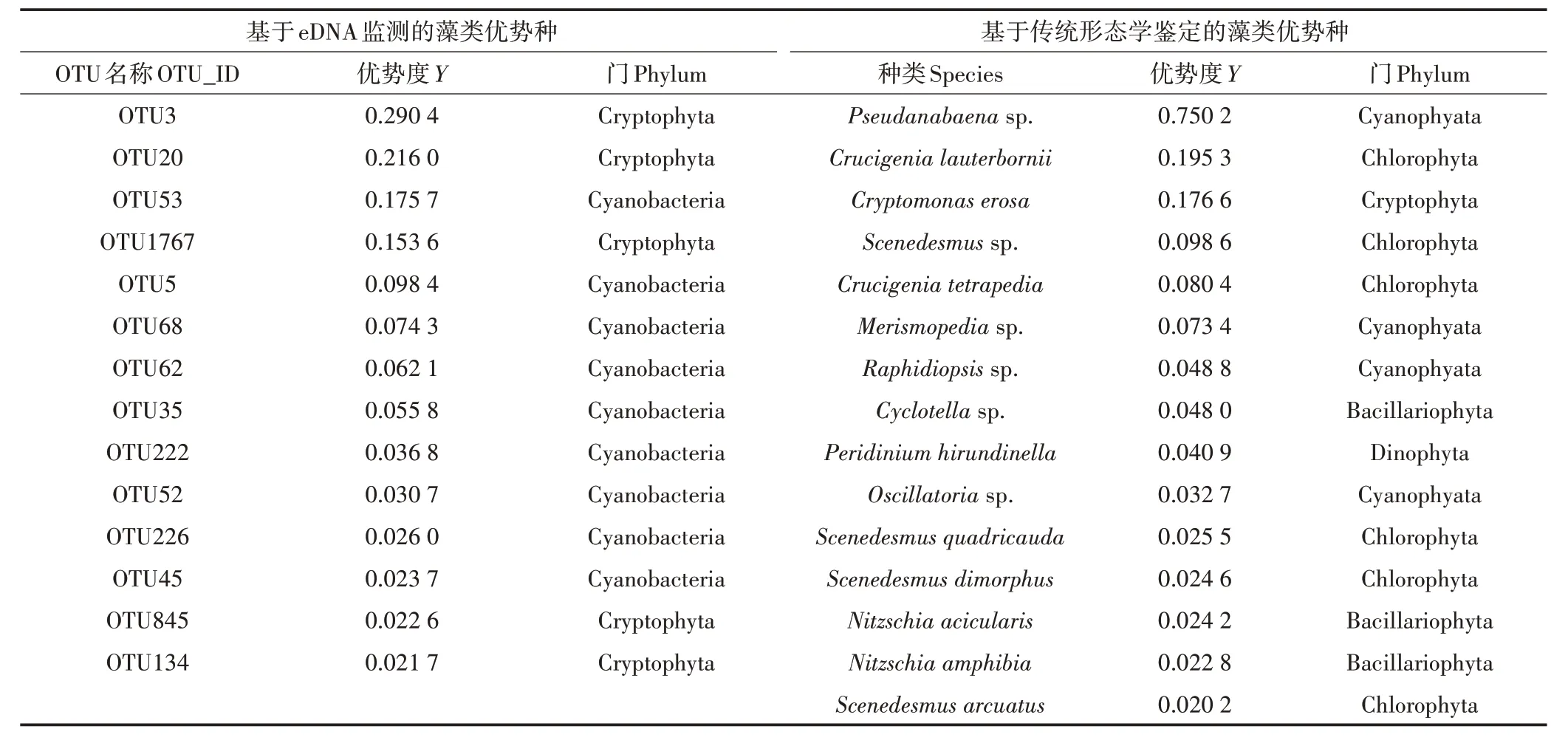
表2 基于eDNA和传统形态学方法确定的藻类优势种Table2 Dominant species of algae determined by eDNA and traditional morphological methods
2.2 白洋淀微型生物组成的时空分布
2.2.1 微型生物群落的季节分布
对检出的原核生物和真核生物分别进行ANOSIM 分析,结果显示:原核生物和真核生物的R值分别为0.648 和0.621,且P值均小于0.01,季节间差异显著(图4)。
如图5 所示,在白洋淀检出的6 个主要微型生物类群中,秋季的香农-威纳指数均明显高于其他3 个季节,说明秋季微型生物的种类最丰富,生物多样性最高,除无脊椎后生动物外,其他微型生物类群夏季的香农-威纳指数仅次于秋季。春季和冬季的生物多样性在各微型生物类群之间的分布不一致,其中原生动物和无脊椎后生动物春季的生物多样性要高于冬季,而其他4 个微型生物类群正好相反。细菌(除蓝藻门)的香农-威纳指数在6 个微型生物类群中最大,介于5.17~7.56 之间,说明该类群的生物多样性最高;无脊椎后生动物最小,范围在0.74~3.10。
序列数对比分析结果表明,白洋淀各微型生物类群分布有明显不同的季节特点(图6)。细菌(除蓝藻门)序列数在四季中均占绝对优势;真菌次之,且四季变化明显,表现为夏季>秋季>春季>冬季。序列占比同样较高的原生动物序列秋季分布要明显高于其他3 个季节,无脊椎后生动物的序列数相对较少,且在冬季分布最多。原核藻类和真核藻类的季节变化特征较为相似,但和其他类群差异较大,它们的序列分布在冬季最多,夏季最少。OTU数量的总体季节变化趋势为秋季>夏季>春季>冬季,与多样性季节变化特点相吻合。
各微型生物类群代表物种在门和属水平的相对丰度变化,也各自呈现出明显的季节特点。放线菌门是细菌(除蓝藻门)的优势群体,其秋季相对丰度要明显低于其他季节,而蓝藻门冬季和秋季的相对丰度显著高于春季和夏季(图7A)。壶菌门是真菌的优势群体,也是真核生物中最丰富的门类,其夏季相对丰度最高,平均值达到49.96%,冬季相对丰度最低,平均值仅有15.92%,季节差异明显。隐藻门在真核藻类中所占比例具有绝对优势,该门类在冬季相对丰度最高,这与真核藻类序列分布在冬季最多的结果相一致(图8A)。和门水平相比,各微型生物类群在属水平上的分布更为复杂,季节差异更为显著,详细见图7B和图8B。
2.2.2 微型生物群落的空间分布
某些生物类群门水平和属水平的相对丰度变化在采样点之间也有明显差异(图7和图8)。隶属于细菌(除蓝藻门)的Epsilobacteracota 菌群,在南刘庄、枣林庄和光淀张庄3 个点位秋季相对丰度较高,尤其是在枣林庄地区,该菌群相对丰度占比要远高于其他采样点。与之相似,属水平上隶属于原核藻类蓝藻门的Planktothrix-NIVA-CYA-15,在采蒲台和王家寨两个点位秋季相对丰度明显高于其他点位。隶属于原生动物领鞭毛虫门的Monosigidae_Group_O_X,在秋季枣林庄点位相对丰度显著高于其他点位。丝足虫类作为真核生物的优势种群,在四季各点位相对丰度的变化呈现出相似趋势,即在采蒲台和圈头这两个地区相对丰度较高,而在其他地区相对丰度较少。
3 讨论
3.1 白洋淀微型生物群落组成主要受时间变化影响
主成分分析结果表明(图9),白洋淀微型生物群落受时间变化的影响要大于受空间变化的影响,尤其是原核生物,在同一季节内不同采样点之间的相似度很高,而季节间的差异较为明显。真核生物同一季节内不同采样点之间的差异要大于原核生物,但相较而言,空间差异没有季节差异显著。因此,白洋淀微型生物群落组成主要受时间变化的影响。
白洋淀地处亚欧大陆东部季风区,暖温带半干旱地区,全年四季分明。在春季和夏季,适宜的水温有利于微型生物群落的生长繁殖;秋季,随着水温的逐渐下降,微型生物的种类逐渐减少;冬季水温最低,微型生物的种类最少。为进一步分析本研究中出现的秋季微型生物群落多样性高于夏季的情况,对采样时间段内白洋淀水温的历史监测值进行了统计,结果表明:白洋淀4 月末(春季)的水温在18 ℃左右,6 月初(夏季)的水温在24 ℃左右,9月中下旬(秋季)的水温在23 ℃左右,11 月中下旬(冬季)的水温在11 ℃左右。本研究中,夏季的采样时间处于春末夏初,水温未达到最高值,是微型生物群落大量繁殖的前期;而秋季采样时间处于夏末秋初,水温和夏季接近,处于微型生物群落大量繁殖的后期。已有研究表明,水温是对微型生物物种数量和丰度影响最大的环境因子[28],因此夏、秋两季采样时间早于典型季节性环境条件发生时间可能是造成本研究秋季多样性指数偏高的主要原因。
阳小兰等[29]的研究发现,白洋淀整体处于轻度富营养化状态,部分区域呈中度富营养化,富营养程度在夏秋两季较高。藻类是水体富营养化的重要指示生物[30],本研究中,藻类(原核藻类和真核藻类)相对丰度在夏季和秋季较低,在冬季最高。推测造成这种现象的原因可能是夏秋两季水温适合包括藻类在内的多种微型生物的繁殖,尤其是真菌类群中的壶菌门,在夏季的平均相对丰度占比达到49.96%,这在一定程度上削弱了部分微型生物,包括藻类的相对丰度,尽管这些生物(如部分藻类)的绝对丰度可能显著高于其他季节,也可能是受eDNA 宏条形码技术无法判定检测到的是否为活体生物[31]的影响,检测到较多死亡个体残留的DNA所致。
空间上,各采样点的微型生物组成、多样性、序列数等信息分布规律性不够突出,变形菌门、拟杆菌门、放线菌门、蓝藻门等原核生物的优势门类及壶菌门、丝足虫类、领鞭毛虫门、隐藻门等真核生物的优势门类的相对丰度在不同采样点均较高,且差异较小。相反,相对丰度较低的某些门类,如原核生物的Epsilonbacteraeota、真核生物的Basidiomycota 等的相对丰度在不同采样点之间差异明显。原因可能有两方面:一是白洋淀生态补水随机性较强,补水不稳定,会频繁扰动水体,使得水动力条件不稳定,底泥与水体的交换频繁,部分死体生物也进入了样本,对不同采样点的微型生物群落检测造成干扰;二是采样点密度较小,未覆盖整个淀区,可能会使监测到的结果具有片面性,今后应根据具体情况合理设计优化布点与采样方案,高密度设置采样点,开展重复校验性检测,进行长期跟踪监测,使之更能反映实际,更有利于分析时空变化的影响。
3.2 eDNA 技术用于白洋淀微型生物群落监测的优势与不足
在针对白洋淀微型生物群落进行的16S rRNA 和18S rRNA 高通量测序研究中,大量细菌(除蓝藻门)、原核藻类、真核藻类、真菌、原生动物类群,少量的无脊椎后生动物类群以及微量的古细菌类群被检出,填补了白洋淀微型生物群落多样性调查的空缺,也印证了eDNA 宏条形码技术用于生物多样性监测的潜力。相较于传统形态学方法,eDNA 宏条形码技术可识别出更多的物种,包括定鞭藻门(Haptophyta)、红藻门(Rhodophyta)、链型植物(Streptophyta)、Katablepharidophyta 等显微镜未识别到的物种,以及以往白洋淀未报道过的藻类[32-34],丰富了白洋淀藻类名录和微型生物群落数据库。王靖淇等[35]、张丽娟等[36]和杨海乐等[37]的研究已表明,eDNA 宏条形码对传统形态学监测结果有较高的还原性,并可检测到一些传统方法无法识别的稀有物种和隐匿物种,说明eDNA 技术在生物多样性监测中有重要应用潜力。
本研究也揭示了eDNA 宏条形码技术存在的一些问题,如无法将所有的OTU 注释到种水平、eDNA检测对传统形态学监测到的物种覆盖度低等。原因在于自然界中有大量未被发现、培养和测序的物种,其基因序列在数据库中不存在,导致数据库信息与自然界实际存在的物种不对等,无法注释[35,38]。测序平台[39]、特异基因、引物的选择[40-41]等实验条件以及季节、水温、流速等自然条件变化也会影响产物的有无和相对丰度。
可见,eDNA 宏条形码技术发展初期,仍需要与传统形态学鉴定方法紧密结合[42],才能构建可靠的多维生物监测系统。但随着eDNA 技术的不断完善和发展,该方法不仅会用于大规模的生物监测,更有望融合到生物多样性监测的标准程序中。
4 结论
(1)白洋淀水样eDNA 宏条形码分析共检测到7个微型生物类群,分别为古细菌、细菌(除蓝藻门)、原核藻类、真核藻类、真菌、原生动物和无脊椎后生动物,注释得到4 569 个OTU,按这些OTU 所能注释到的最高分类水平统计,分属于567 个种。其中,细菌(除蓝藻门)和真菌分别是原核生物和真核生物的优势类群。
(2)白洋淀微型生物群落分布与组成受时间影响更大。原核生物和真核生物均有显著的季节间差异,且各主要微型生物类群的生物多样性、序列数和物种相对丰度都呈现出季节分布特征。优势门类在各采样点的微型生物组成、多样性、序列数等信息分布规律性不够突出,在不同采样点的空间分布没有明显差异,Epsilonbacteraeota、Planktothrix-NIVA-CYA-15等丰度较低的种群呈现出区域分布特征。
(3)eDNA 技术和传统形态学的藻类监测结果具有相似性,且eDNA 技术可检测到更多传统形态学不能识别的藻类,两种监测手段结合可获得更全面的生物组成信息。