玉米KNOX基因家族鉴定及组织和逆境表达分析
赵长江,宋巨奇,都梦翔,徐尚缘,徐晶宇,李佐同
(1 黑龙江八一农垦大学 农学院,黑龙江大庆 163319;2 黑龙江省现代农业栽培技术与作物种质改良重点实验室,黑龙江大庆 163319;3 黑龙江省秸秆资源化利用工程技术研究中心,黑龙江大庆 163319;4 黑龙江省普通高校寒地作物种质改良与栽培重点实验室,黑龙江大庆 163319)
同源异型盒基因KNOX(KNOTTED1-like homeobox)转录因子属于TALE(Three amino acid loop extension superfamily)超家族成员,在植物生长发育过程中起着重要的调控作用[1-2]。KNOX蛋白一般含有4个保守结构域:KNOX1、KNOX2、ELK和Homeobox KN[3-4],位于上游的KNOX1和KNOX2统称为MEINOX[5],MEINOX结构域通过与ELK结构域和下游的Homeobox KN结构域相连接。自第一个KNOX基因在玉米中被报道以来[6],越来越多的KNOX家族基因在不同物种中被相继鉴定,例如拟南芥(Arabidopsisthaliana)、水稻(Oryzasativa)、小麦(Triticumaestivum)、棉花(Gossypiumspp.)、苹果(Malusdomestica)和梨(Pyrusspp.)等[4,7-10]。拟南芥KNOX家族根据其序列和表达模式被分为3类[4],部分基因功能有较为深入的研究。其中,第一亚组基因主要在茎尖分生组织中表达,包含STM(SHOOTMERI-STEMLESS)、KNAT1/BP、KNAT2和KNAT6。STM的突变引起拟南芥缺少分生组织,KNAT1/BP的突变会导致花梗长度减少[1];水稻同亚组OSH15参与控制细胞壁的和木质素的合成[11],OSH1和OSH15 基因的双缺失突变体愈伤组织只能形成叶状结构,无法成芽[12];杨树(PopulusL.)Pttkn1基因调控叶片形态及维管束的形成[13]。而且,该亚组可与同一家族的其他成员或其他家族成员相互作用调控植物生长发育,例如拟南芥KNAT1/BP与STM互作,通过阻止分生组织分化来维持分生活性[14],STM或BP与BLH(BEL1-like homeodomain)家族成员互作[15-16]。第二亚组KNOX蛋白更为保守,拟南芥KNAT3可调节种子萌发和早期发育过程中的脱落酸反应[17],KNAT4可通过控制种子种皮角质层的透性厚度控制种子的物理休眠[18],KNAT7和OFP4相互作用共同调控次生细胞壁的形成[19]。此外,仅存在双子叶植物中的M亚组发现较晚,拟南芥中KNATM参与调控叶近端-远端形态[4]。上述研究表明,植物KNOX家族在生长发育调控中发挥重要作用。尽管第一个植物KNOX基因在玉米中被报道,但是在全基因组水平对KNOX家族的系统分析却鲜有报道。所以,本研究在基因组水平对玉米KNOX家族成员进行鉴定,并对该家族理化性质、系统进化、基因结构、顺式作用元件等信息进行分析,揭示ZmKNOX家族基因在全生育期和盐胁迫条件下的表达模式,为该类基因功能解析提供理论依据。
1 材料和方法
1.1 材料与处理
以东北地区玉米重要自交系‘合344’为供试材料。用10%次氯酸钠对精选的种子消毒30 min,用蒸馏水冲洗至无味后浸泡6~8 h,置于培养箱中22 ℃暗培养24 h催芽;选择萌发1.5 cm左右根的种子摆放于泡沫漂浮板的孔洞中,人工气候室(22 ℃,16 h光/8 h暗)中1/2 Hoagland营养液培养至两叶一心期进行处理。在营养液中添加150 mmol·L-1NaCl,分别于处理0 d、1 d、3 d和7 d取样,用蒸馏水冲洗3遍再将地上和地下部分离,用滤纸吸干组织表面水分后,迅速用液氮冷冻存放于-80 ℃冰箱中。每组样品3个生物学重复。
1.2 方 法
1.2.1 基因家族成员的鉴定玉米基因组数据、蛋白序列及注释文件下载于phyzotome(https://phytozome.jgi.doe.gov/pz/ portal.html)。拟南芥和水稻KNOX蛋白序列分别下载于TAIR(https://www.arabidopsis.org/)、RGAP(http://rice. plantbiology.msu.edu/)。以拟南芥和水稻蛋白序列为种子序列,利用TBtools[20]软件获取玉米蛋白数据库中候选KNOX蛋白,利用NCBI CD-Search、Pfam[21]、SMART[22]在线工具进一步验证,含有KNOX结构域的蛋白序列视为KNOX基因家族成员。
1.2.2 蛋白相关的生信分析利用网站Expasy(https://web.expasy.org/protparam/)分析蛋白理化特性,利用PSORT(http://psort1.hgc.jp/form.html)网站进行亚细胞定位预测分析。利用MEGA X[23]软件,选择最大似然法构建不同物种基因家族成员系统发育进化树,设置如下参数:采用自展法(Bootstrap method),参数为1 000,模型选择最适模型(JTT+G),设置数据缺失处理为比对删除(Pairwise deletion)。
利用MEME网站(https://meme-suite.org/meme/tools/meme)分析蛋白保守基序,参数设置为:输出基序数量为10,基序最小长度为3,最大长度为50;并利用TBtools绘制保守基序图。使用STRING[24](https://string-db.org/)数据库对蛋白互作关系进行预测。
1.2.3 基因相关的生信分析利用MCScanX[25]软件进行共线性关系分析,利用TBtools软件绘制基因在染色体上的分布图和分析基因中的外显子-内含子结构并绘制基因结构图,同时取翻译起始点ATG上游2 000 bp 和1 000 bp序列记为基因启动子,并提交PlantCARE (http://bioinformatics.psb.ugent.be/webtools/ plantcare/html/) 数据库进行顺式作用元件的类型和数量分析。
1.2.4 全生育期组织表达谱及共表达分析玉米全生育期的表达数据来源于Maize eFP Browser(http://bar.utoronto.ca/efp_maize/)数据库。从NCBI-GEO下载GSE117030转录组表达谱数据,使用TBtools中OneStepWGCNA工具进行加权共表达网络构建,筛选样本中6成且FPKM值>1的数据,计算加权系数β值,构建无尺度网络并划分相关模块,并对性状与模块进行关联分析,绘制模块-性状热图。利用TBtools软件的GO Enrichment进行GO富集分析,以玉米基因组为参考数据库,利用Enrichment Bar Plot设置数量参数为30进行可视化。
1.2.5 非生物逆境表达谱及深入验证分析一方面,通过 SRA 数据库(https://submit.ncbi.nlm.nih.gov/about/sra/)下载冷(SRR1238717,SRR1819204,SRR1819205)、热(SRR1238715,SRR1819196和SRR1819198)、盐(SRR1238719)和UV(SRR1238720)转录表达数据,构建玉米KNOX基因非生物胁迫表达数据;另一方面,用本实验室转录数据进一步分析验证玉米KNOX基因表达,即采用TRIZOL试剂(Invitgen)提取玉米叶片和根组织总RNA,纯化后构建cDNA文库,通过聚合酶链反应(PCR)富集cDNA文库,然后使用Illumina HiSeq 2000平台对构建的文库进行RNA-seq测序(SRP176441和SRP307694)。同时抽取KNOX基因构建盐处理下根和叶表达谱,相对表达量计算方法为:首先将3次生物学重复处理的FPKM平均值与对照(0 d)平均值相比,然后二者比值进行log2FC转换,转换值即为相对表达量。
2 结果与分析
2.1 ZmKNOX基因家族成员的鉴定
在玉米基因组水平鉴定22个ZmKNOX家族成员(表1),根据其在染色体上的位置依次命名为ZmKNOX1~ZmKNOX22。ZmKNOX家族基因编码蛋白的分子质量跨度为40.28 kD~14.70 kD,理论等电点范围4.70~9.78。其中,ZmKNOX6氨基酸数量最多为364个,ZmKNOX15氨基酸数量最少为129个;有4个ZmKNOX家族成员的蛋白质等电点大于7,其余成员均小于7。除ZmKNOX7预测定位于细胞质,ZmKNOX5、ZmKNOX11、ZmKNOX12和ZmKNOX15定位于线粒体外,其余家族蛋白质预测定位于细胞核。
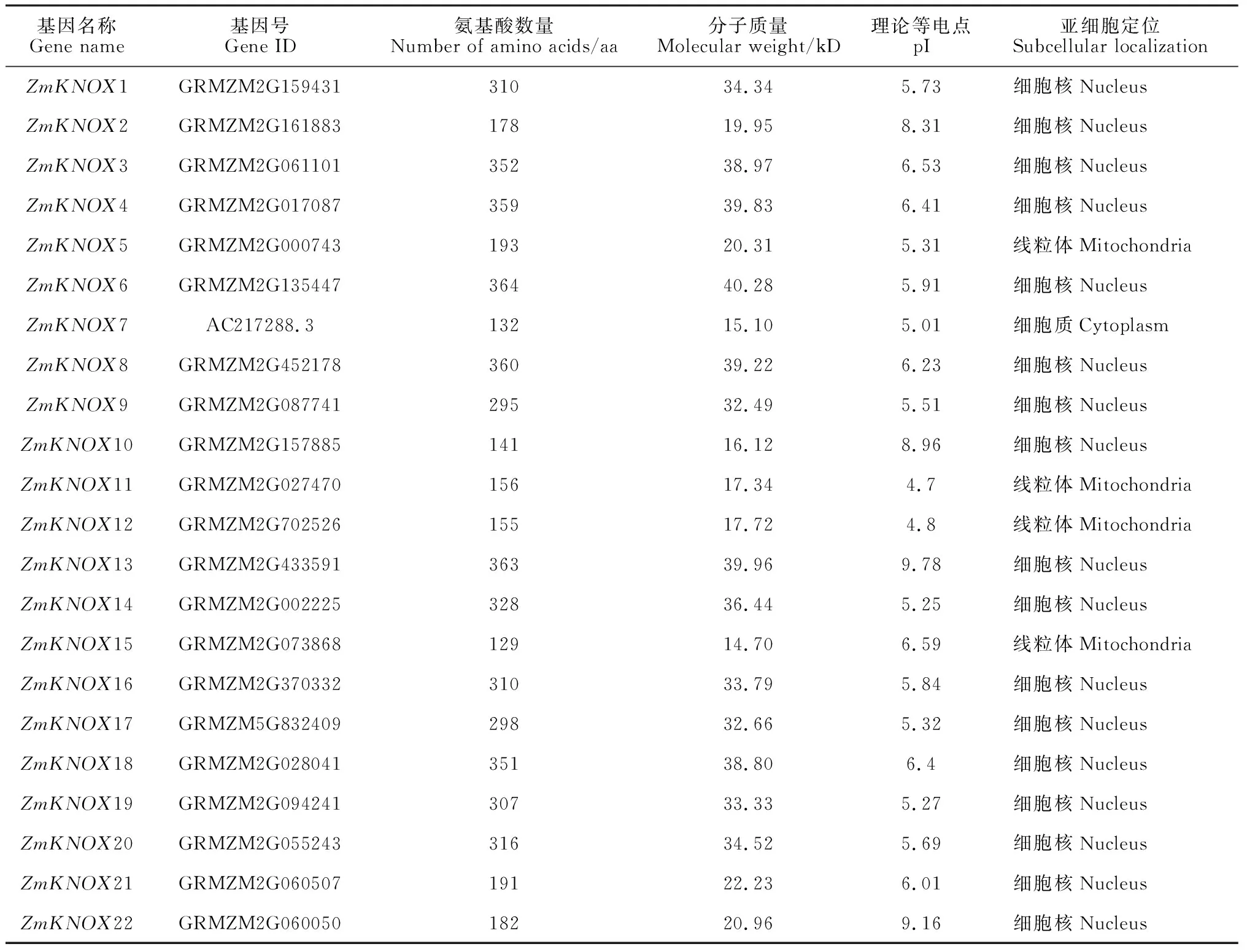
表1 ZmKNOX基因编码蛋白序列理化性质
2.2 ZmKNOX基因家族进化及物种间基因共线性分析
使用ClustalX V.2.0将已知的拟南芥(9个)、水稻(13个)、小麦(36个)、高粱(23个)KNOX家族的蛋白序列和玉米的蛋白序列进行多序列比对,后使用MGEA X软件构建系统进化树(图1)。进化树中显示KNOX家族基因被分为三大类ClassⅠ、ClassⅡ和ClassKNATM,其中ClassⅠ被分为ClassⅠA、ClassⅠB和ClassⅠC三个亚类,ClassⅡ被分为两个亚类ClassⅡA和ClassⅡB。ClassⅠA中含有31个KNOX成员,数量最多,其中玉米9个(ZmKNOX2、4、5、6、8、10、15、18、22);ClassⅠB含有21个KNOX成员,其中玉米3个(ZmKNOX9、17、19);ClassⅠC含有18个KNOX成员,其中玉米5个(ZmKNOX3、7、11、12、14)。ClassⅡA有8个KNOX成员,其中玉米2个(ZmKNOX1、 21);ClassⅡB含有19个KNOX成员,其中玉米3个(ZmKNOX13、16、20)。Class KNATM中含有拟南芥的1个成员和5个高粱成员。此外,通过进化树可以发现,大多数ZmKNOX和高粱聚在一个分支,表明两物种系统发育关系较近。
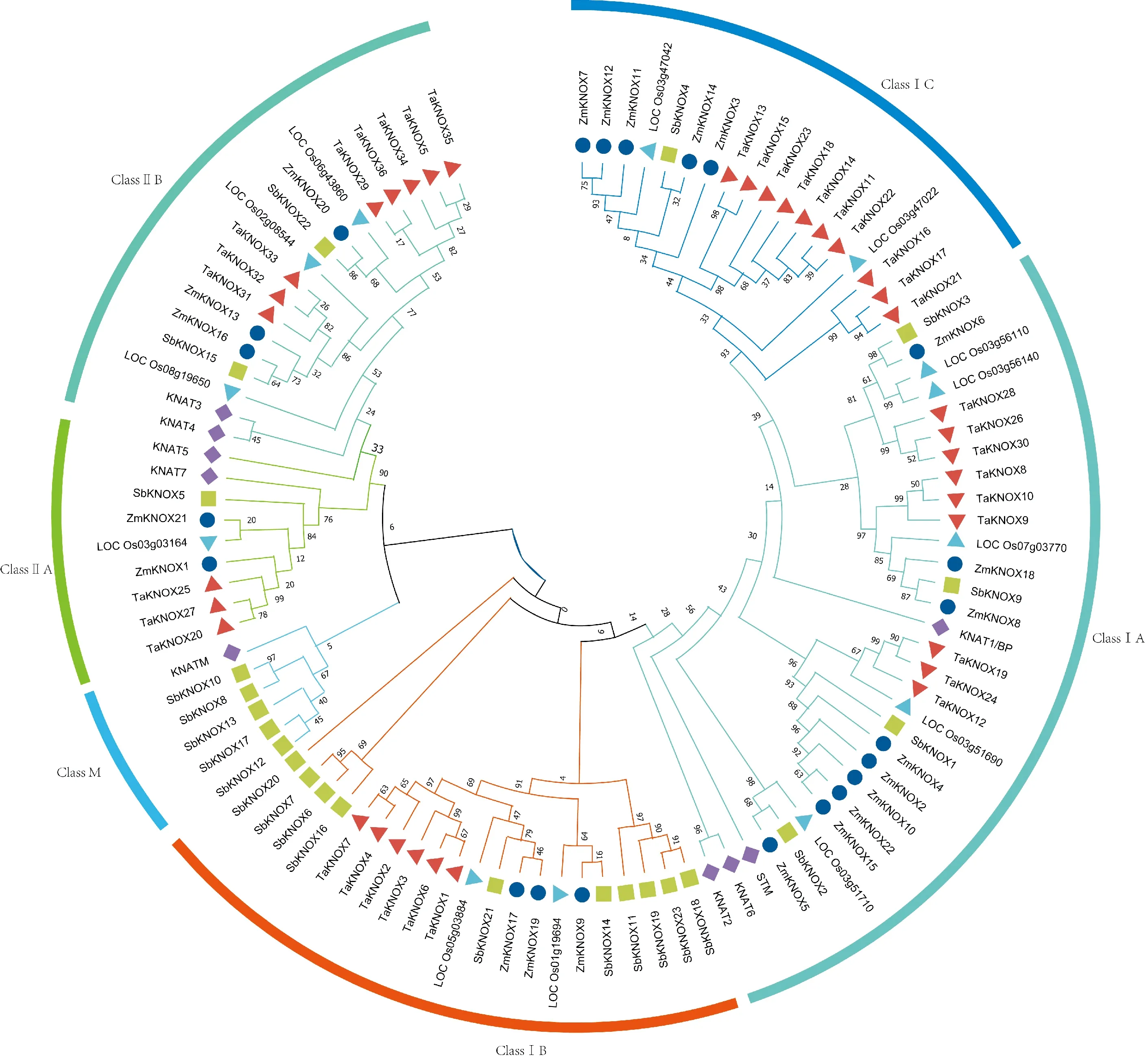
ClassⅠA、B和C代表第一亚类的3个亚族;ClassⅡA和B代表第二亚类的2个亚族;Class M代表M亚族。水稻(Os)和小麦(Ta)分别用青色和红色三角表示;高粱(Sb)、玉米(Zm)和拟南芥(At)分别用绿色方块、蓝色圆圈和紫色菱形表示图1 ZmKNOX家族成员蛋白系统进化树Class Ⅰ A, B and C represent three subgroups of the first subclass; Class Ⅱ A and B represent two subgroups of the second subclass; Class M represents the M subgroup. Among them, Oryza sativa (Os) and Triticum aestivum (Ta) are characterized by a cyan and red triangle, and Sorghum bicolor(Sb) Zea mays(Zm) and Arabidopsis thaliana (At) represented by a green square, a blue circle and a purple diamond, respectivelyFig.1 Protein phylogenetic tree of ZmKNOX family
ZmKNOX家族基因在10条染色体上均有分布(图2),其中6、7、8、10号染色体上只有1个家族成员。ZmKNOX1-6分布在1号染色体,ZmKNOX7-8分布于2号染色体,ZmKNOX9、10分布在3号染色体,ZmKNOX11-13分布于4号染色体,ZmKNOX14-16分布在5号染色体,ZmKNOX17、18、19分别分布于6、7、8号染色体上,ZmKNOX20、21分布在9号染色体,ZmKNOX22定位于10号染色体上。利用MSCAN软件分析玉米、水稻、高粱3个物种KNOX家族基因共线性关系。结果表明11个ZmKNOX家族基因与6个高粱KNOX家族基因存在线性关系,4个ZmKNOX家族基因与3个水稻KNOX家族基因存在线性关系。玉米和高梁两物种间的共线性基因数明显多于玉米和水稻间的,这与我们构建的多物种进化树的结果相佐证。
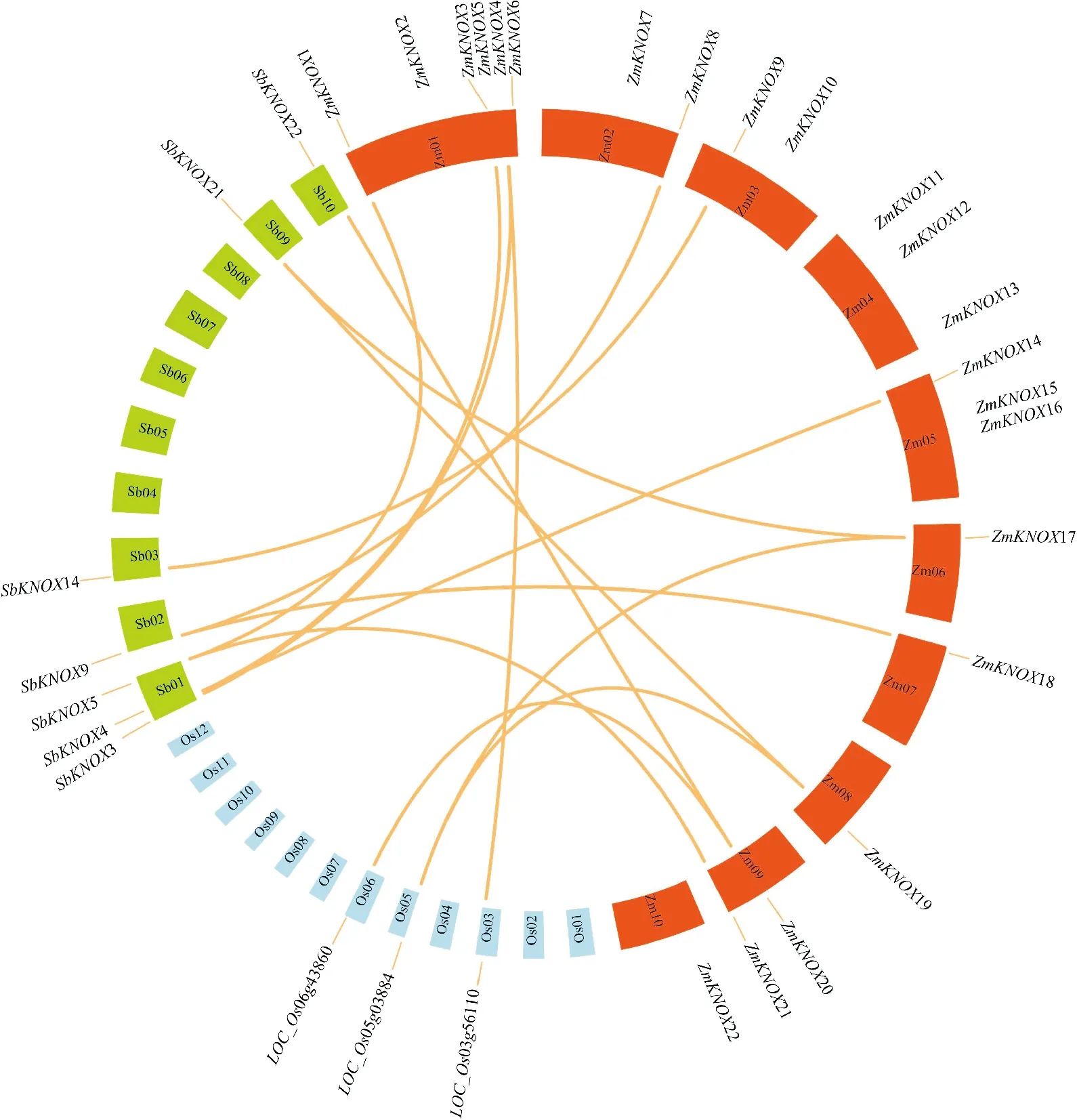
Os1-12代表水稻12条染色体,Zm1-10代表玉米10条染色体,Sb1-10代表高粱10条染色体图2 ZmKNOX基因家族染色体定位及种间共线性分析Os1-12 represents twelve rice chromosomes, Zm1-10 represents ten maize chromosomes, and Sb1-10 represents ten sorghum chromosomesFig.2 Chromosome localization of ZmKNOX gene and their interspecific collinearity analysis
2.3 ZmKNOX蛋白保守基序和基因结构分析
利用MEME网站对ZmKNOX家族蛋白序列保守性进行分析(图3,A)。其中,motif3为KNOX1结构域的保守基序,motif2、motif5和motif8构成KNOX2的保守基序,motif4为ELK的保守基序,motif1为Homeobox KN的保守基序。所有ZmKNOX家族成员均含有KNOX2结构域。通过ZmKNOX家族基因结构(图3,B)分析发现,ClassⅠ组基因内含子0~4个,ClassⅠB组基因内含子均为4个,ClassⅡ组基因内含子数4~6个。

A下方比例尺表示蛋白长度;B中数字代表内含子的数量,下方比例尺表示碱基数图3 ZmKNOX家族保守基序(A,C)和基因结构图(B)The scale below figure A shows protein length; the number in figure B represents intron number, and the scale represents the bases numberFig.3 The conserved motif (A,C) and gene structure (B) of the Zmknox family
2.4 ZmKNOX基因启动子顺式作用元件分析
在ZmKNOX家族中我们分析了基因启动子区生长、光响应、胁迫、激素四大类作用元件的数量和分布(图4)。首先,基于2 000 bp启动子序列进行相关元件的预测分析,生长发育相关的作用元件包括O2-site、motifI、CAT-box等7个元件;光响应相关的包括Sp1、TCT-motif、GATA-motif等25个元件;逆境胁迫相关的包括LTR、MBS、ARE等7个元件;与植物激素相关的包括P-box、TGA-element、ABRE等7个元件。其中,光响应元件分布于ZmKNOX家族所有成员中,除ZmKNOX14、19、21外,其余ZmKNOX基因启动子均含有生长相关的元件,ZmKNOX6、22启动子上数量最多;除ZmKNOX20启动子只含有生物钟相关元件外,其余该家族基因启动子均含有逆境相关元件;ABA相关元件在ZmKNOX6、8、17启动子上数量较多。
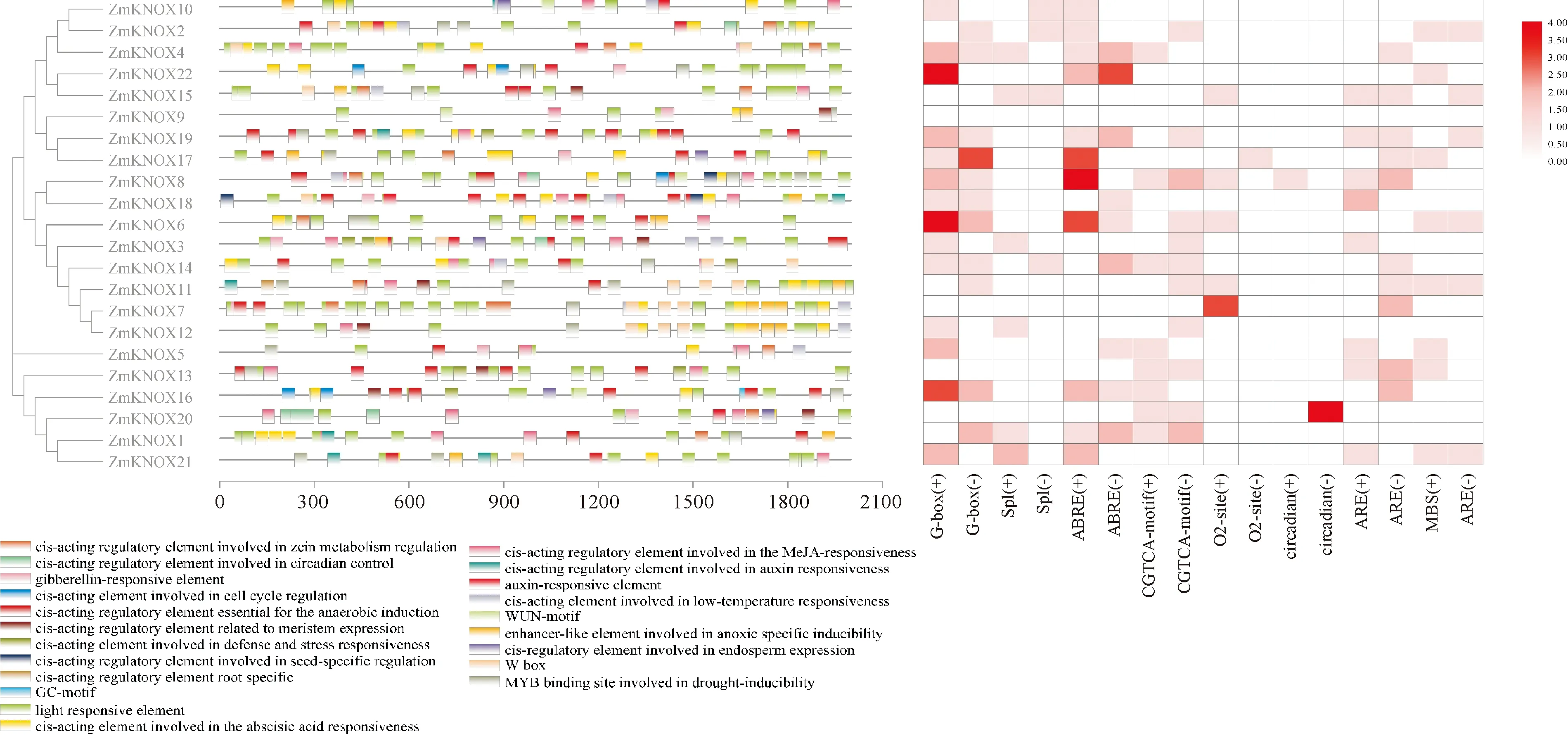
右图中+或-代表DNA双链中两条方向相反的单链图4 ZmKNOX基因家族启动子区顺式作用元件分布及统计数In the figure on the right, + and - represent the two chain in a different directionFig.4 Distribution and number of the cis-acting elements in the promoter region of the ZmKNOX gene family
进而,选取启动子上游1 000 bp范围内各类顺式作用元件中数量最多的2个元件进行分析(图4),结果显示,光(G-box、Sp1)、逆境(ARE、MBS)、激素(ABRE、CGTCA-motif)响应元件分布较为广泛,生长相关的元件(O2-site、Circadian)仅在部分ZmKNOX家族成员启动子出现。揭示ZmKNOX家族可能在玉米生长发育和响应逆境胁迫方面起着重要的作用。
2.5 ZmKNOX基因全生育期及非生物胁迫下表达模式分析
排除ZmKNOX2、7、10、11、12、15、22等7个基因没有相应表达数据外,其余15个ZmKNOX家族基因在全生育期中的表达模式(图5)可以分为:全生育期组成型高表达(5个基因)、低表达(4个基因)和特异性表达(6个基因)3种。而且,基因的生育期表达模式与蛋白进化分类具有一定关联性。ClassⅠ中的ZmKNOX4、8、9、17、18、19在胚胎、种子萌发时期、胚芽鞘、茎尖、茎节间、顶端分生组织、花序表达量较高,说明其表达具有阶段性和组织特异性。全生育期组成型低表达的基因也分布在该亚族中,包括ZmKNOX3、5、6、14。ClassⅡ中ZmKNOX1、13、20、21呈现出全生育期组成型高表达。

图例表示标准化的FPKM值,红色表示高表达水平,蓝色表示低表达水平图5 ZmKNOX基因家族生育期及非生物胁迫表达分析The legend indicates the standardized FPKM value, the red indicates the high expression level, and the blue indicates the low expression levelFig.5 Expression analysis of ZmKNOX gene family during whole growth period and various abiotic stresses
在盐、冷、热和UV条件下ZmKNOX家族基因表现出不同的响应模式,只有ZmKNOX1、3、6、13、14、16、20、21等8个基因参与对供试非生物逆境的应答,总体来看基因表达量变化不大(图5)。基于供试4种逆境的表达聚类,将8个基因分为两类,一类为总体下调表达包括ZmKNOX3、14;其余6个划为一类,为上调表达或基本无变化。其中,ZmKNOX6冷处理下明显上调,ZmKNOX14热处理明显上调表达,盐胁迫下ZmKNOX13明显上调、ZmKNOX3、14明显下调。同时,逆境应答基因中ZmKNOX3、6、14具有生育期低表达的特点,说明家族成员在生长发育和逆境应答方面存在分工和协作。
2.6 玉米基因共表达网络构建及目标模块GO富集分析
根据GEO数据库的GSE50191为基础,构建共表达网络(图6,A、B)。其中turquoise模块含有的基因数目最多,greenyellow模块含有的基因最少。将不同处理看作质量性状与模块进行关联分析并绘制相关性热图。ZmKNOX家族基因与生长发育相关,同时在turquoise模块中发现2条ZmKNOX基因(ZmKNOX16、20),black模块中发现1条ZmKNOX家族基因(ZmKNOX13)。同时在turquoise模块中Ear Primordium 6~8 mm这一性状的相关性最高为0.42,black模块中Root Meristem Zone 5 Days这一性状相关性最高为0.65,因此选择turquoise模块和black模块进行后续分析。GO功能富集分析,turquoise模块中基因的分子功能主要富集于嘌呤核苷酸结合(GO:0017076)和嘌呤核糖核苷三磷酸结合(GO:0035639)等分子结合功能;生物过程富集于细胞器构建(GO:0033043)和细胞周期(GO:0007049)等生长发育相关过程。black模块中基因的分子功能主要富集于泛素结合酶活性(GO:0061631)和泛素蛋白转移酶活性(GO:0004842),生物过程富集于蛋白质泛素化(GO:0016567)和小蛋白偶联修饰蛋白质等蛋白泛素化分子过程。
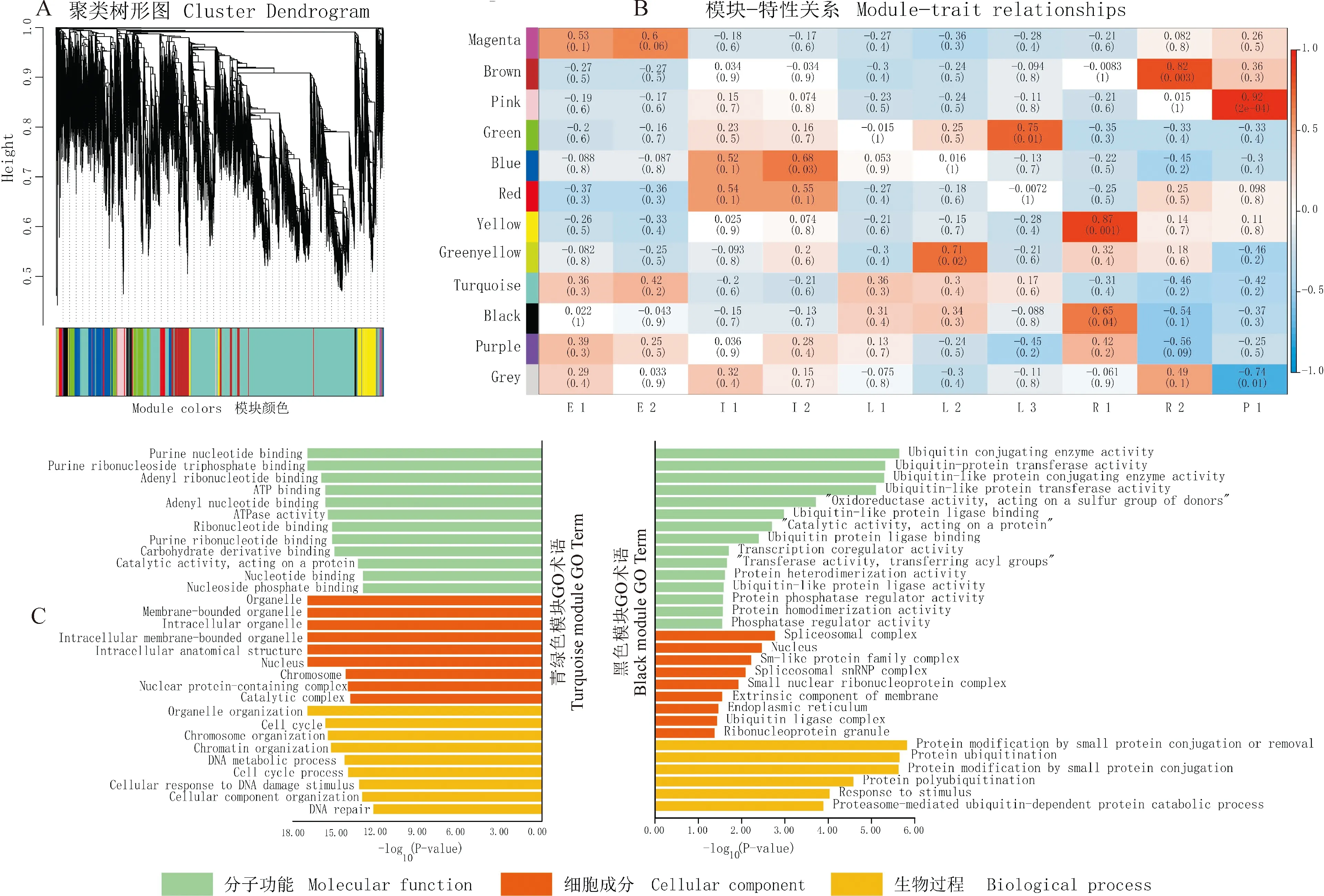
E1.穗原基2~4 mm;E2.穗原基6~8 mm;I1.节间6-7;I2.节间7-8;L1.叶区1(对称);L2.叶区2(气孔);L3.叶区3(生长);R1.根-分生组织区5 d;R2.根-伸长区5 d;P1.主根5 d图6 生长发育期玉米基因共表达及GO富集分析E1. Ear primordium 2-4 mm; E2. Ear primordium 6-8 mm; I1. 6-7 internode; I2. Internode 7-8; L1. Leaf zone 1 (Symmetrical); L2. Leaf zone 2 (Stomatal); L3. Leaf zone 3 (Growth); R1. Root-meristem zone 5 days; R2. Root-elongation zone 5 day; P1. Primary root 5 dayFig.6 Coexpression analysis and GO enrichment based on transcriptional data of maize growth and development periods
2.7 ZmKNOX基因盐胁迫下表达模式分析
通过实验室不同盐处理时间(1 d、3 d和5 d)玉米幼苗和不同取样部位(叶片和根系)的转录数据对ZmKNOX基因进行深入分析(图7)发现,盐处理叶片中ZmKNOX3、4、13、14等4个基因基本都上调表达,其中ZmKNOX3、13不同时间点表达模式基本一致;盐处理根系中ZmKNOX3、4、6、8、17等5个基因差异表达,其中ZmKNOX3、6、17不同时间点表达模式基本一致且多为下调表达,值得一提的是只有ZmKNOX6为上调表达。此外,盐处理下ZmKNOX3在叶片和根中均稳定表达,且表达方向不同,我们也通过实时荧光定量PCR对该基因在叶片中表达进行了验证(结果未显示)。本实验室和网络数据都表明ZmKNOX基因确实参与对盐的响应。

图7 盐胁迫玉米幼苗叶和根中ZmKNOX基因表达分析Fig.7 The expression analysis of ZmKNOX in the leaves and roots of seedlings challenged by NaCl
2.8 ZmKNOX蛋白互作分析
通过String网站预测蛋白互作(图8,A),包括ZmKNOX1、3、4、5、6、8、13、14、16、18、20、21和22等13个ZmKNOX家族成员存在蛋白间互作关系。互作关系网中存在19个节点55组互作关系,ZmKNOX1、21位于整个蛋白互作网络的中心,且可与ZmBELL8、13、14、ZmOFP19、ZmMYB92和Zmsbp10.a等蛋白发生互作。结合生育期组织基因共表达分析,ZmBELL8与ZmKNOX4、18具有相同的组织表达模式(图8,B),在转录水平上支持蛋白互作。
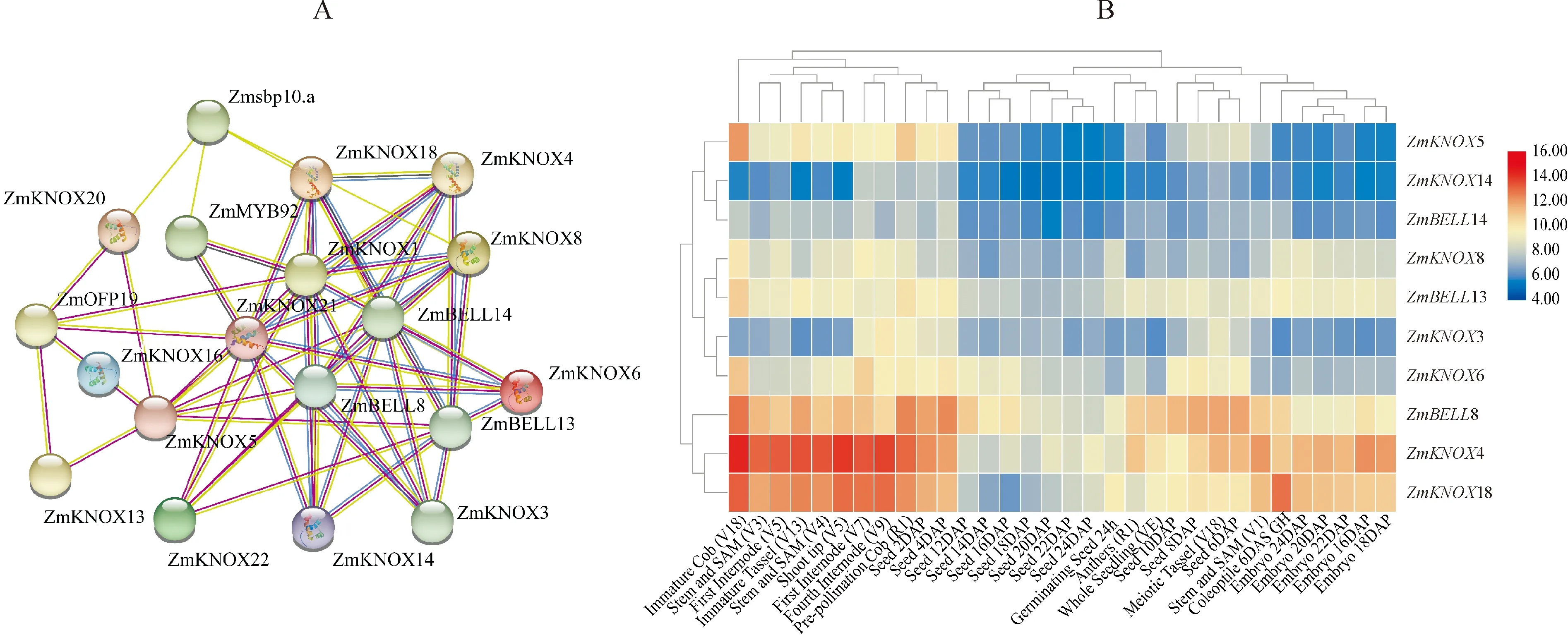
图8 ZmKNOX家族蛋白互作网络预测(A)及相关基因表达分析(B)Fig.8 Prediction of ZmKNOX protein interaction network (A) and their gene expression analysis (B)
3 讨 论
本研究在基因组水平鉴定了22个ZmKNOX家族成员。基于系统进化分析,多数(17个)ZmKNOX基因划分为第一组,该组拟南芥成员主要调控茎尖分生组织的形成,推测该组ZmKNOX基因参与分生组织调控;少部分第二组中ZmKNOX成员可能参与调控植物种子萌发和细胞壁的形成[18,26]。通过对水稻、高粱和玉米KNOX家族基因的共线性分析,发现玉米与高粱间的共线基因数多于玉米与水稻;而且蛋白系统进化分析中玉米和高粱KNOX共聚于更小的分支,上述结果都表明同为C4作物的玉米和高粱间KNOX家族成员间亲缘关系较近。
在ZmKNOX基因启动子区域预测到大量与逆境、生长发育和激素相关的作用元件,表明ZmKNOX家族基因可参与逆境、生长发育和激素调控,甚或参与激素介导的生长发育或逆境调控。本研究中对玉米ZmKNOX基因生育期表达分析发现,第一组ZmKNOX基因主要在顶端分生组织、茎节间和花序中表达,同时在植物器官建成初期的表达量较高;第二组ZmKNOX基因在所有组织中均有表达,特别是ZmKNOX13和ZmKNOX20表达量最高,ZmKNOX1和ZmKNOX21在根部、叶片、苞叶中表达量较高。表明该家族基因可能参与不同生长发育阶段特异组织的调控。同时,通过盐胁迫表达分析发现,尽管ZmKNOX3、ZmKNOX6和ZmKNOX14在全生育期表达量不高甚或可能下调表达,但在盐胁迫下表达量变化明显,参与植物对盐胁迫的应答调控。此外,不同植物中该家族基因功能的研究,也旁证了启动子顺式元件中存在大量激素相关元件的结果,例如苔藓(Physcomitrellapaten)中KNOX基因可以通过异戊烯基转移酶基因PpIPT3促进细胞分裂素的生物合成[27],荔枝(Litchichinensis)中LcKNAT1通过调节乙烯的生物合成来调节脱落[28],以及参与赤霉素功能调控[29-30]。
基于全生育期转录组数据的共表达分析,鉴定出含有ZmKNOX基因的2个模块,分别与turquoise模块中叶耳原基(6~8 mm Ear Primordium)期和black模块中根分生区(5 Days Root Meristem)期明显正相关。其中,叶耳原基期主要涉及嘌呤核苷酸结合、嘌呤核糖核苷三磷酸结合和腺苷酸核糖核酸结合等分子功能,推测包含于turquoise模块的ZmKNOX16和ZmKNOX20可能在RNA合成水平调控叶耳生长发育,负调控根的伸长(Root-Elongation Zone 5 Day);同理,根分生区期主要涉及泛素结合酶活性、泛素蛋白转移酶活性、泛素样蛋白结合酶活性等分子功能,推测black模块中ZmKNOX13可能通过泛素化蛋白降解调控根区分生活性,负调控根的伸长(Root-Elongation Zone 5 Day),表明泛素化与植物生长发育密切相关[31]。同属于全生育期组成型高表达的ZmKNOX16和ZmKNOX20基因在盐逆境处理下无明显变化,而生育期表达量总体不高的基因却发生明显变化,当然组成型高表达的ZmKNOX13也参与逆境应答,表明该家族基因在生长发育调控和逆境应答方面既有分工又有对话协作。值得一提的是,基于共表达分析发现全生育期组成型高表达基因在不同模块中凸显,参与生育期的特异调控。通过蛋白互作预测分析发现ZmKNOX13、ZmKNOX16和ZmKNOX20都可以与生育期相对低表达的ZmKNOX5互作,推测通过同源二聚化调控生长发育,当然上述蛋白还可能通过与ZmOFP19等异源蛋白互作实现对植物生长发育的调控,或是对生长发育与逆境应答的平衡。综上,本研究基于生物信息学分析在玉米基因组水平鉴定了22个ZmKNOX家族成员,通过在线生育期和逆境表达数据和实验室盐处理测序数据分析,揭示了ZmKNOX家族基因参与玉米生长发育和非生物逆境应答,为该类基因功能解析奠定了理论基础。

