CRISPR-Cas9基因编辑技术在念珠菌中的应用研究进展
于淑颖 周梦兰 孙天舒 张丽 徐英春
(1.中国医学科学院北京协和医院检验科,北京 100730;2.中国医学科学院北京协和医学院研究生院,北京 100730;3.中国医学科学院北京协和医院医学科学研究中心,北京 100730;4.侵袭性真菌病机制研究与精准诊断北京市重点实验室,北京 100730)
近年来,真菌感染已经成为严重威胁全球健康的疾病,全球约有3亿人遭受了侵袭性真菌感染,造成每年约有160万人死亡,接近结核病的死亡人数[1]。尤其随着免疫功能低下人群的不断增加,侵袭性真菌感染导致的死亡率高达30%~90%[2]。念珠菌属是真菌感染的主要致病菌,白念珠菌仍然是最常见的菌种,而非白念珠菌侵袭性感染所占的比例明显升高[3-4]。目前,念珠菌对抗真菌药物的耐药问题面临严峻的挑战,光滑念珠菌对棘白菌素类药物的高度耐药性在美国和欧洲地区引起了广泛关注[5]。近年来,我国热带念珠菌和光滑念珠菌对唑类药物的耐药率显著升高,角膜念珠菌和解脂念珠菌等少见菌种也呈现出极高的氟康唑和伏立康唑耐药率[3]。此外,新兴的多重耐药病原体—耳念珠菌对全球的公共卫生构成了威胁[6]。
在这种情况下,亟需深入理解真菌病原体的生物学和病理生理学,并阐明耐药机制。因此,对于高效基因操作工具的需求十分迫切,然而,对真菌的基因操作通常费时且麻烦,特别是在缺乏性周期的二倍体菌种中。此外,转化和/或同源重组效率低,缺乏天然质粒、克隆载体,或者有限的筛选标记等也是阻碍该过程的重要原因[7]。规律间隔成簇短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)和CRISPR 相关基因蛋白(CRISPR-associated protein, Cas9)系统的突破性发现彻底改变了基因编辑的现状。原本,CRISPR-Cas9系统通过成熟的crRNA(CRISPR RNA)与反式激活crRNA(trans-activating crRNA,tracrRNA)碱基配对形成双链RNA,双链RNA指导Cas9在目标基因中引入双链断裂,导致入侵核酸沉默,从而为细菌和古细菌提供针对病毒的适应性免疫[8]。随后,陆续的研究发现,通过单向导RNA(single guide RNA,sg RNA)的巧妙设计,CRISPR-Cas9系统也逐渐应用于哺乳细胞、植物以及真菌等真核细胞的基因编辑研究[9-10]。
本文旨在对CRISPR-Cas9系统的工作原理和其在念珠菌中的应用进行系统的综述,希望CRISPR的发展和应用能够有助于微生物学家解决念珠菌的致病机理和耐药机制等多方面问题,从而进一步促进新的治疗策略的开发。
1 CRISPR-Cas9基因编辑系统的工作原理
简单来说,CRISPR-Cas9基因编辑系统的两大核心组分包括:用于产生DNA双链断裂(double strand breaks,DSB)的Cas9蛋白和能够将Cas9核酸内切酶引导至具有合适原型间隔序列毗邻基序(protospacer adjacent motif, PAM)的染色体中任何基因座的sgRNA(small guide RNA)[11]。Cas9蛋白,是Ⅱ型CRISPR-Cas系统的核酸内切酶,受sgRNA引导在PAM上游3 bp处引入了一个双链断裂[如酿脓链球菌 (Streptococcuspyogenes)的 Cas9蛋白识别的PAM 结构为三核苷酸NGG序列]。sgRNA同时具有靶向特异性和Cas9结合活性,在天然细菌系统中由crRNA 和tracrRNA组成。tracrRNA能够与前体crRNA转录的重复序列互补配对紧密结合,随后被RNA聚合酶Ⅲ识别并加工为成熟的crRNA: tracrRNAs复合体(即sgRNA,功能性双链RNA二级结构),直接引导Cas9核酶结合外源DNA并进行剪切破坏。因此,通过将包含靶向引导序列的crRNA与tracrRNA融合,构建sgRNA,然后结合Cas9蛋白便可致使目标基因发生双链断裂(见图1)[7-8,12-13]。DSB可以通过两种不同的方式修复:非同源末端连接(non-homologous end joining,NHEJ)和同源定向修复(homology-directed repair,HDR)。NHEJ的修复方式容易出错,通常会在DSB处随机引入碱基的插入或者缺失,从而导致目标基因发生移码突变或终止密码子提前出现。如果存在具有同源性区域的供体DNA(donor DNA)作为模板,如二倍体基因组中的等位基因或者外源DNA,则可以通过HDR进行修复,以引入特定点突变或者插入所需序列[12,14]。而CRISPR系统介导的HDR中,具有DSB侧翼同源性的外源修复模板可以精确地将突变引入基因组[15]。
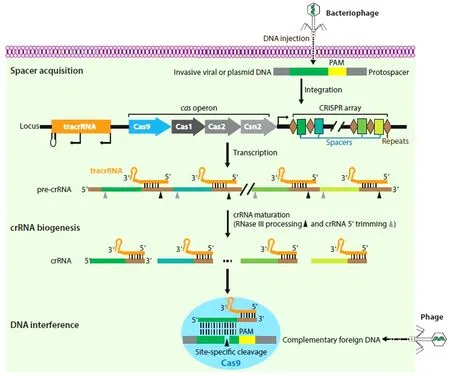
图1 细菌适应性免疫中CRISPR-Cas9介导的DNA干扰[13]
2 CRISPR-Cas9系统在念珠菌中的应用
2.1 白念珠菌
白念珠菌是真菌感染最主要的致病菌,因该菌种是双倍体、生殖周期不完整、缺乏质粒系统以及使用非典型密码子等原因,一直以来亟需便捷的分子遗传工具用于深入探索白念珠菌的致病机制。
得益于具有交配能力的白念珠菌单倍体的发现,Shapiro等开发了一种基于CRISPR-Cas9系统的“基因驱动阵列”(gene drive array,GDA),在白念珠菌中进行基因相互作用的分析。Shapiro等构建了能够表达Cas9并具有一对sgRNA转录框的质粒,sgRNA转录框的两侧连接目标基因上下游的同源序列。sgRNA转录框引导Cas9在目标基因中引入双链断裂后,细胞利用该质粒进行HDR,从而用sgRNA转录框替换掉目标基因,进一步形成“驱动基因”。当带有“驱动基因”的单倍体细胞与野生型单倍体细胞交配时,野生型细胞中的等位基因将会被Cas9剪切,并通过HDR的方式被替换为sgRNA转录框,二倍体纯合双基因敲除菌株就此产生。利用GDA平台,研究人员构建了白念珠菌的双敲除纯合细胞文库,并揭示了药物耐药性和生物膜形成相关的基因间相互作用[16]。
Vyas等[17]开发了经过密码子优化后在念珠菌中稳定表达的CAS9等位基因,首次实现了CRISPR-Cas9系统直接在白念珠菌基因编辑中的成功应用。他们构建了能够表达CAS9和sgRNA的“solo system”和“duet system”两种质粒系统。在“solo system”中整个线性化的质粒整合至ENO1位点,在“duet system”中表达CAS9和sgRNA的两条线性化质粒分别整合至ENO1位点和RP10位点。CAS9的表达由整合位点的启动子驱动,而RNA聚合酶Ⅲ启动子CaSNR52启动sgRNA的表达。质粒上构建有NAT基因,因此使用诺尔丝菌素进行转化菌株的筛选。此外,通过提供带有同源臂的供体DNA,以HDR的修复方式在ADE2基因中提前引入终止密码子,从而破坏该基因。然而,CAS9基因在基因组中的稳定整合可能会增加脱靶效应的发生[7]。
在此基础上,Min等[18]开发的瞬时表达的CRISPR-Cas9系统解决了对脱靶问题的担忧,无需将各元件稳定整合至基因组,即可在白念珠菌中进行有效的基因删除。随后,Ng等[19]发现sgRNA的细胞内水平显著限制了Cas9介导的DNA切割效率,因此,他们尝试分析不同启动子(SNR52、ADH1和tRNA),以及不同的RNA转录后加工方案对CRISPR-Cas9系统编辑效率的影响。结果发现使用RNA聚合酶Ⅱ(ADH)启动子驱动sgRNA的表达,并分别在sgRNA的5’端和3’端连接tRNA(转运核糖核酸)和自我裂解型肝炎病毒(hepatitis delta virus,HDV)核酸酶,可以将CRISPR-Cas9系统在白念珠菌中的编辑效率提高10倍。
念珠菌的基因操作工程中可供选择的筛选标记十分有限,如果能够重复使用单个筛选标记进行连续基因编辑将会大有裨益。Huang等[20]巧妙地设计了一种标记回收的新方法—CRISPR-Cas9诱导的标记切除系统(CRISPR-Cas9-induced marker excision,CRIME),该系统在用于删除目标基因的选择标记两侧设置重复序列,删除目标基因后,CRISPR-Cas9系统再次在标记本身中诱导DSB,然后通过重复序列之间的重组实现标记切除。Huang等[20]利用CRIME系统成功构建了白念珠菌的多突变菌株,并认为该系统可以广泛应用于多种微生物。然而,Nguyen等[21]认为:Huang等研究者构建的CRIME体系虽然能够快速连续的删除基因,但是仍需要使用两个不同的筛选标记,并且涉及标记之间的相互替换,反而使得基因编辑过程更加复杂,无法做到真正的无标记化基因编辑。基于前期研究,Nguyen等人进一步优化了CRISPR系统,使其能够在不同白念珠菌遗传背景下进行标记回收。LEUpOUT系统只能在具有LEU2/leu2Δ特性的菌株中进行基因编辑,而HIS-FLP系统可以用于任何菌株。这两个系统都能够将包含筛选标记、CAS9基因和sgRNA的基因座瞬时整合至LEU2或者HIS位点,完成目标基因的删除后,分别通过重复序列的同源重组(LEUpOUT系统)或者FLP重组酶的激活将基因座再切除下来。上述系统不需要进行分子克隆,不会在基因组中留下永久性标记,就可以高效、快速、精确的完成白念珠菌的基因编辑[7,21]。Vyas等[15]又探索出标记回收的新方法,他们设计的质粒上包含CAS9、sgRNA、NAT和FLP等序列,质粒整合至目标基因后,通过激活FLP重组酶将CRISPR-Cas9系统各元件移除,这种质粒设计允许标记回收,在基因组上仅会留下尽可能少的外源序列,可以在任何遗传背景下进行高效的连续诱变。
通过对CRISPR-Cas9系统的不断改进,该方法已经广泛应用于白念珠菌的单基因或双基因删除、基因标记以及开放阅读框的重组等基因编辑的研究[22]。
2.2 非白念珠菌
CTG进化枝(CTG clade)中的菌种将CGU密码子翻译成丝氨酸而不是亮氨酸,主要包括白念珠菌、近平滑念珠菌复合体、热带念珠菌、葡萄牙念珠菌和耳念珠菌等,而光滑念珠菌不是CTG进化枝的成员。此外,不同的念珠菌依赖于不同的DNA修复机制,白念珠菌和近平滑念珠菌复合体大多数通过HDR的方式完成DNA修复,葡萄牙念珠菌主要依赖于NHEJ,而热带念珠菌和光滑念珠菌同时使用HDR和NHEJ两种方式[7]。CRISPR-Cas9系统已经陆续成功应用于各种非白念珠菌的研究中,为念珠菌基因功能和代谢调控等研究提供了高效、快速和简便的方法。
Grahl等[23]发现在白念珠菌中有效工作的表达构建体不能在其他念珠菌中良好表达,为了规避这种菌种特异性表达的问题,他们构建了无需表达的CRISPR基因编辑系统,并成功应用于葡萄牙念珠菌、光滑念珠菌和耳念珠菌。不同于以往CAS9和sgRNA需在细胞内进行表达,Grahl等将Cas9蛋白和特异性的crRNA: tracrRNA双链RNA在体外组装为核糖核蛋白复合体(ribonucleoprotein complexes,RNP),以NAT基因作为筛选标记,目标基因两侧0.5~1 kb的区域作为标记基因的同源臂。然后将RNP和含有筛选标记的DNA一同电转化至上述3种菌的细胞内,通过同源重组的方式便可诱导目标基因的删除。在这3个菌种中,该方法的转化效率都很高,达到60%~70%。然而,该方法的缺点是不能进行标记回收。
此前,对于葡萄牙念珠菌,Norton等利用瞬时表达的CRISPR-Cas9系统进行基因删除,使用葡萄牙念珠菌的特异性启动子驱动CAS9和sgRNA在该菌种中的有效表达,筛选标记NAT基因两侧连接约1 kb的目标基因的同源臂诱导HDR,可成功应用于单倍体和双倍体菌株的多个基因的删除,编辑效率约为36%。而研究进一步发现,删除NHEJ相关基因KU70和LIG4,该系统的基因编辑效率明显升高至81%[24]。
光滑念珠菌作为非CTG进化枝的一员,相比于白念珠菌,它在系统发育上更接近酿酒酵母。在Grahl等人的研究前,Enkler等[25]就首次将依赖于质粒的CRISPR-Cas9系统应用在光滑念珠菌中,并成功产生功能性缺失的突变体。该研究利用三重营养缺陷ΔHTL菌株(his3,trp1和leu2)作为实验菌株,并分别使用来自酿酒酵母的TEF1启动子(pTEF1)和RNA聚合酶Ⅲ启动子(pSNR52),以及来自光滑念珠菌CYC1启动子(pCYC1)和RNAH1启动子(pRNAH1)驱动CAS9和sgRNA表达。研究结果显示当使用pCYC1和pRNAH1启动CAS9和sgRNA表达,光滑念珠菌通过NHEJ方式对ADE2基因进行破坏的效率高达100%。Vyas等构建的游离型质粒不仅成功应用于白念珠菌,也成功实现了原养型光滑念珠菌的基因编辑。在提供和不提供修复模板的情况下,都能够将终止密码子提前引入ADE2基因,完成基因敲除[15]。Maroc等开发的可诱导型CRISPR-Cas9系统具有一项新优势,即可以将菌株的转化步骤和基因的切割及修复过程分开。这个系统仍然依赖于包含sgRNA和CAS9的质粒系统,使用MET3启动子控制CAS9的表达,可以通过NHEJ和HDR的方式在光滑念珠菌中进行基因编辑。因该系统中CAS9的表达是可诱导的,故可以利用该系统鉴定光滑念珠菌中的必需基因以及研究双链断裂后的修复机制。Maroc等[26]的研究也进一步证实了NHEJ是光滑念珠菌双链断裂后的主要修复方式。
近平滑念珠菌复合体包括近平滑念珠菌(Candidaparapsilosis)、似平滑念珠菌(Candidametapsilosis)和拟平滑念珠菌(Candidaorthopsilosis)。2017年,Lombardi等应用于近平滑念珠菌的CRISPR-Cas9系统依赖于自主复制质粒,可通过修复模板的巧妙设计,进行基因编辑(引入终止密码子或特定突变)、基因删除或者添加特定标签等操作,将修复模板与包含CAS9、sgRNA和NAT基因的质粒共同转化,甚至可以实现仅有目标基因组被破坏、无需添加任何无关序列的 “无痕性”基因编辑。这个系统的优势在于能够在不同遗传背景的近平滑念珠菌中进行基因编辑,Lombardi等利用该系统已经成功完成了20株临床菌株的基因编辑。在缺乏选择性压力的情况下质粒容易丢失,因此可以用于同一遗传背景菌株的连续基因编辑[14]。Zoppo等[27]使用上述CRISPR-Cas9系统首次成功地在拟平滑念珠菌中进行基因编辑,证实CoALS4210基因在拟平滑念珠菌对人类颊上皮细胞的黏附中发挥重要作用,但是与侵袭性感染的发生关系不大。随后,Lombardi等又进一步改良了该质粒,将sgRNA连接的锤头型核酸酶(hammerhead ribozymes,HH)序列改为能够直接被酵母菌核糖核酸酶Z裂解切割的tRNA,因此仅需要替换20nt的靶序列互补区即可靶向不同的目标基因,极大的简化了克隆过程。Lombardi等已经将此改良的CRISPR-Cas9系统pCP-tRNA成功地应用于近平滑、拟平滑和似平滑念珠菌的基因编辑[28]。
同时,Lombardi等构建了另外一个质粒pCT-tRNA用于热带念珠菌的基因编辑,除调控序列和自主复制序列不同外,pCT-tRNA质粒的结构与pCP-tRNA基本一致。其中,驱动CAS、NAT和sgRNA表达的调控元件分别是来自季也蒙毕赤酵母、都柏林念珠菌的和Ashbyagossypii;而自主复制序列ARS2来自白念珠菌,该系统高效的完成了ADE2基因的敲除[7,28]。Zhang等[11]通过依赖于质粒的整合型和瞬时表达型CRISPR-Cas9系统,快速、可靠地进行多基因敲除或突变,该系统还大大提高了体内多个DNA片段的组装效率,并仅通过一个步骤即可将他们整合至目标基因座上。
综上所述,本文提供了CRISPR-Cas9基因编辑技术在念珠菌中应用的最新进展,灵活、有效的CRISPR-Cas9系统对医学真菌学领域产生了巨大的影响。在实验室方面,通过促进基因操作技术的发展,为探究真菌生物学和致病机制、剖析毒力因子的作用、调查新的药物潜在靶点以及研究宿主和病原体间的相互作用奠定了基础;在临床方面,进一步改善了真菌感染的诊断策略和抗真菌耐药的监测手段。此外,目前该系统在模式真菌中应用可以进一步推广至其他致病真菌,从而研究宿主和真菌间的相互作用或者解决念珠菌耐药等问题[7]。

