贵州仡佬族和苗族人群30个插入/缺失位点遗传多态性分析*
刘亚举,胡 萌,石美森,李学博
1.河南省许昌市公安局刑事科学技术研究所,河南许昌 461000;2.铁道警察学院公安技术系,河南郑州 450053;3.中国政法大学刑事司法学院侦查学研究所,北京100192;4.山东省高校证据鉴识重点实验室(山东政法学院),山东济南 250014
插入/缺失(InDel)多态性遗传标记是指基因组中插入或缺失不同大小的DNA片段所形成的多态性遗传标记。InDel在人类基因组中分布广泛,一般平均每隔7.2 kb就会有一个InDel位点,即约20%的DNA多态性是InDel,在法医学应用领域中,由于InDel具有突变率相对较低、扩增片段较小(通常<200 bp)的特点,适用于降解检材的分析和亲缘关系鉴定,同时InDel是二等位基因,不适合用于混合检材的分析,但其兼具了短串联重复序列(STR)和单核苷酸多态性(SNP)两种遗传标记的双重优点,且可以用法医DNA实验室的毛细管电泳平台进行基因分型检测,分型方法简便、快速、准确,易于在法医DNA实验室进行推广和应用。本研究针对贵州仡佬族和苗族人群常染色体上30个InDel位点的遗传多态性进行分析,评估其在法医学中的应用价值,同时为个体识别和亲权鉴定、群体间遗传分布等相关研究提供参考数据。
1 材料与方法
1.1材料来源 根据知情同意原则,从健康体检人群中随机采集贵州仡佬族(220例,其中男175例、女45例)和苗族(207例,其中男157例、女50例)无血缘关系个体外周血0.1 mL,滴入采血卡(武汉骥腾公司),阴干后常温保存。本研究得到相关道德伦理委员会批准。
通过中国知网(www.cnki.net)和NCBI(www.ncbi.nlm.nih.gov)数据库获得5个汉族[北京汉族(210例)、河南汉族(293例)、上海汉族(565例)、成都汉族(129例)、广东汉族(300例)]和13个少数民族[新疆维吾尔族(223例)、西藏藏族(226例)、福建畲族(119例)、四川彝族(54例)、广西壮族(49例)、广西侗族(50例)、广西苗族(50例)、四川藏族(136例)、云南彝族(122例)、新疆锡伯族(223例)、湖北土家族(236例)、新疆哈萨克族(96例)、云南白族(125例)]等18个人群的30个InDel位点等位基因频率作为群体遗传关系比较的数据。
1.2仪器与试剂 Hamilton STAR-LET自动化工作站(AusBio公司,瑞士);平板离心机(Eppendorf公司,德国);9700型PCR扩增仪(AB公司,美国);3500XL型遗传分析仪和ID-X分析软件(AB公司,美国);GeneMarker®HID分析软件(SoftGenetics公司,美国);Investigator®DIPplex试剂盒(Qiagen公司,德国)。
1.3方法 采用5% Chelex-100快速提取法提取标本DNA。在Eppendorf Mastercycler pro S银座热循环仪上进行复合扩增,Investigator®DIPplex试剂盒反应体系为10 μL,其中Reaction Mix A 2.0 μL,Primer Mix 2.0 μL,Multi Taq2 DNA Polymerase 0.25 μL,纯水4.75 μL,模板DNA 1.0 μL(0.5~1.0 ng/μL)。PCR扩增条件:94 ℃预变性4 min;94 ℃变性30 s,61 ℃复性120 s,72 ℃延伸75 s,30个循环;最后68 ℃延伸60 min,10 ℃保温。取1.0 μL扩增产物与DNA size standard 550(标记橙色荧光素BTO)0.5 μL和10.0 μL去离子甲酰胺混合,95 ℃变性3 min后,冰浴3 min,置3500XL型遗传分析仪上进行全自动毛细管电泳,Data Collection软件收集数据,用GeneMarker®HID分析软件进行等位基因分型,并采用GeneMapper ID-X v1.5分析软件复核。每批标本检测均采用XX28和超纯水分别作为阳性对照和阴性对照。
1.4统计学处理 用PowerStats v12.xls软件统计分析30个InDel位点的等位基因频率、个体识别率(DP)、累积个体识别率(TDP)、多态信息含量(PIC)和非父排除率(PE)等法医学相关参数;采用Cervus 3.0软件计算三联体累积非父排除率(CPEtrio)和二联体累积非父排除率(CPEduo)。采用Arlequin v3.5软件计算30个InDel位点的观察杂合度(Ho)和期望杂合度(He),并进行各位点的Hardy-Weinberg平衡及连锁不平衡检验;采用Haploview软件分析Hardy-Weinberg平衡P值。采用GENEPOP v4.2软件计算各群体间遗传距离和遗传分化系数(Fst)等参数。
2 结 果
2.1贵州仡佬族和苗族人群30个InDel位点的群体遗传学参数 在贵州220例仡佬族和207例苗族无关个体血样中,Investigator®DIPplex荧光检测试剂盒中的30个InDel位点都得到了有效扩增,且各位点间扩增产物平衡。经Bonferroni校正,30个InDel位点的基因型频率分布均符合Hardy-Weinberg平衡(P>0.05),各位点的群体遗传学参数见表1。

表1 贵州仡佬族和苗族群体30个InDel位点的群体遗传学参数
2.2贵州仡佬族和苗族人群30个InDel位点的连锁不平衡检验 经Arlequin v3.5软件分析后发现,贵州仡佬族和苗族人群的30个InDel位点之间相互独立,各位点间均不存在连锁不平衡现象(P>0.05)。采用乘积定律计算,TDP分别为0.999 999 999 34和0.999 999 996 86,CPEduo分别为0.951 2和0.952 8,CPEtrio分别为0.995 6和0.995 7。
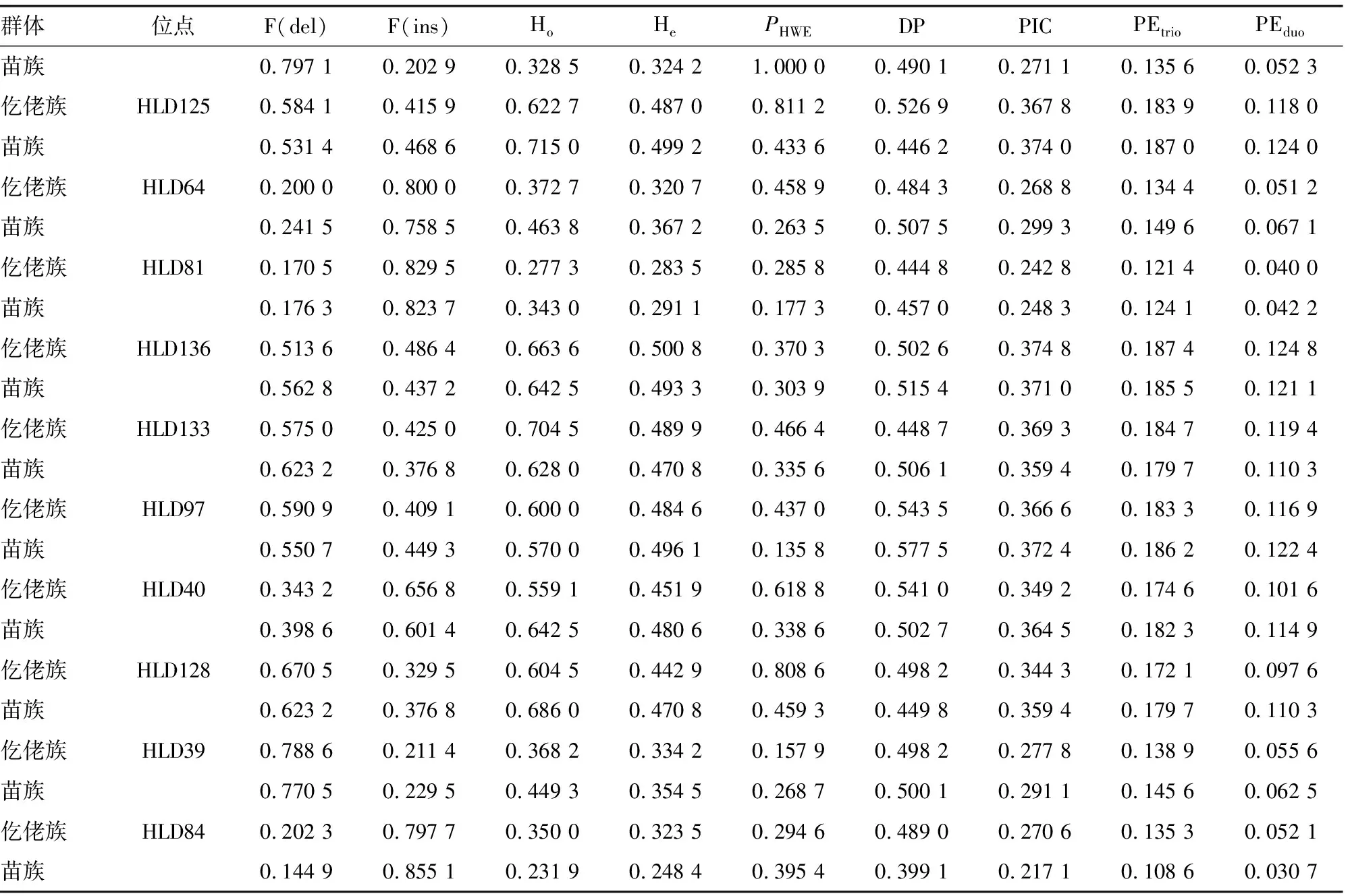
续表1 贵州仡佬族和苗族群体30个InDel位点的群体遗传学参数
2.3贵州仡佬族和苗族与其他群体的遗传分化系数Fst及遗传距离 应用本研究获得的30个InDel位点基因频率数据分析了贵州仡佬族和苗族人群与18个比较群体的Nei′s DA遗传距离,见表2;通过缺失频率进行计算的20个群体之间的遗传分化系数Fst值,见表3。结果显示,仡佬族和苗族与成都汉族的遗传距离较近,分别与新疆维吾尔族和哈萨克族的遗传距离较远。
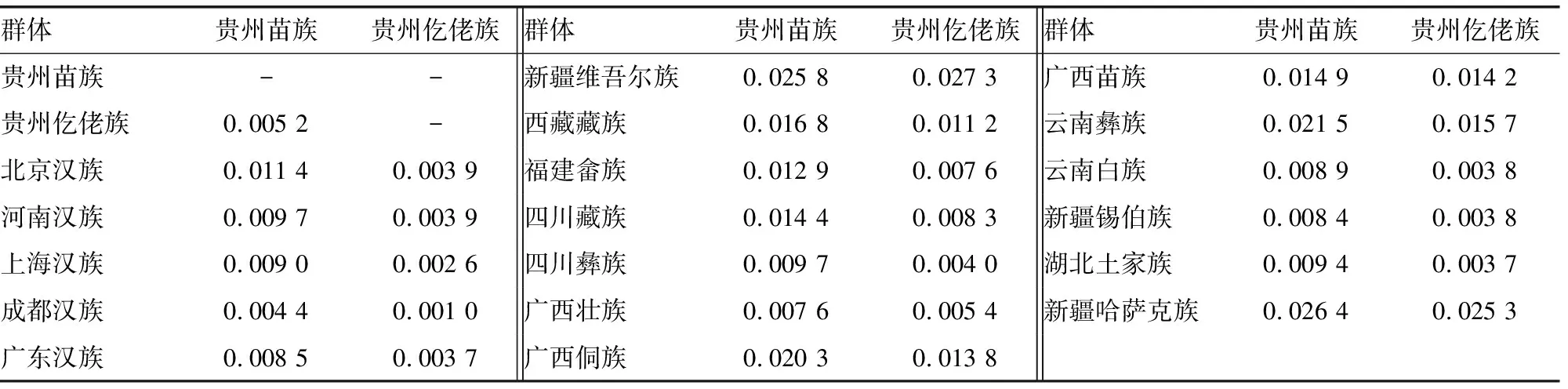
表2 贵州仡佬族、苗族群体与其他18个群体的遗传距离
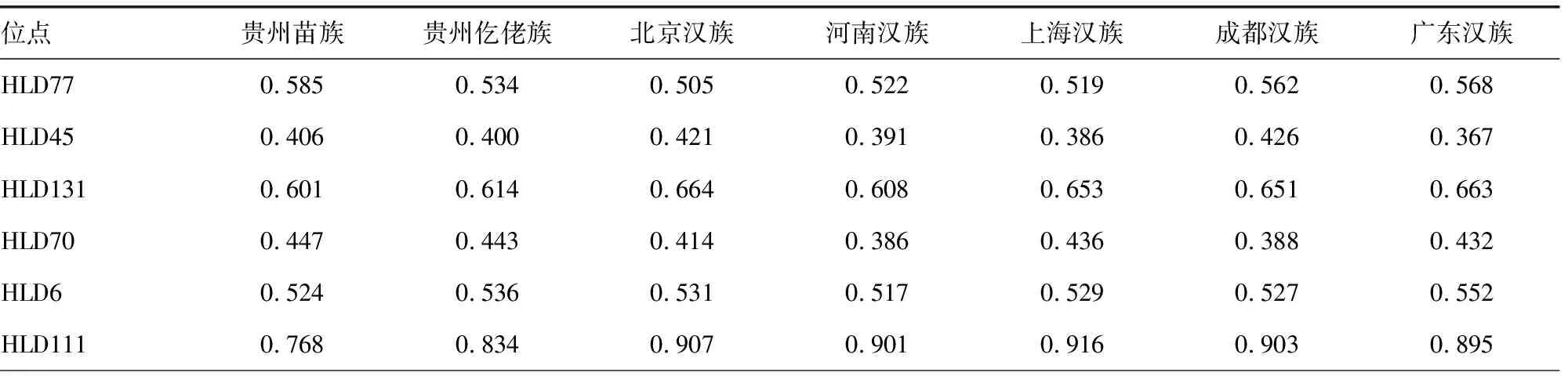
表3 30个InDel 遗传标记在20个群体中的遗传分化系数Fst值

续表3 30个InDel 遗传标记在20个群体中的遗传分化系数Fst值

续表3 30个InDel 遗传标记在20个群体中的遗传分化系数Fst值
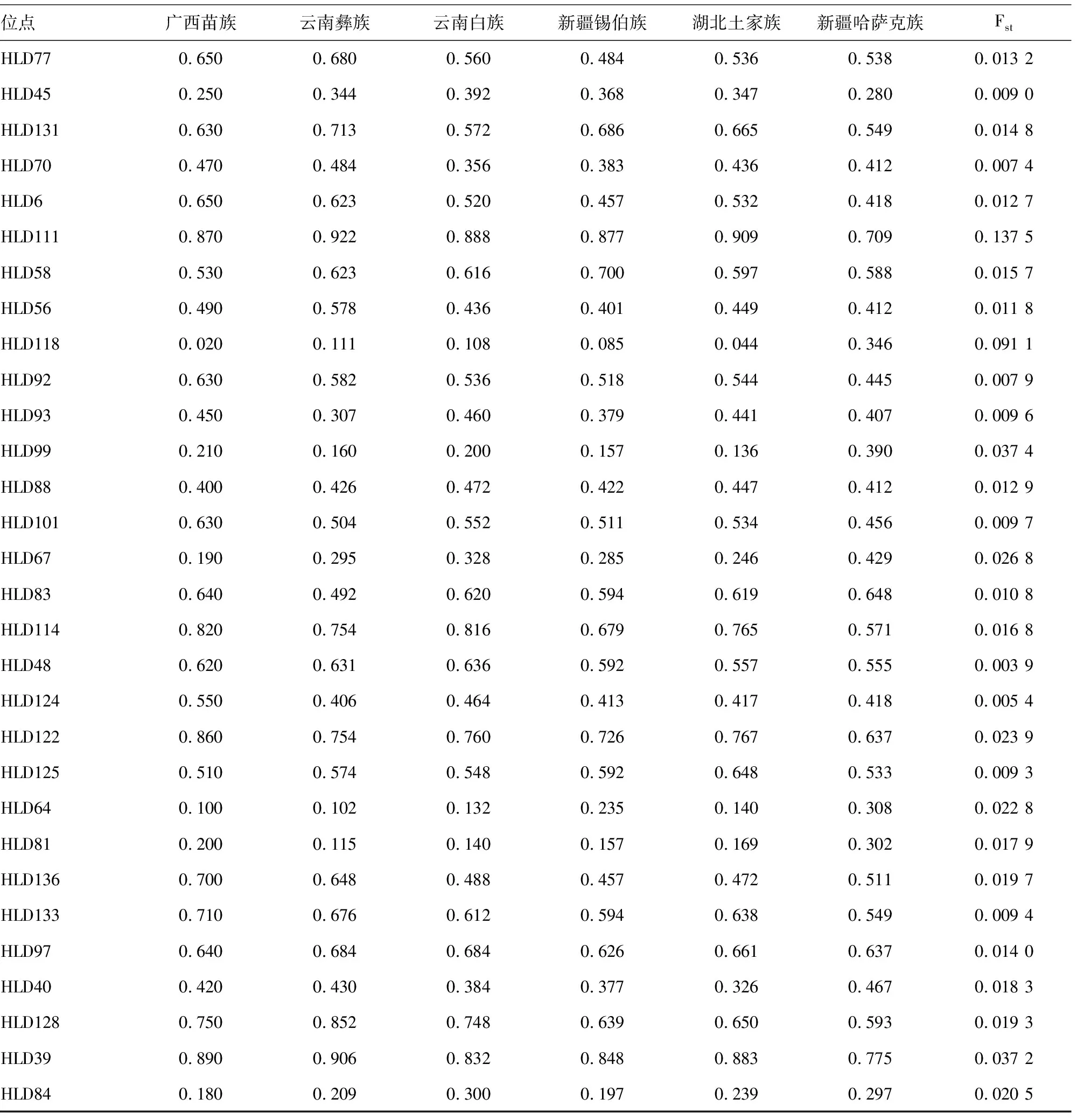
位点广西苗族云南彝族云南白族新疆锡伯族湖北土家族新疆哈萨克族FstHLD770.6500.6800.5600.4840.5360.5380.013 2HLD450.2500.3440.3920.3680.3470.2800.009 0HLD1310.6300.7130.5720.6860.6650.5490.014 8HLD700.4700.4840.3560.3830.4360.4120.007 4HLD60.6500.6230.5200.4570.5320.4180.012 7HLD1110.8700.9220.8880.8770.9090.7090.137 5HLD580.5300.6230.6160.7000.5970.5880.015 7HLD560.4900.5780.4360.4010.4490.4120.011 8HLD1180.0200.1110.1080.0850.0440.3460.091 1HLD920.6300.5820.5360.5180.5440.4450.007 9HLD930.4500.3070.4600.3790.4410.4070.009 6HLD990.2100.1600.2000.1570.1360.3900.037 4HLD880.4000.4260.4720.4220.4470.4120.012 9HLD1010.6300.5040.5520.5110.5340.4560.009 7HLD670.1900.2950.3280.2850.2460.4290.026 8HLD830.6400.4920.6200.5940.6190.6480.010 8HLD1140.8200.7540.8160.6790.7650.5710.016 8HLD480.6200.6310.6360.5920.5570.5550.003 9HLD1240.5500.4060.4640.4130.4170.4180.005 4HLD1220.8600.7540.7600.7260.7670.6370.023 9HLD1250.5100.5740.5480.5920.6480.5330.009 3HLD640.1000.1020.1320.2350.1400.3080.022 8HLD810.2000.1150.1400.1570.1690.3020.017 9HLD1360.7000.6480.4880.4570.4720.5110.019 7HLD1330.7100.6760.6120.5940.6380.5490.009 4HLD970.6400.6840.6840.6260.6610.6370.014 0HLD400.4200.4300.3840.3770.3260.4670.018 3HLD1280.7500.8520.7480.6390.6500.5930.019 3HLD390.8900.9060.8320.8480.8830.7750.037 2HLD840.1800.2090.3000.1970.2390.2970.020 5
3 讨 论
贵州是个多民族省份,现有少数民族49个,世居17个。仡佬族是贵州最古老的民族之一,且贵州是仡佬族的发源地。贵州仡佬族和苗族人口分别占全国同一民族总人口的97%和50%以上,分属于汉藏语系的壮侗语族和苗瑶语族。目前,国内外学者对仡佬族和苗族白细胞抗原(HLA-G)、线粒体DNA和STR等遗传标记的多态性进行了研究[1-8],而本研究对427例仡佬族和苗族健康无关个体的30个InDel位点的遗传多态性进行分析,同时探讨其与其他群体之间的遗传学关系,有助于分析InDel遗传标记在不同民族或同一民族不同群体间的遗传、进化的相互关系,为科学应用InDel遗传标记提供理论基础。
Hardy-Weinberg遗传平衡检验结果一方面可以反映基因分型的质量,另一方面也反映研究对象的代表性是否较好。本研究结果显示,经过Bonferroni法校正,30个InDel位点的基因型频率分布在贵州仡佬族和苗族群体中符合Hardy-Weinberg遗传平衡,说明贵州仡佬族和苗族群体在符合随机采样的情况下,群体中个体的基因频率可从一代传到另一代且维持不变,保持遗传的平衡稳定性,本次标本随机性和采集数目符合统计学要求。连锁不平衡检验结果表明,30个InDel位点在贵州仡佬族和苗族群体中不存在连锁不平衡现象(P>0.05),提示这些位点是相互独立的遗传标记,可以应用于法医学鉴定。
本研究根据30个InDel位点的等位基因频率进行了贵州仡佬族和苗族与其他群体的遗传距离分析,结果显示,贵州仡佬族和苗族与成都汉族的遗传距离较近,分别与新疆维吾尔族和哈萨克族的遗传距离较远。刘亚举等[7-8]基于常染色体STR和Y-STR遗传标记研究了仡佬族和苗族的遗传结构及与其他群体的遗传关系,发现仡佬族和苗族与湖北、湖南、重庆汉族有较近的遗传距离,与西藏藏族、甘肃裕固族、青海藏族有较远的遗传距离。李彬彬等[3]和姚永刚等[9]调查线粒体DNA 9bp缺失频率,结果发现仡佬族为25%、苗族为32.4%,与目前认为南方民族人群的缺失频率高于北方民族人群的观点是一致的[9]。何燕等[5]基于贵州苗族等人群的线粒体DNA多态性研究表明,组成“南方谱系”的B、M7、N9、R9等在贵州苗族等3个民族中频率为51.22%,同时构建了20个人群mtDNA单倍群频率主成分,分析结果表明贵州苗族是南方民族。刘烜等[10]研究了贵州仡佬族等贵州境内多个民族个体外周血标本Y染色体单核苷酸多态(Y-SNP),其中仡佬族H8单倍型(M134)和苗族H8(M134)、H11(M95)、H12(M88)频率较高,也表明仡佬族和苗族均是南方民族。这些研究表明,InDel作为遗传标记物,以等位基因频率计算遗传距离可用于群体遗传学的研究,和地域实际的物理距离区分度相似,与民族的起源、进化、迁徙的研究数据结果一致[11-12]。
遗传分化系数Fst值是进行群体间遗传分化概括分析最常用的方法之一,是评价基因座等位基因频率在群体间的分布差异程度。WRIGHT认为群体间Fst值在0.00~<0.05表示群体间不存在分化,Fst值在0.05~<0.15则为中度分化,Fst值在0.15~0.25则为高度分化,Fst值>0.25则分化度极大。WEI等[13]对中国汉族、藏族、维吾尔族和哈萨克族群体30个InDel位点的Fst分析结果显示,HLD111和HLD118位点遗传分化贡献率相对较高。LARUE等[14]对北美地区的非洲裔、高加索人、亚裔和西南部西班牙裔的Fst分析结果也证实,HLD111和HLD118等位点遗传分化贡献率相对较高。本研究中,HLD111和HLD118位点Fst值分别为0.137 5、0.091 1,在20个群体中遗传分化贡献率相对较高,提示这两个位点在不同民族,尤其是不同人种中基因型分布有很大的差异,反映群体差异性大,在群体遗传学和种群识别方面有重要的应用价值。
综上所述,由于贵州仡佬族和苗族大多在偏远地区聚族而居,生活环境相对封闭,各民族之间隔离程度较高,民族特异性保存较好,这些遗传特性成为人类遗传多样性的重要组分,可作为遗传学上很好的研究材料。这些民族的遗传物质是全人类遗传多样性的重要组成部分,研究这些民族的起源和进化对于探索中华民族乃至东亚人群的起源也具有重要意义。

