肺栓塞患者差异甲基化位点及区域分析*
曹国磊, 何丽丽, 马荣辉, 唐 乐, 曹嘉瑞, 牛海文, 罗 琴
新疆医科大学附属肿瘤医院呼吸与神经内科,乌鲁木齐 830011
肺血栓栓塞症(pulmonary thromboembolism,PTE)是呼吸系统疾病中比较常见的一种血管疾病,是由于各种原因导致的血栓栓子堵塞肺动脉干或其分支,引起肺循环障碍的临床、病理生理综合征[1-2]。研究显示,近年来国内PTE的患病率、疾病构成比均显著增长[3]。高达15%的PTE患者在患病后1月内死亡,30%的患者在未来10年内复发[4]。
DNA甲基化是在DNA甲基转移酶(DNA methyltransferases,DNMT)的催化作用下,二核苷酸中C核苷酸上第5个碳原子发生甲基化转变,成为甲基化的胞嘧啶[5]。研究证明,DNA的异常甲基化在恶性肿瘤、自身免疫性疾病及心血管疾病中发挥着重要作用[6],但关于其与PTE关系的研究却罕见报道。本研究拟通过Illumina Infinium Methylation EPIC Bead Chip芯片(850K芯片)甲基化检测平台在全基因组水平检测PTE患者与正常对照者每个CpG位点的甲基化水平,并对比分析PTE患者与正常对照组之间差异甲基化位点、区域及其目的基因,进一步揭示PTE的发病机制。
1 材料与方法
1.1 一般资料
纳入标准:经肺动脉CT血管造影确诊肺动脉存在血栓,无心脑血管疾病史,无内分泌疾病史。对照组选取标准:选取与PTE组性别比例、年龄相当,既往无心脑血管、血液、内分泌、肝肾疾病,且无慢性阻塞性肺疾病(COPD)、哮喘等病史的相同地区人群作为正常对照组。所有入组人员均签署相关知情同意书,并经新疆医科大学附属肿瘤医院伦理委员会批准(伦理审批号为:2019BC007)。收取2019年1月至2019年12月就诊的肺栓塞患者和正常对照人群外周血标本各10例,进行DNA提取。
1.2 甲基化检测
采用Illumina Infinium Methylation EPIC Bead Chip芯片(850K芯片)甲基化检测平台检测分析各样本中每个CpG位点的甲基化水平并行差异位点、区域分析。
1.2.1 DNA的提取及样品质检 使用试剂盒(北京天根生化科技有限公司产品)提取外周血中的基因组DNA,用分光光度计定量,并将样品调到标准浓度50 ng/μL,共计20 μL,然后用0.8%的琼脂糖凝胶电泳。样品电泳主带清晰,通常不小于10 kb,没有明显降解,总量5 μg以上,可进行下游的甲基化芯片实验。
1.2.2 亚硫酸盐转化及DNA扩增 根据Illumina官方推荐的Zymo EZ DNA Methylation Kit优化方法进行亚硫酸盐转化。
1.2.3 制备MSA3板 在样本中加入0.1 mol/L NaOH使DNA变性为单链,经中和后加入全基因组扩增试剂,在37℃恒温条件下过夜孵育。
1.2.4 DNA的片段化、沉淀、重悬 扩增后产物经过酶解处理,得到片段化DNA。DNA片段加入异丙醇在4℃下离心沉淀。沉淀后的DNA在空气中干燥后,加入杂交缓冲试剂使DNA沉淀重新溶解。
1.2.5 DNA芯片制备 将重悬后的DNA样本与准备好的芯片杂交,置于杂交炉内过夜。在杂交过程中,片段化后的DNA经过变性,与特异位点的50个碱基(连接在芯片的微珠上)退火。洗去未杂交和非特异杂交的DNA,以捕获到的DNA为模板,在芯片上进行单碱基的延伸反应,在芯片上加上可检测的荧光基团,从而区分样本的甲基化状况。将反应完成的芯片放入XC4试剂中,使其表面包裹上一层粘性透明液体,再将其放入真空环境下干燥1 h,从而将芯片包被,以使其信号稳定较长的时间。
1.2.6 芯片扫描和数据提取 提前下载相应的manifest文件,将处理好的芯片放入扫描仪,利用激光激发芯片上单碱基延伸产物的荧光基团,扫描仪获取由荧光基团发出的荧光,并生成原始数据,记录扫描结果存放的位置。由此所得的数据直接导入R包Champ软件进行分析,从而得到每个样本每个位点的甲基化水平数据。
1.2.7 差异甲基化位点分析 将芯片原始扫描数据idat文件直接导入R包Champ进行分析,得到每个样品每个CpG位点的原始信号值、样品分组等信息。然后使用一系列R软件包计算出每个样品每个CpG位点的甲基化水平,进行甲基化差异分析,以P<0.05为差异有统计学意义。
1.2.8 差异甲基化区域分析 采用Bumphunter的方法寻找差异甲基化区域(differentially methylated regions,DMR),该方法首先根据位点距离信息来聚簇(cluster),然后再在每个cluster中筛选出甲基化水平一致的连续候选区段,并根据候选区段构建线性统计模型,筛选出明显差异的区段(P<0.05)。
1.3 统计学分析
采用R包Limma 3.32.10进行差异甲基化分析,然后根据P值筛选差异位点(P<0.05)。采用Limma moderatedt-statistics和empirical Bayes方法检验差异位点的显著性。采用Bumphunter的方法根据候选区段构建线性统计模型,筛选出明显差异的区段(P<0.05)。
2 结果
2.1 差异甲基化位点分析
将PTE与正常对照组所有甲基化位点进行对比分析,在全基因组水平共筛选到95286个有意义的差异CpG位点(P<0.05),其中37983个为高甲基化,57303个为低甲基化(图1)。对应28273个目的基因,其中13785个为高甲基化,14488个为低甲基化。我们分别列出两组差异最大的高甲基化与低甲基化CpG位点各10个(表1),显著差异CpG位点在CpG岛的分布情况为:CpG岛29%(27483)、Shore 18%(16700)、Shelf 6%(6011)、Open sea 47%(45092)。
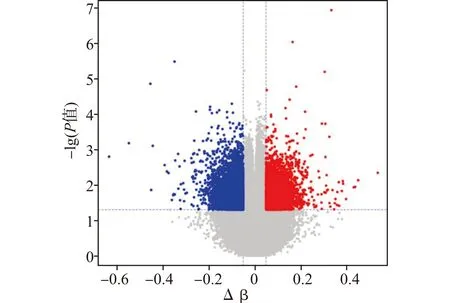
图中标注为红色的是高甲基化位点,标注为蓝色的是低甲基化位点,标注为灰色的是无显著差异位点。图1 差异CpG位点分布火山图Fig.1 Volcano map of the distribution of differential CpG sites

表1 PTE组与正常对照组差异甲基化位点对比Table 1 Comparison of differential methylation sites between the PTE group and the normal control group
2.2 差异甲基化区域分析
采用Bumphunter的方法寻找PTE与正常对照组差异甲基化区域,结果筛选出两组差异甲基化区域DMR-1、DMR-2,分别位于3号染色体(195489708-195490309)和6号染色体(49681178-49681774)上(均P<0.05)。DMR-1宽度601 bp,DMR-2宽度596 bp,DMR-1包含8个CpG位点,其所属的簇包含8个CpG位点,DMR-2包含11个CpG位点,其所属的簇包含11个CpG位点,且PTE组DMR-1与DMR-2所有CpG位点甲基化水平均较正常对照组高(表2)。DMR-1所含CpG位点为cg23170091、cg18918831、cg16034541等,目的基因为MUC4;DMR-2所含CpG位点为cg01706515、cg14997592、cg26715042等,目的基因为CRISP2(表3)。

表2 PTE组与正常对照组差异甲基化区域对比Table 2 Comparison of differentially methylated areas between the PTE group and the normal control group
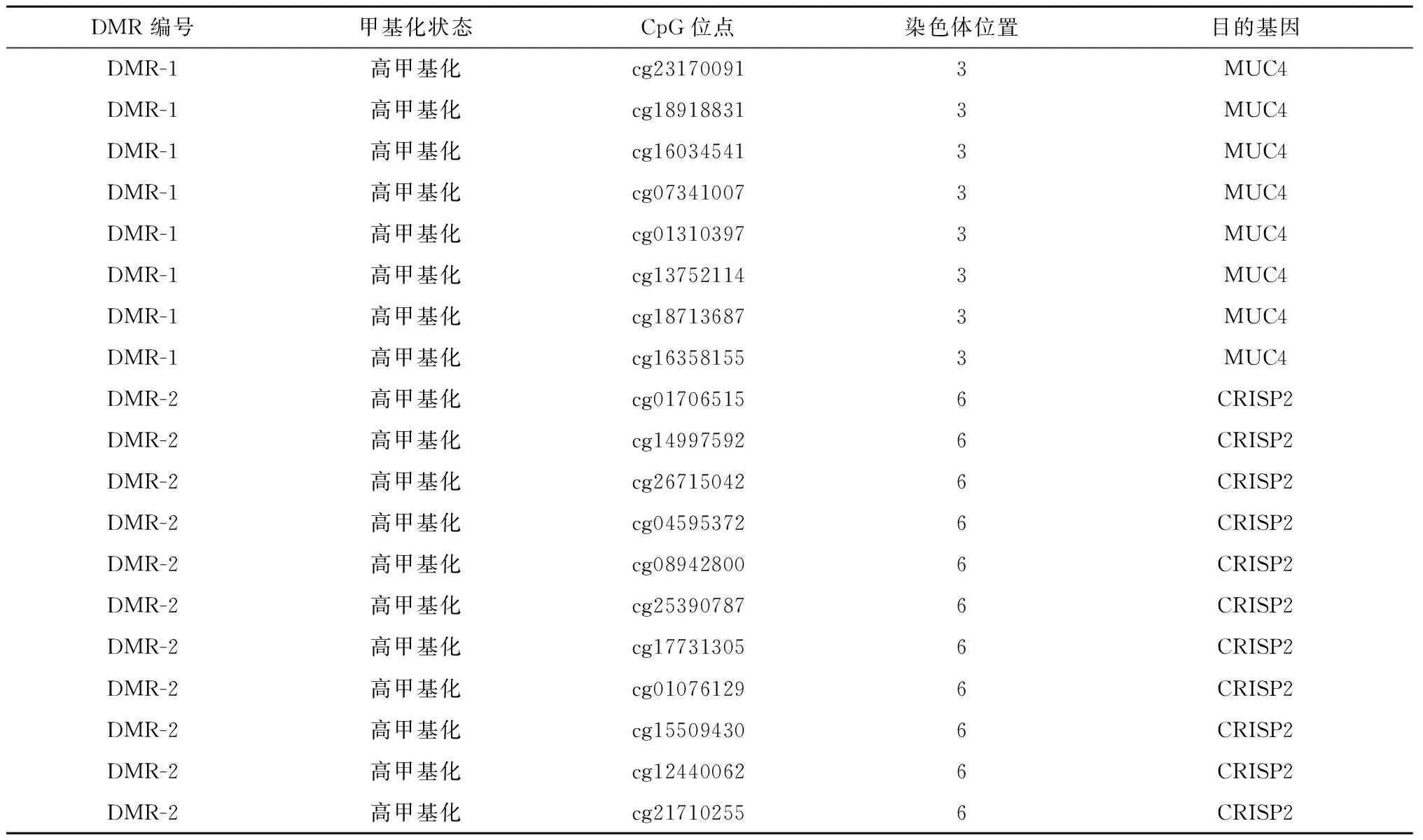
表3 PTE组与正常对照组差异甲基化区域所含甲基化位点及目的基因Table 3 Methylation sites and target genes contained in the differentially methylated regions of the PTE group and the normal control group
3 讨论
已有许多研究证据表明DNA异常甲基化在慢性阻塞性肺疾病、支气管哮喘、肺癌、特发性肺纤维化等肺部疾病中的重要作用。如Sundar等[7]研究发现,吸烟者和慢性阻塞性肺疾病患者肺组织中BID和NOS1AP基因的表达与DNA甲基化状态的改变关系显著,继而影响细胞的衰老、凋亡等。而Yokoyama等[8]研究证明,TET1所介导的DNA低甲基化可调控肺腺癌MUC4基因的表达,但关于PTE与DNA甲基化的关系目前尚未见报道。
本研究利用甲基化检测平台在全基因组水平检测PTE组患者及正常对照组人群CpG位点甲基化水平,对比分析PTE组与正常对照组差异甲基化位点及区域。结果在全基因组水平共筛选到95286个有意义的差异CpG位点(P<0.05),其中高甲基化的位点cg11738485,cg06417478,cg04657146等差异较大,低甲基化的位点cg06758191、cg18105134、cg21790413等差异较大,对应目的基因分别为HOOK2(cg06417478)、PROZ(cg18105134)。HOOK2(微管系连蛋白2)是一种蛋白质编码基因,有研究表明2型糖尿病和肥胖患者HOOK2基因的甲基化与2型糖尿病的发生相关[9],其在血栓性疾病中的作用目前未见报道。而PROZ(蛋白质Z)是一种维生素K依赖的蛋白质,具有促凝与抗凝双向调节作用,Santacroce等[10]对197例深部静脉血栓(DVT)和197例健康人群的研究表明低PROZ血浆水平与DVT相关。这种关系是否建立在PROZ基因甲基化的基础之上有待我们进一步研究证实。
众所周知,基因的转录过程有赖于CpG岛的甲基化状态,若是甲基化状态出现了异常,便会影响到基因的转录过程。然而,DNA异常甲基化不仅发生在CpG岛,也可能发生在CpG Shore、CpG Shelf等区域。本研究发现,显著差异的CpG位点在CpG岛的分布情况为:CpG岛29%(27483)、CpG Shore18%(16700)、CpG Shelf 6%(6011)、Open sea 47%(45092)。除广阔的Open sea区域之外,发生在CpG岛的差异CpG位点依然是最多的,达到了29%,远高于CpG Shore和CpG Shelf。这与Dudziec等[11]有关CpG岛和CpG shores的研究结果相一致。
差异甲基化区域(DMR)指基因组上不同组间甲基化值有明显差异的一个片段。本研究筛选出2组差异甲基化区域DMR-1,DMR-2,发现PTE组DMR-1与DMR-2所有CpG位点甲基化水平均较正常对照组高。DMR-1所对应目的基因为MUC4,DMR-2所对应目的基因为CRISP2。关于MUC4基因,研究主要集中在与肿瘤发生、发展的关系,并可作为肿瘤发展过程的标记物。在表观遗传学方面,曾有学者报道MUC4的表观遗传学有赖于5′-末端的2个CpG岛,在某些肿瘤中这种调节显得尤为重要[12]。而肿瘤患者因其体内血液高凝状态等本身便是血栓高危因素,有研究显示,肺癌患者发生静脉血栓的风险是普通人群的22倍,并使患者病死率增加2~8倍[13]。据此,我们推测,或许是由于MUC4基因的异常甲基化导致了恶性肿瘤的发生,继而引起了血栓的形成;抑或是MUC4基因的异常甲基化本身便与肺栓塞有着直接的关系。无论是哪种可能,DMR-1中的这些差异CpG位点都值得我们去关注并继续研究。关于CRISP2基因,目前研究主要集中在其与弱精症之间的关系,但本研究却发现了CRISP2基因的高甲基化与肺栓塞的关系,为广大学者提供了新的思路。
综上所述,肺栓塞患者CpG位点的高甲基化可能参与了肺栓塞的发生,差异较大的CpG位点cg06417478、cg18105134及其目的基因HOOK2、PROZ和DMR-1、DMR-2中包含的差异CpG位点及其目的基因MUC4、CRISP2的异常甲基化或许在肺栓塞的发生中起到重要的作用。课题组拟进一步扩大样本量,同时对以上基因相关mRNA、蛋白等进行验证,以期深入揭示表观遗传学机制在肺栓塞发生、发展中的作用,为肺栓塞的研究提供新的思路。

