铜绿丽金龟气味结合蛋白AcorOBP11的表达纯化及功能分析
秦健辉,李金桥,赵旭,李克斌,曹雅忠,尹姣
铜绿丽金龟气味结合蛋白AcorOBP11的表达纯化及功能分析
秦健辉,李金桥,赵旭,李克斌,曹雅忠,尹姣
中国农业科学院植物保护研究所植物病虫害生物学国家重点实验室,北京 100193
【】通过研究铜绿丽金龟()气味结合蛋白11(odorant binding protein 11,OBP11)与寄主植物挥发物的结合特性,探讨AcorOBP11的功能,为阐明铜绿丽金龟识别寄主植物的嗅觉分子机制打下基础。设计特异性引物,通过RT-PCR克隆,分析其序列特征并与相似序列进行对比;将目的基因连入原核表达载体pET28a后,重组质粒转入大肠杆菌感受态细胞BL21(DE3)。利用IPTG诱导AcorOBP11重组蛋白的表达,超声破碎菌体,取上清液过镍柱,利用重组肠激酶切除his标签后再次过镍柱纯化获得目的蛋白;纯化后的蛋白以1-NPN为荧光探针开展荧光竞争结合试验,测定AcorOBP11蛋白与37种寄主植物挥发物的结合特性;利用Modeller对AcorOBP11进行同源建模,以冈比亚按蚊气味结合蛋白AgamOBP48(PDB ID:4ij7)作为模板,获得其三维结构;利用Autodock半柔性对接将蛋白与化合物对接,模拟AcorOBP11与6种候选寄主植物挥发物的结合情况。克隆获得了全长基因,开放阅读框共639 bp,N末端有17个氨基酸组成的信号肽,具有8个保守的半胱氨酸位点,属于Plus-C OBP亚家族,进化树结果表明其与HparOBP9进化关系最近。成功连入pET28a表达载体,在0.25 mmol·L-1IPTG、37℃条件下诱导表达,并通过两次镍柱纯化得到目的蛋白。竞争结合试验结果表明,AcorOBP11结合谱较广,对-紫罗兰酮、异戊醛、丙烯酸-2-乙基己酯、乙酸龙脑酯、柠檬烯、反-2-己烯醛、2-乙基己醛、异戊酸等13种寄主植物挥发物有较好的结合能力。其中,与寄主植物挥发物-紫罗兰酮结合能力最好,竞争解离常数为22.78 μmol·L-1。构建了AcorOBP11三维结构图并进行蛋白构象评估后,与6种化合物进行分子对接,结果表明AcorOBP11与-紫罗兰酮结合自由能最低,为-24.74 kJ·mol-1,表明其与-紫罗兰酮的结合能力最强,与荧光竞争结合结果一致;从对接图还可以看出,-紫罗兰酮不仅与AcorOBP11在Cys163处形成氢键,而且其疏水端进入了蛋白的疏水腔,两个甲基与蛋白表面高度契合,因此,-紫罗兰酮与蛋白间结合能力最强。AcorOBP11能够识别多种寄主植物挥发物,推测其在铜绿丽金龟成虫定位寄主植物中发挥重要作用,此研究结果为生态防控铜绿丽金龟提供了新思路。
铜绿丽金龟;气味结合蛋白11;原核表达;荧光竞争结合;同源建模;分子对接
0 引言
【研究意义】铜绿丽金龟()属于鞘翅目,丽金龟科,是我国重要的地下害虫,其成虫主要危害榆树、柳树、苹果树、桃树、苘麻等植物叶片,造成巨大的经济损失。长期以来,化学农药是防治铜绿丽金龟成虫的主要手段,但长期使用化学农药不仅会杀伤天敌、增强害虫抗药性,还会造成一定的环境问题。研究铜绿丽金龟嗅觉蛋白对寄主植物挥发物的识别机制可明确铜绿丽金龟对寄主植物的选择机理,为铜绿丽金龟的生态防控提供新思路。【前人研究进展】目前,开发成虫引诱剂是地下害虫绿色防控的研究热点,其中植物挥发性信息物质是常见的引诱剂之一。一些寄主植物挥发性成分已被证明对金龟子有显著的诱集作用[1]。如丁酸苯乙酯、丁香酚、香叶醇以3﹕7﹕3比例混合时对日本丽金龟()有很好的引诱效果,引诱剂现已商品化[2];由苯乙醛、苯甲醛和苯甲醇混配而成的引诱剂能强烈吸引扁绿丽金龟()成虫[3];邻苯二甲酸二丁酯、肉桂醛、顺-3-己烯基乙酸酯三者混配对华北大黑鳃金龟()成虫有较好的引诱活性[4]。同样地,铜绿丽金龟对甲基庚烯酮、顺-3-己烯乙酸酯等植物挥发物也产生明显的趋性行为反应[5]。昆虫主要通过高度灵敏的嗅觉系统来识别环境中的寄主植物挥发物,进而产生定位寄主、取食等一系列的行为反应,其中嗅觉蛋白是其发挥嗅觉功能的基础[6]。嗅觉蛋白主要在成虫触角中表达,主要包括气味结合蛋白(odorant binding protein,OBP)、化学感受蛋白(chemosensory protein,CSP)、气味受体(odorant receptor,OR)、离子型受体(ionotropic receptor,IR)、味觉受体(gustatory receptor,GR)、感觉神经元膜蛋白(sensory neuron membrane protein,SNMP)和气味降解酶(odorant-degrading enzyme,ODE)[7]。其中OBP是昆虫专一性识别外界气味物质的第一步生化反应[8]。研究表明,暗黑鳃金龟()HparOBP1和HparOBP2主要识别寄主植物挥发物[9],HparOBP15a可结合多种寄主植物挥发物与两种性信息素[10];华北大黑鳃金龟HoblOBP1、2、3、4、13识别寄主植物挥发物,HoblOBP5、8、9和24可以识别产卵信息素[11-14]。另外,通过分子模拟和蛋白突变解析了华北大黑鳃金龟HoblOBP1和HoblOBP1/OBP2异源二聚体对寄主植物挥发物识别的分子机制[15],为阐明鳃金龟的嗅觉识别机制打下了基础。【本研究切入点】作为田间优势种群且成虫危害严重的铜绿丽金龟的嗅觉蛋白研究较少,仅探讨了AcorOBP1识别寄主植物小叶女贞挥发物的功能[16]。因此,本研究通过克隆铜绿丽金龟OBP11的全长基因(),原核表达并纯化获得重组蛋白,利用荧光竞争结合试验明确其与37种寄主植物挥发物的结合特征,结合同源建模和分子对接探讨蛋白与不同化合物的结合机理。【拟解决的关键问题】研究AcorOBP11与寄主植物挥发物的结合特性,为阐明铜绿丽金龟在定位寄主植物过程中的嗅觉识别机制打下基础。
1 材料与方法
试验于2019年在中国农业科学院植物保护研究所完成。
1.1 供试材料
铜绿丽金龟采集于河北省石家庄市,成虫饲养于养虫盒中(60 cm×50 cm×40 cm),室温(25±1)℃,光周期为16L﹕8D。将羽化5—7日龄成虫的触角用镊子取下后迅速置于液氮中速冻,于-80℃保存备用。
Trizol试剂购自生工生物工程(上海)有限公司,FastKing一步法除基因组cDNA第一链合成预混试剂、胶回收试剂盒购于天根生化科技(北京)有限公司,限制性内切酶HⅠ、d Ⅲ购于日本Takara公司,1×Taq PCR Master Mix、DNA Marker、感受态细胞DH5、BL21(DE3)感受态细胞购于北京博迈德基因技术有限公司,PEASY-T载体、Trans-T1感受态细胞、重组肠激酶购于北京全式金生物技术有限公司,T4 DNA Ligase购于Promega公司,Kan、Amp、X-gal、IPTG购于Sigma公司,引物由生工生物工程(上海)有限公司合成。
1.2 RNA提取及cDNA第一链的合成
收集铜绿丽金龟雌、雄成虫的触角各60对,平均分装到3个1.5 ml离心管中,将收集到的样品立即置于预冷的无RNA酶研钵中研磨,根据TRIzol试剂提供的说明书提取触角总RNA,加入适量DEPC水溶解沉淀。利用核酸蛋白浓度测定仪测定RNA浓度及质量,用1.0%琼脂糖凝胶电泳检测RNA完整性。按照FastKing一步法除基因组cDNA第一链合成预混试剂说明书进行反转录合成cDNA第一链,作为RT-PCR的扩增模板。
1.3 AcorOBP11克隆及序列分析
根据GenBank登录的(KM251651)基因全长序列,利用Primer Premier 5.0设计特异性引物,引物加上H Ⅰ和d Ⅲ酶切位点(下划线表示)及保护碱基,上下游引物分别为AcorOBP11F:5′-CGTTCGAGGATCAGGCT TATAAT-3′;AcorOBP11R:5′-CCCTTAGCA CTTGACAGGTGGTG-3′。
以1.2反转录合成的cDNA为模板,在PCR仪中进行扩增。RT-PCR反应体系为50 μL,其中cDNA模板2 μL,上下游引物(10 μmol·L-1)各2 μL,其余用1×Taq PCR Master Mix补齐。扩增程序:95℃ 5 min;95℃ 30 s,45℃ 30 s,72℃ 30 s,30个循环;72℃ 10 min。用1.0%琼脂糖凝胶电泳检测扩增产物,按照普通琼脂糖凝胶回收试剂盒说明书回收符合预期大小的DNA片段。将回收产物与PEASY-T载体连接,转化到Trans-T1感受态细胞中,在含有IPTG、X-gal和AMP的LB固体培养基中进行蓝白斑筛选,挑选5个阳性菌落至10 μl ddH2O中,进行菌落PCR,利用琼脂糖凝胶电泳验证目的基因是否成功插入。将正确的阳性菌落置于LB液体培养基中振荡培养12—16 h(37℃,220 r/min),送至生工生物工程公司测序。测序结果通过DNAMAN进行序列比对进一步验证。
利用ClustaX2.1进行AcorOBP11与其他昆虫OBP的多序列比对,通过MEGA7软件中的最大似然法构建系统进化树。
1.4 AcorOBP11基因重组表达载体构建及原核表达纯化
利用限制性内切酶HⅠ和d Ⅲ将含有目的片段的重组克隆质粒PEASY-T-AcorOBP11和表达载体pET28a进行双酶切,用普通琼脂糖凝胶回收试剂盒分别回收表达载体和目的片段。利用T4 DNA连接酶将目的片段连接到表达载体pET28a上,再转化到DH5感受态细胞中,挑取阳性克隆培养,送至生工生物工程公司测序验证。将测序正确的重组质粒pET28a/AcorOBP11转入大肠杆菌BL21(DE3)表达感受态细胞,挑取单菌落,培养测序。将测序正确的菌液按1﹕100(v/v)的比例接种于新鲜的LB液体培养基(含终浓度50 μg·mL-1Kan)中,37℃,220 r/min振荡培养至OD600值为0.8—1.0时,加入终浓度为0.25 mmol·L-1IPTG溶液进行诱导,37℃,220 r/min摇床中诱导4 h。离心收集菌体,加入裂解液将菌体重新悬浮,超声波破碎,12 000 r/min离心30 min,收集上清和沉淀,用SDS-PAGE检测蛋白的表达。使用镍柱亲和层析方法纯化蛋白,使用咪唑磷酸缓冲液进行梯度洗脱,纯化后蛋白用重组肠激酶切除His标签,再次过柱纯化,置于50 mmol·L-1Tris-HCl(pH 7.4)缓冲液中过夜透析,定量至2 μmol·L-1,置于-20℃备用。
1.5 荧光竞争结合试验
分别选择来源于榆树、苹果树、桃树等铜绿丽金龟寄主植物的37种植物挥发物(表1)作为候选配体化合物,以色谱级甲醇为溶剂将各气味溶液配制成1 mmol·L-1的试样。使用970 CRT荧光分光光度计(上海精密科学公司)进行荧光竞争结合试验,供试容器为1 cm宽的石英比色皿,激发狭缝10 nm,灵敏度为1,设定激发光波长为337 nm,扫描波长的范围设定为350—520 nm。
取2 mL浓度为2 µmol·L-1的AcorOBP11重组蛋白溶液加入石英比色皿中,逐次加入2 µmol·L-11-NPN直至荧光强度值达到饱和值。分别记录每个浓度梯度产生的荧光强度最大值,利用Scatchard方程计算重组蛋白与1-NPN的结合常数K1-NPN。
在比色皿中加入2 mL浓度为2 μmol·L-1的AcorOBP11蛋白溶液,再加入1-NPN使其终浓度为2 μmol·L-1。将配制好的气味标样逐次加入到比色皿中,使终浓度由2 μmol·L-1递增至40 μmol·L-1,记录荧光强度峰值的变化。假设AcorOBP11活性为100%,且在饱和状态下配体与蛋白按照1﹕1的比例结合。根据公式Ki=[IC50]/(1+[1-NPN]/K1−NPN)计算重组蛋白AcorOBP11与配体的结合常数Ki。[IC50]为AcorOBP11/1-NPN复合物的荧光强度下降50%的配体浓度。[1-NPN]为自由的1-NPN探针的浓度,K1−NPN为AcorOBP11/1-NPN的结合常数。
1.6 AcorOBP11同源建模及分子对接
根据AcorOBP11的氨基酸序列,利用Swiss-model(https://swissmodel.expasy.org/interactive)从蛋白质数据库中搜索模板,选取相似度较高且已解析三维结构的蛋白质作为模板。利用Modeller9.24程序将AcorOBP11氨基酸序列与模板分子序列进行比对,使目标序列与模板对齐,根据比对结果构建5个可能的模型构象,利用Modeller评分函数molecule PDF(molpdf)方法对模型进行评估,选择DOPE score最小的结构作为模板,并利用共轭梯度算法和分子动力学对模板进行优化,产生5个优化后的模板蛋白。利用Procheck(https://servicesn.mbi.ucla.edu/ PROCHECK/)对优化后的模板蛋白主链键长、键角及二面角的合理性、侧链的立体化学稳定性等特征进行评估[17-18],根据拉氏图结果选择最优的蛋白构象进行后续的分子对接试验。
利用Autodock 4.2.6模拟AcorOBP11与寄主挥发物的结合情况。选取荧光竞争结合试验中与AcorOBP11结合较好的-紫罗兰酮、异戊醛、丙烯酸-2-乙基己酯、乙酸龙脑酯、柠檬烯、反-2-己烯醛6种化合物开展分子对接。利用ChemDraw 18.0绘制配体的二维结构,然后通过Chem3D 18.0软件将其转换为三维结构,并使用MM2力场优化配体的构型以获得最稳定的构象。利用Autodock 4.2.6为AcorOBP11添加极性氢原子和电荷,其中AutoGrid主要负责计算网格中的相关能量,而AutoDock负责构象搜索和评估。设置GridBox大小为110×110×110,栅格间距设置为0.375 Å。采用半柔性对接,将蛋白质设置为刚性大分子,将配体设置为柔性分子。使用Lamarckian遗传算法寻找配体与受体最佳的结合位置状态。利用半经验的自由能计算方法计算配体与受体之间的结合能力。对接结果生成50个最佳构象,选取结合能最小的构象用Pymol 2.3.4和LigPlus进行可视化分析。
2 结果
2.1 AcorOBP11序列分析
的开放阅读框(ORF)为639 bp,编码212个氨基酸,预测分子量为23.8 kD,等电点为6.92,氨基酸序列的N末端有17个氨基酸组成的信号肽(图1-A),将AcorOBP11与其他昆虫OBP的氨基酸序列进行序列比对(图1-B),发现其在第6个保守的半胱氨酸后面含有2个保守的半胱氨酸残基和1个脯氨酸残基,属于Plus-C OBP亚家族。构建系统发育树(图1-C),结果显示AcorOBP11与HparOBP9进化关系最近,自展值为100。
2.2 AcorOBP11蛋白表达纯化
将含有pET28a/AcorOBP11质粒的BL21(DE3)单克隆菌落接种培养后,取1 mL菌液作为阴性对照,经终浓度0.25 mmol·L-1IPTG诱导后,SDS-PAGE结果显示,含有His-tag的重组蛋白分子量约在30 kD,与预测的重组蛋白大小一致。将诱导后的菌液超声破碎后,检测包涵体和上清,目的蛋白主要在上清表达。蛋白纯化后用重组肠激酶切除His-tag,获得单一条带的目的蛋白,大小在25 kD左右,符合目的蛋白大小(图2)。
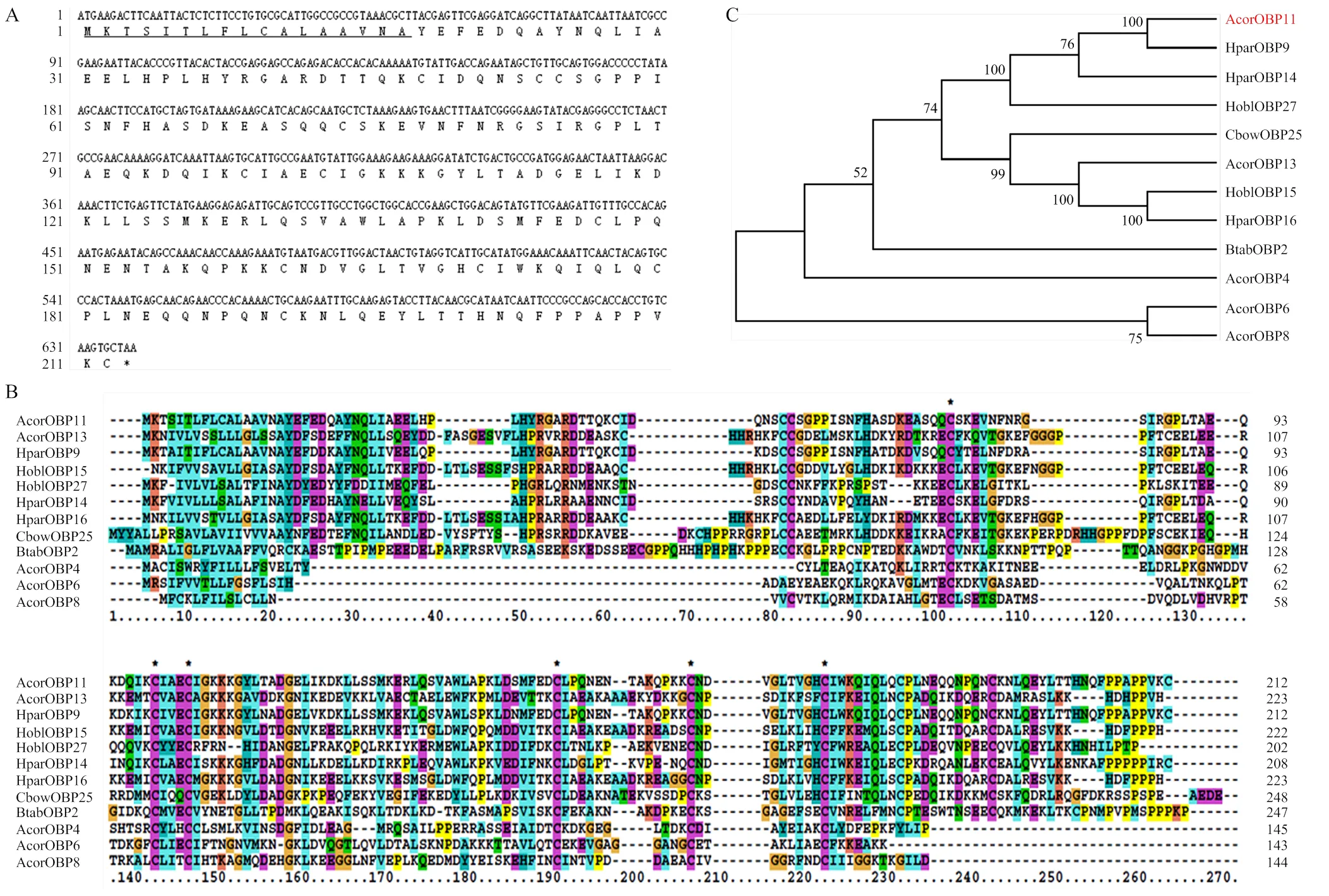
A:AcorOBP11核苷酸序列及其推导氨基酸序列,信号肽用单下划线表示Nucleotide sequence and amino acid sequence of AcorOBP11. The predicted signal peptide is underlined;B:AcorOBP11与其他昆虫OBP的序列比对Sequences alignment of AcorOBP11 with other insect OBPs;C:AcorOBP11与其他昆虫OBP的系统发育树Phylogenetic tree of AcorOBP11 and other insect OBPs
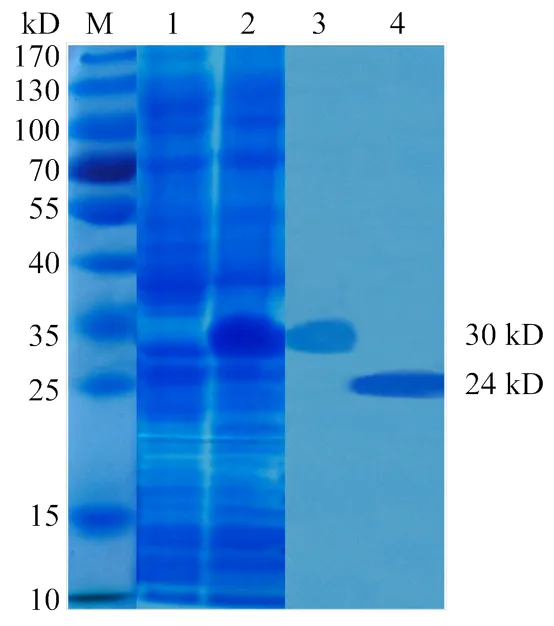
M:蛋白质分子量标准物Protein molecular weight marker;1:未诱导的pET28a-AcorOBP11菌体Non-induced pET28a-AcorOBP11 in Escherichiacoli;2:经过IPTG诱导表达的pET28a-AcorOBP11-His目的蛋白Expressed protein pET28a-AcorOBP11-His after induction by IPTG;3:经过镍柱纯化获得pET28a-AcorOBP11蛋白Purified protein pET28a-AcorOBP11 through Ni-NTA column;4:经过重组肠激酶切除His标签后的pET28a-AcorOBP11蛋白Purified protein pET28a-AcorOBP11 with His-tag cleaved by recombinant enterokinase
2.3 AcorOBP11与气味配体的结合
AcorOBP11与1-NPN的结合曲线如图3-A所示,表明AcorOBP11与1-NPN具有良好的结合特性。利用Scatchard方程可计算出复合物AcorOBP11/1-NPN的结合常数为8.819 μmol·L-1。
铜绿丽金龟与37种植物挥发物的荧光竞争性结合试验结果表明(表1),AcorOBP11结合谱较广,能够识别供试的绝大多数寄主植物挥发物,但其与不同寄主植物挥发物的结合能力存在显著差异(图3-B),对13种寄主植物挥发物具有较强的结合能力(IC50<40 μmol·L-1)。其中,AcorOBP11与植物挥发物-紫罗兰酮结合能力最强,解离常数Ki值为22.78 µmol·L-1,其次是异戊醛(Ki=24.49 µmol·L-1)、丙烯酸-2-乙基己酯(Ki=25.11 µmol·L-1)和乙酸龙脑酯(Ki=26.36 µmol·L-1)。总体而言,AcorOBP11更倾向于结合醛类化合物,除了上述异戊醛,还对反-2-己烯醛、2-乙基己醛、庚醛、丁醛、辛醛有较强的结合能力。
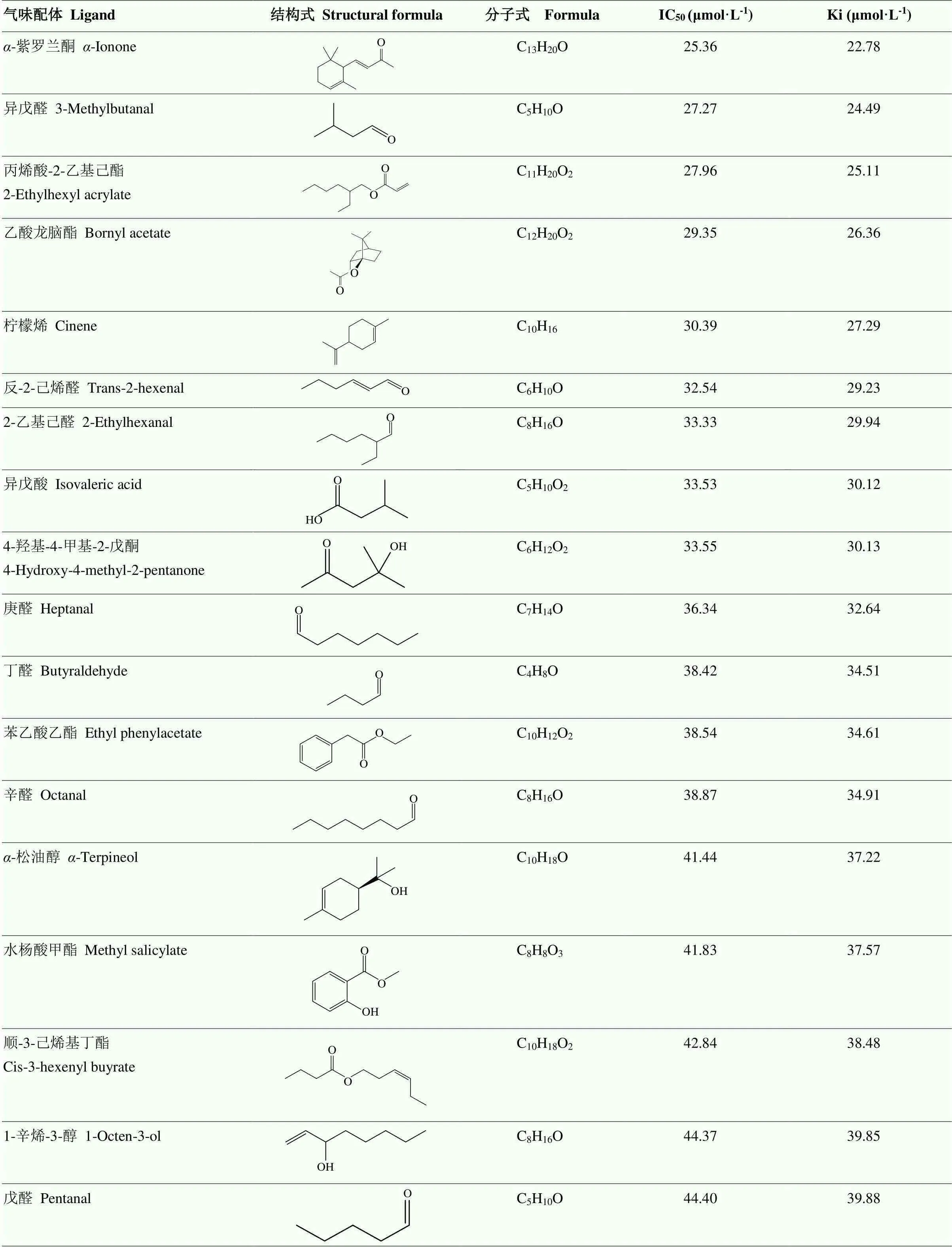
表1 AcorOBP11与配体的结合特性
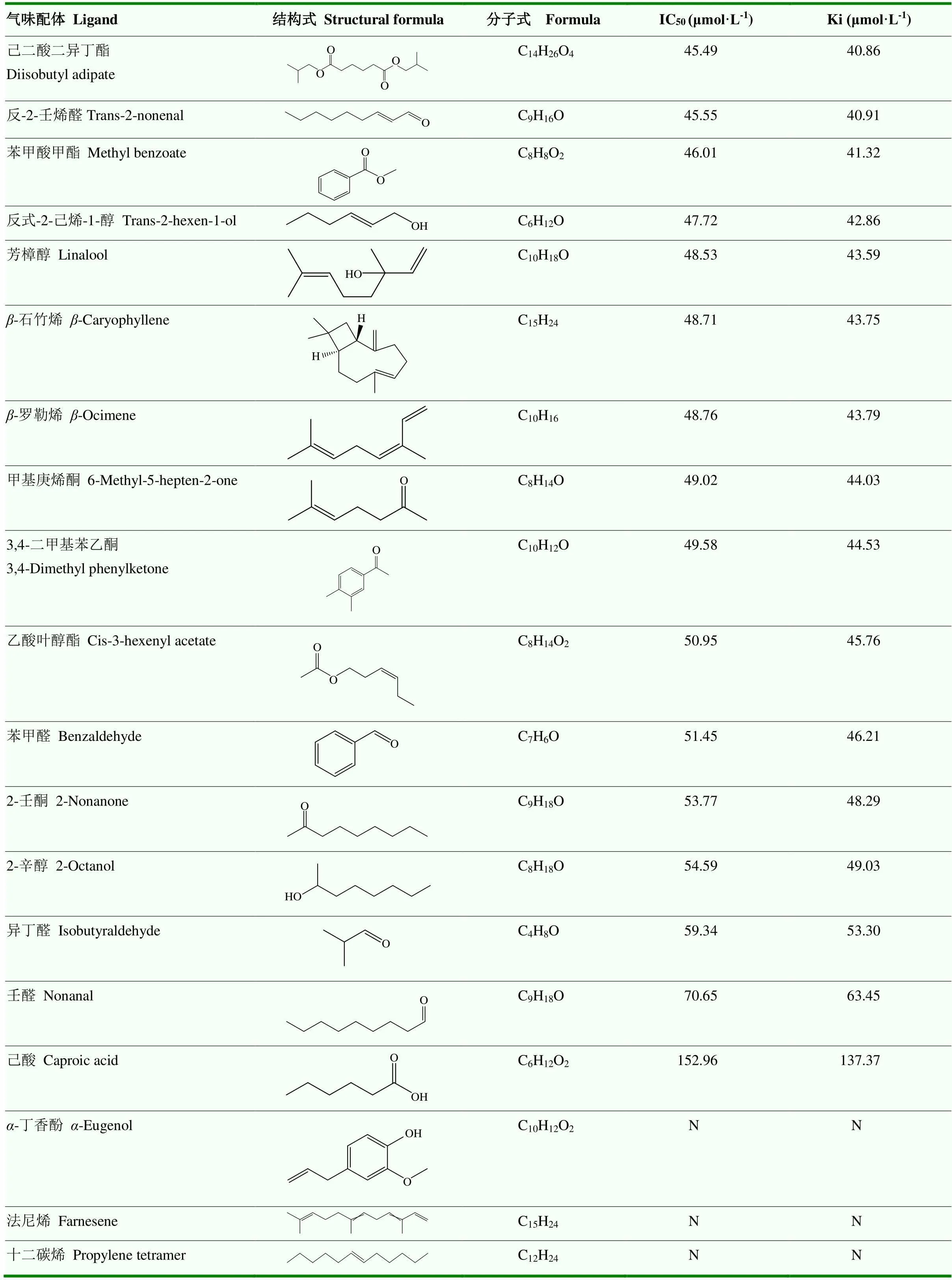
续表1 Continued table 1
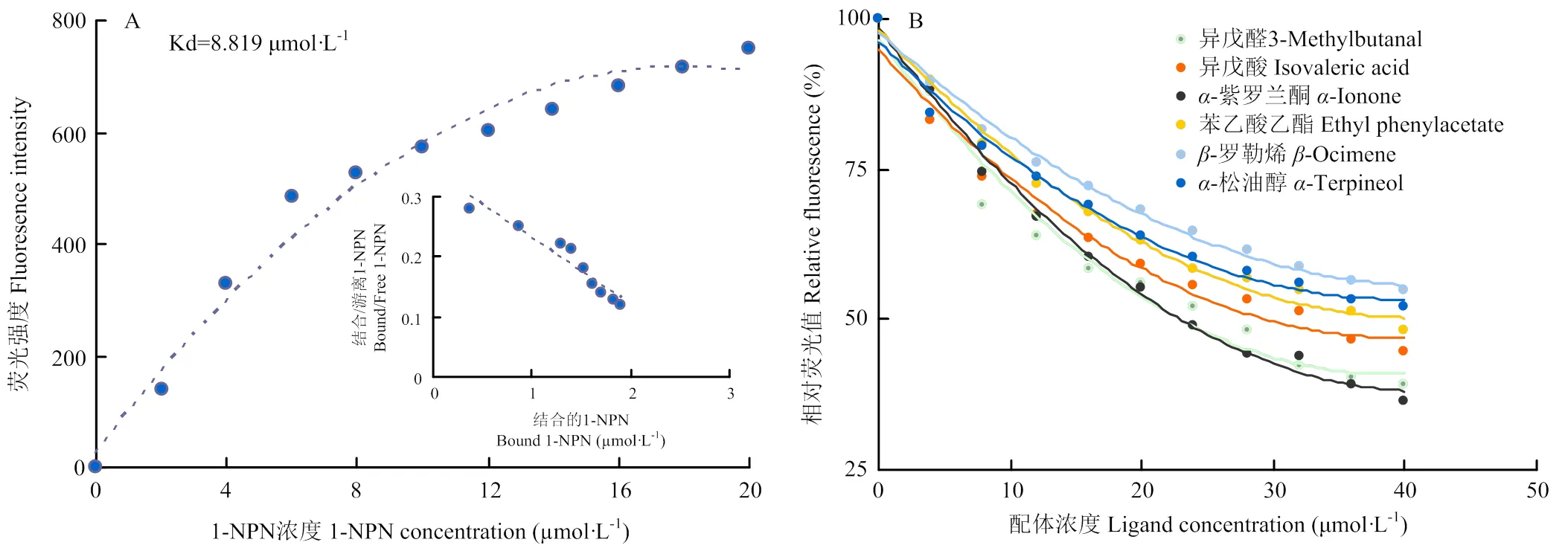
A:AcorOBP11和1-NPN的结合曲线以及Scatchard方程Binding curves and Scatchard plots ofAcorOBP11 and 1-NPN;B:AcorOBP11与6种植物挥发物的结合曲线Binding curves of AcorOBP11 to six plant volatiles
2.4 AcorOBP11同源建模及分子对接
根据Swiss-model模板搜索的结果,选择冈比亚按蚊()气味结合蛋白AgamOBP48(PDB ID:4ij7)作为模板,构建了AcorOBP11三维结构图(图4-A),其主要包括6个螺旋,分别由51—69(1)、82—91(2)、102—113(3)、117—137(4)、146—162(5)、172—184(6)位氨基酸组成。利用Procheck评估蛋白构象的合理性,分析拉氏图(图4-B)可知,AcorOBP11序列中位于最佳区域和较适合区域的氨基酸残基总和>90%,其中87.7%的氨基酸残基位于最佳区域,9.4%的氨基酸残基位于较合适区,表明构建的AcorOBP11三维结构是合理的。
由构建的AcorOBP11三维结构和6种化合物进行分子对接结果可知(表2),AcorOBP11与6种化合物均具有较低的结合能,其中与-紫罗兰酮结合能最低,为-24.74 kJ·mol-1,表明其结合能力最强。由蛋白与配体结合的二维图可以看出(图5),-紫罗兰酮、乙酸龙脑酯、异戊醛、丙烯酸-2-乙基己酯和反-2-己烯醛5种化合物分别与蛋白结合腔中的Cys163、Lys51、Glu52、Ala53、Asn176等氨基酸残基形成了氢键,柠檬烯虽然未与结合腔中的氨基酸残基形成氢键,但与12个氨基酸形成疏水作用。另外,由蛋白与配体结合的三维图(图6)可以看出,-紫罗兰酮的疏水端进入了蛋白的疏水腔中,两个甲基与蛋白表面契合,几何匹配度极高,因此,-紫罗兰酮与蛋白间结合能力最强;乙酸龙脑酯、丙烯酸-2-乙基己酯、异戊醛也分别与结合腔表面凹槽形状匹配;反-2-己烯醛与柠檬烯则完全进入了蛋白结合腔内部,周围分子间作用力很紧密,因此结合力较强。
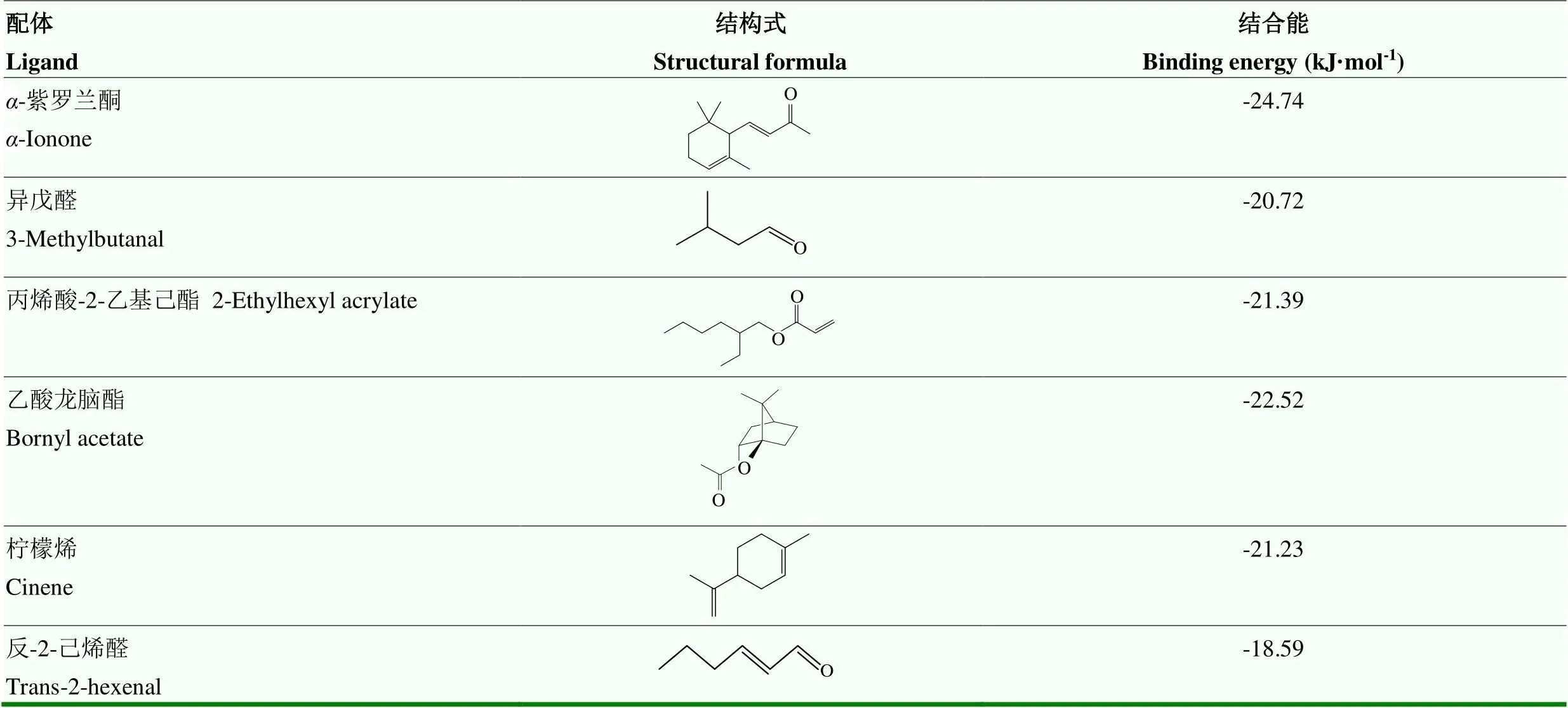
表2 AcorOBP11与配体的结合能
3 讨论
铜绿丽金龟利用灵敏的嗅觉系统来识别环境中的寄主植物挥发物等气味化合物,气味结合蛋白是其行使嗅觉功能的重要嗅觉蛋白[19]。本文克隆了,其编码的氨基酸序列在第6个半胱氨酸后存在2个保守的半胱氨酸残基和1个保守的脯氨酸残基,属于Plus-C OBP亚家族,具有鞘翅目气味结合蛋白的典型特征。寄主植物挥发物在昆虫对寄主植物的定位、识别、取食和产卵等生命活动中发挥重要作用[20]。本研究选取了37种铜绿丽金龟寄主植物挥发物,AcorOBP11与其中13种化合物具有较好的结合能力,如反-2-己烯醛、反-2-己烯醇、辛醛、庚醛等化合物均能引起铜绿丽金龟强烈的行为反应[5,21],表明AcorOBP11在铜绿丽金龟对寄主植物的识别定位中发挥着重要作用。然而,水杨酸甲酯、-松油醇、石竹烯、罗勒烯、3,4-二甲基苯乙酮、苯甲醛、壬醛、法尼烯等植物挥发物虽然也能引起铜绿丽金龟成虫的行为趋性,但是与AcorOBP11结合较弱或不结合,推测这些寄主植物挥发物由铜绿丽金龟其他OBP识别,不同OBP分工明确,协同配合,共同完成其对寄主植物的识别过程[5,21]。
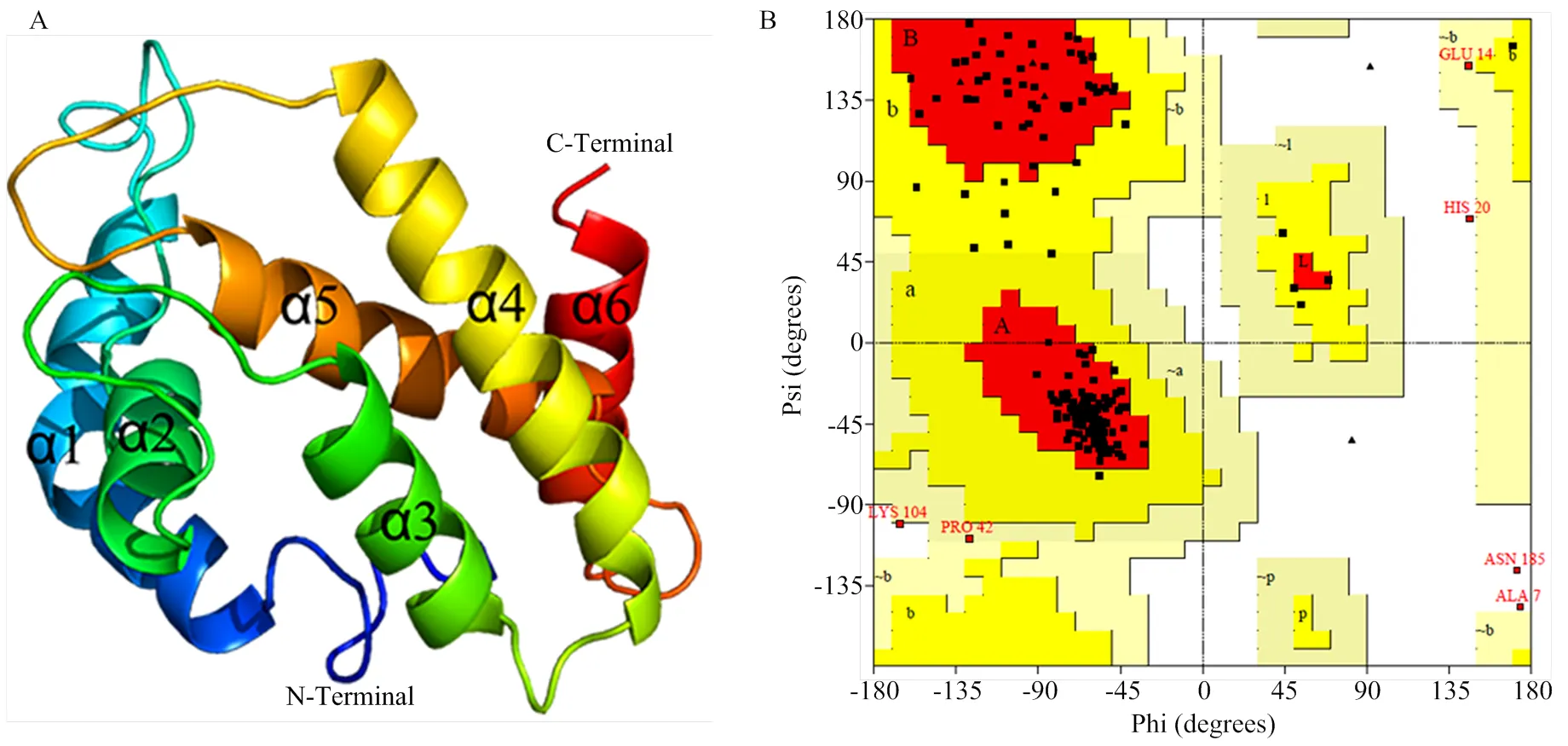
图4 AcorOBP11的三维结构模型(A)及拉氏图(B)
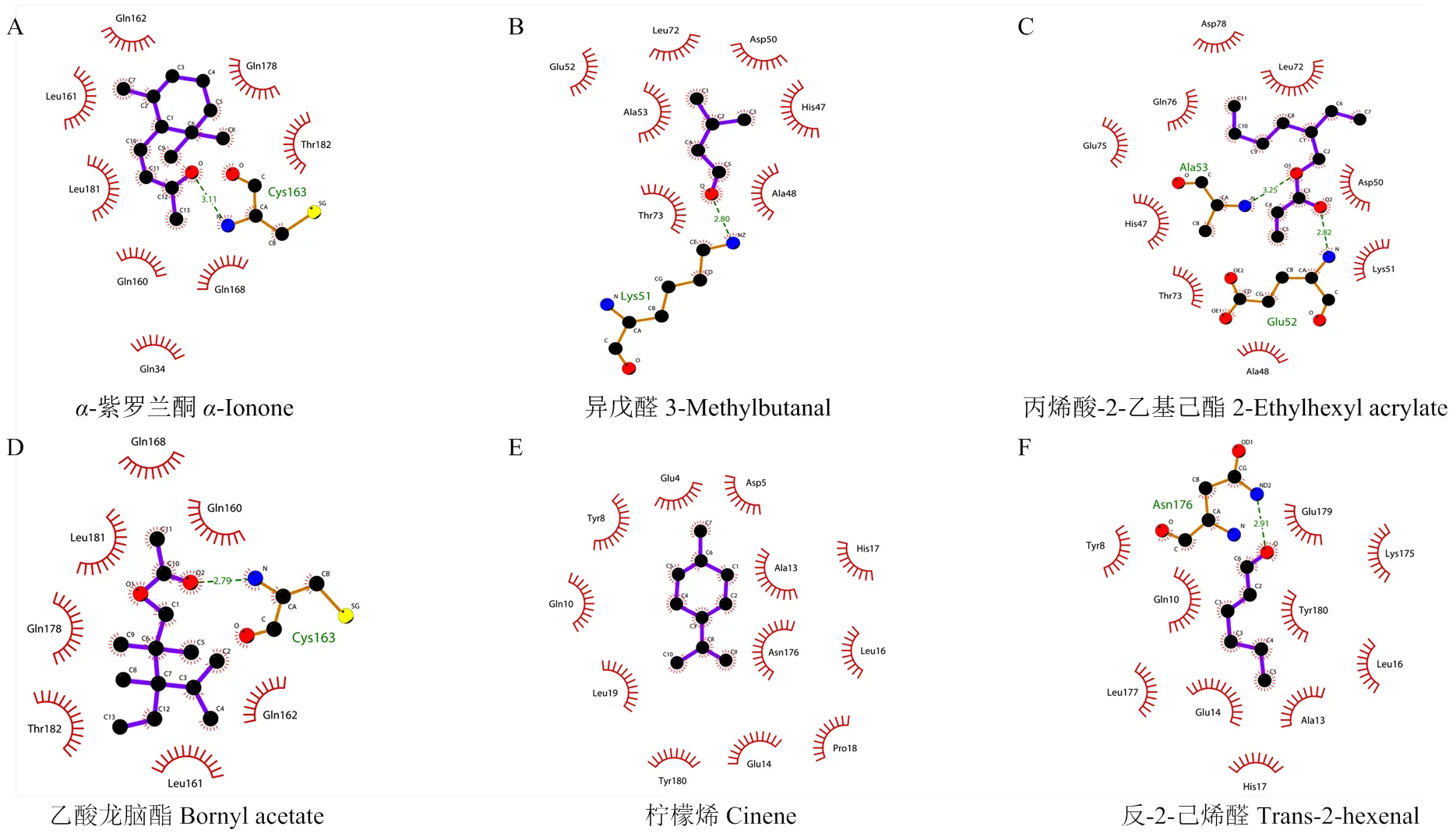
图5 AcorOBP11与配体相互作用的关键氨基酸残基(二维结构)
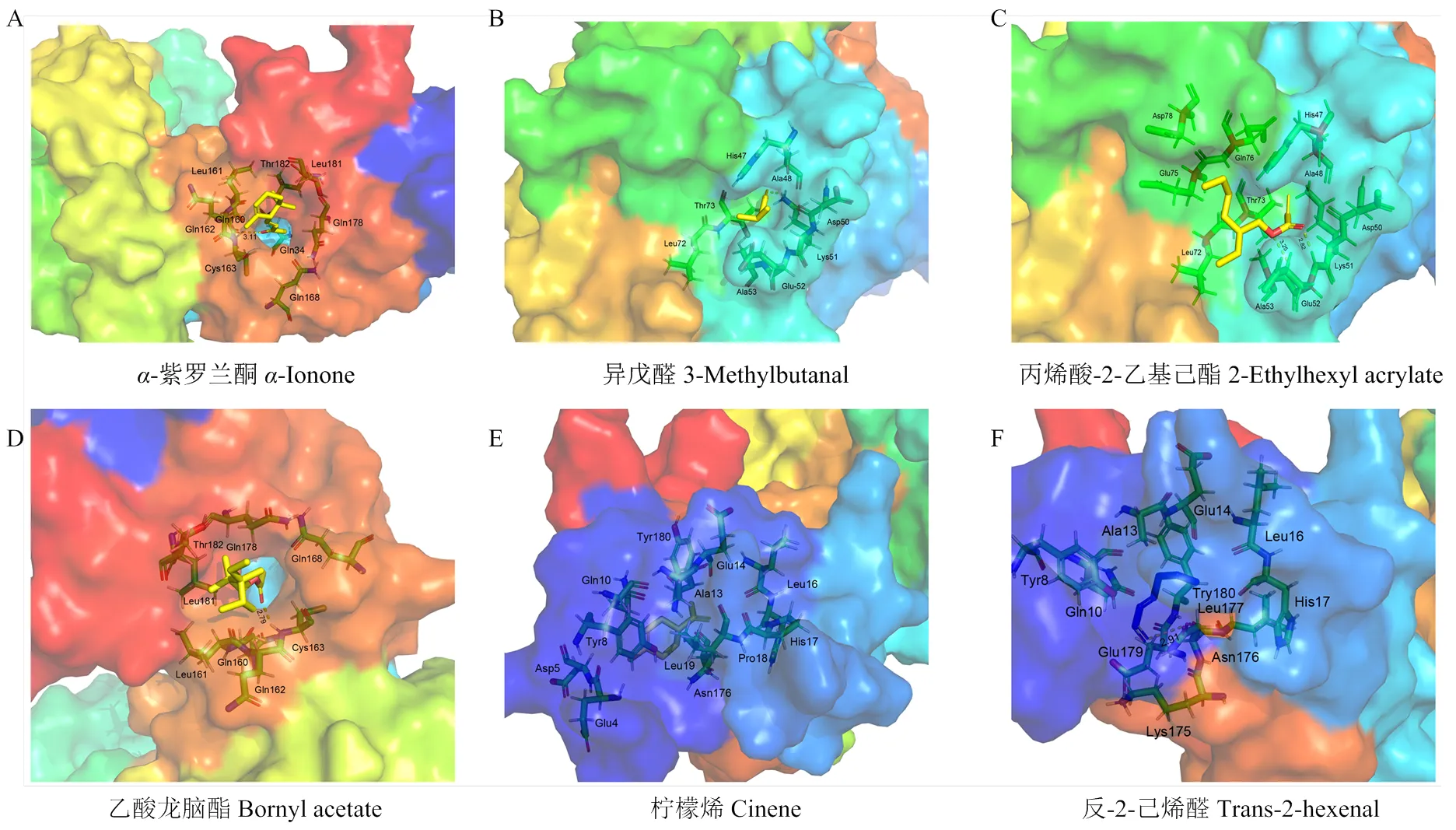
图6 AcorOBP11与配体相互作用的氨基酸残基(三维结构)
选取的37种寄主植物挥发物包含酮类、醛类、酯类、烯类、酸类、醇类和酚类化合物,其中AcorOBP11与醛类化合物结合种类最多,与酮类、酯类、烯类、酸类都可结合,但不能与醇类和酚类化合物结合,说明化合物的官能团可影响AcorOBP11与寄主植物挥发物的结合。DENG等研究表明,气味分子的碳原子数影响配体与蛋白的结合能力,在一定范围内,随着碳原子数的增加,配体与重组蛋白的结合能力逐渐减弱[11,22-23];JU等研究认为气味配体的结合能力与配体的碳原子个数之间无明显线性关系[9,24-25]。本研究通过荧光竞争结合和分子对接试验表明,配体与AcorOBP11的结合能力与碳链长度不呈线性相关。AcorOBP11既可以结合-紫罗兰酮(C13)、乙酸龙脑酯(C12)、丙烯酸-2-乙基己酯(C11),也可以结合丁醛(C4)、异戊醛(C5)、异戊酸(C5)。根据气味结合蛋白的三维结构模拟可以看出,其表面可形成形状各异的凹槽,推测化合物通过诱导契合原理结合在形状与之匹配的凹槽中,因此同一蛋白可能会结合不同大小及官能团的配体[26]。
荧光竞争结合和分子对接试验中,AcorOBP11均与-紫罗兰酮结合能力最强,主要是由于-紫罗兰酮与AcorOBP11在Cys163处形成长3.11 Å的氢键,推测氢键是一种较强的分子间作用力,可使蛋白与配体结合更加稳定。尽管其他化合物也与蛋白疏水腔形成了氢键,但-紫罗兰酮不仅与蛋白形成氢键,其疏水端进入了蛋白的结合腔中,两个甲基与蛋白表面高度契合,因此,推测蛋白结合腔与气味配体间的契合程度可以增加蛋白结合配体的稳定性。
分子对接从理论分析角度计算蛋白与配体的作用,与荧光竞争结合试验结果一致,研究结果明确了AcorOBP11蛋白的识别谱,推测AcorOBP11在铜绿丽金龟寄主植物的定位过程中发挥作用,不仅为解析铜绿丽金龟的嗅觉识别机制打下了基础,也为其生态防控提供了新思路。后续将通过RNA干扰、行为生测等进行功能的验证,利用分子动力学模拟、蛋白突变等手段探究蛋白与化合物的结合过程,深度解析AcorOBP11的功能;并进一步开展气味受体识别植物挥发物的功能研究,以期系统解析铜绿丽金龟定位寄主植物的分子机制。
4 结论
AcorOBP11能结合多种寄主植物挥发物,表明其可能在铜绿丽金龟找寻与定位寄主植物的过程中发挥重要作用。研究结果可为解析铜绿丽金龟嗅觉识别机制打下理论基础,并为害虫的无害化防控提供分子靶标。
[1] MUELLER D, PIERCE L, BENEZET H, Krischik V. Practical application of pheromone traps in food and tobacco industry. Journal of the Kansas Entomological Society, 1990, 63(4): 548-553.
[2] KLEIN M G, TUMLINSON J H, LADD T L, DOOLITTLE R E. Japanese beetle (Coleoptera: Scarabaeidae): Response to synthetic sex attractant plus phenethyl propionate: Eugenol. Journal of Chemical Ecology, 1981, 7(1): 1-7.
[3] LEAL W S, ONO M, HASEGAWA M, SAWADA M. Kairomone from dandelion,, attractant for scarab beetle. Journal of Chemical Ecology, 1994, 20(7): 1697-1704.
[4] 李晓峰, 曹雅忠, 尹姣, 张帅, 李金桥, 李克斌. 华北大黑鳃金龟植物源引诱剂配方筛选及引诱效果. 植物保护学报, 2020, 47(1): 35-45.
LI X F, CAO Y Z, YIN J, ZHANG S, LI J Q, LI K B. Prescription screening and trapping effect of plant volatile attractants to northern China scarab beetleJournal of Plant Protection, 2020, 47(1): 35-45. (in Chinese)
[5] 谢明惠, 陈浩梁, 衣建坤, 李克斌, 张杰, 苏卫华. 铜绿丽金龟对植物源挥发物的触角电位和行为反应. 植物保护, 2015, 41(1): 33-38.
XIE M H, CHEN H L, YI J K, LI K B, ZHANG J, SU W H. Electroantennographic and behavioral responses ofto plant volatiles. Plant Protection, 2015, 41(1): 33-38. (in Chinese)
[6] FrANCIS F, LOGNAY G, HAUBRUGE E. Olfactory responses to aphid and host plant volatile releases: ()-beta-farnesene an effective kairomone for the predator. Journal of Chemical Ecology, 2004, 30(4): 741-755.
[7] LEAL W S. Odorant reception in insects: Roles of receptors, binding proteins, and degrading enzymes. Annual Review of Entomology, 2013, 58: 373-391.
[8] VOGT R G, RIDDIFORD L M. Pheromone binding and inactivation by moth antennae. Nature, 1981, 293(5828): 161-163.
[9] JU Q, QU M J, WANG Y, JIANG X J, LI X, DONG S L, HAN Z J. Molecular and biochemical characterization of two odorant-binding proteins from dark black chafer,. Genome, 2012, 55(7): 537-546.
[10] 房迟琴, 张鑫鑫, 刘丹丹, 李克斌, 张帅, 曹雅忠, 樊东, 尹姣. 暗黑鳃金龟气味结合蛋白HparOBP15a基因的克隆及功能分析. 昆虫学报, 2016, 59(3): 260-268.
FANG C Q, ZHANG X X, LIU D D, LI K B, ZHANG S, CAO Y Z, FAN D, YIN J. Cloning and functional analysis of an odorant-binding protein HparOBP15a gene from(Coleoptera: Melolonthidae). Acta Entomologica Sinica, 2016, 59(3): 260-268. (in Chinese)
[11] DENG S S, YIN J, ZHONG T, CAO Y Z, LI K B. Function and immunocytochemical localization of two novel odorant-binding proteins in olfactory sensilla of the scarab beetleFaldermann (Coleoptera: Scarabaeidae). Chemical Senses, 2012, 37(2): 141-150.
[12] WANG B, GUAN L, ZHONG T, LI K B, YIN J, CAO Y Z. Potential cooperations between odorant-binding proteins of the scarab beetleFaldermann (Coleoptera: Scarabaeidae). PLoS ONE, 2013, 8(12): e84795.
[13] Yin J, Wang C Q, Fang C Q, ZHANG S, CAO Y Z, LI K B, LEAL W S. Functional characterization of odorant-binding proteins from the scarab beetlebased on semiochemical- induced expression alteration and gene silencing. Insect Biochemistry and Molecular Biology, 2019, 104: 11-19.
[14] Wang C Q, Li J Q, Li E T, NYAMWASA I, LI K B, ZHANG S, peng y, wei z j, YIN J. Molecular and functional characterization of odorant-binding protein genes inFaldermann.International Journal of Biological Macromolecules, 2019, 136: 359-367.
[15] Wei H S, Duan H X, Li K B, ZAHNG S, wei z j, YIN J. The mechanism underlying OBP heterodimer formation and the recognition of odors inFaldermann. International Journal of Biological Macromolecules, 2020, 152: 957-968.
[16] 王超群, 赵莹, 曹雅忠, 魏红爽, 李克斌, 张帅, 彭宇, 尹姣. 铜绿丽金龟气味结合蛋白AcorOBP1的表达和结合特性分析. 昆虫学报, 2017, 60(4): 363-371.
WANG C Q, ZHAO Y, CAO Y Z, WEI H S, LI K B, ZHANG S, PENG Y, YIN J. Expression and binding characterization of odorant binding protein 1 (OBP1) in(Coleoptera: Scarabaeoidea). Acta Entomologica Sinica, 2017, 60(4): 363-371. (in Chinese)
[17] LASKOWSKI R A, MACARTHUR M W, MOSS D S, THORNTON J M.: a program to check the stereochemical quality of protein structures.Journal of Applied Crystallography, 1993, 26(2): 283-291.
[18] LASKOWSKI R A, RULLMANNN J A, MACARTHUR M W, KAPTEIN R, THORNTON J M. AQUA and PROCHECK-NMR: programs for checking the quality of protein structures solved by NMR. Journal of Biomolecular NMR, 1996, 8(4): 477-486.
[19] 王桂荣, 郭予元, 吴孔明. 昆虫触角气味结合蛋白的研究进展. 昆虫学报, 2002, 45(1): 131-137.
WANG G R, GUO Y Y, WU K M. Progress in the studies of antenna odorant binding proteins of insects.Acta Entomologica Sinica, 2002, 45(1): 131-137. (in Chinese)
[20] Field L M, Pickett J A, Wadhams L J. Molecular studies in insect olfaction. Insect Molecular Biology, 2010, 9(6): 545-551.
[21] 鞠倩, 郭晓强, 李晓,蒋相国, 倪婉莉, 曲明静. 铜绿丽金龟对寄主植物挥发物的触角电生理及行为反应. 植物保护学报, 2016, 43(2): 281-287.
JU Q, GUO X Q, LI X, JIANG X G, NI W L, QU M J. EAG and behavioral responses of copper-green chaferMotschulsky (Coleoptera: Rutelidae). Journal of Plant Protection, 2016, 43(2): 281-287. (in Chinese)
[22] YIN J, FENG H L, SUN H Y, XI J H, CAO Y Z, LI K B. Functional analysis of general odorant binding protein 2 from the meadow moth,L. (Lepidoptera: Pyralidae).Plos One, 2012, 7(3): e33589.
[23] LI S, PICIMBON J F, JI S D, KAN Y C, QIAO C L, ZHOU J J, PELOSI P. Multiple functions of an odorant-binding protein in the mosquito. Biochemical and Biophysical Research Communications, 2008, 372(3): 464-468.
[24] LI G W, CHEN X L, LI B L, ZAHNG G H, LI Y P, WU J X. Binding properties of general odorant binding proteins from the oriental fruit moth,(Busck) (Lepidoptera: Tortricidae). Plos One, 2016, 11(5): e0155096.
[25] 李广伟, 陈秀琳, 尚天翠. 光肩星天牛气味结合蛋白AglaOBP12的基因克隆、表达及配体结合特征. 昆虫学报, 2017, 60(10): 1141-1154.
LI G W, CHEN X L, SHANG T C. cDNA cloning, expression and ligand binding properties of the odorant binding protein AglaOBP12 in the Asian longhorned beetle,(Coleoptera: Cerambycidae). Acta Entomologica Sinica, 2017, 60(10): 1141-1154. (in Chinese)
[26] KOSHLAND D E. Application of a theory of enzyme specificity to protein synthesis. Proceeding of the National Academy of Sciences of the United States of America, 1958, 44(2): 98-104.
Expression, purification and functional analysis of Odorant Binding Protein 11 (OBP11) in
QIN JianHui, LI JinQiao, ZHAO Xu, LI KeBin, CAO YaZhong, YIN Jiao
State Key Laboratory for Biology of Plant Diseases and Insect Pests, Institute of Plant Protection, Chinese Academy of Agricultural Sciences, Beijing 100193
【】The objective of this research is to explore the function ofodorant binding protein 11 (AcorOBP11) by studying the binding characteristics of AcorOBP11 with the host plant volatiles, and to lay the foundation for clarifying the olfactory molecular mechanism of AcorOBP11.【】Reverse transcription PCR (RT-PCR) was used to clone the full-length ORF ofby specific primers,and sequences with high similarity were downloaded for sequence comparison and analysis by BLAST. Prokaryotic expression of AcorOBP11 was conducted byprotein expression system,thewas inserted into the expression vector pET28a, and the recombinant plasmid was transferred intocompetent cells BL21 (DE3). The target protein was purified by using recombinant enterokinase to remove the His-tag and purified again by using a nickel column. The purified protein was diluted with 50 mmol·L-1Tris-HCl (pH 7.4) to a dilution of 2 μmol·L-1, and the odorant was diluted with chromatographic grade methanol to a final concentration of 1 mmol·L-1, and 1-NPN was used as a fluorescent probe to determine the binding characteristics of AcorOBP11 to 37 host plant volatiles by fluorescence competitive binding assay. Modeller was used to obtain the three-dimensional structure of AcorOBP11 by using AgamOBP48 (PDB ID: 4ij7) as a template. Autodock semi-flexible docking was used to simulate the binding of AcorOBP11 to host plant volatiles.【】The full-length ORF ofwas amplified, which is 639 bp in total, including 17 amino acids of signal peptide at the N-terminal. With eight conserved cysteine sites, AcorOBP11 belongs to the Plus-C OBP subfamily. The evolutionary tree results indicated that it had the closest evolutionarily relationship with HparOBP9.was successfully inserted into pET-28a and expressed at 0.25 mmol·L-1IPTG and induced expression at 37℃. the target protein was obtained by nickel column purification twice. The results of competitive binding experiments showed that AcorOBP11 has a good binding ability to 13 kinds of host plant volatiles, such as-ionone, 3-methylbutanal, 2-ethylhexyl acrylate, bornyl acetate, cinene, trans-2-hexenal, 2-ethylhexanal, isovaleric acid. Among them,-ionone has the best binding ability, and its competitive dissociation constant is 22.78 μmol·L-1. The molecular docking results showed that AcorOBP11 has the lowest binding free energy with-ionone, which is -24.74 kJ·mol-1, and forms a hydrogen bond at Cys163, indicating that it has the strongest binding ability with-ionone, which is consistent with the fluorescence competition binding result.【】AcorOBP11 can recognize a variety of host plant volatiles, and it is speculated that it plays an important role in locating host plants for.The results will provide new insights forecological control.
; odorant binding protein 11 (OBP11); prokaryotic expression; fluorescence competitive binding; homology modeling; molecular docking

10.3864/j.issn.0578-1752.2021.14.008
2020-10-31;
2020-12-05
国家重点研发计划(2018YFD0201000)
秦健辉,E-mail:17863800974@163.com。通信作者尹姣,E-mail:jyin@ippcaas.cn
(责任编辑 岳梅)

