谷子条纹叶突变体A36-S的细胞学特性分析及基因定位
张硕,智慧,唐婵娟,罗明昭,汤沙,贾冠清,贾彦超,刁现民
谷子条纹叶突变体的细胞学特性分析及基因定位
张硕1,2,智慧1,唐婵娟1,罗明昭1,汤沙1,贾冠清1,贾彦超1,刁现民1
1中国农业科学院作物科学研究所,北京 100081;2湖北省农业科学院粮食作物研究所,武汉 430064
【】谷子是C4模式植物,其叶色突变体是研究C4光合途径的良好材料。通过研究谷子条纹叶突变体的细胞学特性并对突变基因进行定位,为克隆突变基因、解析谷子叶绿体合成及发育机理、进一步理解C4光合调控机制奠定基础。谷子条纹叶突变体是由育种创制的中间材料A36自然变异而来。对比及其正常表型等基因系的表型特征,调查二者的株高、叶宽、叶长、穗重、千粒重、结实率等农艺性状指标;测定和的叶绿素含量、净光合速率、胞间CO2浓度、气孔导度、蒸腾速率等光合指标,分析的光合特性;观察和对照品种豫谷1号的叶片半薄横截切片和超薄切片,分析叶片解剖结构特征,分别统计叶肉细胞和维管束鞘细胞中叶绿体的数量和面积,从而分析叶绿体合成及发育情况;构建×SSR41的F2分离群体,统计群体中正常表型单株与条纹叶单株的数量,进行遗传分析;分别构建F2分离群体正常单株与条纹叶单株的DNA混池,采用集团分离分析法(BSA法)进行突变基因的定位;筛选、开发多个SSR标记及In-Del标记,扫描F2群体中条纹叶单株,进行进一步基因定位。谷子条纹叶突变体在全生育期表现出叶片不规则白色条纹的表型。农艺性状分析表明,相比其近等基因系,在株高、叶宽、穗重、千粒重、结实率等表型上均显著下降。光合指标测定表明叶片中叶绿素含量明显降低,尤其是叶绿素b含量下降更为严重,同时净光合速率也明显下降。叶片解剖结构观察发现,与对照豫谷1号相比,的Kranz结构变化并不明显,但叶绿体数量和大小都显著低于对照。观察叶绿体超微结构,发现的不同细胞间叶绿体发育状况差异较大,依据叶绿体发育情况可将叶片细胞可分为3类:Ⅰ类细胞具有正常发育的叶绿体;Ⅱ类细胞叶绿体基粒及片层结构减少;Ⅲ类细胞则叶绿体结构严重异常甚至不含有叶绿体。遗传分析表明表型受隐性单基因控制,利用F2分离群体将突变基因定位在第4染色体7.66—27.90 Mb区间内。谷子条纹叶突变体表现为农艺性状及光合能力下降,叶片细胞叶绿体的数量、大小及结构均表现出显著异常。条纹叶性状受隐性单基因控制,利用分子标记将候选基因定位于第4染色体7.66—27.90 Mb区间内。
谷子;条纹叶突变体;叶绿体;基因定位
0 引言
【研究意义】谷子((L.) P. Beauv.)属于禾本科(Granineae)黍亚科(Panicoideae)狗尾草属(),是中国北方传统粮食作物[1]。谷子为二倍体植物,基因组小(仅430 Mb左右)。近年来,随着谷子高质量基因组序列的获得[2-4],以及一些矮秆早熟品种高效遗传转化的突破[5],谷子作为功能基因组研究的模式作物而逐渐受到重视[6-7]。谷子同时也是典型的C4植物,随着环境问题及粮食安全问题的日趋严重,科学家提出将C4光合途径引入C3水稻及其他C3主要粮食和经济作物中,从而提高其产量和耐逆性的设想。谷子与传统的C4模式植物玉米和高粱相比,生育期短且基因组简单,加之其与水稻具有高度的基因组共线性,使其成为研究C4光合途径、构建水稻C4光合系统的新的模式植物。突变体是基因挖掘与功能研究的重要工具,目前,许多国内外机构都在广泛开展谷子及其野生近缘种狗尾草((L.) P. Beauv.)C4光合相关突变体的筛选及研究工作[8-10]。叶色突变体通常具有光合能力下降、光合器官结构异常等特点,是研究光合作用的重要材料。利用谷子叶色突变体开展光合生理学及细胞学观察,进而开展叶色调控基因的功能研究,对于进一步理解C4光合途径机理具有重要意义。【前人研究进展】植物叶色突变体具有多种类型,调控叶色的基因及机理也各不相同。目前,研究较为广泛且深入的叶色调控基因主要包括以下几类:(1)参与叶绿素合成及代谢的基因,比较典型的包括镁离子螯合酶D/I/H亚基的编码基因、和[11-12]、叶绿素a加氧酶基因[13]、叶绿素b还原酶基因[14]等,这些基因的变异往往导致叶片发生白化、黄化或持绿等均匀的叶色变化,有些还伴随叶绿体结构的异常;(2)参与叶绿体形成及发育的基因,这些基因中有些是直接编码某些叶绿体蛋白或结构组分的,例如叶绿体蛋白酶基因[15]、类囊体结合蛋白基因[16]等,这些基因变异会导致叶绿体结构异常,有些则参与调控叶绿体基因的转录,从而影响叶绿体的形成及发育,这类基因的突变很多都会导致白化甚至致死等严重后果,例如叶绿体核糖体L13蛋白基因[17]、质体RNA聚合酶相关蛋白基因[18]、参与叶绿体RNA编辑及剪切的[19]等;(3)一些参与钾、铁等金属离子吸收及运输的基因,这些基因突变后,通常会影响叶绿体的正常发育,导致叶色变异,例如铁螯合物运输基因[20]、钾离子外排逆向转运蛋白基因[21]等;(4)一些参与核酸合成及代谢的基因,在其功能丧失后,机体为保证核基因及线粒体基因的正常复制,会被迫牺牲叶绿体基因的复制与转录,从而导致叶绿体合成发育受到抑制,发生条纹或白化等叶色异常,例如尿嘧啶核苷酸激酶基因[22]、核糖核苷酸还原酶小亚基基因/[23]等。【本研究切入点】目前,已经在谷子上克隆了若干调控叶色的基因,包括镁离子螯合酶D亚基编码基因[24]、核糖核苷酸还原酶大亚基基因[25]、脱氧胞苷脱氨酶基因[26]、金属肽链内切酶[27]、类PsbP蛋白编码基因[28]等。然而,对于C4模式植物谷子而言,目前克隆的叶色调控基因尚少,C4植物叶绿体合成、发育及光合作用相关基因的研究基础仍然缺乏。【拟解决的关键问题】本研究中谷子突变体是来源于育种创制材料A36的一个自然突变体,叶片具有不规则白色条纹。通过对的光合参数、叶片细胞叶绿体合成发育情况进行观察分析,以探究叶色变异造成的光合生理和细胞学影响,并利用BSA图位克隆技术对条纹叶致变基因进行定位,以期为谷子光合作用相关基因的鉴定和功能分析提供一定的参考。
1 材料与方法
1.1 试验材料
谷子突变体由育种创制的中间材料A36(1066A×创62后代稳定系)自然变异而来,其叶片全生育期表现出不规则白条纹,经多代自交后表型稳定。与A36系回交多代后,分离得到的正常表型等基因系,在农艺性状及光合参数测定中作为对照。豫谷1号为华北夏谷区广泛栽培的品种,也是最早完成基因组测序的谷子品种,在以往各研究中广泛作为参照品种使用,在本研究中用作叶片解剖结构和叶绿体超微结构观察的对照。
1.2 表型及农艺性状调查
突变体及其正常表型的等基因系于2017年夏季种植于中国农业科学院作物科学研究所网室(北京,116°20′E,39°56′N)进行表型观察,并于抽穗后调查株高、叶长、叶宽、穗长、穗粗等农艺性状,待成熟且籽粒自然风干后,测定穗重、千粒重、结实率等产量相关性状。每组性状测定时取5个重复,结果采用检验进行统计分析。
1.3 叶绿素含量测定
取和拔节期相同叶位的新鲜叶片各0.2 g,剪碎后于95%乙醇中避光震荡48 h直至叶片完全变白,充分提取叶绿素。采用Lichtenthaler[29]的方法,分别测定665和649 nm波长下的OD值,并计算叶绿素a与叶绿素b的含量。计算公式为:Chl a=13.95OD665—6.88OD649,Chl b=24.96OD649—7.32OD665。设定3个重复,结果采用检验进行统计分析。
1.4 光合参数测定
选择晴朗天气,于上午9:00—11:00,利用Li-6400便携式光合测定仪分别测定抽穗期与旗叶的净光合速率、胞间CO2浓度、气孔导度及蒸腾速率。设定5组重复,结果采用检验进行统计分析。
1.5 叶片横截切片制作及叶绿体数量、面积的统计
取苗期表型特征明显的叶片及豫谷1号叶片,剪成2 mm×1 mm的长方形碎片,于2.5%的戊二醛固定液中抽真空,并过夜固定。固定好的材料经脱水透明后,包埋于树脂中,并使用切片机进行叶片横截切片,切片厚度10 μm。切片用0.2%甲苯胺蓝染色后,于显微镜下观察叶片解剖结构。统计视野中所有叶肉细胞及维管束鞘细胞中叶绿体的数量,分别统计了117个豫谷1号叶片细胞和318个叶片细胞,并对叶绿体数量进行统计分析。对横截切片进行拍照后,利用Image-Pro plus 6.0软件分别统计照片中所有维管束鞘细胞叶绿体和叶肉细胞叶绿体的面积,计算平均值,并利用检验进行统计分析。
1.6 透射电镜观察
取苗期表型特征明显的叶片及豫谷1号叶片,小心剪成2 mm×1 mm的长方形碎片,于2.5%戊二醛固定液中抽真空固定,并包埋于树脂中。之后用超薄切片机切片并用醋酸双氧铀(uranyl acetate,UA)染色。使用JEM-1230透射电镜观察,并照相。
1.7 遗传分析及基因定位
以作为母本,以谷子品种SSR41作为父本,进行杂交。观察F1表型并自交产生F2。对F2分离群体中正常表型及条纹叶植株数量进行统计,并取F2中所有条纹叶后代共881株的叶片用于基因定位。
基因定位首先采用集团分离分析法(BSA法),选取条纹叶表型的F2代20株,采用CTAB法提取DNA,测定浓度后等量混合构建混池。在谷子基因组9条染色体上筛选与SSR41间存在多态性的SSR标记共60个,以、SSR41、F1、混池DNA为模板,利用这些标记分别进行扩增,根据带型判断标记与目标基因的连锁情况。找到连锁标记后,在连锁标记上下游选取多态性标记,扫描所有的F2条纹叶单株,并统计连锁交换事件,以确定目标基因所在区间。
2 结果
2.1 A36-S条纹叶突变体的表型特征分析
谷子突变体是由A36系自然突变而来的条纹叶突变体,其叶片全生育期表现出白色不规则条纹(图1)。是与1066A恢复系经多代回交后获得的等基因系,其叶片表型正常,理论上具有与相同的遗传背景。通过调查比较与的农艺性状,发现株高和叶宽显著下降(分别下降15.08%和25.40%),而植株茎节数和叶长与没有显著差异。在产量性状上,的穗长、主穗重、穗粒重、千粒重、结实率均显著低于(表1),其中结实率下降幅度最大,达到38.34%,说明的育性受到较严重影响,产量下降主要原因是育性降低。
2.2 A36-S突变体的光合特性分析
为分析条纹叶表型对光合特性的影响,对拔节期与叶片叶绿素含量进行测定。结果显示,与相比,的叶绿素a含量显著下降(=0.031,下降8.99%),而叶绿素b含量极显著下降(=7.170×10-4,下降52.33%)(图2-A)。的叶绿素a/b值为1.357,而的叶绿素a/b值为2.591。上述结果表明,的叶绿素含量受到影响,尤其是叶绿素b受到影响更加严重,叶绿素a/b值升高。

A:抽穗期植株;B:抽穗期叶片;C:抽穗期叶片细节;D:成熟后穗部
对抽穗期与的光合参数进行测定,发现的净光合速率相比显著降低了29.09%(图2-B);虽然的气孔导度平均值比有所下降(9.89%),胞间CO2浓度平均值有所上升(19.02%),但在统计学上,与的这两项指标无显著差异(图2-C和图2-D);与的蒸腾速率没有显著差异(图2-E)。这些结果说明叶片光合能力明显降低。
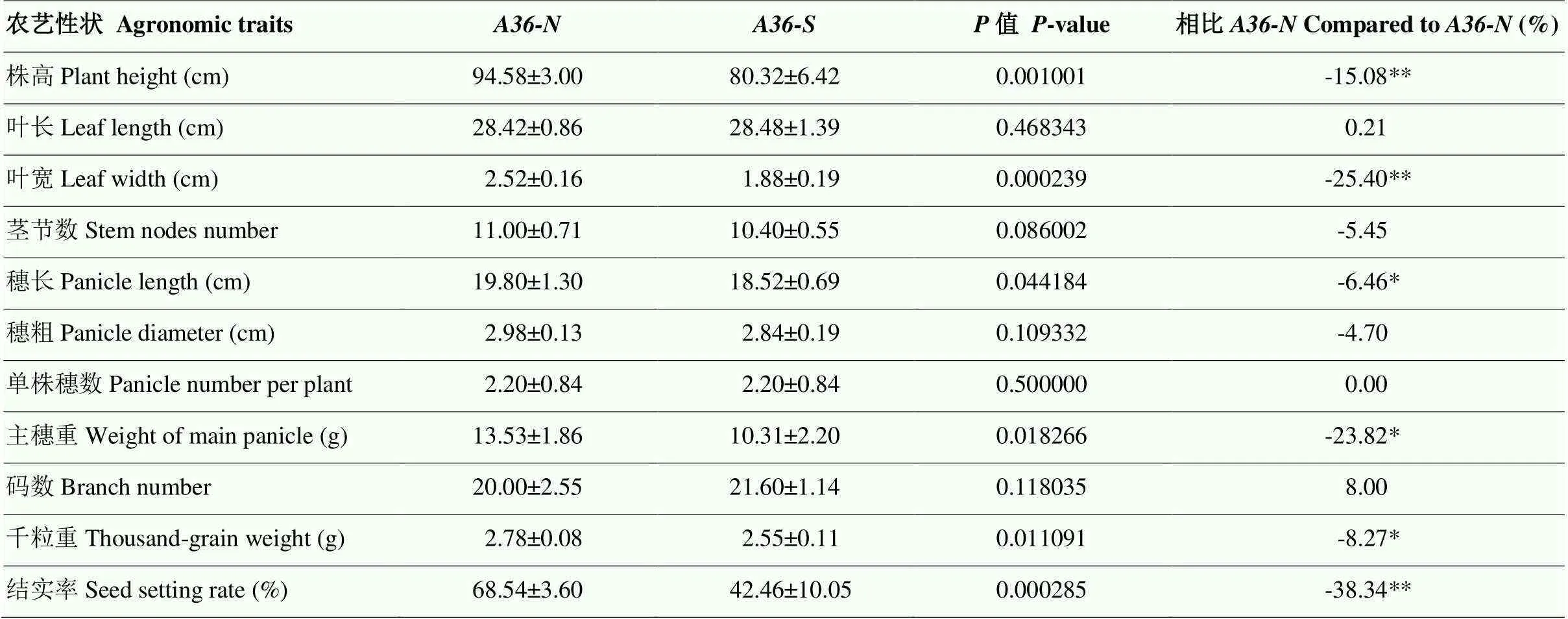
表1 A36-S与A36-N的主要农艺性状对比
*表示在<0.05水平差异显著;**表示在<0.01水平差异极显著 *: significant difference at0.05 level; **: significant difference at0.01 level

图2 A36-S与A36-N的叶绿素含量及光合参数
2.3 A36-S突变体的叶片解剖结构分析
为了进一步分析导致突变体叶片条纹表型的细胞学原因,对及具有正常叶片的谷子品种豫谷1号的苗期叶片进行横截半薄切片观察。谷子属于NADP-ME亚型C4植物,叶片具有典型的Kranz结构,即维管束鞘细胞形态饱满,内含许多体积大且不含基粒的叶绿体(图3-A,蓝色箭头指示豫谷1号维管束鞘细胞叶绿体),一层叶肉细胞整齐地围绕维管束鞘细胞排列,内含许多具丰富基粒的叶绿体(图3-A,红色箭头指示豫谷1号叶肉细胞叶绿体)。相比而言,的叶片Kranz结构变化并不明显,但叶绿体的发育情况明显异常。虽然部分细胞含有正常数量的叶绿体(图3-B,红色和蓝色箭头分别指示正常叶肉细胞及维管束鞘细胞叶绿体),然而也有许多维管束鞘细胞(图3-B,蓝色星号)及叶肉细胞(图3-B,红色星号)不含有叶绿体,这可能是叶片呈现白色条纹的主要原因。
对切片不同视野中所有叶肉细胞及维管束鞘细胞中叶绿体数量进行统计(图4-A)。豫谷1号叶片细胞中叶绿体数量分布较离散,从1个到7个不等,叶绿体数中值为4个;相比而言,叶片细胞叶绿体数量从0个到8个不等,中值为2个,但数据在0 个处密集分布,说明部分细胞中叶绿体数量正常,但大量细胞中不含有叶绿体。通过对数据进行了统计学分析,结果表明,细胞中叶绿体数量显著低于豫谷1号(=4.98×10-17)。进而,对豫谷1号和的叶绿体大小进行了比较。由于维管束鞘细胞与叶肉细胞的叶绿体体积存在明显差异,因此对这两种类型细胞的叶绿体分别进行统计,结果表明,的维管束鞘细胞叶绿体和叶肉细胞叶绿体面积均显著小于豫谷1号(图4-B)。上述结果说明在叶绿体形成及发育方面均受到抑制。

A:豫谷1号叶片横截切片(标尺50 μm);B:A36-S叶片横截切片(标尺50 μm);红色箭头指示正常叶肉细胞叶绿体;蓝色箭头指示正常维管束鞘细胞叶绿体;红色星号指示不含叶绿体的叶肉细胞;蓝色星号指示不含叶绿体的维管束鞘细胞

图4 豫谷1号与A36-S叶绿体数量和面积的统计
2.4 A36-S突变体的叶绿体超微结构分析
对及豫谷1号苗期叶片叶绿体超微结构进行观察,由于谷子叶片维管束鞘细胞与叶肉细胞的叶绿体存在结构及功能差异,因此对二者分别进行观察分析(图5)。豫谷1号的维管束鞘细胞叶绿体具有排列整齐的基质片层,且包含许多淀粉颗粒(图5-A);叶肉细胞叶绿体则含有大量的基粒,淀粉粒含量也十分丰富(图5-B)。的情况则较为复杂,不同细胞间叶绿体发育情况存在较大差异。根据细胞中叶绿体的发育情况将其分为3类:
Ⅰ类细胞:包含发育正常的叶绿体。的有些细胞中,叶绿体数量相对正常,叶绿体的超微结构也与对照豫谷1号无明显差异(图5-C为维管束鞘细胞及内部的叶绿体,图5-D为叶肉细胞及内部的叶绿体),推断这些细胞位于条纹叶的绿色部分。
Ⅱ类细胞:叶绿体结构基本完整,但基粒及片层结构减少。表现为维管束鞘细胞叶绿体片层结构排列松散(图5-E),叶肉细胞叶绿体基粒数量及囊状堆叠层数明显减少(图5-F),并且叶绿体中仅含有少量或不含有淀粉粒(图5-E和图5-F),说明光合作用积累的糖类减少,光合能力下降。
Ⅲ类细胞:不含叶绿体或含有严重异常的叶绿体。表现为有些细胞中完全没有叶绿体形成,有些细胞虽含有叶绿体(图5-G),但叶绿体不含有基质片层及基粒结构,仅含有一些泡状结构(图5-H,白色箭头指示泡状结构)。
综上所述,尽管部分细胞中叶绿体结构正常,A36-S的叶绿体形成及发育依然受到较严重影响。
2.5 A36-S突变基因的定位
为定位突变基因,以为母本构建了F2代分离群体。选择的杂交父本SSR41具有正常叶片表型,其株高、生育期等性状与相近似,且育性良好。前期研究表明,SSR41与豫谷1号、辽谷1号、大青秸等谷子品种都具有较高的遗传多态性,因此,长期以来被作为构建作图群体的主要亲本材料,广泛进行杂交组合的组配,并成功定位了若干基因[25-27, 30]。选择SSR41作为作图群体父本。对F2代分离群体进行遗传统计发现,群体中正常后代455株,条纹叶后代130株,分离比例符合3﹕1(表2),说明的条纹叶性状由隐性单基因控制。

A:豫谷1号维管束鞘细胞叶绿体;B:豫谷1号叶肉细胞叶绿体;C:A36-S的Ⅰ类维管束鞘细胞;D:A36-S的Ⅰ类叶肉细胞;E:A36-SⅡ类维管束鞘细胞叶绿体;F:A36-SⅡ类叶肉细胞叶绿体;G:A36-S的Ⅲ类细胞;H:A36-S Ⅲ类细胞中的叶绿体,白色箭头指示泡状结构;SG:淀粉颗粒;SL:基质片层;GR:基粒;CLP:叶绿体
采用集团分离分析法(BSA法)进行突变基因的初定位。选取F2分离群体中条纹叶单株20株,提取DNA构建混池。在谷子基因组9条染色体上均匀选取与SSR41间多态性良好的SSR标记60个,用于基因初定位。结果显示,使用第4染色体标记SiCAAS4019(9 999 146 bp)、SiCAAS4054(15 511 536 bp)、SiCAAS4033(23 843 301 bp)扩增混池DNA得到条带与母本带型一致(图6),说明突变基因与这几个标记紧密连锁。

表2 A36-S与SSR41杂交F2代分离比统计

♀:A36-S突变体;♂:SSR41;F1:A36-S×SSR41;P:F2群体条纹叶单株混池DNA
利用上述3个标记检测40株F2条纹叶单株,发现所有单株均与母本带型一致。因此,为确定定位区间边界,在这三个标记的上下游再次选取和开发了多个SRR及In-Del标记,用来检测上述40株F2条纹叶单株(表3),将突变基因定位于InDel 14与SiCAAS4034之间26.2 Mb区间内。
为了进一步定位突变基因,一方面扩大群体,将F2条纹叶单株增加至881株,另一方面根据现有基因组In-Del数据信息,在定位区间内开发In4-3、In4-17、In4-20、In4-21、In4-29、In4-32、In4-33共7对In-Del标记(标记物理位置及引物序列见表4)。对881个F2条纹叶单株进行扫描,统计各标记处杂合带型的数量(表4)。将突变基因定位于In4-20(7 661 828 bp)与In4-32(27 896 330 bp)之间20.13 Mb区间内。
3 讨论
3.1 对A36-S叶绿素a/b值升高原因的分析
本研究中,与对照相比,的叶绿素含量显著下降,且叶绿素b下降的幅度更大,叶绿素a/b值明显升高。发现谷子的几个叶色变异材料[24]、[27]、黄金苗[32]等也存在类似的情况。有报道指出叶绿素a/b值与总叶绿素含量及光合速率呈负相关,即总叶绿素含量越高,叶绿素b的占比越大,叶绿素a/b值越低,叶片光合速率越高[33]。这可能与叶绿素a和叶绿素b在光合作用原初反应中的功能差异有关。叶绿素a与叶绿素b都负责吸收与传递光能,而少数激发态的叶绿素a还具有将光能转化为电能及化学能的作用。因此,叶绿素b相对含量升高有利于在光照不足状态下增加光能的吸收,叶绿素a相对含量升高有利于在强光状态下提高光合速率[34]。由此推测,等叶色突变体中叶绿素a/b值的升高,可能是植物应对叶片光合能力下降的一种适应性机制。
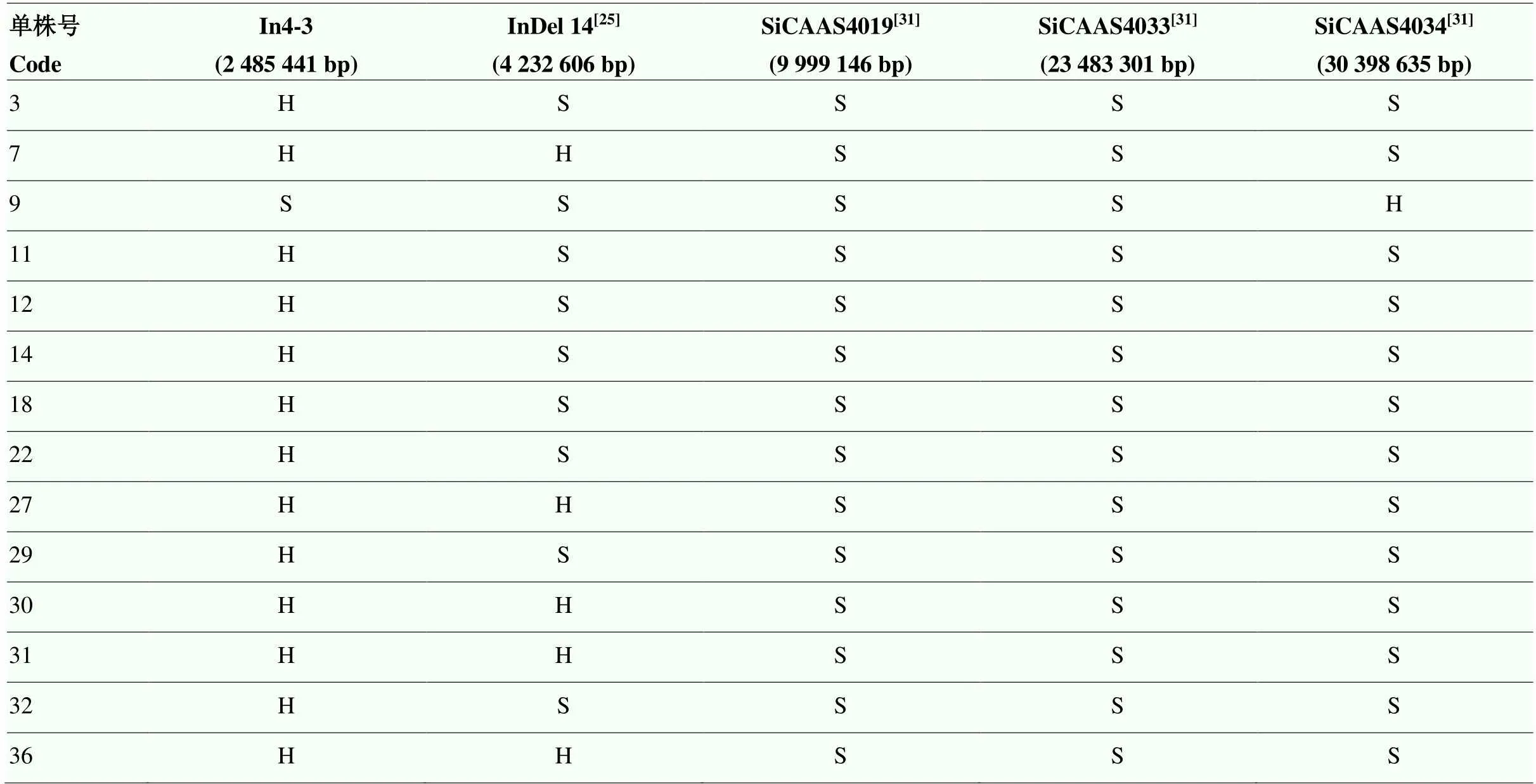
表3 40株F2代条纹叶单株标记检测结果
S:母本带型;H:杂合子带型 S: The band of female parent; H: The band of heterozygous individual
加粗字体:突变基因定位区间 Bold fonts: The locating region of the mutant gene
另外,还有研究指出,由于分子结构不同,叶绿素a与叶绿素b对于不同胁迫的敏感程度不同,例如叶绿素a对活性氧的增加更加敏感[35],叶绿素b对于酸胁迫更加敏感[36]。中的基因突变有可能通过损害某一代谢通路对植株造成某种胁迫,使得叶绿素b的含量受到更严重的影响,这一推测还需要在完成基因克隆后进一步证实。
3.2 A36-S突变基因可能参与调控叶绿体合成发育的上游途径
谷子突变体叶片呈现不规则白色条纹(图1-B和图1-C)。对叶片解剖结构及叶绿体超微结构观察发现,叶绿体的大小和数量与豫谷1号相比均显著下降(图4),且部分叶绿体结构异常,有些细胞中甚至不含有叶绿体(图5),说明的某些细胞中,叶绿体在早期合成及发育时即受到抑制,这可能是叶片呈现白色条纹的原因。目前谷子报道的相似表型突变体有[25]、[26],二者均表现为不规则白色条纹叶,并且叶片中同样包含部分不含有叶绿体的叶肉细胞及维管束鞘细胞,叶绿体发育也受到了阻碍,二者的突变基因分别为核糖核苷酸还原酶大亚基编码基因和脱氧胞苷脱氨酶基因[25-26],但这两个基因均不在本研究的初定位区间内,可排除为或等位突变体的可能性。
水稻等其他禾本科植物中也报道了许多与表型类似的白条纹叶突变体,例如水稻[37]、[38]、[39]、[40]、[41]等,这些突变体均表现出叶片不规则白色条纹、叶绿体发育不良、部分细胞叶绿体缺失等表型,它们的突变基因分别参与叶绿体核糖体合成、叶绿体RNA剪切、核酸代谢等通路,通过控制叶绿体关键基因的合成、转录、翻译上游途径来调控叶绿体的合成及发育。结合谷子及的表型和突变基因功能,推测A36-S的突变基因也可能参与核酸代谢、叶绿体基因转录及翻译调控等叶绿体合成及发育的上游途径。
另外,在叶片横截切片及叶绿体超微结构观察中使用的对照品种豫谷1号与突变体之间存在一定的遗传背景差异,有可能会影响叶绿体大小和数量。然而相比豫谷1号,表现出大量细胞叶绿体缺失,叶绿体结构明显异常,C4植物固有的叶片解剖结构不完整等严重缺陷,遗传背景的差异很难导致这样的缺陷。因此,认为在叶片解剖结构及叶绿体发育上的异常的确是由基因突变导致的。在后续研究中,为使结果更加准确严谨,将以近等基因系为对照,进一步研究在叶片解剖结构、叶绿体大小数量及结构上的变化,以确认突变基因功能。
3.3 A36-S突变基因的精细定位
本研究将突变基因定位在第4染色体20.13 Mb区间内。没能完成精细定位的主要原因是作图群体中交换株过少,尽管群体数量已经足够多,但在定位区间内染色体重组的频率非常低。是一个来源于育种中间材料的自然突变体,推测在定位区间内存在与作图亲本SSR41差别较大的复杂的染色体结构差异,干扰这一段染色体与SSR41对应染色体片段的正常配对,从而导致重组交换率偏低。而这种染色体结构变异可能是由于自然突变导致的,也可能本来就存在与的野生型中。为解决这一问题,本研究计划一方面使用与遗传关系更近的材料重新构建作图群体,进行基因定位,但这很有可能导致在定位区间内找不到足够的多态性标记;另一方面对及其近等基因系进行初定位区间的高通量测序,通过比对二者在区间内的序列差异来寻找突变基因。
3.4 A36-S突变基因功能与C4光合途径的关系
决定C4植物高效光合作用的关键之一在于其具有独特的Kranz解剖结构及CO2浓缩机制。目前已报道一些可能调控Kranz结构及维管束鞘细胞叶绿体发育的蛋白,例如玉米GLK转录因子可诱导水稻产生pro-Kranz结构并促使维管束鞘细胞中叶绿体的发育[42],SCR/SHR转录因子调控维管束鞘细胞发育和叶脉排列模式[43]。通过叶片横截切片观察,发现的维管束鞘细胞与叶肉细胞中都发生了叶绿体的数量减少、面积下降以及结构异常,说明突变基因对叶绿体的影响不存在细胞类型特异性。而的Kranz结构不完整是由于维管束鞘细胞及叶肉细胞中叶绿体的丧失导致的,Kranz结构的细胞数量、细胞排列方式等没有明显异常。因此我们推测的突变基因可能并不直接参与C4解剖结构的构建,而是通过控制叶绿体合成及发育间接参与其中。而在C4生化途径上发生了哪些变化,C4通路上关键基因的表达是否受到影响尚未证实。谷子是C4模式植物,其叶绿体发育异常突变体对于C4光合途径仍然具有较高的研究价值。下一步应在初定位的基础上进行基因精细定位并克隆候选基因,进一步研究基因生物功能,以及其与C4途径的相互关系,对于加深人们对叶绿体合成发育调控机理以及C4光合作用的理解具有一定意义。
4 结论
与正常表型的等基因系相比,谷子条纹叶突变体在株高、叶宽、穗重、千粒重、结实率等农艺性状上显著下降,叶片叶绿素含量及净光合速率显著降低。叶片细胞中叶绿体数量减少且体积变小,不同细胞间叶绿体发育状况差异较大:部分细胞具有正常发育的叶绿体,部分细胞叶绿体基粒及片层结构减少,有些细胞则叶绿体结构严重异常甚至不含有叶绿体。的突变表型受隐性单基因控制,利用分子标记将该基因定位于第4染色体7.66—27.90 Mb区间内。
[1] 贾冠清, 刁现民. 谷子((L.) P. Beauv.)作为功能基因组研究模式植物的发展现状及趋势. 生命科学, 2017, 29(3): 292-301.
JIA G Q, DIAO X M. Current status and perspectives of researches on foxtail millet ((L.) P. Beauv.): A potential model of plant functional genomics studies. Chinese Bulletin of Life Science, 2017, 29(3): 292-301. (in Chinese)
[2] Bennetzen J L, Schmutz J, Wang H, Percifield R, Hawkins J, Pontaroli A C, Estep M, Feng L, Vaughn J N, Grimwood J, Jenkins J, Barry K, Lindquist E, Hellsten U, Deshpande S, Wang X W, Wu X M, Mitros T, Triplett J, Yang X H, Ye C Y, Mauro-Herrera M, Wang L, Li P H, Sharma M, Sharma R, Ronald P C, Panaud O, Kellogg E A, Brutnell T P, Doust A N, Tuskan G A, Rokhsar D, Devos K M. Reference genome sequence of the model plant. Nature Biotechnology, 2012, 30: 556-561.
[3] Zhang G Y, Liu X, Quan Z W, Cheng S F, Xu X, Pan S K, Xie M, Zeng P, Yue Z, Wang W L. Genome sequence of foxtail millet () provides insights into grass evolution and biofuel potential. Nature Biotechnology, 2012, 30: 549-554.
[4] Jia G Q, Huang X H, Zhi H, Zhao Y, Zhao Q, Li W J, Chai Y, Yang L F, Liu K Y, Lu H Y, Zhu C R, Lu Y Q, Zhou C C, Fan D L, Weng Q J, Guo Y L, Huang T, Zhang L, Lu T T, Feng Q, Hao H F, Liu H K, Lu P, Zhang N, Li Y H, Guo E H, Wang S J, Wang S Y, Liu J R, Zhang W F, Chen G Q, Zhang B J, Li W, Wang Y F, Li H Q, Zhao B H, LI J Y, DIAO X M, HAN B. A haplotype map of genomic variations and genome-wide association studies of agronomic traits in foxtail millet (). Nature Genetics, 2013, 45: 957-961.
[5] Yang Z Y, Zhang H S, Li X K, Shen H M, Gao J H, Hou S Y, Zhang B, Mayes S, Bennett M, Ma J X, Wu C Y, Sui Y, Han Y H, Wang X C. A mini foxtail millet with an-like life cycle as a C4system. Nature Plants, 2020, 6(9): 1167-1178.
[6] Li P H, Brutnell T P.and, model genetic systems for the. Journal of Experiment Botany, 2011, 62: 3031-3037.
[7] Diao X M, Schnable J, Bennetzen J L, Li J Y. Initiation ofas a model plant. Frontiers of Agricultural Science and Engineering, 2014, 1: 16-20.
[8] Caemmerer S von, Quick W P, Furbank R T. The development of C4rice: current progress and future challenges. Science, 2012, 336: 1671-1672.
[9] Luo M Z, Zhang S, Tang C J, Jia G Q, Tang S, Zhi H, Diao X M. Screening of mutants related to the C4photosynthetic Kranz structure in foxtail millet. Frontiers in Plant Science, 2018, 9: 1650.
[10] 罗明昭, 唐婵娟, 张硕, 智慧, 汤沙, 贾冠清, 刁现民. 利用低CO2浓度培养箱筛选谷子() C4光合作用相关突变体. 植物遗传资源学报, 2018, 19(3): 554-560.
Luo M Z, Tang C J, Zhang S, Zhi H, Tang S, Jia G Q, Diao X M. Screening of C4photosynthesis-related mutants in foxtail millet () by employmeny of low CO2concentration incubator. Journal of Plant Genetic Resources, 2018, 19(3): 554-560. (in Chinese)
[11] Zhang H T, Li J J, Yoo J H, Yoo H C, Cho S H, Koh H J, Seo H S, Paek N C. Riceandencode ChlD and ChlI subunits of Mg-chelatase, a key enzyme for chlorophyll synthesis and chloroplast development. Plant Molecular Biology, 2006, 62(3): 325-337.
[12] Zhou S X, Sawicki A, Willows R D, Luo M Z. C-terminal residues of oryza sativa GUN4 are required for the activation of the ChlH subunit of magnesium chelatase in chlorophyll synthesis. FEBS Letters, 2012, 586: 205-210.
[13] Yang Y L, Xu. J, Huang L C, Leng Y J, Dai L P, Rao Y C, Chen L, Tu Z J, Hu J,Ren D Y, Zhang G H, Zhu L, Guo L B, Qian Q, Zeng D L., encoding chlorophyllide a oxygenase 1, impacts leaf senescence and indirectly affects grain yield and quality in rice. Journal of Experimental Botany, 2016, 5: 1297-1310.
[14] Kusaba M, Ito H, Morita R, Iida S, Sato Y, Fujimoto M, Kawasaki S, Tanaka R, Hirochika H, Nishimura M, Tanaka A. Rice NON-YELLOW COLORING1 is involved in light-harvesting complex II and grana degradation during leaf senescence. The Plant Cell, 2017, 19: 1362-1375.
[15] Dong H, Fei G L, Wu C Y, Wu F Q, Sun Y Y, Chen M J, Ren Y L, Zhou K N, Cheng Z Y, Wang J L, Jiang L, Zhang X, Guo X P, Lei C L, Su N, Wang H Y, Wan J M. A ricemutant reveals new insights into the role and assembly of plastid caseinolytic protease in higher plants. Plant Physiology, 2013, 162: 1867-1880.
[16] Li J J, Pandeya D, Nath K, Zulfugarov I S, Yoo S C, Zhang H T, Yoo J H, Cho S H, Koh H J, Kim D S, Seo H S, Kang B C, Lee Z H, Paek N C. ZEBRA-NECROSIS, a thylakoid-bound protein, is critical for the photoprotection of developing chloroplasts during early leaf development. The Plant Journal, 2010, 62: 713-725.
[17] Song J, Wei X J, Shao G N, Sheng Z H, Chen D B, Liu C L, Jiao G A, Xie L H, Tang S Q, Hu P S. The rice nuclear geneencoding a chloroplast ribosome L13 protein is needed for chloroplast development in rice grown under low temperature conditions. Plant Molecular Biology, 2014, 84: 301-314.
[18] Lv Y S, Shao G N, Qiu J H, Jiao G A, Sheng Z H, Xie L H, Wu Y W, Tang S Q, Wei X J, Hu P S., encoding a PEP-associated protein, is required for chloroplast biogenesis under heat stress in rice. Journal of Experiment Botany, 2017, 68: 5147-5160.
[19] Tang J P, Zhang W W, Wen K, Chen G M, Sun J, Tian Y L, Tang W J, Yu J, An H Z, Wu T T, Kong F, Terzaghi W, Wang C M, Wan J M. OsPPR6, a pentatricopeptide repeat protein involved in editing and splicing chloroplast RNA, is required for chloroplast biogenesis in rice. Plant Molecular Biology, 2017, 95: 345-357.
[20] Lee S, Chiecko C J, Kim S A, Walker E L, Lee Y, Guerinot M L, Ann G G. Disruption ofleads to iron inefficiency in rice plants. Plant Physiology, 2009, 150: 786-800.
[21] Sheng P K, Tan J J, Jin M N, Wu F Q, Zhou K N, Ma W W, Heng Y Q, Wang J L, Guo X P, Zhang X, Cheng Z J, Liu L L, Wang C M, Liu X M, Wan J M., encoding a putative potassium efflux antiporter, affects chloroplast development and drought tolerance in rice. Plant Cell Reports, 2014, 33: 1581-1594.
[22] Zhu X Y, Guo S, Wang Z W, Xing Y D, Zhang T Q, Shen W Q, Sang X C, Ling Y H, He G H. Map-based cloning and functional analysis of, which controls leaf colour in rice (). BMC Plant Biology, 2016, 16: 134-148.
[23] Qin R, Zeng D D, Liang R, Yang C C, Akhter D, Alamin M, Jin X L, Shi C H. Rice gene, encoding the small subunit of ribonucleotide reductase, is required for chlorophyll synthesis and plant growth development. Gene, 2017, 627: 351-362.
[24] Li W, Tang S, Zhang S, Shan J G, Tang C J, Chen Q N, Jia G Q, Han Y H, Zhi H, Diao X M. Gene mapping and functional analysis of the novel leaf color gene(L.) P. Beauv.]. Physiologia Plantarum, 2015, 157: 24-37.
[25] Tang C J, Tang S, Zhang S, Luo M Z, Jia G Q, Zhi H, Diao X M., encoding a large subunit of ribonucleotide reductase, is crucial for plant growth, chloroplast biogenesis, and cell cycle progression in. Journal of Experimental Botany, 2019, 70(4): 1167-1182.
[26] Zhang S, Tang S, Tang C J, Luo M Z, Jia G Q, Zhi H, Diao X M.is required for cell cycle leaf organ development, chloroplast biogenesis and influences C4photosynthesis in(L.) P. Beauv. Frontiers in Plant Science, 2018, 9: 1308.
[27] Zhang S, Zhi H, Li W, Shan J G, Tang C J, Jia G Q, Tang S, Diao X M.is involved in the regulation of leaf senescence and photosystem II efficiency in(L.) P. Beauv. Frontiers in Plant Science, 2018, 9: 1103.
[28] 王秋兰, 王智兰, 韩芳, 杜晓芬, 连世超, 韩康妮, 周雪, 李慧娟, 张林义, 王军, 郭二虎. 谷子条纹叶突变体的鉴定及候选基因分析. 华北农学报, 2020, 35(1): 214-221.
Wang Q L, Wang Z L, Han F, Du X F, Lian S C, Han K N, Zhou X, Li H J, Zhang L Y, WANG J, Guo E H. Identified and candidate gene analysis of a white stripe leaf mutant
[29] Lichtenthaler H K. Chlorophylls and carotenoids: pigments of photosynthetic biomembranes. Methods in Enzymology, 1987, 148: 350-382.
[30] XIANG J S, TANG S, ZHI H, JIA G Q, WANG H J, DIAO X M.encoding a novel WRKY transcription factor, regulates panicle development, stem elongation, and seed size in foxtail millet [(L.) P. Beauv.]. Plos One, 2017, 12 (6): e0178730.
[31] Zhang S, Tang C J, Zhao Q, Li J, Yang L F, Qie L F, Fan X K, Li L, Zhang N, Zhao M C, Liu X T, Chai Y, Zhang X, Wang H L, Li Y T, Li W, Zhi H, Jia G Q, Diao X M. Development of highly polymorphic simple sequence repeat markers using genome- wide microsatellite variant analysis in foxtail millet [(L.) P. Beauv.]. BMC Genomics, 2014, 15: 78.
[32] 李传宗, 智慧, 汤沙, 贾冠清, 唐婵娟, 贾彦超, 刁现民. 黄金苗谷子苗期黄色的生理基础和黄苗基因初定位. 植物遗传资源学报, 2020, 21(5): 1068-1077.
Li C Z, Zhi H, Tang S, Jia G Q, Tang C J, Jia Y C, Diao X M. Physiological basis and and linkage analysis of elite foxtail millet variety Huangjinmiao that shows yellow seedling leaves. Journal of Plant Genetic Resources, 2020, 21(5): 1068-1077. (in Chinese)
[33] 周黄磊, 黄升谋. 库源关系对水稻叶绿素含量及叶绿素a/b值的影响. 绿色科技, 2017, 24: 147-149.
Zhou H L, Huang S M. Effects of sink source relationship on chlorophyll content and photosynthetic characteristics of rice. Journal of Green Science and Technology, 2017, 24: 147-149. (in Chinese)
[34] 战吉成, 王利军, 黄卫东. 弱光环境下葡萄叶片的生长及其在强光下的光合特性. 中国农业大学学报, 2002, 7(3): 75-78.
Zhan J C, Wang L J, Huang W D. Effects of low light environment on the growth and photosynthetic characteristics of grape leaves. Journal of China Agricultural University, 2002, 7(3): 75-78. (in Chinese)
[35] 张明生, 谈锋. 水分胁迫下甘薯叶绿素a/b比值的变化及其与抗旱性的关系. 种子, 2001, 4: 23-25.
Zhang M S, Tan F. Relationship between ratio of chlorophyll a and b under water stress and drought resistance of different sweet potato varieties. Seed, 2001, 4: 23-25. (in Chinese)
[36] 王建华, 徐同. 模拟酸雨对棉花子叶叶绿素a、b含量及其比值的影响. 湖北农学院学报, 1991, 11(2): 1-10.
Wang J H, Xu T. Effects of simulated acid rain on content of chlorophyll a, b and their ratio in cotton cotyledon. Journal of Hubei Agricultural College, 1991, 11(2): 1-10. (in Chinese)
[37] Ge C L, Wang L, Ye W J, Wu L W, Cui Y T, Chen P, Pan J J, Zhang D, Hu J, Zeng D L, Dong G J, Qian Q, Guo L B, Xue D W. Single-point mutation of an histidine-aspartic domain containing gene involving in chloroplast ribosome biogenesis leads to white fine stripe leaf in rice. Scientific Reports, 2017, 7: 2398.
[38] Wang Y, Ren Y L, Zhou K N, Liu L L, Wang J L, Xu Y, Zhang H, Zhang L, Feng Z M, Wang L W, Ma W W, Wang Y L, Guo X P, Zhang X, Lei C L, Wan J M.encodes a novel P-type PPR protein required for chloroplast biogenesis during early leaf development. Frontiers in Plant Science, 2017, 8: 1116.
[39] Niu M, Wang Y H, Wang C M, Lyu J, Wang Y L, Dong H, Long W H, Wang D, Kong W Y, Wang L W, Guo X P, Sun L T, Hu T T, Zhai H Q, Wang H Y, Wan J M. ALR encoding dCMP deaminase is critical for DNA damage repair, cell cycle progression and plant development in rice. Journal of Experimental Botany, 2017, 68: 5773-5786.
[40] Liu L L, You J, Zhu Z, Chen K Y, ·Hu M M, Gu H, Liu Z W, Wang Z Y, Wang Y H, Liu S J, Chen L M, Liu X, Tian Y L, Zhou S R, Jiang L, Wan J M., encoding a deoxyribonucleoside kinase, is involved in chloroplast development in rice. Plant Cell Reports, 2020, 39: 19-33.
[41] Ye W J, Hu S K, Wu L W, Ge C W, Cui Y T, Chen P, Wang X, Xu J, Ren D Y, Dong G J, Qian Q, Guo L B.(), encoding a nucleoside diphosphate kinase 2 (OsNDPK2), regulates chloroplast development and abiotic stress response in rice (L.). Molecular Breeding, 2016, 36: 57.
[42] Wang P, Khoshravesh R, Karki S, Tapia R, Balahadia C P, Bandyopadhyay A, Quick W P, Furbank R, Sage T L, Langdale J A. Re-creation of a key step in the evolutionary switch from C3to C4leaf anatomy. Current Biology, 2017, 27(21): 3278-3287.
[43] Slewinski T L, Anderson A A, Zhang C K. Scarecrow plays a role in establishing Kranz anatomy in maize leaves. Plant Cell Physiology, 2012, 53(12): 2030-2037.
Cytological Characters Analysis and Low-resolution Mapping of Stripe-Leaf Mutantin Foxtail Millet
ZHANG Shuo1,2ZHI Hui1, TANG ChanJuan1, LUO MingZhao1, TANG Sha1, JIA GuanQing1, JIA YanChao1, DIAO XianMin1
1Institute of Crop Science, Chinese Academy of Agricultural Science, Beijing 100081;2Food Crop Research Institute, Hubei Academy of Agricultural Science, Wuhan 430064
【】Foxtail millet is a C4model plant, and its leaf color mutants are good materials for C4photosynthetic pathway research. Through the cytological characters analysis and gene initial mapping of the stripe-leaf mutantin foxtail millet, it laid the foundation for cloning the mutant gene, analyzing the chloroplast biogenesis and development, and further understanding the C4photosynthetic regulation mechanism in foxtail millet. 【】The stripe-leaf mutant of foxtail milletwas naturally mutated from intermediate material A36 created by breeding. Comparing the phenotypic characteristics ofand its isogenic line, which showed normal phenotypes, and investigating the agronomic traits, such as plant height, leaf width, leaf length, panicle weight, thousand-grain weight, and seed setting rate. To analyze the photosynthetic characters of, the chlorophyll content, net photosynthetic rate, intercellular CO2concentration, stomatal conductance, and transpiration rate ofandwere determined. By observing the leaf transverse section and ultrathin section ofand the control variety Yugu1, the leaf anatomical structure characters were analyzed, by counting the numbers and areas of the chloroplasts in mesophyll cells and bundle sheath cells respectively, the chloroplast biogenesis and development were assessed. An F2segregation population of×SSR41 were created, and genetic analysis was conducted by counting the number of normal phenotype single plant and stripe-leaf single plant in the population. The DNA mixed pools of normal single plants and stripe-leaf single plants of the F2segregation population were constructed separately, and the method of Bulked Segregation Analysis (BSA) was used to locate the mutant gene. By screening the stripe-leaf plants in the F2generation using SSR and In-Del markers, the mutant gene were furtherly located.【】The stripe-leaf mutant of foxtail milletshowed the phenotype of irregular white stripe-leaf in the whole growth period. Agronomic traits analysis showed that compared with its isogenic line,decreased significantly in plant height, leaf width, panicle weight, thousand-grain weight, and setting percentage. Photosynthetic index measurement showed that the chlorophyll contents ofwere also reduced significantly, especially the chlorophyll b content declined more severely, additionally, the net photosynthetic rate was also decreased significantly. Observation of the leaf anatomical structure showed that the chloroplasts number and area were significantly lower than that of the contrast Yugu1, while the changes in Kranz structure were not obvious. Furtherly, the ultrastructure of chloroplast was observed and showed that the chloroplast development situation in different cells was quite different. And the leaf cells ofcould be classified into three types: type Ⅰ cells had normal chloroplasts, type Ⅱ cells had chloroplasts with reduced grana and lamellar structures, while type Ⅲ cells had severely abnormal chloroplasts or even had no chloroplast. Genetic analysis suggested that the stripe-leaf trait ofwas controlled by a single recessive gene, and the mutant gene was located to the region from 7.66 Mb to 27.90 Mb of chromosome 4 by F2population. 【】Stripe-leaf mutant of foxtail milletrepresented decreased agronomic traits and photosynthetic capacity, and the number, size, and ultrastructure of leaf cell chloroplast were significantly abnormal. The stripe-leaf trait ofwas controlled by a single recessive gene, which was mapped to a region from 7.66 Mb to 27.90 Mb of chromosome 4.
(L.) P. Beauv.; stripe-leaf mutant; chloroplast; gene mapping

10.3864/j.issn.0578-1752.2021.14.003
2020-12-11;
2021-02-01
国家重点研发计划(2019YFD1000704,2019YFD1000700)、中国农业科学院科技创新工程协同创新任务“作物高光效育种生物学基础及核心材料创制”(CAAS-XTCX2016002)、国家现代农业产业技术体系(CARS-06-13.5-A4)、中国农业科学院科技创新工程(特色农作物优异种质资源发掘与创新利用创新团队)、湖北省农科院青年科学基金(2020NKYJJ02)
张硕,E-mail:zhangshuo0728@126.com。通信作者刁现民,E-mail:diaoxianmin@caas.cn
(责任编辑 李莉)

