生物信息学分析构建自噬基因相关的肝癌预后评估模型
刘新会 刘斐烨
肝癌是最常见的恶性肿瘤之一,是肿瘤相关死亡的第二大原因[1]。目前,消融、肝切除术和肝移植是治疗肝癌的常用方法,尽管近年来手术和系统性治疗取得了进展,但肝癌患者的5年生存率仍低于30%[2]。病理分级和BCLC分期是影响肝癌患者预后的重要因素,但现有研究发现即使是相同病理分级和分期的肝癌患者,其复发和死亡风险仍各不相同[3-5]。目前临床仍无可以精准预测肝癌患者预后的指标。自噬是一种细胞在营养缺乏条件下的分解代谢过程,大量证据表明,自噬在不同类型的癌症中上调或下调,发挥着阻碍肿瘤启动或促进肿瘤进展的双重作用[6-9]。同样,自噬在肝癌发展和生长过程中发挥着促肿瘤或抗肿瘤的双重作用[10]。值得注意的是,虽然多种自噬基因在肝癌中的作用已被认识,但自噬基因表达水平能否预测肝癌患者的预后仍不明确,且多数研究都集中在检测单一自噬基因上,对肝癌中所有自噬基因的表达水平和预后预测价值仍不明确。基于此,本研究分析癌症基因组图谱(the cancer genome atlas,TCGA)肝癌数据,明确肝癌组织中所有自噬基因的表达水平及其在预测肝癌预后中的作用,构建自噬基因相关的肝癌预后评估模型,其效果独立于肝癌患者的临床特征,能更精准预测肝癌患者的生存时间。
1 材料和方法
1.1 数据收集和差异表达分析 从TCGA肝癌数据库(https://www.cancer.gov/about-nci/organization/research/structural-genomics/tcga,tcga-lihc)下载374例肝癌组织和50例癌旁组织的原始RNA测序数据及其临床特征。采用R语言Limma包分析肝癌组织与癌旁组织间自噬基因的表达差异,显著差异表达基因(different expression genes,DEGs)定义为:和矫正P<0.05。采用R语言软件绘制DEGs的火山图和热图。
1.2 DEGs的基因本体论(gene ontology,GO)和京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)富集分析 为进一步分析DEGs的潜在分子功能、生物学过程和细胞成分的富集情况,采用R语言软件进行GO富集分析[11],采用clusterProfiler进行KEGG富集分析[12],进一步说明与DEGs相关信号通路的前景。以P<0.05为cut-off值,采用ggplot2和GO-plot R语言软件作图。
1.3 自噬基因相关的肝癌预后评估模型建立 通过单因素生存分析,明确与肝癌患者总生存时间(overall survival,OS)显著相关的自噬基因。随后,将前15个基因纳入多因素Cox回归分析,发现可独立指导肝癌预后的自噬基因。选取这些基因的表达值与权重的线性组合,建立肝癌预后评估模型,采用该模型将肝癌患者分为高危和低危亚群。通过Kaplan-Meier生存曲线和log-rank检验进一步验证高危和低危肝癌患者OS的差异。此外,对肝癌患者OS进行多因素Cox回归分析,包括肿瘤大小分期(T分期)、肿瘤淋巴结状态(N分期)、肿瘤转移分期(M分期)、病理分级和临床分期,同时计算HR和95%CI。所有分析都使用3.0.0版本R软件进行。
2 结果
2.1 肝癌组织和癌旁组织之间自噬相关的DEGs 本研究检测了TCGA肝癌数据库中203个自噬基因在肝癌组织和癌旁组织之间的表达,见表1。结果发现了60个自噬相关的DEGs,其中4个基因在肝癌组织中表达下调,而56个基因在肝癌组织中表达上调,这60个DEGs的热点图见图1(插页)。采用KEGG和GO富集分析进一步分析DEGs的生物学功能,见表2~3。KEGG富集的通路包括自噬、HPV感染、细胞凋亡和p53信号通路。GO富集的通路包括自噬、自噬调控和凋亡信号通路。

表1 TCGA肝癌数据库中60个自噬相关DEGs

表2 DEGs的KEGG富集分析

表3 DEGs的GO富集分析

图1 TCGA数据库肝癌组织和癌旁组织中60个自噬相关显著差异表达基因(DEGs)的火山图
2.2 自噬基因相关的肝癌预后评估模型 对TCGA肝癌数据库中的203个自噬基因进行了单因素生存分析。结果发现其中81个基因与肝癌预后显著相关,进一步选择前15个基因进行多因素Cox回归分析,发现4个自噬基因(HDAC1、ATIC、EIF2S1和 RAB7A)与肝癌患者的OS显著相关,见表4。以多因素Cox回归系数为权重,基于基因的表达值构建自噬基因相关的肝癌预后评估模型。计算公式具体如下:风险评分=(0.385×HDAC1表达水平)+(0.430×ATIC 表达水平)+(0.596×EIF2S1表达水平)+(0.449×RAB7A表达水平)。按中位风险评分将肝癌患者分为高危组(171例)和低危组(172例)。与低危组相比,高危组患者的OS明显缩短(P<0.05),见图2。
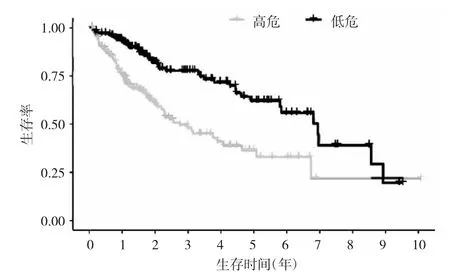
图2 自噬基因相关的肝癌预后评估模型预测高危或低危肝癌患者的生存曲线
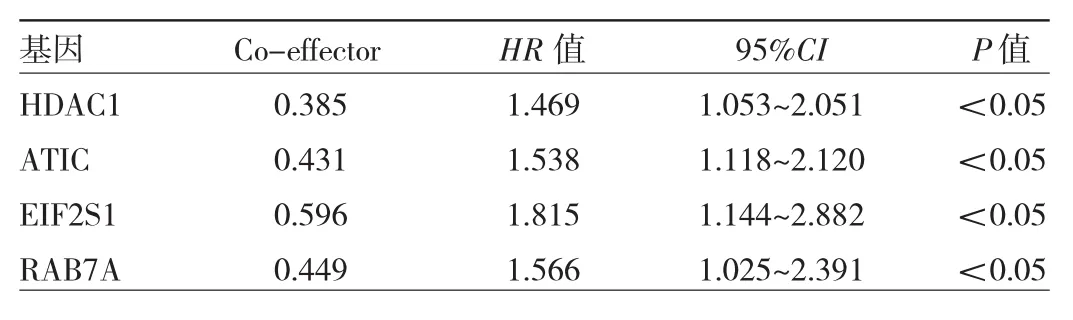
表4 肝癌患者自噬基因的多因素Cox回归分析
2.3 自噬基因相关的肝癌预后评估模型独立预测肝癌患者预后 采用单因素分析,进一步明确自噬基因相关的肝癌预后评估模型及其他临床病理因素对肝癌患者预后预测作用,见表5。自噬基因相关的肝癌预后评估模型(P<0.05)与其他临床病理因素,包括T分期(P<0.05)、M 分期(P<0.05)、肿瘤临床分期(P<0.05)与肝癌患者OS显著相关,而N分期和病理分级与肝癌患者OS无关(均P>0.05)。进一步进行多因素Cox回归分析,以确定自噬基因相关的肝癌预后评估模型对肝癌患者预后的预测价值是否独立于这些临床病理因素,见表6。尽管自噬基因相关的肝癌预后评估模型、T分期、M分期、肿瘤临床分期与肝癌患者OS显著相关,但只有自噬基因相关的肝癌预后评估模型可独立指导肝癌患者的预后(HR=1.536,95%CI:1.341~1.760,P<0.05)。
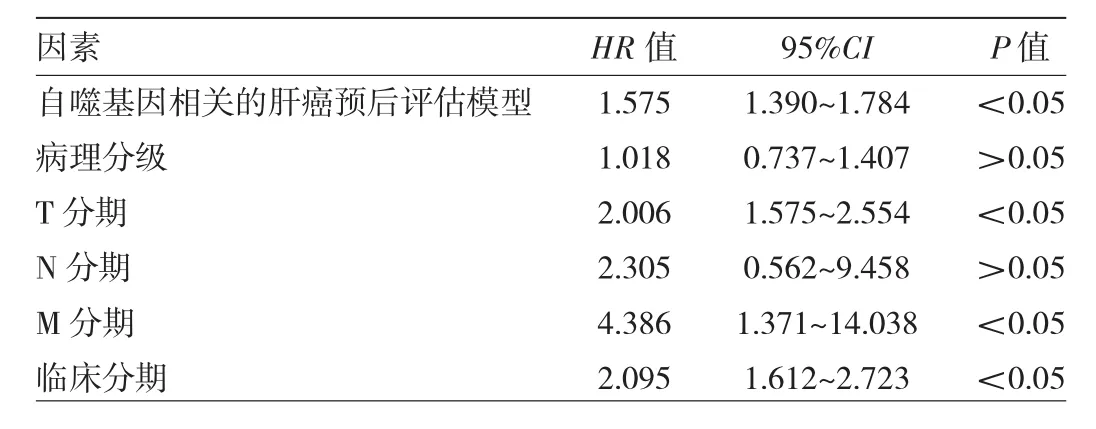
表5 影响肝癌患者预后的单因素分析结果

表6 影响肝癌患者预后的多因素Cox回归分析结果
3 讨论
目前,病理分级和BCLC分期是评估肝癌预后重要的预测指标。然而,有研究报道,即使是相同病理分级和BCLC分期的肝癌患者的复发和死亡风险也各不相同[5]。因此,需要更多的肝癌预后评估模型来帮助临床医生选择精准化的肝癌预防和治疗策略。越来越多的证据证明自噬在发生、发展中发挥着抑癌或促癌的双重作用[10,13],但自噬基因表达水平能否预测肝癌患者的预后仍不明确。
本研究通过对TCGA肝癌数据库自噬基因的表达谱分析,确定自噬基因在肝癌中的表达水平及其在预测肝癌预后中的作用。在203个自噬基因中检测到60个肝癌组织和癌旁组织间的DEGs,其中包括了已经报道的 TP73[14]、CDKN2A[15]、SPHK1[16]、FOS[17]等基因,这和本研究结果一致。但除此之外,本研究还发现一些新的肝癌组织与癌旁组织间DEGs,这些基因目前在肝癌中的作用仍不明确,例如CAPN10、RB1CC1、RPTOR等,这仍需要进一步的研究验证。
本研究同时分析明确了这些DEGs在自噬、凋亡和p53信号通路中富集,这提示自噬在肝癌发生、发展中可能共同参与了细胞凋亡、p53通路激活等相关信号通路。近年来已有研究表明,肝癌的发生、发展可能与自噬参与的炎症损伤和氧化应激的积累有关。例如,自噬基因PARP1可以和HBX协同抑制DNA损伤时DNA修复基因的募集,导致损伤进而参与肝癌的发生、发展[18],这和本研究发现自噬基因PARP1在肝癌中显著高表达一致。
更重要的是,本研究发现81个自噬基因与肝癌患者预后显著相关。其中包括了既往已经发现的和肝癌预后显著相关的 TSC1[19]、HDAC1[20]、PEA15[21]、SESN2[22]等基因。但仍有部分基因尽管已有研究发现其参与肝癌的发生、发展,但其能否预测肝癌患者预后仍不明确。例如FOXO1在肝癌中已有研究表明可能发挥抑制肝癌增殖和侵袭的作用[23-24],但是其能否作为肝癌的预后指标仍不明确,本研究不仅发现在肝癌组织中FOXO1显著低表达,而且其表达较低和肝癌预后较好显著相关。除此之外,本研究还发现了一些新的和肝癌患者预后显著相关的自噬基因,如 RAB24、DRAM1、BNIP1、TBK1 等,这些基因仍需要进一步研究明确其在肝癌中的作用及具体机制。
在进一步的多因素Cox回归分析中,基于4个自噬基因(HDAC1、ATIC、EIF2S1和 RAB7A)成功构建自噬基因相关的肝癌预后评估模型。该模型对肝癌患者的预后评估可独立于肝癌患者的T分期、M分期、肿瘤临床分期等其他临床特征。有研究发现HDAC1可以通过调控细胞有丝分裂参与肝癌的发生、发展,并能预测肝癌患者的死亡率[25-26]。ATIC通过调节AMPK-mTOR-S6 K1信号通路促进肝癌的增殖,并与肝癌预后较差有关[27]。这和本研究结果一致,但RAB7A和EIF2S1在肝癌发生、发展中发挥的作用尚未明确,值得进一步研究。
综上所述,本研究通过TCGA肝癌数据库生物信息学分析确定了自噬基因在肝癌的表达水平和对肝癌患者的预后影响,发现了60个在肝癌中显著表达差异和81个与肝癌预后显著相关的自噬基因,这些基因在肝癌中的作用及机制仍需要进一步的实验室研究进行验证。除此之外,本研究构建了自噬基因相关的肝癌预后评估模型,可独立预测肝癌患者预后,这值得在临床肝癌患者中进一步验证,以用于帮助临床医生选择精准化的肝癌预防和治疗策略。

