红螯螯虾养殖群体遗传多样性的SSR分析
金 玲,陈奕彬,李色东,5,刘 萍,李 健,吕建建
(1.上海海洋大学,水产科学国家级实验教学示范中心,上海 201306;2.中国水产科学研究院黄海水产研究所,农业农村部海洋渔业可持续发展重点实验室,青岛 266071;3.青岛海洋科学与技术国家实验室,海洋渔业科学与食物产出过程功能实验室,青岛 266235;4.广东恒兴饲料实业股份有限公司,广东湛江 524022;5.湛江市海洋与渔业发展研究中心,广东湛江 524039)
红螯螯虾(Cheraxquadricarinatus)原产于澳大利亚北部,俗称澳洲淡水龙虾,属于节肢动物门甲壳纲十足目拟螯虾科,是世界上最名贵的淡水经济虾种之一[1]。它具有个体大,食性杂,生长快,病害少,适应能力强等养殖优势。因其可食用部分比率高、肉味鲜美、营养价值高而广受欧美等国家欢迎[2,3]。我国内地最早于1992年广东珠海进行试养,随后在福建、江苏、浙江、江西、广西等地进行了小规模试养,产业整体处于初级阶段。目前,良种稀缺、亲虾品质良莠不齐等因素严重制约了红螯螯虾养殖业的发展[4]。
遗传多样性是遗传选育的基础[5]。目前国内关于经济甲壳类物种的不同地理群体遗传多样性分析已有不少的报道,如中国对虾(Fenneropenaeuschinensis)[6]、三疣梭子蟹(Portunustrituberculatus)[7]、中华绒螯蟹(Eriocheirsinensis)等[8]。开展遗传多样性和遗传结构分析对于了解红螯螯虾种质资源现状、育种潜力及制定育种方案具有重要意义[9]。
微卫星(Microsatellite)又称简单重复序列(Simple sequence repeats,SSR),因其多态性高、属于共显性遗传、操作便捷等特点,目前已被广泛应用于水产动物的遗传图谱构建、遗传多样性分析、遗传结构和亲缘关系分析等研究[10-12]。Baker等[13]最早开发了红螯螯虾7个微卫星位点。随后,谢艳楠[14]开发了28个微卫星位点,并对福建厦门、广东广州、上海崇明3个不同地理位置的红螯螯虾养殖群体进行了遗传结构分析。然而,自2011年以来,我国红螯螯虾群体遗传多样性是否发生改变或降低,亟需提供最新的遗传多样性分析数据。
本研究利用微卫星分子标记对我国广东和台湾两个主要的红螯螯虾养殖群体进行分析,旨在了解两个群体的遗传多样性信息和遗传结构,同时为后续的红螯螯虾种质资源评估及利用提供数据参考。
1 材料与方法
1.1 实验材料
实验所用的红螯螯虾群体分别来自广东和台湾。每个群体30尾,雌雄比为1 ∶1。取背部适量的肌肉样品共60份,于-80 ℃冰箱保存备用。
1.2 实验方法
1.2.1 基因组DNA的提取
采用TransGen Biotech的基因组DNA提取试剂盒,根据其说明书提取红螯螯虾DNA,并用1% 琼脂糖凝胶电泳检测DNA的完整性,微量分光光度计测其浓度,-20℃保存备用。
1.2.2 微卫星引物的筛选与条件优化
本实验所用引物序列均为之前已报道的SSR引物,由上海生工生物工程股份有限公司合成。利用梯度PCR方法筛选SSR引物并确定其最适退火温度,最终获得10对符合实验要求的引物(表1)。
1.2.3 PCR反应及检测
具体实施步骤如下:由3条引物进行PCR扩增,第1条是5′端带有M13尾的正向引物(5′-CACGACGTTGTAAAACGAC-3′);第2条是SSR反向引物;第3条是带有荧光标记的M13 通用引物(5′端用FAM、HEX、ROX荧光基团标记)。以上所用引物均由上海生工生物工程股份有限公司合成。引物准备就绪后利用筛选出的10个位点对60个红螯螯虾基因组DNA进行PCR扩增。PCR反应体系为20 μL:Taq酶(5 U/μL) 0.2 μL,10×Buffer 2 μL,dNTP(2.5 mmol/L) 3.2μL,M13正向引物(10 μmol/L) 0.4μL,反向引物(10 μmol/L) 1.6 μL,M13荧光引物(10 μmol/L) 1.6 μL,DNA模板小于1 μg,ddH2O补齐至20 μL。PCR扩增程序:94 ℃变性5 min;94 ℃变性30 s;引物在各自退火温度下复性30 s;72 ℃延伸1 min;34个循环;72 ℃延伸10 min;4 ℃保存。对PCR产物进行凝胶检测并观察其条带,将符合条件的PCR产物送至上海生工生物工程股份有限公司进行基因分型。
1.3 数据分析
运用PopGene3.2[15]软件来分析等位基因数(Na)、有效等位基因数(Ne)、Shannon指数(H)等;观测杂合度(Ho)、期望杂合度(He)、多态信息含量(PIC)以及Hardy-Weinberg平衡(P)检验等遗传多样性参数则利用Cervus 3.0.7软件完成;用公式D=(Ho-He)/He计算遗传偏离系数(D)[16];用FSTAT[17]软件计算近交系数(Fis)以及群体间的遗传分化指数(Fst)。
2 结果
2.1 SSR标记在两个群体中的分型
本研究对广东和台湾红螯螯虾养殖群体进行微卫星分型(图1)。10个微卫星位点在2个养殖群体中共检测到83个等位基因,各个位点的Na介于3~17之间,其中位点HAo22数量最多(Na=17),位点HAo04数量最少(Na=3);Ne范围为1.180~4.699,平均Ne为2.784(表2)。广东和台湾养殖群体在10个位点上的平均Na均为6;平均Ne分别为2.619和2.829(表3)。
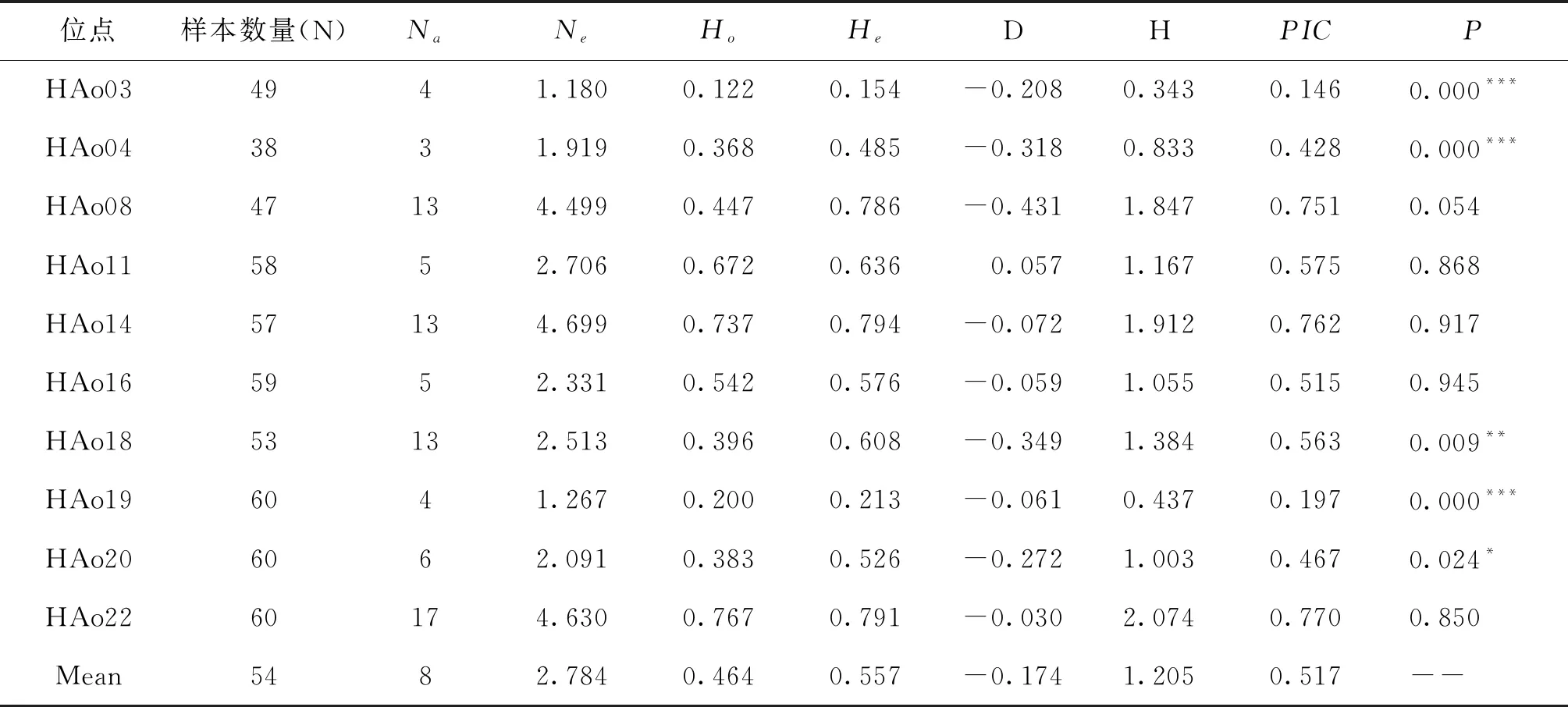
表2 10个微卫星位点上的遗传多样性参数
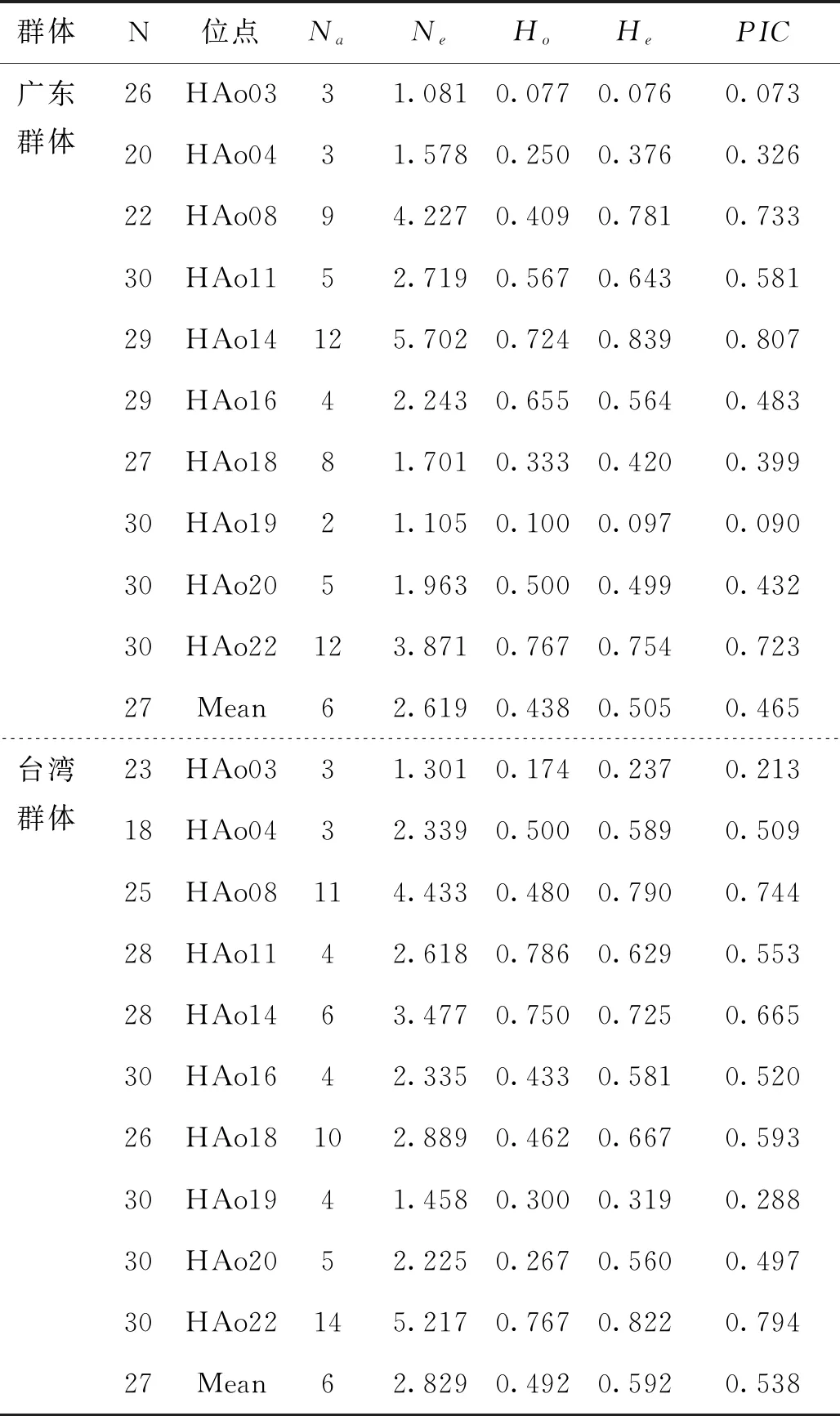
表3 2个红螯螯虾群体在10个微卫星位点上的遗传多样性参数比较
2.2 两个群体的遗传多样性分析
两个红螯螯虾群体的平均Ho为0.464,平均He为0.557;PIC介于0.146~0.770之间,平均PIC为0.517,其中位点HAo22最高,位点HAo03最低(表2)。结果表明10个位点中6个位点显示高度多态性(PIC>0.50),2个位点显示中度多态性(0.25 表4 2个红螯螯虾群体10个微卫星位点的遗传多样性指数及近交系数 对红螯螯虾2个群体的遗传分化指数Fst值进行分析,广东群体和台湾群体间的Fst为0.0969,属于中等程度的分化(0.05 遗传多样性分析结果表明,广东群体平均PIC为0.465,呈中度多态性,而台湾群体的平均PIC为0.517,呈高度多态性。整体而言,国内两个群体的遗传多样性水平较高。谢雁南等[14]利用微卫星分子标记对厦门、广州、崇明3个红螯螯虾养殖群体进行了遗传多样性分析,结果显示三个群体的平均PIC为0.451 ~ 0.480。该结果与本研究结果相似,表明经过近十年的累代养殖,国内红螯螯虾养殖群体仍保持在较高的遗传多样性水平。然而,相较Baker等[18]已报道的澳大利亚野生种群,国内养殖群体的遗传多样性水平确有一定程度的下降,说明在今后的选育或保种过程中,仍需制定科学、合理的方案,以维持较高的群体多样性水平。 Shannon指数H是判定遗传多样性水平的重要指标[19],根据Shannon多样性指数,广东群体和台湾群体分别为1.069、1.192,进一步说明两群体的遗传多样性较为丰富。Fst是反映群体间遗传分化程度的重要参数。本研究中,红螯螯虾群体间的Fst值为0.0969,为中等程度遗传分化(0.05 本研究通过微卫星标记对广东和台湾红螯螯虾养殖群体进行分析,得到其群体间的遗传多样性等信息。研究结果为红螯螯虾的遗传多样性评估、遗传选育提供数据参考,有助于推动红螯螯虾种质资源的合理保护与利用。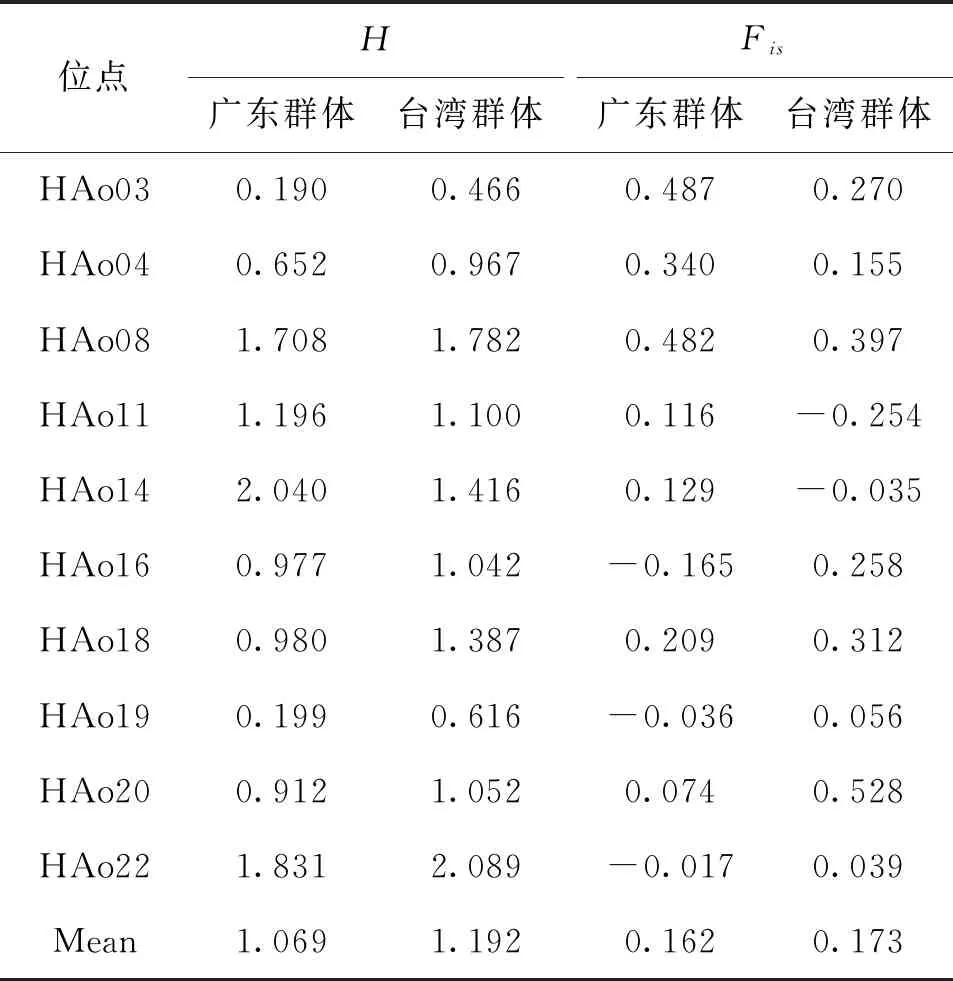
2.3 群体间的遗传分化及近交系数评估
3 讨论

