基于文本挖掘筛选COVID-19症状相关核心基因及分析潜在药物
李俊玲,何太平,王晓辉,杨峥嵘 (1.广东医科大学公共卫生学院,广东东莞 52808;2.广东医科大学公共卫生学院,广东湛江 52402;.深圳市疾病预防控制中心,广东深圳 508055)
SARS‑CoV‑2是于2020年1月通过基因测序确定的一种新型冠状病毒,该病毒引起新型冠状病毒肺炎(COVID‑19),简称“新冠肺炎”,并造成了世界范围的广泛流行[1]。SARS‑CoV‑2的持续传播,对国际公共卫生构成了巨大威胁[2],全球科学家都在积极探索新冠肺炎治疗和预防的方法,其中药物研究是一个热点。我们通过大数据的挖掘以及生物信息学的分析筛选出新冠肺炎症状相关的核心基因,并鉴定出潜在对症治疗药物,希望能为新冠肺炎药物的研发提供一些帮助。
1 材料和方法
1.1 文本挖掘确定基因列表
查阅文献确定了临床诊断中与COVID‑19 密切相关的7 个主要临床症状,分别是“发烧”“咳嗽”“重症肺炎”“呼吸困难”“呼吸窘迫”“乏力”“肌肉酸痛”,随后通过文本挖掘数据库(http://pubmed2ensembl.ls.manchester.ac.uk/)搜索这7 个关键词,得到7 组与这些关键词相关的基因列表,以供下一步分析。
1.2 基因功能注释和通路富集分析
基因注释功能分析主要包括基因的生物学过程、分子功能和细胞组分这三个方面。通路富集分析能够从分子水平确定基因参与最主要的信号转导途径。这两种分析方法主要是基于超几何分布检验,通过P值大小筛选出这些基因主要显著富集在何种生物学过程、分子功能、细胞组分及通路。利用R 软件中的clusterProfiler 包对基因进行功能注释和通路富集的统计分析和可视化[3]。
1.3 蛋白互作网络分析
用STRING(https://string‑db.org/)数据库来确定蛋白之间相互作用的信息,用Cytoscape 软件来可视化STRING 数据库中导出的蛋白互作数据信息,再利用互作网络中分值由大到小的顺序确定核心基因[4]。
1.4 药物基因相互作用分析
DGIDB 数据库是探索药物与基因之间的相互关系的分析平台,将上述分析后产生的核心基因作为靶向基因并使用DGIDB 数据库来筛选出潜在治疗药物,并最终用R软件中的ggalluvival包来可视化药物‑基因‑通路之间的相互关系[5]。
2 结果
2.1 确定基因列表
通过文本挖掘数据库对7个COVID‑19主要症状进行检索,得到7 组基因集,涉及的基因数量分别为:768、563、456、627、879、1 148、809 个,对这7 组基因集取交集,得到包含97个基因的基因列表(图1)。
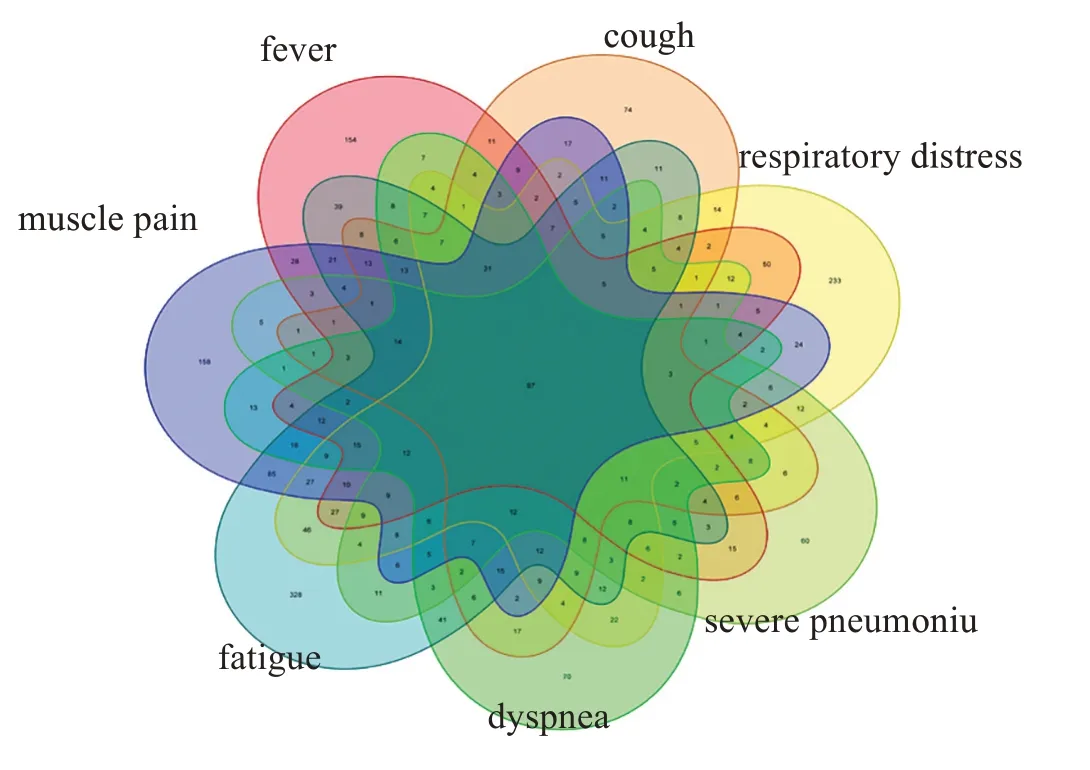
图1 与COVID‑19症状密切相关的基因列表
2.2 基因功能注释和富集通路分析
使用R 软件对97 个基因进行基因功能注释和通路富集分析。基因功能注释显示(如图2,仅显示P值最小的前6 个条目)这些基因的生物学过程主要集中在Stat5 蛋白酪氨酸磷酸化的正调控(P=3.86E‑11)、活化T 细胞增殖的正调控(P=7.35E‑06)等96 个功能簇;这些基因的细胞组分构成主要是细胞外隙(P=1.79E‑18)、胞外区(P=1.00E‑12)和质膜外侧(P=7.64E‑09)等21 个功能簇;这些基因的分子功能方面主要集中在细胞因子受体结合(P=6.15E‑12)、生长因子活性(P=6.14E‑11)等35个功能簇。
富集通路分析显示这些基因一共富集到47 条通路,同样选择6 条P值最小的通路(如图3),分别是细胞因子与细胞因子受体的相互作用(P=5.00E‑12)、JAK‑STAT 信号通路(P=6.93E‑10)、疟疾相关免疫反应(P=1.35E‑09)、炎症性肠病(IBD)(P=5.93E‑09)、T细胞受体信号通路(P=6.18E‑07)、同种异体移植排斥(P=1.34E‑06)以及PI3K‑Akt信号通路(P=2.57E‑06)。

图3 通路富集分析柱状图
2.3 构建互作网络并选取核心基因
利用String 数据库对97 个基因进行相互作用分析,构建出了一个拥有85 个节点,917 条边的蛋白互作网络图(如图4)。利用Cytoscape 软件筛选出85 个基因节点中与其他基因之间相互联系最为密切的,分值最高的前30个基因(如图4中颜色较深部分),为下一步数据分析做准备。

图4 基因相互作用网络图
2.4 药物基因相互作用分析
我们用上述筛选到的30 个基因进一步分析药物与基因之间的相互作用,最终筛选到9 个核心基因,涉及到22 个潜在药物,可能对COVID 具有一定的对症治疗作用(如表1)。

表1 基于核心基因治疗COVID‑19的潜在药物汇总表
3 讨论
COVID‑19 作为一种新发的传染病,至今尚无确定的特效药物。对于大多数轻症及无症状感染者,此疾病具有一定的自愈能力或者不需要特别的治疗,但是对于一些危重患者来说,情况却很严峻。有研究表明,即使在积极的治疗情况下,进入ICU的COVID‑19重症患者28 d 内病死率高达61.5%,其中有47.0%的重症死亡患者并无基础疾病[6]。因此,迫切需要筛选有效的COVID‑19 药物来降低病死率。文本挖掘等生物信息学分析工具为药物筛选提供了一条快速通道,通过KEGG 通路富集分析,按照P值由小到大的顺序鉴定出3 条与COVID‑19 症状密切相关的通路;进一步通过蛋白互作分析以及基因与药物间的相互作用,筛选出9个与COVID‑19症状密切相关的基因。
KEGG 通路富集分析结果表明,与COVID‑19 症状高度相关的通路与细胞因子及其受体间的相互作用相关。多项临床实验表明,在SARS‑CoV2的感染过程中,COVID‑19患者的淋巴细胞和NK细胞计数显著降低,细胞因子水平却显著升高[7],出现“细胞因子风暴”,使宿主免疫反应过度,造成急性肺部损伤、多器官衰竭以及不良的预后等严重的后果[8]。但细胞因子发挥其生物学功能是需要通过与靶细胞表面的相应受体结合才能将信号转导到细胞内部,因此,细胞因子与其受体的相互作用是重要的治疗靶点。细胞因子及其受体互作网络极其复杂,需要分析处于核心的细胞因子。在严重的SARS‑CoV‑2感染病例中,IL‑6水平显著升高,是最常被检测出来并被报道[9‑11],而IL‑6 受体与IL‑6结合进一步促进IL‑6 的生物学作用,加剧“细胞因子风暴”的进程。我们鉴定到的药物司妥昔单抗可以有效地阻断两者的结合,避免激活信号传导通路[12],可能是COVID‑19严重感染病例的有效治疗手段。
其次是JAK‑STAT 信号通路,在炎症反应时,细胞因子与其受体相互作用增强,进一步激活JAK,发生JAK 的自磷酸化以及STATs的二聚化,随后二聚化后的STATs 进入到细胞核中参与细胞的免疫调节等生物学过程,进一步促进“细胞因子风暴”[13]。因此,通过JAK 抑制剂治疗由SARS‑CoV‑2 引起的“细胞因子风暴”可能是一种有效策略。经过检索文献发现鲁索替尼作为JAK的抑制剂,相比其他药物耐受性较好并且在老年人群体中也适用,可能对COVID‑19 患者出现的免疫反应过度症状有比较好的效果[14]。
另一条与COVID‑19 症状高度相关的通路是疟疾相关免疫反应信号通路,疟原虫感染及其治疗药物均有其特点,最古老的治疗药物为氯喹,后来逐步改进到磷酸氯喹、羟氯喹等衍生药物。在武汉、荆州、广州、上海、北京、重庆、宁波等多家医院进行的试点实验表明磷酸氯喹可以有效地抑制肺炎的恶化,缩短COVID‑19 的病程[15]。国家卫建委发部的《新型冠状病毒肺炎诊疗方案(试行第八版中)》也指出磷酸氯喹可以继续试用,在临床应用上进一步评价它的疗效。综合分析来看,磷酸氯喹可以有效地调节与COVID‑19相关的病理学通路。
在进一步的基因与药物相互作用分析中,我们发现KIT 基因所筛选到的靶向药物最多且评分最高。KIT 基因是一种Ⅲ类酪氨酸酶受体,它的表达异常可能会使宿主细胞发生多种肿瘤[16‑18]。我们经过一系列的生物信息学分析发现KIT 与COVID‑19 具有很强的相关性。伊马替尼是我们鉴定到评分最高的KIT抑制剂,它在针对严重性呼吸窘迫综合征以及中东呼吸综合征冠状病毒的体外实验中显示出具有抗病毒活性[19]。今年6 月份,国外相关临床研究显示,一位38 岁的确诊女性病例在经过羟氯喹和利托那韦的双重治疗后病情却再次复发之际,改用伊马替尼进行治疗后情况好转并顺利出院[20]。这提示伊马替尼治疗COVID‑19具有继续进行临床研究的价值。
基因筛选评分其次的是ACE基因,它与ACE2基因是肾素‑血管紧张素系统(RAS)中的两个不可或缺的调制器,两者之间相互保持平衡对于维持RAS的稳定具有重要的作用,预计可以有效地降低COVID‑19的死亡率和发病率。ACE2由于与冠状病毒表面的胞膜蛋白有很好的亲和力使得使其消耗而表达水平下降[21‑22],此时ACE 和ACE2 之间的表达不平衡促使血管紧张素II的水平不受限制,增加血管的通透性并引起血管收缩,从而导致急性肺损伤并促进纤维化。一项包含1 128名COVID‑19 伴高血压患者的多中心回顾性研究显示,使用ACE 抑制剂的住院患者比不使用的死亡风险降低[23],我们研究中鉴定到的一些ACE抑制剂,如卡托普利等药物可能在一定程度上具有治疗和预防由COVID‑19引起的急性肺损伤的问题。
基因筛选评分第三是ESR1 基因,是一种介导雌激素发挥生物学效应的配体依赖转录因子。COVID‑19 流行病学资料显示,不同性别、年龄群体对于新冠病毒所表现出的炎症反应是不同的。有研究表明,除了个体差异,雌激素可能是造成这一差别的重要原因[24]。雌激素可以调节中性粒细胞、巨噬细胞等免疫细胞的发育,使B 细胞介导的适应性免疫产生特异性抗体[25],抑制NF‑kB 通路介导的炎症反应,可能降低肺部损伤。这个机理在动物实验中已得到证实[26],国外已有学者提出雌激素可能会降低COVID‑19 的死亡率[27‑28],因此对老年女性新冠患者尝试用外源雌激素治疗的方法可能更具有实际意义。
除了上述描述的评分较高的3 个基因之外,还有其他一些与新冠症状密切相关的细胞因子及受体,包括TNF、VEGFA、IL‑1B、IL5、TGFB1,同时也确定了一些基因对应的靶向药物。综上,我们利用生物信息学的分析方法筛选出一些COVID‑19 症状相关的核心基因,之后通过基因功能富集分析和通路富集分析对这些基因的生物学功能、涉及的信号通路做了进一步的分析,同时分析出与核心基因相互作用的潜在药物。希望此类研究能为COVID‑19 的预防和治疗提供一定的方向指引。

