新型冠状病毒核酸荧光型RT-RAA检测方法的建立及其评价
高 越,刘伯玉,任翠平,高 勇,朱 禹,杨 莉,温 萌,钱 振,芦宝静,柳 燕
新型冠状病毒肺炎(coronavirus disease 19,COVID-19)是由严重急性呼吸综合征冠状病毒2型(severe acute respiratory syndrome coronavirus 2,SARS-CoV-2)引起的急性传染病。此次疫情在全球范围内快速蔓延,WHO已将其认定为国际公共卫生紧急事件。冠状病毒(coronavirus,CoVs)是一种有包膜的单链正链RNA病毒,隶属于冠状病毒科。病毒基因组中16个非结构蛋白由5′端的开放阅读框架(ORF)1a/b编码,而结构蛋白包括刺突蛋白(S)、包膜蛋白(E)、膜蛋白(M)和核衣壳蛋白(N)则由3′端的其他ORF编码。由于潜伏期长,感染者的早期症状无特异性,且存在无症状感染者,极易造成漏诊或误诊,因此,建立更便捷、快速的分子检测方法,为临床提供精准诊断具有重要意义。
该研究建立的反转录重组酶介导核酸扩增(reverse transcription recombinase aided amplification,RT-RAA)技术是一种等温快速扩增检测方法,通过是选择SARS-CoV-2的S
基因和orf1ab
基因,设计特异性等温扩增引物和探针,建立了SARS-CoV-2快速诊断的分子检测方法,并进行初步评价。1 材料与方法
1.1 材料
1
.1
.1
样本 收集2020年1月20日—2月28日的临床确诊感染SARS-CoV-2患者咽拭子标本30例来自于项目合作单位安徽省阜阳市第二人民医院,COVID-19的诊断依据是中国国家卫生委员会发布的新的《冠状病毒肺炎防治方案(第五版)》;另外临床呼吸道感染咽拭子标本30份为2019年底前本实验室收集,经核酸检测分别包括H9N2(3例)、H7N9(2例)、H3N2(5例)、H1N1(6例)、B型流感(6例)、RSV(3例)、RV(5例)。纳入研究的所有患者都知情同意,经安徽医科大学人类生物伦理学委员会批准,批准号为2020H015。1
.1
.2
主要仪器及试剂 体外转录试剂盒RiboMAXLarge Scale RNA Production System-T7(美国Promega公司,货号:P1300);二级生物安全柜、GENCHECK-1荧光检测仪(杭州众测生物科技公司);实时荧光定量 PCR 仪(美国Bio-Rad公司,型号:CF-X96TM);RNA核酸提取RNeasy Mini Kit (德国QIAGEN公司,货号:74106);磁珠核酸提取试剂(江苏泰州硕世生物科技有限公司,货号:SDK60104);全自动核酸提取仪(苏州华益美生物科技有限公司,型号:SLAD14800);新型冠状病毒 2019-nCoV 核酸检测试剂盒(荧光 PCR 法)(上海伯杰医疗科技有限公司,货号:ZC-HX-201-2);重组质粒由生工生物工程(上海)股份有限公司合成;质粒提取试剂盒(美国AXYGEN公司,货号:AP-MN-250);RT-RAA核酸扩增试剂盒荧光型(杭州众测生物科技有限公司,货号:S004ZC)。1.2 方法
1
.2
.1
引物探针设计及筛选 从GenBank数据库下载SARS-CoV-2病毒全序列,通过Bioedit (v7.0.9.0)软件进行多重序列比对,根据对比结果找出具有高同源性的保守区域,最终选择SARS-CoV-2保守性较高的S
基因和orf1ab
基因作为靶基因,并与其他冠状病毒序列进行比较,将GenBank登录号为MN908947.3的SARS-CoV-2 Wuhan-Hu-1基因组序列作为引物、探针设计的候选序列,用Oligo (v7.56)软件进行探针和引物设计。依据恒温快速扩增技术设计原则对两个靶基因分别设计上、下游引物各 3条,以及4条探针进行第一轮筛选,为进一步提高其扩增效率,依据引物筛选策略,在第一轮筛选结果的基础上将待选引物碱基位置及数量做进一步调整。经补充设计orf1ab
基因4条上游引物和4条下游引物,S
基因2条上游引物和2条下游引物进行第二轮筛选,确定可良好工作的引物和探针。见表1。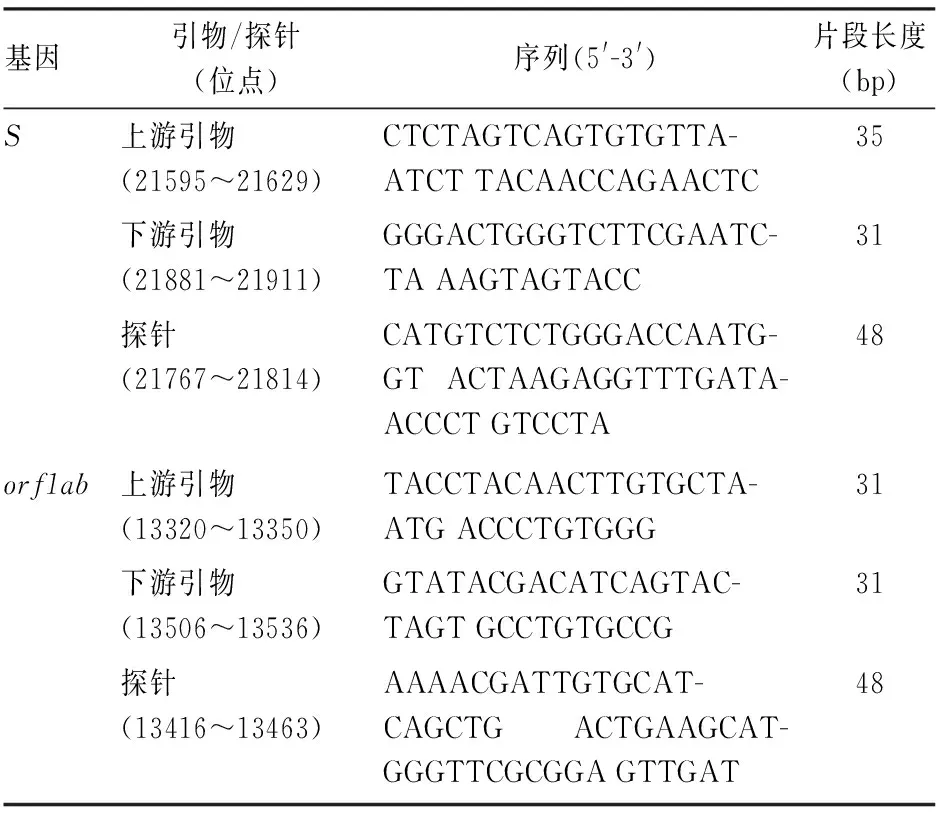
表1 SARS-CoV-2引物和探针序列
1
.2
.2
探针修饰 探针的修饰方法包括:荧光标记基团为FAM,荧光淬灭基团为 BHQ1。S
基因的探针荧光报告基团修饰在探针序列距5′端碱基数31 bp 的位置上;荧光淬灭基团修饰在探针序列距 3′端碱基数 13 bp 的位置上,荧光报告基团与淬灭基团之间间隔 4 个碱基(TTGA),其中第2个碱基T用四氢呋喃残基替换,序列尾部加上C3 spacer修饰。orf1ab
基因的探针中荧光报告基团修饰在探针序列距5′端碱基数31 bp 的位置上,荧光淬灭基团修饰在探针序列距 3′端碱基数 14 bp 的位置上,荧光报告基团与淬灭基团之间间隔3个碱基(GGG),其中第2个碱基G用四氢呋喃残基替换,序列尾部加上C3修饰,经过修饰的探针如表2所示。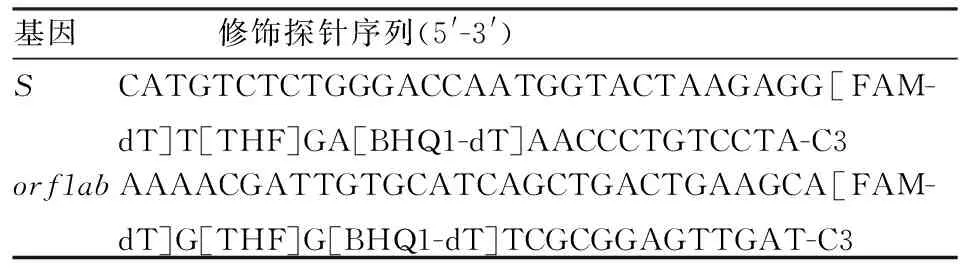
表2 SARS-CoV-2修饰探针序列
1
.2
.3
体外转录RNA 针对SARS-CoV-2的S
基因和orf1ab
基因分别设计一对带有T7启动子的引物,其序列如表3所示,利用表中引物分别对含有相应S
基因和orf1ab
基因序列的质粒进行PCR扩增,采用RiboMAXLarge Scale RNA Production System-T7试剂盒对SARS-CoV-2进行体外转录,分别获得S
基因和orf1ab
基因的 RNA 模板,采用 Nanodrop 2000分光光度计测量纯化后RNA产物的质量浓度,计算出S
基因orf1ab
基因的浓度为3.68×10、4.02×10拷贝数/μl。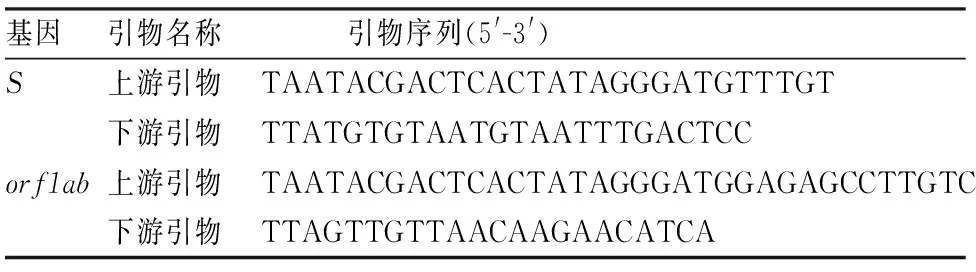
表3 质粒PCR扩增引物序列
1
.2
.4
Real-time PCR检测SARS-CoV-2 采用磁珠核酸提取试剂盒提取样本核酸,将提取的核酸模板液20 μl转移至已分装扩增混合液的PCR反应管中,盖上管盖,混匀,离心后放入多通道核酸检测仪。根据试剂说明书,用FAM通道检测orf1ab
基因,HEX/VIC通道检测N基因,ROX通道检测内参基因;阳性和阴性的判断标准参照试剂盒说明书,在内参基因Ct值≤38的情况下:①两种基因Ct值均≤38,样本为阳性; ②两种基因均无Ct值或Ct值>38,样本为阴性; ③单个基因为阳性时,为可疑标本。1
.2
.5
RT-
RAA反应体系的建立及优化 商品化荧光RAA试剂盒包含A、B缓冲液。每反应管总体积为50 μl, RT-RAA等温扩增试剂的反应体系如表4所示。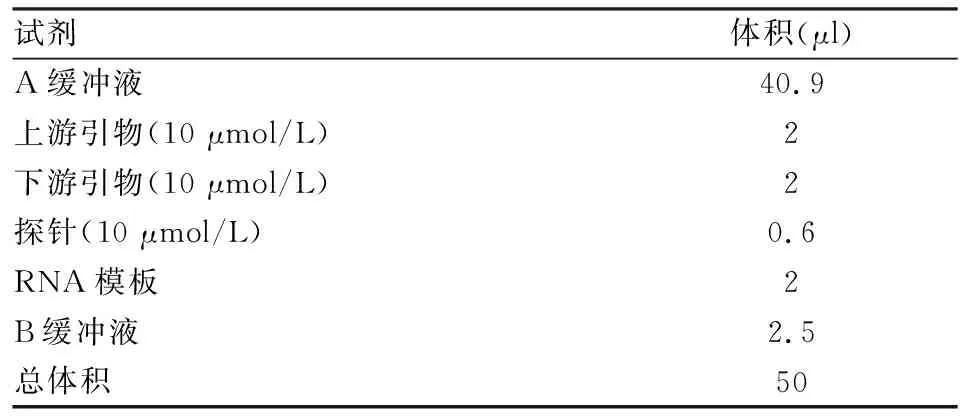
表4 RT-RAA反应体系
如上配置好反应体系后,盖上管盖,上下颠倒充分混匀5~6次,低速离心10 s,将检测单元管放入恒温扩增荧光检测仪,选用经过优化的RT-RAA程序:40.5 ℃,每 30 s读取一次荧光值,循环40次,共20 min。启动仪器。阳性和阴性的判断标准:阳性结果一般以显示典型的扩增曲线判定,阴性结果和对照一般显示为水平直线(即未采集到荧光信号)或轻微斜线。
1
.2
.6
灵敏性检测 将SARS-CoV-2体外转录RNA稀释为10、10、10、10、1 拷贝数/μl共5个浓度梯度,并使用无核酸酶的水作为阴性对照,用已优化好的反应体系进行等温扩增,测试方法的灵敏度。1
.2
.7
特异性检测 将COVID-19疫情之前,经核酸检测验证为其他呼吸道病毒感染的流感的30例咽拭子标本提取RNA,分别将S
基因和orf1ab
基因的体外转录RNA 模板作为阳性对照,使用无核酸酶的水作为阴性空白对照,对 RT-RAA等温快速扩增技术进行特异性评价,通过收集荧光信号判断其他病毒是否有扩增。1
.2
.8
重复性检测 用已优化好的反应体系分别将S
基因引物和orf1ab
基因引物的最低检出限2 拷贝数/反应管的SARS-CoV-2体外转录RNA模板进行3次重复实验,并使用无核酸酶的水作为阴性对照,测试该方法的重复性。1
.2
.9
临床标本验证 提取本次研究检测的60例临床样本中RNA,用建立的RT-RAA等温快速扩增方法对临床标本进行检测,并与Real-time PCR的检测结果进行比较,计算Kappa值(采用SPSS 13.0统计软件),分析两种检测方法检测结果的一致性,并计算出各引物的阳性符合率和阴性符合率,以此判断本研究建立的SARS-CoV-2 RT-RAA方法的临床检测效果。1
.3 统计学处理
应用SPSS 13.0软件进行统计学处理,两组间的一致性比较采用Kappa检验,α=0.05检验水准下,Kappa值越接近1,说明两组间的一致性越好。2 结果
2.1 SARS-CoV-2 RT-RAA方法的建立及优化
本研究根据orf1ab
和S
基因设计多对引物和探针,为了确定最合适的引物、探针和反应温度,分别对S
基因和orf1ab
基因的引物和探针进行了一系列的排列组合,并用浓度为10拷贝数/μl的体外转录的SARS-CoV-2 RNA模板在39~42 ℃之间做温度梯度探索,结果显示在40.5 ℃时,表1所列的引物探针能在最短的时间荧光强度达到最高,因此确定本研究建立的RAA方法的最佳反应温度为40.5 ℃。RAA荧光法最佳反应条件为40.9 μl A缓冲液、上游引物、下游引物(10 μmol/L)各2 μl,0.6 μl探针(10 μmol/L),2 μl模板至47.5 μl,最后向检测单元管盖上加入2.5 μl的B缓冲液,并混匀。2.2 RT-RAA方法的灵敏性
对于所稀释的各浓度的体外转录RNA,其S
基因和orf1ab
基因均可检出,且2种基因的最低检出限均可达到2 拷贝数/反应管。S
基因检出最低检测线的用时为11 min,orf1ab
基因检出最低检测线的用时为9 min。见图1。以上表明,本研究建立的RT-RAA 检测方法灵敏性好,且检测速度快。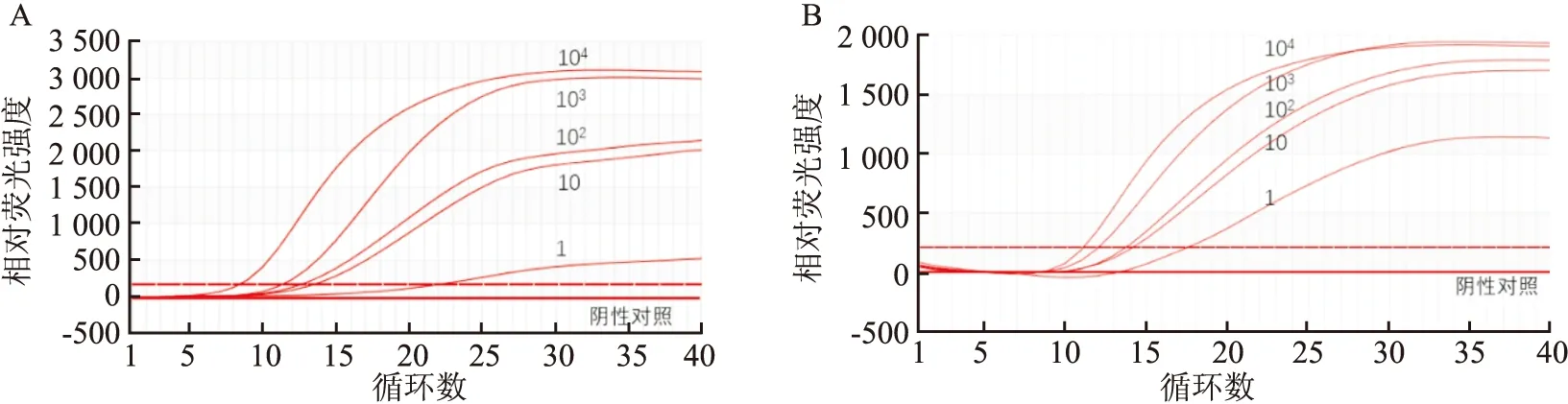
图1 RT-RAA等温扩增技术灵敏性检测A:S基因;B:orf1ab基因
2.3 RT-RAA方法的特异性
仅有SARS-CoV-2阳性对照有扩增曲线,为阳性,其他病毒及阴性对照均无扩增,显示本研究建立的RT-RAA等温扩增技术具有良好的特异性。见图2。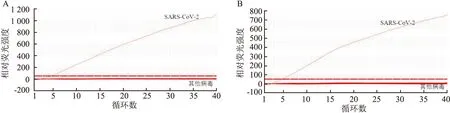
图2 RT-RAA等温扩增技术特异性评价A:S基因;B:orf1ab基因
2.4 RT-RAA方法的重复性
S
基因引物和orf1ab
基因引物的SARS-CoV-2体外转录RNA模板进行3次重复实验的结果如图3所示,结果显示2种引物扩增反应起峰时间一致,曲线形态接近,阴性对照无扩增,具有良好的重复性。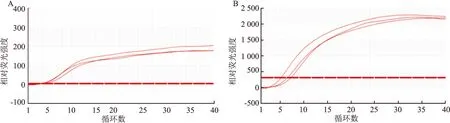
图3 RT-RAA等温扩增技术重复性评价A:S基因;B:orf1ab基因
2.5 RT-RAA方法检测临床标本的符合率
本次研究检测的共60例临床样本,结果如表5与表6所示。与Real-time PCR结果相比,S
基因的阳性总符合率为96.66%,阴性总符合率为100%;orf1ab
基因的阳性总符合率为100%,阴性总符合率为100%。经SPSS13.0软件进行Kappa检验,S
基因的Kappa值为 0.967,orf1ab
基因的Kappa值为1,提示本研究建立的RT-RAA等温快速扩增技术与Real-time PCR检测结果的一致性极好,两种引物均有较好的临床检测效果。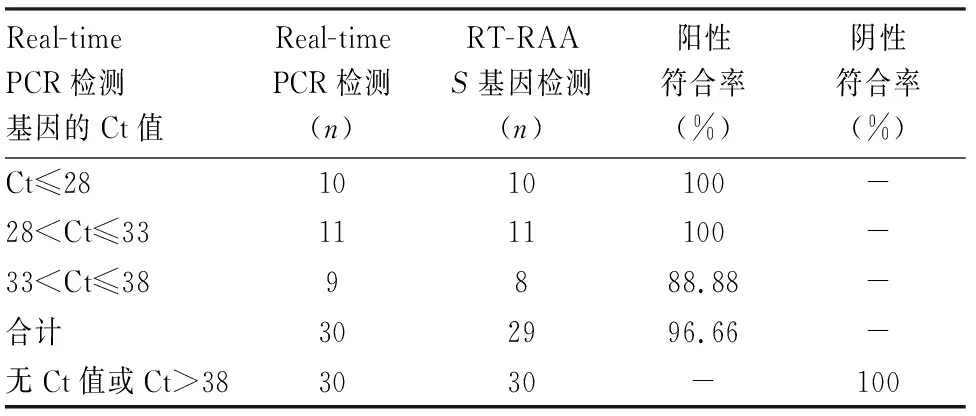
表5 RT-RAA等温扩增技术检测临床标本S基因结果验证情况
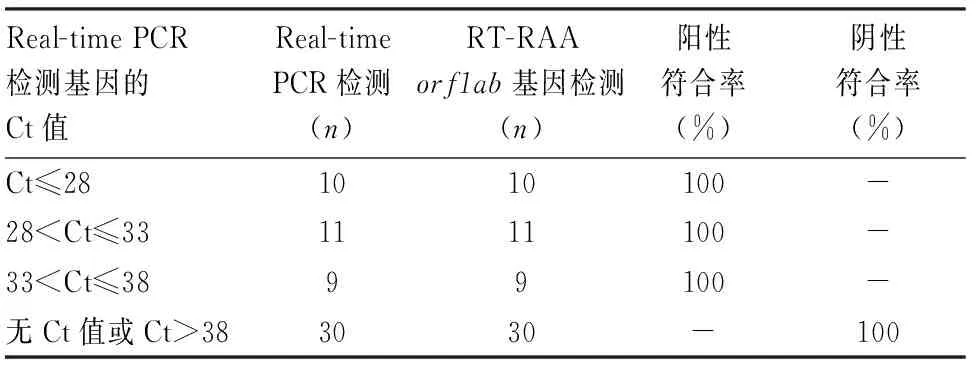
表6 RT-RAA等温扩增技术检测临床标本orf1ab基因结果验证
3 讨论
SARS-CoV-2疫情的爆发对国际卫生构成重大威胁。当前全球六大洲、200多个国家和地区受到新型冠状病毒肺炎疫情不同程度的影响。目前,尚未有针对该病毒的特异治疗方法且相关疫苗正在研发中。因此,建立一种针对SARS-CoV-2的现场便携式快速检测方法,弥补一些昂贵的检测设备带来的检测能力不足的缺陷,对SARS-CoV-2的防控具有重大价值。
该病毒最初是通过RT-PCR和咽拭子测序确认。后来,检测SARS-CoV-2的Real-time PCR方法迅速建立,并在国内外得到广泛应用。但该方法需要昂贵的实验仪器,且大多数的Real-time PCR试剂盒的灵敏度较低(10拷贝数/μl),有报道称患者出院后一段时间又出现核酸复检阳性,这可能由多种原因造成,如患者出院时核酸浓度未达到Real-time PCR检出限导致阴性误判、标本采集及运输过程的不规范操作、扩增仪孔内温度差异、核酸提取过程中的损耗等影响检测结果。因此,需要现场开展更高的核酸灵敏度检测以有效解决上述问题带来的不利影响。
已有报道建立了RPA扩增方法用于检测新型冠状病毒的特异性S
基因以及N
基因。但目前国内使用的RPA试剂均需进口,经济成本高、不稳定等因素多,而RAA技术是一种具有我国自主知识产权的等温核酸扩增技术,不受国外专利限制,且试剂成本低廉,不需昂贵的仪器。并且,相对于RPA使用的噬菌体T4uvsX重组酶,RAA使用的细菌或真菌中获得的重组酶不仅来源更为广泛,而且在37 ℃恒温下便可与引物紧密结合形成酶-引物聚合体,反应的温度适应性更强。本研究建立的RT-RAA 检测方法通过荧光探针实现检测,以S
基因和orf1ab
基因作为靶标,通过对反应体系的优化,对于SARS-CoV-2 RNA的模板,其两种引物均可稳定检出,灵敏性达到2拷贝数/反应管,且重复性较好。同时与其他相关病毒无交叉反应,特异性好,与Real-time PCR方法相比,临床样本S
、orf1ab
基因的阳性符合率为96%、100%,阴性符合率为100%;通过Kappa检验表明与Real-time PCR检测结果有较好的一致性。S
基因的阳性符合率没有达到100%,其灵敏度未达到理论值的1拷贝数/μl,可能是由于该研究所用的体外转录RNA中的RNA纯度较高,因此未能完全模拟出对于Real-time PCR检出33
