广西PRRSV分离株Y12018全基因组扩增和遗传进化分析
王 政,解长占,于成东,于 桐 ,李亭玉,李金凤,孟 媛 ,鲁会军,金宁一,张雪梅
(1.延边大学 农学院,吉林 延吉 133002;2.军事医学研究院 军事兽医研究所,吉林 长春 130122;3.吉林农业大学 动物科学技术学院,吉林 长春 130118;4.吉林大学 动物医学学院,吉林 长春 130062)
猪繁殖与呼吸系统综合征(porcine reproductive and respiratory syndrome,PRRS)是由猪繁殖与呼吸综合征病毒(porcine reproductive and respiratory syndrome virus,PRRSV)引起的一种以受孕母猪繁殖障碍和仔猪呼吸综合征为主要特征的高度接触性传染病。不同性别、年龄阶段和品种的猪均可被感染,感染妊娠母猪出现流产、死胎、弱胎和木乃伊胎等情况,流产率达30%,1月龄以内的仔猪相对较易被感染,感染仔猪伴有呼吸困难、发育迟缓和败血症等现象[1-2]。自1987年首次报道以来,全球除仅有少数国家如新西兰、瑞士及澳大利亚无疫情报道外,在其他地区和国家仍然呈地方流行性趋势[3]。我国PRRSV流行至今大致分为以下3个阶段:美洲型经典PRRSV流行期;高致病性PRRSV流行期;多种PRRSV流行的稳定控制期。当前PRRS依然给我国及世界的养猪业带来巨大的经济损失,对养猪业的影响和危害依然存在。
在1996年国际病毒分类委员会的报告中,将PRRSV归属至动脉炎病毒科(Arteriviridae),动脉炎病毒属(Arterivirus),该属病毒共同存在的特点即发生变异和重组的频率较高,并且具有巨噬细胞内复制的能力,能够诱发自然宿主持续性感染[4]。PRRSV为不分节段的单股正链RNA病毒,全长约15 kb,其开放性阅读框(ORF)依次命名为ORF1a(ORF1a-TF)-ORF1b-ORF2a-ORF2b-ORF3-OR-F4-ORF5a-ORF5a-ORF6-ORF7,5′端有帽子结构,3′端有UTR和Poly(A)尾巴。其中ORF1a和ORF1b主要编码与病毒复制和转录相关的酶,ORF2~5 分别编码 GP2、GP3、GP4、GP5 蛋白,ORF6编码N端非糖基化的膜蛋白M,ORF7编码病毒核衣壳蛋白N[5-7]。RNA病毒基因组因存在点突变、缺失和插入等变异方式具有很高的突变频率,PRRSV的各不同分离株间的核苷酸序列存在广泛变异且不同基因类型的变异速率各不相同,变异速率达每年10-2/位点,高于一般RNA病毒的每年突变速率(10-3~10-5/位点)[8]。
2001年美国出现的PRRSV变异株MN184A、MN184B和MN184基因组均存在 Nsp2氨基酸的131个不连续缺失,对应经典毒株基因组位置分别为324~434,486和505~523,美国国家动物疾病中心(NADC)于2008年首次分离具有相同缺失模式的毒株并将其命名为NADC30[9-11]。我国关于NADC30的相关报道较少,自2012年起我国陆续出现与美国NADC30毒株遗传进化关系较为相近的毒株,并将其统一命名为类NADC30毒株(NADC30-like)。我国NADC30-like毒株的出现很可能是由美国输入的NADC30毒株与我国境内的主要流行毒株(VR-2332、HP-PRRSV和疫苗毒株等)发生重组并经过进化演变而来。流行病学调查显示,自2014年起,北京、天津、河北、吉林、黑龙江、广西、江苏、浙江、四川和福建等我国大部分地区的类NADC30毒株临床检出率相对较高且呈上升趋势,疫情已涉及我国大部分省市,NADC30-like很可能已经成为我国境内的主要流行毒株。NADC30-like毒株的出现增加了PRRSV防治难度和临床症状的复杂性,对于该类毒株的监测和全基因组特征分析对流行预警和疾病防控具有重要战略意义。
1 材料与方法
1.1 毒株、菌株及细胞从广西壮族自治区某发病猪场分离疑似PRRS阳性样品;Trans1-T1感受态和Marc-145细胞由本实验室保存。
1.2 主要试剂Random Primer(9mer)、dNTP Mixture、DL2000核酸Marker等购自TaKaRa公司;TransStart Fastpfu Fly DNA Polymerase、5×TransStart Fastpfu Fly Buffer等购自全式金公司;pClone 007 Blunt Vector购自TSINKE公司;M-MLV RT 5×Buffer、M-MLV Reverse transcriptase等均购自 Promega公司;UNIQ-10柱式Trizol总RNA提取试剂盒购自生工生物工程股份有限公司;胶回收试剂盒和质粒小提试剂盒购自Invitriogen公司。
1.3 引物合成根据GenBank数据库提供的PRRSV NADC30-like毒株的核苷酸序列,设计10对扩增PRRSV全长所需的引物(表1),并将引物送由吉林省库美生物科技有限公司合成。

表1 类NADC30毒株全基因组扩增引物序列
1.4 病毒总RNA的提取和反转录将PRRSV分离株接种于Marc-145细胞中,37℃吸附1 h,后弃去上清,加入DMEM(含2% FBS)后继续培养,出现明显CPE(达75%以上)后收集细胞培养物,反复冻融3次后,利用总RNA提取试剂盒按说明书提取病毒基因组RNA后常规方式反转录为cDNA,将其置于-20℃储存备用。
1.5 目的基因分段扩增将得到的cDNA作为模板,使用引物进行PCR扩增。PCR反应体系(25 μL):cDNA模板1 μL;上、下游引物各1 μL;Fly酶0.5 μL;5×Fly Buffer 5 μL;dNTP 2.5 μL;ddH2O 14 μL。PCR仪扩增程序:95℃预变性2 min;95℃变性20 s;退火20 s(温度均采取相对应引物的最适退火温度);72℃延伸90 s;40个循环结束后再72℃延伸10 min。PCR产物经1.2%琼脂糖凝胶电泳检测。
1.6 扩增产物的克隆及测序将大小正确的琼脂糖凝胶电泳产物利用试剂盒进行胶回收,具体操作步骤详见说明书,目的条带与pClone 007 Blunt Vector连接后将连接产物转化到大肠杆菌Trans1-T1感受态细胞中,然后筛选重组阳性质粒,接种于含有Amp+的LB液体培养基中,37℃震荡培养约12 h,最后小提回收重组质粒送至库美生物科技有限公司进行序列测定。
1.7 全基因序列拼接及分析利用DNAMAN 6.0将分段获得并成功测序的核苷酸序列进行拼接,然后将其进行校正后得到完整的全基因序列,然后采用DNAStar 7.1和MEGA 5.2等软件将全基因序列与GenBank数据库中下载的具有代表性的10株参考毒株的核苷酸序列及相对应编码氨基酸的序列进行同源性比对和分析,并用MEGA 7.0软件构建进化树。参考毒株相关信息详见表2。
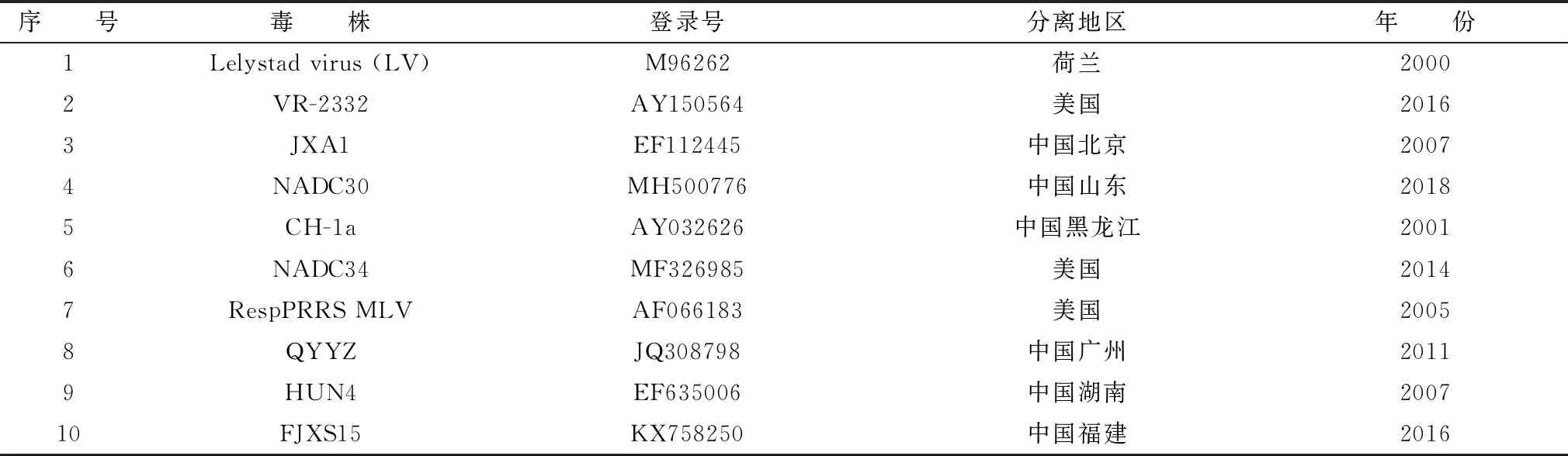
表2 参考毒株
2 结果
2.1 PRRSV Y12018株全基因扩增结果利用UNIQ-1-柱式Trizol总RNA提取试剂盒提取疑似样品的总RNA,并转录为cDNA,用10对引物进行PCR扩增,扩增出病毒基因组的各目的片段与预期一致(图1),将测序结果利用DNA Star 7.1 SeqMan 拼接为Y12018全基因组序列,拼接校对后的基因组全长为15 017 bp(除去polyA尾),包括长度为191 bp的5′UTR和145 bp 的3′UTR。
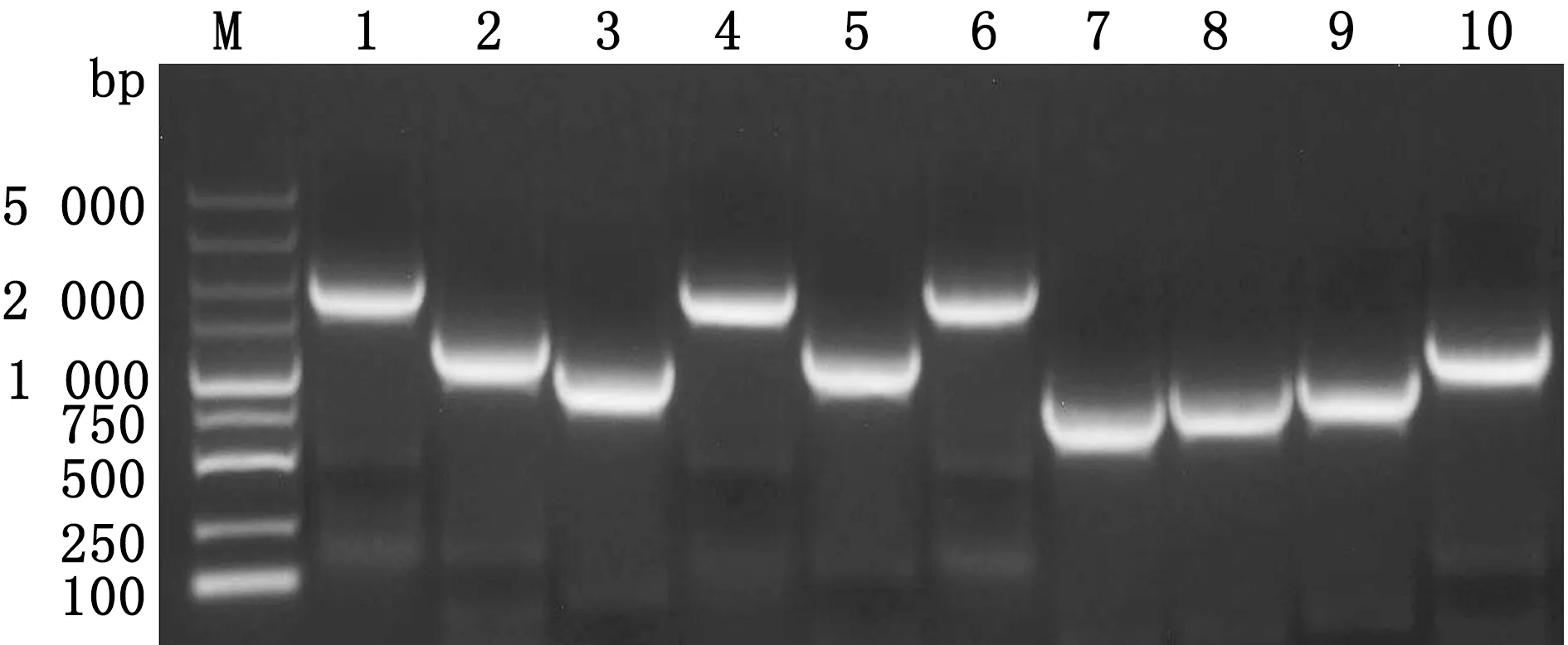
M.DL5000 DNA Marker;1~10.PRRSV-1F、PRRSV-2F、PRRSV-3F、PRRSV-4F、PRRSV-5F、PRRSV-6F、PRRSV-7F、PRRSV-8F、PRRSV-9F、PRRSV-10F
2.2 全基因组遗传进化分析及同源性对比利用MEGA 5.2和DNA Star 7.1将Y12018全基因组序列与GenBank上收录的国内外10株参考毒株基因组序列构建遗传进化树并进行核苷酸同源性分析(图2)。结果表明,PRRSV毒株分为2个基因型,以Lelystad virus (LV)为代表的欧洲型分支,美洲型毒株又可分为4个分支,以VR-2332和RespPRRS-MLV为代表的分支,以我国分离的CH-1a为代表的分支,以JXA1、HUN4和FJXS15为代表的HP-PRRSV-like分支,以NADC30和NADC34为代表的分支。遗传进化树结果显示Y12018与NADC30的遗传关系最为接近。Y12018分离株与 Lelystad virus (LV) 同源性最低为58.5%,与美国分离的NADC30毒株的同源性最高为94.4%,与美洲型VR-2332毒株同源性为82.5%。

图2 基于Y12018 全基因核苷酸序列的遗传进化分析
2.3 GP5基因进化树分析及同源性对比利用MEGA5.2构建进化树,系统进化树显示Y12018分离株与NADC30、NADC34和FJXS15属于同一分支,与NADC30亲缘关系最近(图3)。利用DNAStar 7.1软件中的MegAlign将分离株Y12018的GP5基因核苷酸序列与参考序列对比,结果显示Y12018分离株与Lelystad virus (LV) 同源性为59.5%,与NADC30、FJXS15和NADC34的同源性分别为92.9%,92.4%和85.8%,对比NADC30氨基酸序列参在31个位点的改变。
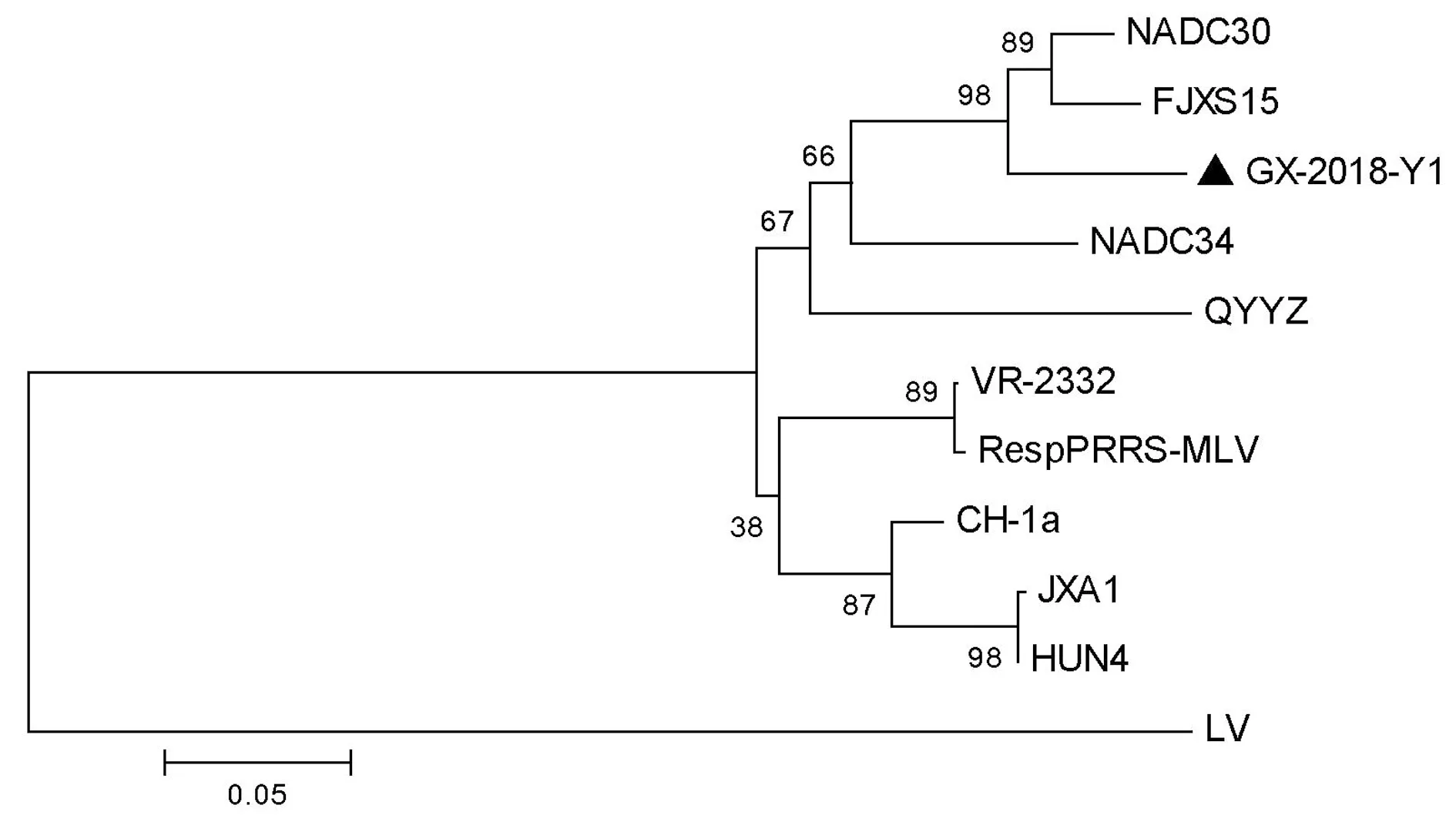
图3 基于Y12018 GP5基因核苷酸序列的遗传进化分析
2.4 Nsp2基因进化树分析及同源性对比利用MEGA 5.2构建进化树,系统进化树显示分离株Y12018与NADC30和NADC34属于同一分支,与NADC30亲缘关系最接近。利用DNAStar 7.1软件中的MegAlign将分离株Y12018的Nsp2基因的核苷酸序列与参考序列对比,结果显示Y12018分离株与Lelystad virus (LV) 同源性为50.0%,与NADC30和NADC34的同源性分别为93.5%和79.7%。Nsp2氨基酸序列对比结果显示:分离株Y12018的Nsp2基因对应氨基酸存在“111+1+19”的缺失模式,即分别对应VR-2332分别为第324~434、486和505~523位置,缺失情况与NADC30一致,表明分离株Y12018属于NADC30-like亚群(图4A~D);同时与参考序列NADC30对比得出分离株Y12018的Nsp2氨基酸序列存在2个氨基酸位点的缺失,对应VR-2332的位置为617与618。(图4E)。

图4 Nsp2基因的氨基酸序列比对分析(缺失部分用方框标出)
2.5 主要开放阅读框核苷酸同源性对比利用DNAStar 7.1软件中的MegAlign将分离株Y12018的各开放阅读框(5′UTR、ORF1a、1b、2~7和3′UTR)的核苷酸序列与GenBank数据库中收录的10株参考序列进行对比,各开放阅读框核苷酸同源性的对比结果见表3。

表3 Y12018与10株参考毒株不同开放阅读框核苷酸同源性的比较 %
3 讨论
自PRRSV在世界上首次被报道至今已30余年,PRRSV毒株不断发生进化具有多样性,无疑加大了该病的防治难度,给我国养猪业带来了巨大的经济损失。目前的田间流行毒株主要包括PRRSV野毒株、重组毒株、疫苗毒株和HP-PRRSV毒株等[12],类NADC30毒株的出现又成为新的流行毒株且具有易和其他毒株发生重组的特点,使得NADC30-like毒株在我国的流行呈现加剧趋势[1,13-14]。
GP5作为PRRSV重要的糖基化膜蛋白,大小为130~200aa,具有较大的C端结构域,存在6个B淋巴细胞表位和3个T淋巴细胞表位,可诱导中和性抗体的产生且其中和活性优于其他的结构蛋白所产生的中和性抗体[15-16]。该分离株Y12018与NADC30的GP5同源性为92.9%,氨基酸序列对比结果发现分离株Y12018存在31个位点的改变。Nsp2基因位于ORF1区域编码病毒主要的非结构蛋白,具有较好的免疫原性同时也存在高度变异等特点,可通过该区段的变异从而分析PRRSV的变异情况[17]。本研究中分离株Y12018与NADC30的Nsp2同源性为93.5%,氨基酸序列大部分缺失情况与NADC30一致,同时与NADC30相比缺失的2个氨基酸对应VR-2332为587~588位点。相关研究结果表明非结构蛋白Nsp2氨基酸的缺失可能与病毒毒力之间密切相关,因此基因差异与致病力之间存在的关系还有待进一步研究。
通过对分离株Y12018的全基因进行扩增,并将全基因组、GP5和Nsp2等基因核苷酸序列与参考毒株同源性对比分析,结果表明该分离株为NADC30-like毒株。本研究可为进一步探究广西地区PRRSV的遗传和进化关系提供理论依据,并为后续致病性研究奠定基础。

