基于适配体的光学和电化学法对食源性致病菌检测的研究进展
惠媛媛,彭海帅,王毕妮,张富新,刘玉芳,贾蓉,任荣
基于适配体的光学和电化学法对食源性致病菌检测的研究进展
惠媛媛,彭海帅,王毕妮,张富新,刘玉芳,贾蓉,任荣
陕西师范大学食品工程与营养科学学院,西安 710119
由于食品的多样性和食品基质的复杂性,食品安全问题已成为全社会共同关注的热点问题,其中微生物引起的食源性疾病居全球食品安全问题之首。食源性致病菌的检测是食源性疾病预防与控制的关键环节。平板计数法被评为微生物检测的金标准,但在致病菌检测过程中信号的放大需要通过单个细胞生长成菌落来实现,因此检测周期较长(3—7 d)。此外,现有的准确检测致病菌的技术有聚合酶链式反应(PCR)和酶联免疫吸附法(ELISA)等,但由于预处理时间长、操作复杂、检测结果存在假阳性等问题,不适合对致病菌进行现场快速有效的检测。核酸适配体是利用指数富集的配体系统进化技术(SELEX)从核酸分子文库中得到的寡核苷酸片段,具有良好的特异性、稳定性、易于修饰和亲和力高等特点,在毒素、抗生素、重金属和致病菌等其他小分子的检测中有很大的潜力。目前,国内外学者已开发了各种基于适配体的检测方法。本文综述了食品中常见的食源性致病菌及其传统的检测方法和近十年来用于致病菌检测的光学和电化学适配传感器,涵盖每种方法的检测策略、检测时间、检测范围和检测限等信息,也提出了适配体传感器在食品安全检测中存在的主要问题,并对其未来研究的发展趋势进行了展望,这些将为今后的研究工作提供一定的基础。
食源性致病菌;适配体;快速检测;食品安全
近20年来,随着食源性疾病发病率的显著增加,食源性疾病已成为世界性的重大公共卫生问题。美国疾病预防与控制中心(CDC)估计,美国每年由食源性致病菌造成大约7 600万例疾病,3 215万例住院治疗,5 200例死亡[1-2]。澳大利亚每年因食源性疾病带来的经济损失可达26亿澳元。食源性疾病的大量暴发可追溯到食用被致病菌污染的食品。致病菌又称病原微生物,据报道,已被确认的食源性致病菌多达31种,常见的有空肠弯曲杆菌、沙门氏菌、副溶血性弧菌、大肠埃希氏菌、金黄色葡萄球菌及单核增生李斯特菌等[3]。食源性致病菌的检测对于防止其危害具有重要意义,其中用于致病菌检测的生物识别元件有抗体、噬菌体、抗菌肽和适配体等。
近年来,国内外学者构建了各种各样基于适配体的生物传感器。本文对用于食源性致病菌检测的光学及电化学适配体传感器进行了全面的综述。首先,对引起食源性疾病的主要致病菌进行了介绍并简述了传统的致病菌检测方法。然后,根据近10年的学术文献,总结了基于适配体的电化学和光学方法在食源性致病菌检测中的研究现状,其中涵盖每种方法的检测策略。分析每种方法的检测范围、检测限及检测时间等,并在此基础上,讨论基于适配体的方法在致病菌检测中所存在的问题和挑战及对未来的研究展望。
1 常见的食源性致病菌
1.1 空肠弯曲杆菌(Campylobacter jejuni)
空肠弯曲杆菌是一种螺旋形、有鞭毛、微需氧的革兰氏阴性菌,广泛散布在各种动物体内,其中以家禽、野禽和家畜带菌最多[4]。该菌在水、牛奶中4℃下能存活3—4周,在鸡粪中保持活力可达96 h,对酸碱也具有耐受力,易在胃肠道中生存[5]。空肠弯曲杆菌可引起人患各种疾病,如急性肠胃炎,在急性肠胃炎病例中,弯曲杆菌病可能导致严重的自身免疫性后遗症,例如反应性关节炎,此外,每1 000例病例中约有1例为格林-巴利综合征(GBS)[6-9]。
1.2 沙门氏菌(Salmonella typhimurium)
沙门氏菌是一种杆状、有鞭毛、需氧或兼性厌氧的革兰氏阴性杆菌,是一种经常在鸡肠道内发现的细菌。常见的肉类、奶蛋类食品和蔬菜都会被沙门氏菌污染,在世界各地的食物中毒中,沙门氏菌引起的中毒病例是仅次于弯曲杆菌的第二大致病菌。在美国,沙门氏菌每年导致120万人患病,2.3万人住院,450人死亡[10],直接经济损失超过24亿美元。在中国,每年由沙门氏菌造成的食物中毒事件占全部食物中毒事件的40%—60%[11]。消费者食用被沙门氏菌污染的食物12—72 h后即可出现腹泻、腹痛、恶心、呕吐、头痛、发热、便血等症状,重症时出现打寒战、惊厥、抽搐和昏迷,严重可致人死亡[12]。
1.3 大肠杆菌(Escherichia coli,E. Coli)
大肠杆菌(又名大肠埃希氏菌)在自然界分布广泛,大多数不致病,主要附生在人或动物的肠道里,为正常菌群,少数的大肠杆菌具有毒性,可引起严重腹泻和败血症等疾病[13-14],尤其对婴儿、幼畜、幼禽。目前,引起人类感染性腹泻的大肠杆菌又可被分为5类,包括致病性大肠埃希氏菌(EPEC)、肠产毒性大肠埃希氏菌(ETEC)、肠道侵袭性大肠埃希氏菌(EIEC)、肠黏附性大肠杆菌(EAEC)及肠出血性大肠埃希氏菌(EHEC)[15]。肠出血性大肠杆菌肠炎是由EHEC感染引起的感染性腹泻,为人兽共患性疾病,临床上可见出血性结肠炎、溶血性尿毒综合征、血栓性血小板减少性紫癜等[16-18]。经证实,EHEC O157:H7是导致该病流行的主要致病菌株。
1.4 金黄色葡萄球菌(Staphylococcus aureus,S. aureus)
金黄色葡萄球菌是一种球形、无芽孢、荚膜及鞭毛、需氧或兼性厌氧的革兰氏阳性菌,具有耐盐性,在含10%—15%的氯化钠环境中仍可生长。金黄色葡萄球菌在自然界广泛存在,可通过直接接触、空气传播等多种途径对食品造成污染,而且该菌可产生耐热性的肠毒素,在食品加工过程中不容易被灭活[19],因此,金黄色葡萄球菌是引起细菌性食物中毒的重要食源性致病菌之一。美国疾控中心报告显示,该菌引起的食物中毒数量占细菌性食物中毒数量的1/3,仅次于大肠杆菌。在我国,由金黄色葡萄球菌引起的食物中毒(food poisoning,)病例约占食物中毒病例的20%。人感染金黄色葡萄球菌后,可引发恶心、反复呕吐、腹痛、腹泻等急性中毒症状,严重时可引起局部化脓感染,如肺炎、盆腔炎、心包炎,甚至败血症、脓毒症等全身性感染[20-22]。
1.5 单核细胞增生李斯特菌(Listeria monocytogenes,LM)
单核细胞增生李斯特氏菌简称单增李斯特氏菌,是一种直或稍弯,两端钝圆,常呈“V”字型排列,偶有球状、双球状,兼性厌氧、无芽孢,一般不形成荚膜,但在营养丰富的环境中可形成荚膜的革兰氏阳性菌[23-24]。能够适应酸性食品、高盐食品、低温(2—4℃)等多种条件,能够在食物接触表面增殖(如不锈钢),同时能抵抗高浓度的抗菌素和防腐剂[25-26],且与其他致病菌相比,能在寒冷环境中繁殖,并能迅速污染食品(如牛奶、冷冻的家禽产品、熏鱼、蔬菜、海鲜、冰淇淋、沙拉等)[19,27]。WHO将列为20世纪90年代食品中四大致病菌之一,它可导致李斯特菌病,该病是一种罕见的具有高发病率、住院率(94%)和死亡率的传染病,尤其是在老年人、免疫功能低下者、孕妇和婴儿中[28-31]。
2 食源性致病菌的传统检测方法
目前,虽然已有先进的安全控制技术可以在很大程度上降低食品中的致病菌(如良好农业规范(GAP)、食品良好生产规范(GMP)及危害分析和关键控制点(HACCP)),但无法完全消除,因此致病菌的检测仍然非常重要,它是预防和减少食品安全事件发生的关键。传统的致病菌检测和鉴定方法包括微生物培养法、免疫学检测法、分子生物学法等(表1)。
2.1 微生物培养法
微生物培养法被评为最经典的食源性致病菌检测技术之一,包括増菌、选择性增菌、镜检以及血清学验证等一系列步骤。不同的选择性培养基被用来检测特定的细菌,选择性培养基可以包含抑制剂(为了阻止或延迟非目标菌株的生长)或特定的基质,只有目标菌能够降解或赋予生长菌落一种特定的颜色(沙门氏菌检测的彩虹琼脂),然后用光学方法进行检测,主要是通过目测。由于培养法检测限低、可靠性高、操作简单,被认为是微生物检测的金标准,但在检测过程中,信号的放大需要通过单个细胞生长成菌落来实现,因此检测周期较长(如:对可疑的LM的生化鉴定一般需要7 d才能得到结果)[32-34]。此外,环境中可存活的菌株可以进入休眠状态,变为不可培养(活的但不可培养的(VBNC)),这可能导致对病原体数量的低估或无法从污染样本中分离出病原体[35-36]。
2.2 基于PCR的检测方法
聚合酶链式反应技术(polymerase chain reaction,PCR)是20世纪80年代发展起来的一种在体外快速扩增特定核酸片段的分子生物学技术。PCR技术依据生物体内DNA半保留复制的原理,由模板DNA、人工合成的寡核苷酸引物、4种dNTP及缓冲液组成的反应混合物在热稳定的DNA聚合酶催化下,经过变形、退火、延伸等循环步骤,快速扩增目的核酸片段,其被广泛应用于分类鉴别、转基因检测、食品过敏原检测及食源性病原微生物等领域[37]。目前,应用于检测的PCR方法有实时荧光PCR、多重PCR和逆转录-聚合酶链反应(RT-PCR)等,PCR技术除可单独进行检测外,还可与分子信标技术、表面等离子体共振(SPR)传感技术等结合应用于细菌检测中。在PCR检测中,随着DNA模板扩增产物的指数增长,病原体的靶段可以放大几百万倍,大大提高了检测灵敏度,同时快速PCR技术的发展可将检测时间缩短至20 min。然而PCR技术不能区分活的和死的病原体,因为DNA同时存在于活菌和死菌中[38]。此外,食品样品中可能存在一些抑制化合物,如腐植酸,可能影响PCR扩增,产生假阴性结果。

表1 传统食源性病原体检测方法的基本原理、优势和局限性
2.3 免疫学检测法
免疫学检测方法也已被成功地用于食源性致病菌的检测与鉴定,它们是基于抗体与特定抗原通过一个或多个表位的亲和反应。在致病菌的免疫学检测方法中(如:乳胶凝聚法、免疫扩散法、酶联免疫吸附法(ELISA)、免疫磁珠法及免疫沉淀法),ELISA是最常用、最成熟的技术[39],酶联免疫吸附试验所用酶不同,但一般为碱性磷酸酶、辣根过氧化物酶(HRP)和半乳糖苷酶。例如,检测单核增生李斯特菌、大肠杆菌和空肠弯曲杆菌是通过辣根过氧化物酶标记这些病原体的抗体。与传统的培养技术相比,免疫学检测时间非常短,但基于抗体的检测仍然缺乏实时检测微生物的能力。此外,免疫法检测中抗体的制备比较繁琐,且抗体的获得具有局限性,对于那些太小而没有结合位点的分子或者免疫原性差、毒性高的分子,抗体的获得难以实现,且抗体对被测病原体的亲和力低,检测常有局限性[40-41]。
3 基于适配体的生物传感器在食源性致病菌检测中的应用
近年来,适配体在食源性致病菌中的应用越来越多,研究也越来越深入。国内王周平课题组一直致力于食源性致病菌适配体的筛选与应用研究,利用Cell-SELEX技术,先后得到了阪崎肠杆菌、副溶血性弧菌、鼠伤寒沙门氏菌和金黄色葡萄球菌等食源性致病菌的适配体序列。表2为文献中报道的食源性致病菌的核酸适配体序列。适配体作为一种新型仿生识别分子,受到国内外学者的青睐,被誉为“第四代抗体”,但相较于抗体,适配体具有更明显的优势,如:(1)容易合成。适配体在体外可以通过化学方法大量合成,但抗体需要通过动物或细胞培养来合成,通常耗时、耗力;(2)修饰方便。适配体可以依照不同的功能对其加以化学修饰,如巯基化修饰、荧光标记、生物素标记及酶标记等。适配体经过适当修饰后可提高其化学稳定性,而且不影响与靶标之间的亲和力;(3)稳定性好。适配体由具有抗变性的核苷酸组成,可承受较大范围的pH和温度,因此可保存较长时间,而抗体多为易变性的蛋白质;(4)应用灵活。适配体的尺寸比传统抗体小,分子量适中,可以与多种靶标进行结合,如金属离子、真菌毒素、抗生素、蛋白质、病毒、癌细胞等。鉴于适配体的各种优点,国内外学者已将开发了各种基于适配体的生物传感器对食源性致病菌进行检测,包括电化学适配体感器以及各种光学适配体传感器(表3为用于食源性致病菌检测的各种传感器的性能比较)。
3.1 电化学适配体传感器
电化学适配体传感器是近几年来适配体传感器中发展最为快速广泛的分支之一,具有选择性好、灵敏度高、操作简便、成本低廉等优点,被广泛应用于病原体的识别与定量。为了将适配体与靶标之间的相互作用转化为可测量的电化学信号,人们开发了不同的电化学方法,包括阻抗法、伏安法、电位法和电导法,同时为了增强传感器的灵敏度,各种信号放大策略被应用到传感器的构建中,如酶催化信号放大、目标物循环放大、纳米材料信号放大、DNA等温扩增放大(最典型的为滚环扩增(rolling circle amplification,RCA)),其中纳米材料信号放大应用最为广泛,各种纳米材料(如金属纳米颗粒、碳纳米材料、二氧化硅纳米颗粒等)都可以被应用到电化学适配体传感器的构建中。
碳纳米材料包括富勒烯、碳纳米管(CNTs)、石墨烯(GR)、还原氧化石墨烯(rGO)和碳量子点(CQDs)等。rGO是一种新型二维碳材料,它不仅具有石墨烯的片状结构而使其比表面积增大,而且较石墨烯而言,羟基、羰基、羧基等含氧官能团的减少提高了电子传递速率,促进其他金属颗粒附着在表面。MUNIANDY等[62]基于还原氧化石墨烯二氧化钛(rGO-TiO2)纳米复合材料构建了一种用于检测沙门氏菌的方法。通过静电相互作用,将适配体固定在rGO-TiO2纳米复合材料上,沙门氏菌与适配体特异性结合后,形成物理屏障,抑制了电子传递。DPV信号随着沙门氏菌浓度的增加而降低,在最佳条件下,该方法的检测范围为101—108cfu·mL-1,检测限为10 cfu·mL-1,检测时间较短(1 h,包括食物样品制备和电化学分析)。该方法与微生物培养法相比,检测结果准确;同时,适配体上的磷酸盐基团与还原氧化石墨烯表面的TiO2基团相互作用,不需要对适配体进行标记,降低了检测成本。基于同样的原理,APPATURI[63]在玻碳电极表面修饰还原氧化石墨烯碳纳米管(rGO-CNT)纳米复合材料,氨基化的适配体通过共价偶联固定在电极表面。在最佳试验条件下,该方法的检测范围为101—108cfu·mL-1,检测限为10 cfu·mL-1,检测时间为10 min。笔者课题组[12]基于还原氧化石墨烯甲苯胺蓝(Tb)(rGO-Tb)复合纳米材料及酶切循环信号放大策略构建了一种用于沙门氏菌检测的方法。通过金硫键将巯基化的互补链1固定在金电极表面,沙门氏菌适配体与互补链1通过碱基互补配对修饰在电极表面,利用I信号放大效应和适配体对沙门氏菌的特异性识别,循环带离适配体,再通过互补链2与互补链1的杂交将互补链2-AuNPs-Tb-rGO负载到电极表面,通过监测电极表面的电化学信号完成对沙门氏菌的检测。该方法的检测范围为6×10-2—6×106cfu·mL-1,检测限为200 cfu·mL-1,检测时间为1 h。
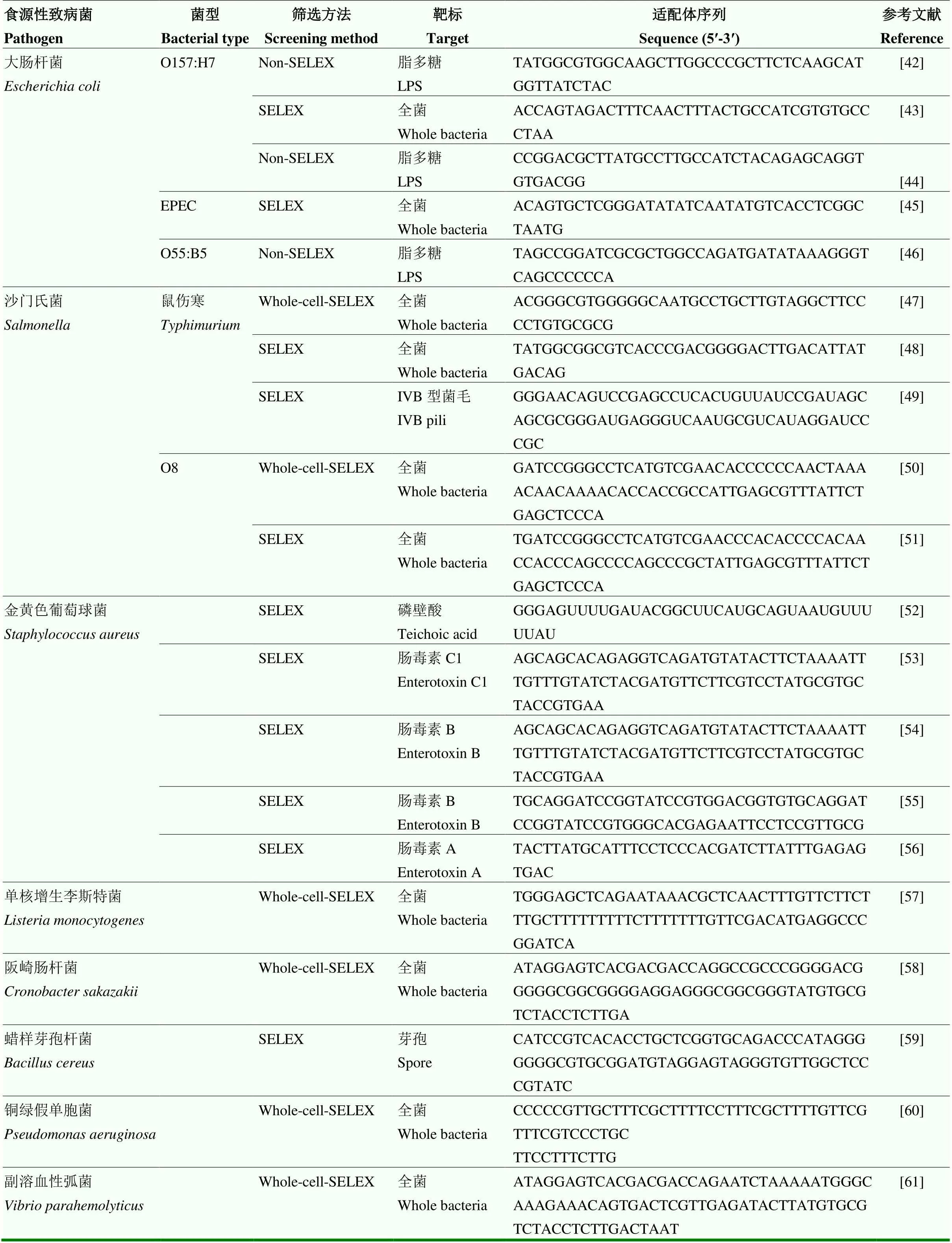
表2 食源性致病菌的适配体序列
最近,三维形状的有机-无机杂化花状纳米复合材料引起了广泛的关注,花状纳米复合材料,具有大的比表面积,良好的生物相容性,优异的电子传导能力,能提供更多的活性位点,保证更多纳米材料的负载,并且纳米材料可以吸附到花瓣间隙中,保持纳米材料的稳定性。BU等[64]以MB@MI花状纳米复合材料作为信号标记,构建了一种用于检测大肠杆菌O157:H7的传感器,将巯基化的适配体通过金硫键固定在电极表面,靶标与适配体特异性结合后再加入MB@MI有机无机纳米复合材料(MI是一种新型的大肠杆菌O157:H7生物识别抗菌肽),此时形成一个“三明治夹心”复合结构。该方法的检测范围为102—107cfu·mL-1,检测限为32 cfu·mL-1。
导电聚合物(CPs)是由聚合物链和与链非键合的一价对阴离子(p-型掺杂)或对阳离子(n-型掺杂)组成的一类聚合物材料,它不仅具有聚合物轻质、柔性和可加工的特点,还具有金属和半导体的导电特性。聚吡咯是一种常见的CPs,具有易合成、稳定性高及导电性好等独特性能。SHEIKHZADEH等[65]使用苯胺-吡咯共聚物检测沙门氏菌,在金电极表面滴涂苯胺-吡咯共聚物膜,氨基化的适配体通过共价偶联固定在共聚物膜上,当目标菌存在时,与适配体形成靶向配合物,在LiClO4溶液中记录电化学信号,该方法的检测范围为102—108cfu·mL-1,检测限为3 cfu·mL-1。
特殊的纳米颗粒也适用于无标签检测,WANG等[66]提出了一种基于磁性纳米颗粒同轴毛细管的阻抗型电化学适配体传感器检测大肠杆菌O157:H7。使用抗体修饰的磁性纳米颗粒分离目标菌,适配体和脲酶修饰到金纳米颗粒上,注射到毛细管中与目标菌发生反应,形成磁性纳米颗粒-抗体-目标菌-适配体-脲酶复合物,脲酶可水解尿素生成铵,脲酶催化尿素的水解反应降低了电极的阻抗,阻抗的变化与目标菌的浓度有关。该传感器可用于10 mL大容量目标菌的检测,检测范围为101—104cfu·mL-1,检测限为10 cfu·mL-1,但该方法需3 h才能完成靶标的检测。
RCA是一种在恒温下发生的核酸扩增技术。其体系包括单链环状DNA模板、一个能与环状DNA模板结合的引物及DNA聚合酶。在扩增反应中,引物先与单链环状DNA结合,当有环状DNA模板和聚合酶时,在DNA聚合酶作用下,以环状DNA为模板合成其互补链,当合成至引物结合位点时,DNA聚合酶的链置换活性把已合成的引物延伸链置换下来,继续以环状DNA为模板进行合成,形成滚环扩增。该技术不需要特殊的实验仪器,这使得它比其他信号放大方法更经济、更简便。TENG等[67]基于RCA信号放大策略构建了一种用于副溶血性弧菌检测的适配体传感器。抗体被固定在金电极表面捕获靶标,适配体再与靶标结合形成“三明治夹心”结构,该结构可进行RCA信号增强,RCA产物与AuNPs-ssDNA杂交作为检测探针,互补的sDNA探针与检测探针杂交形成大量的dsDNA结构,以亚甲基蓝(MB)为指示剂,实现电化学信号的增强。该方法对副溶血性弧菌的检测范围为2.2—2.2×108cfu·mL-1,检测限为2 cfu·mL-1。GE等[85]也使用RCA信号放大策略开发了一种用于沙门氏菌检测的方法,该方法的检测范围为20—2×108cfu·mL-1,检测限为16 cfu·mL-1。
3.2 比色适配体传感器
比色法主要以底物反应前后颜色变化为参照,利用肉眼对比颜色深浅或紫外分光光度计来定性或定量,无需复杂的检测设备,且检测成本低、速度快、操作简便。比色适配体传感器按照显色方法的不同分为3种:胶体金法、酶催化型以及无酶催化型。
3.2.1 基于胶体金的比色适配体传感器 胶体金(AuNPs)在正常情况下呈酒红色,当体系中加入适配体时,适配体可以与AuNPs结合,此时AuNPs在高浓度的盐溶液中不会发生聚集,颜色不发生改变。当待测液中有靶标时,靶标与适配体特异性结合,将适配体从AuNPs表面带离,此时高浓度的盐会引起AuNPs聚集,颜色从酒红色变为蓝色。基于此原理,WU等[68]报道了一种检测大肠杆菌O157:H7的方法,在靶标存在时,适配体与靶标结合,加入高盐溶液后,AuNPs会聚集,导致颜色从酒红色变为蓝色,其检测限为105cfu·mL-1。基于同样的策略,MA等[69]报道了一种简单、快速、方便的检测沙门氏菌的方法。该方法的线性检测范围为102—107cfu·mL-1,检测限为56 cfu·mL-1。KIM等[70]也报道了一个类似的方法用于现场检测鸡胴体样品中的空肠弯曲杆菌和大肠弯曲杆菌,该方法的准确度优于传统琼脂培养法(p=0.016),检测时间仅需30 min,节省了时间、人力和成本,同时,这也是首次利用比色传感器对自然污染样品中的食源性病原体进行检测的验证研究。
3.2.2 酶催化型比色适配体传感器 辣根过氧化物酶(HRP)具有较高的底物特异性和催化效率,可以催化3,30,5,50-四甲基联苯胺(TMB)和2,2-联氮-二(3-乙基-苯并噻唑-6-磺酸)二铵盐(ABTS)等底物的氧化,产生比色信号。G-四联体是DNA的二级结构,可以和氯化血红素(Hemin)结合,形成具有过氧化氢酶活性的DNA模拟酶。Sun等[71]基于DNA模拟酶构建了检测副溶血弧菌的方法,适配体通过生物素亲和素固定在磁珠上,适配体的互补链与其进行碱基互补配对,当待测液中存在靶标时,靶标与适配体特异性结合,适配体的二级结构发生改变,导致适配体的互补链与磁珠分离,靶标浓度越大,进入上清液的G-四联体互补序列就越多,G-四联体与Hemin结合,形成的G4-DNAzyme可以催化H2O2将无色的TMB氧化成蓝色的氧化TMB,进而通过颜色和吸光度的变化实现对靶标的检测,在最佳条件下,检测范围为102—107cfu·mL-1,检测限为10 cfu·mL-1。
3.2.3 无酶催化型比色适配体传感器 纳米材料不仅具有光、电、磁学特性,还具有天然酶(如过氧化物酶、过氧化氢酶)的催化活性,目前,已合成了多种金属氧化物纳米材料作为过氧化物模拟酶,包括CeO2NPs、Co3O4NPs、ZnFe2O4NPs、Au@Pd和Au@ Pt等。与天然酶相比,纳米材料模拟酶具有表面积大、稳定性高及催化可控等优点。WU等[72]开发了一种基于ZnFe2O4还原氧化石墨烯(ZnFe2O4/rGO)纳米颗粒的比色传感器用于检测沙门氏菌。当待测物中含有靶标时,适配体-ZnFe2O4/rGO复合物与靶标特异性结合,ZnFe2O4/rGO可催H2O2氧化TMB,生成典型的蓝色产物。该方法的检测范围为11—1.1×105cfu·mL-1,检测限为11 cfu·mL-1。DEHGHANI等[73]使用具有过氧化物酶活性的Au@Pd纳米材料构建了用于检测空肠弯曲杆菌的方法。当待测物中含有靶标时,适配体与靶标特异性结合,然后将溶液离心,将含游离适配体的上清液加入到Au@pd中,游离适配体与纳米颗粒表面的相互作用抑制过氧化物酶活性,降低TMB氧化。该方法的检测范围为102—106cfu·mL-1,检测限为10 cfu·mL-1。
3.3 荧光适配体传感器
荧光是被激发的分子、染料或纳米材料在回到基态时发出的光,基于适配体的荧光检测主要分为两种,即非标记型核酸适配体荧光检测法和标记型核酸适配体荧光检测法。
3.3.1 标记型荧光适配体检测法 标记荧光检测法的构建原理一般有两种,一种是捕获探针与靶标结合后,导致荧光标记物所处的微环境发生变化,引起相应的荧光分子发射性质的变化,例如荧光强度、荧光偏振等。另一种则是捕获探针与靶标结合后,使得核酸上面标记的荧光物质同另一种荧光物质或者猝灭物质靠近或远离,从而导致其发生电子、能量转移的发生或关闭,引起荧光强度的改变。基于第一种原理,GUO等[74]以1-(4-羟基苯基)-1,2,2-三苯乙基醚(TPE-OH)为材料,将其包封于牛血清白蛋白(BSA)中形成TPE-OH@BSA纳米颗粒,兔IgG抗体被偶联到TPE-OH@BSA纳米颗粒表面,适配体偶联磁珠捕获靶标,抗体-TPE-OH@BSA复合物进一步识别靶标,形成一种“三明治夹心”结构,由于TPE-OH能在牛血清白蛋白(BSA)微球中聚集,产生明亮的荧光信号,通过检测上清液中的荧光强度实现对靶标的检测。该方法对单核增生李斯特菌的检测范围为10—106cfu·mL-1,检测限为10 cfu·mL-1。在第二种原理中,荧光淬灭基团如BHQ(Black hole quencher),其猝灭效率高,但修饰成本较高。研究发现,金纳米球、碳纳米管、石墨烯、MoS2等纳米材料可通过共振能量转移和光诱导电子转移(PET)机理猝灭标记在适配体上的荧光基团。基于MoS2纳米材料对荧光的猝灭,SINGH等[75]构建了一种检测沙门氏菌的方法,使用荧光素(FAM)标记适配体(Apt-FAM),当待测液中存在靶标时,适配体与靶标特异性结合,荧光存在;当待测液中无靶标时,MoS2-Ns可与Apt-FAM结合,猝灭其荧光,通过前后荧光强度的改变实现对靶标的检测。该方法的检测范围为105—107cfu·mL-1,检测限为10 cfu·mL-1。基于羰基铁粉(CIP)多壁碳纳米管(MWCN)(CIP@MWCNT)纳米复合材构对荧光的猝灭,YANG等[76]构建了用于检测大肠杆菌O157:H7的方法,FAM标记的适配体可通过Π-Π堆积修饰在CIP@MWCNT上,CIP@MWCNT可使FAM标记的适配体发生荧光猝灭,当适配体由于靶标的存在而脱离CIP@MWCNT纳米复合材料表面时,荧光恢复。该方法的检测范围为7.15—103cfu·mL-1,检测限为3.15×102cfu·mL-1,检测时间为1 h。以纳米材料为猝灭剂可大大降低使用成本,但是选择性差,难以排除非特异性吸附的干扰。
3.3.2 非标记型荧光适配体检测法 由于对适配体进行荧光标记成本较高,且使用荧光标记可能对靶标与适配体之间亲和性产生一定影响,因此,非荧光标记的适配体传感器得以发展。非标记的适配体荧光检测法常需要一些特殊性质的荧光物质,如溴化乙锭(EG)、孔雀石绿(MG)、噻唑橙色(TO)、SYBR Green I、SYBR Gold、AccuBlue™等染料,这些物质在游离或者是与单链DNA共存时只显示微弱的荧光信号,与双链DNA共存时其荧光信号显著增强,即当靶标存在时,靶标与适配体的结合会改变检测体系的荧光信号,通过检测荧光信号的变化能间接完成对靶标的检测。基于AccuBlue™染料,DUAN等[77]构建了一种检测沙门氏菌的荧光传感器,该传感器具有信号开启和信号关闭两种模式,模式一:在靶标存在时,靶标与适配体特异性结合,随后加入适配体的互补DNA(cDNA)和AccuBlue™染料,cDNA与游离的适配体杂交形成双链DNA(dsDNA),AccuBlue™插入到dsDNA中,荧光强度显著增强。模式二:适配体与cDNA杂交形成dsDNA,加入AccuBlue™染料,产生强的荧光。当靶标存在时,靶标与适配体特异性结合,cDNA与dsDNA双链分离,AccuBlue™染料被释放,荧光强度降低。该方法对靶标的检测范围为50—106cfu·mL-1,检测限为25 cfu·mL-1。SRINIVASAN等[78]报道了两种检测沙门氏菌的方法。方法一基于SYBR Green I(SG)染料,SG在溶液中的荧光强度很弱,但其可插入到适配体(双链DNA)中,导致荧光信号大大增强,当待测液中存在靶标时,其与适配体特异性结合后释放出SG,荧光强度线性下降,该策略对于靶标的最低检测限为733 cfu·mL-1。方法二基于自带荧光的罗丹明B(RB)染料,RB可通过静电作用吸附到带负电的金纳米粒子(AuNPs)的表面,分散的AuNPs具有良好的生物相容性并能够通过与RB间的荧光共振能力转移以及分子间碰撞使得其荧光被淬灭。适配体通过金硫键连接在AuNPs表面可保护AuNPs免受盐诱导的聚集,从而在AuNPs存在时RB荧光被猝灭,当待测液中存在靶标时,靶标与适配体特异性结合,AuNPs的稳定性发生改变,盐诱导AuNPs聚集,导致因荧光共振转移而猝灭的RB荧光发生恢复,RB的恢复程度与靶标的浓度呈现良好的线性关系,该方法对沙门氏菌的检测范围为1 530—96 938 cfu·mL-1,检出限为464 cfu·mL-1。
3.4 化学发光适配体传感器
化学发光(Chemiluminescence,CL)也是一种可靠的检测方法,具有线性范围宽、仪器简单、选择性好等优点。HAO等[79]基于滚环扩增的化学发光适配体传感器,将Fe3O4磁性纳米颗粒(MNPs)修饰的适配体作为捕获探针,将Co2+增强的N(4-氨基丁基)-N-(乙基异构体)(ABEI)功能性花状金纳米颗粒(AuNFs)和互补链(cDNA)复合物(Co2+/ABEI- AuNFs-cDNA)作为信号探针,当待测液中存在靶标时,靶标与适配体的特异性结合以及适配体和cDNA碱基互补配对可以形成捕获探针-靶标-信号探针“三明治夹心”结构,通过测定体系的化学发光强度可以实现对沙门氏菌的检测。在最佳条件下,该方法的检测范围为32—3.2×106cfu·mL-1,检测限为10 cfu·mL-1,磁性纳米颗粒的使用有利于靶标的快速分离和纯化,从而减少了总分析时间。该课题组利用同样的技术原理对金黄色葡萄球菌进行检测,检测范围为50—1.5×105cfu·mL-1,检测限为15 cfu·mL-1[80]。KHANG等[86]基于鸟嘌呤化学发光建立了一种检测大肠杆菌O157:H7的方法。当待测液中存在大肠杆菌O157:H7时,6-羧基荧光素(6-FAM)修饰的适配体与大肠杆菌O157:H7特异性结合,在2S-(1-四氢嘧啶-2-酮)-3-甲基丁酸(TPA)存在时,加入鸟嘌呤化学发光试剂(如3,4,5-三甲氧基苯基乙二醛水合物、四丙基氢氧化铵)后,大肠杆菌O157:H7-适配体复合物发出强烈的光。随着大肠杆菌O157:H7浓度的增加,光强度成比例增强。该方法检测范围为104—107cfu·mL-1,检出限低至4.5×103cfu·mL-1。相比于传统的EIAs,该方法检测速度快,且无需多次长时间孵育和洗涤等耗时步骤。
3.5 表面增强拉曼散射传感器
表面增强拉曼散射技术(surface enhanced raman spectra,SERS)是将表面增强用于拉曼散射效应的一种高灵敏分析检测技术。该技术不仅能提供靶标的结构信息,而且可以实现单分子水平检测。基底在表面增强拉曼光谱分析中起重要作用,性质稳定、重现性好的基底是成功进行表面增强拉曼分析的重要前提。目前,各种形貌以及各种支撑材料如金银纳米棒、金银核壳材料、碳纳米管以及石墨烯-贵金属复合基底被用作SERS光谱的增强基底。ZHOU等[81]建立了一种金纳米棒增强基底的SERS适配体传感器。适配体修饰的磁性纳米颗粒作为捕获探针,适配体和罗丹明B同时修饰的金纳米棒作为信号探针,当待测液中存在大肠杆菌O157:H7时,信号探针与捕获探针上的适配体与靶标特异性结合,形成“三明治夹心”结构。该方法对大肠杆菌O157:H7的检测范围为101—104cfu·mL-1,检测限为3 cfu·mL-1(图5-A)。采用同样的原理,ZHANG等[82]使用金纳米颗粒作为增强基底对鼠伤寒沙门氏菌和金黄色葡萄球菌同时进行检测。适配体修饰的Fe3O4磁性金纳米颗粒(Fe3O4@GNPs)作为捕获探针,巯基苯甲酸和5,5-二硫代氧基(2-硝基苯甲酸)两种拉曼信号和适配体修饰的金纳米颗粒作为信号探针。该方法对鼠伤寒沙门氏菌和金黄色葡萄球菌的检测范围为102—107cfu·mL-1,检测限分别为15和35 cfu·mL-1,约需3 h能实现对靶标的定量检测。虽然金、银等金属纳米颗粒被广泛用作SERS增强基底,但金属与分子的直接接触会导致信号的变形,因此,金属纳米颗粒总是涂有保护层如石墨烯、二氧化硅、聚合物等材料形成核壳结构以提高基底的稳定性和再现性。DUAN等[83]用Au@Ag核壳纳米材料为增强基底对鼠伤寒沙门氏菌进行检测。适配体修饰的Au@Ag作为捕获探针,5-羧基-罗丹明(ROX)修饰的适配体作为信号探针。信号探针与捕获探针上的适配体与靶标特异性结合,形成“三明治夹心”结构。该方法对靶标具有良好的线性响应,其检测范围为15—1.5×105cfu·mL-1,检测限为15 cfu·mL-1。DUAN等[87]以聚二甲基硅氧烷(PDMS)膜为增强基底,适配体固定在Au-PDMS膜上作为捕获探针,用4-MBA、尼罗蓝A(NBA)标记的金纳米颗粒作为信号分子,信号分子通过静电作用吸附于膜表面,当待测液中存在靶标时,靶标与适配体特异性结合,形成“捕获基底-靶标-信号分子探针”夹心结构,通过测定拉曼强度从而实现对副溶血性弧菌和鼠伤寒沙门氏菌的同时检测。该方法对副溶血性弧菌和鼠伤寒沙门氏菌的检测限分别为18和27 cfu·mL-1。
3.6 表面等离子体共振传感器
基于表面等离子体共振(SPR)的适配体传感器在食源性致病菌的检测中得到了广泛的应用。首先,SPR是一种无标签的检测系统(不需要任何蛋白或荧光标记)。其次,它对目标物可进行直接和实时检测。第三,SPR提供了探索动力学研究的机会,这是许多其他检测系统所没有的。在表面等离子体共振(SPR)适配体传感器中,适配体常常被固定在金属表面(通常是金),当待测液中含有靶标时,靶标与适配体特异性结合,导致传感器表面离子共振装置的折射率发生位移,反射光会发生变化,通过测定光变化信号从而实现对靶标的检测。XU等[84]设计了一种新型的Ω形局域光纤探针表面等离子体共振(FOLSPR)传感器用来检测鼠伤寒沙门氏菌,检测范围为5.0×102—1.0×108cfu·mL-1。
4 结论与展望
本文综述了电化学适配体传感器和光学适配体传感器在食源性致病菌检测中的研究进展。适配体传感器具有操作简便、灵敏度高、特异性好等优点,为致病菌的现场快速检测提供了可能。但整体而言,仍然存在一些不完美的地方,如:(1)纳米材料的保存稳定性。在传感器的构建中,为了提高灵敏度,常使用各种纳米材料,但纳米材料的生物活性会随着温度、pH的不同而改变。另外,随着时间的推移,它们会因变性、降解或聚集而变得低效,这将极大地影响传感器的检测性能。(2)处理复杂食品基质的准确性。食品样品是一个复合基质体系,不同营养物质会对致病菌的检测产生干扰。另外,适配体与样品组分的非特异性结合可能导致假阳性结果。因此,在多数情况下,适配体传感器在检测前需要对样品进行预处理,多数情况下使用磁珠或磁性纳米颗粒,虽然免疫磁分离可用于从食物基质中分离浓缩致病菌,但延长了检测时间,此外,将磁选机与便携式设备整合在一起并不容易。(3)基于适配体的生物传感器在实验室条件下性能良好,但在相对恶劣的条件下,适配体很容易被破坏和降解,此外,适配体有时会非特异性地与食品中其他组分结合,导致结果错误或不精确。
因此,未来仍需在以下几个方面努力:(1)由于各国政府对大肠杆菌O157:H7、沙门氏菌和单核增生李斯特菌等致病菌是零容忍的,提高生物传感器的灵敏度仍然是检测食品中致病菌的首要目标。因此,开发新型材料,特别是纳米级材料,并将纳米材料与各种信号放大策略联用,为同时检测多种病原体提供可能。(2)特异性是生物传感器检测致病菌需考虑的另一个关键问题,要想达到与培养方法相似的特异性,选择更特异的生物识别分子或将抗体、适配体、噬菌体或具有识别能力的抗生素等生物识别分子联用是未来需要继续努力的方面。(3)基于适配体的生物传感器构建的关键之一是靶标与适配体的特异性结合。为了更加有效地使用适配体,对于新筛选的适配体,需要研究其与靶标的结合机制、结合位点及折叠结构(如G-四链体结构、凸环结构和发夹结构)等方面的知识。(4)目前,生物传感器只能检测样品中致病菌的总体数量,不能区分活菌和死菌,检测结果可能会出现假阳性,因此,开发新的策略使生物传感器能够区分活菌和死菌也是学者研究的重点内容。(5)在实际样品中,通常不仅仅是一种病原体,如果在不同的试管中分别检测不同的病原体,会消耗更多的试剂、样本和时间。如果在一个试管中同时检测不同的病原体,它们会相互干扰,降低检测的准确性,如何同时高效、快速、准确地检测多种靶标,是今后研究的重点。总之,随着科技的不断发展和技术的不断进步,致病菌的检测正朝着自动化、微型化方向发展,在未来拇指大小的传感器可以同时实现特异性、自动化检测成千上万个不同样品。
[1] SCALLAN E, HOEKSTRA R M, WIDDOWSON M A, HALL A J, GRIFFIN P M. Food-born illness acquired in the United States response. Emerging Infectious Diseases, 2011, 17(7): 1339-1340.
[2] RAHMAN S M E, PARK J H, WANG J, OH D H. Stability of low concentration electrolyzed water and its sanitization potential against food-borne pathogens. Journal of Food Engineering, 2012, 113(4): 548-553.
[3] SCHARFF R L. Economic burden from health losses due to food-borne illness in the United States. Journal of Food Protection, 2012, 75(1): 123-131.
[4] YEH H Y, HIETT K L, LINE J E. Reactions of chicken sera to recombinantflagellar proteins. Archives of Microbiology, 2015, 197(2): 353-358.
[5] YAMAZAKI W, TAGUCHI M, KAWAI T, KAWATSU K, SAKATA J, INOUE K, MISAWA N. Comparison of loop-mediated isothermal amplification assay and conventional culture methods for detection ofandin naturally contaminated chicken meat samples. Applied & Environmental Microbiology, 2009, 75(6): 1597-1603.
[6] HUMPHREY T, O'BRIEN S, MADSEN M. Campylobacters as zoonotic pathogens: A food production perspective. International Journal of Food Microbiology, 2007, 117(3): 237-257.
[7] GENG Y Y, LIU G H, LIU L B, DENG Q E, ZHAO L W, SUN X X, WANG J F, ZHAO B H, WANG J C. Real-time recombinase polymerase amplification assay for the rapid and sensitive detection ofin food samples. Journal of Microbiological Methods, 2019, 157: 31-36.
[8] PIKE B L, PATRICIA G, POLY F, MARTYN K. Global distribution ofpenner serotypes: a systematic review. PLoS ONE, 2013, 8(6): e67375.
[9] GALLAY A, BOUSQUET V, SIRET V, PROUZET-MAULEON V, DE VALK H, VAILLANT V. Risk factors for acquiring sporadicinfection in France: results from a national case-control study. Journal of Infectious Diseases, 2008, 197(10): 1477-1484.
[10] Pamphlet (or booklet). National enteric disease surveillance:surveillance overview. CDC, 2011.
[11] SILVA S, TEIXEIRA P, OLIVEIRA R, AZEREDO J. Adhesion to and viability ofon food contact surfaces. Journal of Food Protection, 2008, 71(7): 1379-1385.
[12] 徐连应, 王毕妮, 张富新. 基于复合纳米材料和酶切信号放大电化学适体传感器检测沙门氏菌. 中国农业科学, 2017, 50(21): 4186-4195.
XU L Y, WANG B N, ZHANG F X. An electrochemical aptasensor for detection of salmonella based on composite nanomaterial and enzymatic recycling for amplification. Scientia Agricultura Sinica, 2017, 50(21): 4186-4195. (in Chinese)
[13] 山珊, 赖卫华, 陈明慧, 崔希. 农产品中大肠杆菌O157:H7的来源及分布研究进展. 食品科学, 2014, 35(1): 289-293.
SHAN S, LAI W H, CHEN M H, CUI X. Research progress in sources and distribution ofO157:H7 in agricultural products.Food Science, 2014, 35(1): 289-293. (in Chinese)
[14] GALLAY A, BOUSQUET V, SIRET V, PROUZET-MAULEON V, DE VALK H, VAILLANT V, Simon F, Le Strat Y, Mégraud F, Desenclos J C. Risk factors for acquiring sporadicinfection in France: Results from a national case-control study. Journal of Infectious Diseases, 2008, 197(10): 1477-1484.
[15] JAJERE S M. A review ofwith particular focus on the pathogenicity and virulence factors, host specificity and adaptation and antimicrobial resistance including multidrug resistance. Veterinary World, 2019, 12(4): 504-521.
[16] ZHOU C, ZOU H M, LI M, SUN C J, REN D X, LI Y X. Fiber optic surface plasmon resonance sensor for detection ofO157:H7 based on antimicrobial peptides and AgNPs-rGO. Biosensors & Bioelectronics, 2018, 117: 347-353.
[17] ZEINHOM M M A, WANG Y, SONG Y, ZHU M J, Lin Y H, Du D. Portable smart-phone device for rapid and sensitive detection ofO157:H7 in yoghurt and egg. Biosensors & Bioelectronics, 2018, 99(15): 479-485.
[18] ZHENG L Y, CAI G Z, WANG S Y, LIAO M, LI Y B, LIN J H. Microfluidic colorimetric biosensor for rapid detection ofO157:H7 using gold nanoparticle aggregation and smart phone imaging. Biosensors & Bioelectronics, 2019, 124/125:143-149.
[19] GALLAY A, BOUSQUET V, SIRET V, PROUZET-MAULEON V, DE VALK H, VAILLANT V, SIMIO F, STRAT Y L, MEGRAUD F, DESENCLOS J C. Risk factors for acquiring sporadicinfection in France: Results from a national case-control study. Journal of Infectious Diseases, 2008, 197(10): 1477-1484.
[20] TAO X Q, LIAO Z Y, ZHANG Y Q, FU F, HAO M Q, SONG Y, SONG E. Aptamer-quantum dots and teicoplanin-gold nanoparticles constructed FRET sensor for sensitive detection ofs.Chinese Chemical Letters, 2021, 32(2): 791-795.
[21] KLEIN E, SMITH D L, LAXMINARYNY R. Hospitalizations and deaths caused by methicillin-resistant, United States, 1999-2005. Emerging Infectious Diseases, 2007, 13(12): 1840-1846.
[22] WAKABAYASHI Y, UMEDA K, YONOGI S, NAKAMURA H, YAMAMOTO K, KUMEDA Y, KAWATSU K. Staphylococcal food poisoning caused byharboring staphylococcal enterotoxin genes. International Journal of Food Microbiology,2018, 265: 23-29.
[23] 孙炜佳. 适配体识别—分子马达传感器的构建及在食源性致病菌检测中的应用研究[D]. 无锡: 江南大学, 2017.
SUN W J. The study on the detection for food-borne pathogens based on aptamer molecular motor sensor[D]. Wuxi: Jiangnan University, 2017. (in Chinese)
[24] SILVA N F D, NEVES M M P S, MAGALHES J M C S, FREIRE C, DELERUE-MATOS C. Emerging electrochemical biosensing approaches for detection ofin food samples: An overview. Trends in Food Science & Technology, 2020, 99: 621-633.
[25] RADHAKRISHNAN R, POLTRONIERI P. Fluorescence-free biosensor methods in detection of food pathogens with a special focus on. Biosensors, 2017, 7(4): 63-69.
[26] CHENG C N, PENG Y, BAI J L, ZHANG X Y, LIU Y Y, FAN X J, FAN J X, NING B A, GAO Z X. Rapid detection ofin milk by self-assembled electrochemical immunosensor. Sensors and Actuators B: Chemical, 2014, 190: 900-906.
[27] TAYLOR B J, QUINN A R, KATAOKA A. Listeria monocytogenes in low-moisture foods and ingredients.Food Control, 2019, 103: 153-160.
[28] KIM J K, HARRISON M A. Transfer ofO157:H7 to romaine lettuce due to contact water from melting ice. International Journal of Food Microbiology, 2008, 71(2): 252-256.
[29] ABADIAS M, USALL J, ANGUERA M, SOLSON C, VINAS I. Microbiological quality of fresh, minimally-processed fruit and vegetables, and sprouts from retail establishments.International Journal of Food Microbiology, 2008, 123(1/2):121-129.
[30] WILLIAMSON D A, COOMBS G W, NIMMO G R.‘Down Under’: Contemporary epidemiology ofin Australia, New Zealand, and the South West Pacific. Clinical Microbiology & Infection, 2014, 20(7):597-604.
[31] JIESTEBAN J I, OPORTO B, ADURIZ G, JUSTE RA, HURTADO A. A survey of food-borne pathogens in free-range poultry farms. International Journal of Food Microbiology, 2008, 123(1/2):177-182.
[32] ARAVANIS A M, DEBUSSCHERE B D, CHRUSCINSKI A J, GILCHRIST K H, KOBILKA B K, KOVACS G T A. A genetically engineered cell-based biosensor for functional classification of agents. Biosensors & Bioelectronics, 2001, 16(7/8): 571-577.
[33] DE BOER E, BEUMER R R. Methodology for detection and typing of food-borne microorganisms.International Journal of Food Microbiology, 1999, 50(1/2): 119-130.
[34] VELUSAMY V, ARSHAK K, KOROSTYNSKA O, OLIWA K, ADLEY C. An overview of food-borne pathogen detection: In the perspective of biosensors. Biotechnology Advances, 2010, 28(2): 232-254.
[35] PORTER J, EDWARDS C, PICKUP R W. Rapid assessment of physiological status inusing fluorescent probes. Journal of Applied Psychology,1995, 79(4): 399-408.
[36] TOZE S. RCR and the detection of microbial pathogens in water and wastewater. Water Research, 1999, 33(17): 3545-3556.
[37] HE L, YANG H W, XIAO P F, SINGH R, HE N Y, LIU B,LI Z Y. Highly selective, sensitive and rapid detection ofO157:H7 using duplex PCR and magnetic nanoparticle-based chemiluminescence assay. Journal of Biomedical Nanotechnology, 2017, 13(10): 1243-1252.
[38] DAVYDOVA A, VOROBJEVA M, PYSHNYI D, ALTMAN S, VLASSOV V, VENYAMINOVA A. Aptamers against pathogenic microorganisms. Critical Reviews in Microbiology, 2016, 42(6): 847-865.
[39] 李玉珍, 林亲录, 肖怀秋. 酶联免疫吸附技术及其在食品安全检测中的应用研究进展. 中国食品添加剂, 2006(3): 108-112.
LI Y Z, LIN Q L, XIAO H Q. Enzyme-linked immunosorbent assay and study advance of ELISA in food safety detections. China Food Additve, 2006(3): 108-112. (in Chinese)
[40] BENNETT R W.enterotoxin and its rapid identification in foods by enzyme[1]linked immunosorbent assay-based methodology. Journal of Food Protection, 2005, 68(6): 1264-1270.
[41] SHARMA S K, JOSEPH J L, EBLEN B S, WHITING R C. Detection of type A, B, E, and F clostridium botulinum neurotoxins in foods by using an amplified enzyme-linked immunosorbent assay with digoxigenin-labeled antibodies. Applied and Environmental Microbiology, 2006, 72(2): 1231-1238.
[42] KIM H R, SONG M Y, KIM B C. Rapid isolation of bacteria-specific aptamers with a non-SELEX-based method. Analytical Biochemistry, 2020, 591: 113542.
[43] AMRAEE M, OLOOMI M, YAVARI A, BOUZARI S. DNA aptamer identification and characterization forO157:H7 detection using cell based SELEX method. Analytical Biochemistry, 2017, 536: 36-44.
[44] WU W H, ZHANG J, ZHENG M Q, ZHONG Y H, YANG J, ZHAO Y H, WU W P, YE W, WEN J, WANG Q, LU J X. An aptamer-based biosensor for colorimetric detection ofO157:H7. PLoS ONE, 2012, 7(11): e48999.
[45] 段诺, 张田力, 吴世嘉, 王周平. 肠致病性大肠杆菌适配体筛选研究. 食品安全质量检测学报, 2015, 6(12): 4803-4809.
DUAN N, ZHANG T L, WU S J, WANG Z P. Selection of an aptamer targeted to enteropathogenic. Journal of Food Safety & Quality, 2015, 6(12): 4803-4809. (in chinese)
[46] KIM S E, SU W Q, CHO M S, LEE Y, CHOE W S, LEE Y, CHOE W S. Harnessing aptamers for electrochemical detection of endotoxin. Analytical Biochemistry, 2012, 424(1): 12-20.
[47] MOON J, KIM G, LEE S, PARK S. Identification of-specific DNA aptamers developed using whole-cell SELEX and FACS analysis.Journal of Microbiological Methods, 2013, 95(2): 162-166.
[48] JOSHI R, JANAGAMA H, DWIVEDI H P, SENTHIL KUMAR T M, JAYKUS L A, SCHEFERS J, SREEVATSAN S. Selection, characterization and application of DNA aptamers for the capture and detection ofserovars. Molecular and Cellular Probes, 2009, 23(1): 20-28.
[49] PAN Q, ZHANG X L, WU H Y, HE P W, ZHANG M S, HU J M, XIA B, WU J Q. Aptamers that preferentially bind type IVB pili and inhibit human monocytic-cell invasion byserovar typhi. Antimicrobial Agents and Chemotherapy, 2005, 49(10): 4052-4060.
[50] LIU G Q, YU X F, XUE F, CHEN W, YE Y K, YANG X J, LIAN Y Q, YAN Y, ZONG K. Screening and preliminary application of a DNA aptamer for rapid detection ofO8. Microchimica Acta, 2012, 178(1/2): 237-244.
[51] DUAN N, WU S J, CHEN X J, HUANG Y K, XIA Y, MA X Y, WANG Z P. Selection and characterization of aptamers againstusing whole-bacterium Systemic Evolution of Ligands by Exponential Enrichment (SELEX). Journal of Agricultural & Food Chemistry, 2013, 61(13): 3229-3234.
[52] HAN S R, LEE S W.selection of RNA aptamer specific to. Annals of Microbiology, 2014, 64(2): 883-885.
[53] HUANG Y K, CHEN X J, Duan N, WU S J, WANG Z P, WEI X L, WANG Y F. Selection and characterization of DNA aptamers againstenterotoxin C1Food Chemistry, 2015, 166: 623-629.
[54] DEGRASSE J A. A Single-stranded DNA aptamer that selectively binds toaureus enterotoxin B. PLoS ONE, 2012, 7(3): e33410.
[55] HEDAYATI C M, AMANI J, SEDIGHIAN H, AMIN M, SALIMIAN J, HALABIAN R, IMANI FOOLADI A A. Isolation of a new ssDNA aptamer againstenterotoxinB based on CNBr-activated sepharose-4B affinity chromatography. Journal of Molecular Recognition, 2016, 29(9): 436-445.
[56] WANG K Y, WU D, CHEN Z, ZHANG X H, YANG X Y, YANG C Y J, LAN X P. Inhibition of the superantigenic activities ofenterotoxin Aby an aptamer antagonist. Toxicon, 2016, 119: 21-27.
[57] SUH S H, DWIVEDI H P, CHOI S J, JAYKUS L A. Selection and characterization of DNA aptamers specific forspecies. Analytical Biochemistry, 2014, 459: 39-45.
[58] 韩晓晓. 阪崎肠杆菌适配体制备及其应用[D]. 无锡: 江南大学, 2013.
HAN X X. Selection of aptamers forr and application[D]. Wuxi: Jiangnan University, 2013. (in Chinese)
[59] FISCHER C, HUENNIGER T, JARCK J H, FROHNMEYER E, KALLINICH C, HAASE I, HAHN U, FISCHER M. Food sensing: Aptamer-based trapping ofcereus spores with specific detection via real time PCR in milk. Journal of Agricultural and Food Chemistry,2015, 63(36): 8050-8057.
[60] SOUNDY J, DAY D. Selection of DNA aptamers specific for live. PLoS ONE, 2017, 12(9): e0185385.
[61] SONG S X, WANG X Y, XU K, LI Q, NING L F, YANG X B. Selection of highly specific aptamers tousing cell-SELEX powered by functionalized graphene oxide and rolling circle amplification. Analytica Chimica Acta, 2019, 1052: 153-162.
[62] MUNIANDY S, TEH S J, APPATURI J N, THONG K L, LAI C W, IBRAHIM F, LEO B F. A reduced graphene oxide-titanium dioxide nanocomposite based electrochemical aptasensor for rapid and sensitive detection of. Bioelectrochemistry,2019, 127: 136-144.
[63] APPATURI J N, PULINGAM T, THONG K L, MUNIANDY S, LEO B F. Rapid and sensitive detection ofwith reduced graphene oxide-carbon nanotube based electrochemical aptasensor. Analytical Biochemistry, 2019, 589: 113489.
[64] BU S J, WANG K Y, LI Z Y, WANG C Y, HAO Z, LIU W S, WAN J Y. An electrochemical biosensor based on methylene blue-loaded nanocomposites as signal-amplifying tags to detect pathogenic bacteria. Analyst, 2020, 145(12): 4328-4334.
[65] SHEIKHZADEH E, CHAMSAZ M, TURNER A P F, JAGER E W H, BENI V. Label-free impedimetric biosensor fordetection based on poly [pyrrole-co-3-carboxyl-pyrrole] copolymer supported aptamer. Biosensors & Bioelectronics, 2016, 80: 194-200.
[66] WANG L, HUANG F C, CAI G Z, YAO L, ZHANG H L, LIN J H. An electrochemical aptasensor using coaxial capillary with magnetic nanoparticle, urease catalysis and PCB electrode for rapid and sensitive detection ofO157:H7. Nanotheranostics, 2017, 1(4): 403-414.
[67] TENG J, YE Y W, YAO L, YAN C, CHENG K W, XUE F, PAN D D, LI B G, CHEN W. Rolling circle amplification based amperometric aptamer/immuno hybrid biosensor for ultrasensitive detection of.Microchimica Acta,2017, 184(9):3477-3485.
[68] WU W H, LI M, WANG Y, OUYANG H X, WANG L, LI C X, CAO Y C, MENG Q H, LU J X. Aptasensors for rapid detection ofO157:H7 and. Nanoscale Research Letters, 2012, 7(1): 658.
[69] MA X Y, SONG L J, ZHOU N X, XIA Y, WANG Z P. A novel aptasensor for the colorimetric detection ofbased on gold nanoparticles.International Journal of Food Microbiology, 2017, 245: 1-5.
[70] KIM Y J, KIM H S, CHON J W, KIM D H, HYEON J Y, SEO K H. New colorimetric aptasensor for rapid on-site detection ofandin chicken carcass samples. Analytica Chimica Acta, 2018, 1029: 78-85.
[71] SUN Y H, DUAN N, MA P F, LIANG Y, ZHU X Y, WANG Z P. Colorimetric aptasensor based on truncated aptamer and trivalent dnazyme fordetermination. Journal of Agricultural and Food Chemistry, 2019, 67(8): 2313-2320.
[72] WU S J, DUAN N, QIU Y T, LI J H, WANG Z P. Colorimetric aptasensor for the detection ofusing ZnFe2O4-reduced graphene oxide nanostructures as an effective peroxidase mimetics. International Journal of Food Microbiology, 2017, 261: 42-48.
[73] DEHGHANI Z, HOSSEINI M, MOHAMMADNEJAD J, BAKHSHI B, REZAYAN A H. Colorimetric aptasensor forcells by exploiting the peroxidase like activity of Au@Pd nanoparticles. Microchimica Acta,2018, 185(10): 448.
[74] GUO Y Y, ZHAO C, LIU Y S, NIE H, GUO X X, SONG X L, XU K, LI J, WANG J. A novel fluorescence method for the rapid and effective detection ofusing aptamer- conjugated magnetic nanoparticles and aggregation-induced emission dots. Analyst,2020, 145(11): 3857-3863.
[75] SINGH P, GUPTA R, SINHA M, KUMAR R, BHALLA V. MoS2based digital response platform for aptamer based fluorescent detection of pathogens.Microchimica Acta,2016, 183(4): 1501-1506.
[76] YANG T, YANG X Y, GUO X J, FU S Q, ZHENG J P, CHEN S H, QIN X, WANG Z H, ZHANG D Y, MAN C X, JIANG Y J. A novel fluorometric aptasensor based on carbon nanocomposite for sensitive detection ofO157:H7 in milk. Journal of Dairy Science,2020, 103: 7879-7889.
[77] DUAN N, WU S J, MA X Y, XIA Y, WANG Z P. A universal fluorescent aptasensor based on accublue dye for the detection of pathogenic bacteria. Analytical Biochemistry, 2014, 454(1): 1-6.
[78] SRINIVASAN S, RANGANATHAN V, DEROSA M C, MURARI B M. Label-free aptasensors based on fluorescent screening assays for the detection of. Analytical Biochemistry, 2018, 559: 17-23.
[79] HAO L L, GU H J, DUAN N, Wu S J, Ma X Y, XIA Y, WANG H T, WANG Z P. A chemiluminescent aptasensor based on rolling circle amplification and Co2+/n-(aminobut3r1)-n-(ethylisolumino1) functional flowerlike gold nanoparticles fordetection. Talanta,2017, 164:275-282.
[80] HAO L L, GU H J, DUAN N, WU S J, MA X H, XIA Y, TAO Z, WANG Z P. An enhanced chemiluminescence resonance energy transfer aptasensor based on rolling circle amplification and WS2nanosheet fordetection. Analytica Chimica Acta, 2017, 959: 83-90.
[81] ZHOU S S, LU C,LI Y Z, XUE L, ZHAO C Y, TIAN G F, BAO Y M, TANG L H, LIN J H, ZHENG J K. Gold Nanobones Enhanced utrasensitive Surface-Enhanced Raman scattering aptasensor for detectingO157:H7ACS Sensors, 2020, 5(2): 588-596.
[82] ZHANG H, Ma X Y, LIU Y, DUAN N, WU S J, WNAG Z P, XU B C. Gold nanoparticles enhanced SERS aptasensor for the simultaneous detection ofandBiosensors & Bioelectronics, 2015, 74: 872-877.
[83] DUAN N, CHANG B Y, ZHANG H, WANG Z P, WU S J.detection using a surface-enhanced raman scattering- based aptasensor. International Journal of Food Microbiology, 2016, 218: 38-43.
[84] XU Y, LUO Z W, CHEN J M, HUANG Z J, WANG X, AN H F, DUAN Y X. Ω-shaped fiber-optic probe-based localized surface plasmon resonance biosensor for real-time detection of. Analytical Chemistry,2018, 90(22): 13640-13646.
[85] GE C, YUAN R, YI L, YANG J L, ZHANG H W, LI L X, NIAN W Q, YI G. Target-induced aptamer displacement on gold nanoparticles and rolling circle amplification for ultrasensitive liveelectrochemical biosensing. Journal of Electroanalytical Chemistry, 2018, 826: 174-180.
[86] KHANG J, KIM D, CHUNG K W, LEE J H. Chemiluminescent aptasensor capable of rapidly quantifyingO157:H7. Talanta, 2016, 147: 177-183.
[87] DUAN N, SHEN M F, SHUO Q, WANG W Y, WU S J, WANG Z P. A SERS aptasensor for simultaneous multiple pathogens detection using gold decorated PDMS substrate. Spectrochimica Acta Part A: Molecular and Biomolecular Spectroscopy, 2020, 230: 118130.
Research Progress of Food-Borne Pathogen Detection Based on Electrochemical and Optical Aptasensors
HUI YuanYuan, PENG HaiShuai, WANG BiNi, ZHANG FuXin, LIU YuFang, JIA Rong, REN Rong
School of Food Engineering and Nutrition Sciences, Shaanxi Normal University, Xi’an 710119
Food safety has become a heated topic attracting widespread attention from the society due to the diversity of food and complexity of food production system. Food-borne diseases caused by microorganisms have the highest rates in food safety problem. The detection of food-borne pathogenic bacteria is the key link for the food-borne disease prevention and control. The plate counting method is rated as the gold standard for microbial detection, but the signal amplification was achieved by the growth of individual bacterial cells into visible colony during the detection of pathogenic bacteria, so the detection time is longer (3-7 days). Although polymerase chain reaction (PCR) and enzyme-linked immunoabsorbent assay (ELISA) are now applied in the detection of food-borne pathogenic bacteria, they are not suitable for timely and rapid on-site detection due to time-consuming pretreatment, complex operations and false positive results. Aptamers (Apt) are oligonucleotides that are isolated from combinatorial DNA library via systemic evolution of ligands by exponential enrichment (SELEX) technology, which present great potential in the field of detection of toxin, heavy metal, antibiotics, food pathogens and other small molecules due to their small size, easy synthesis and modification. Currently, many researchers of domestic and overseas have developed various detection methods by using aptamer as bio-recognition elements. This paper reviewed common food-borne pathogenic bacteria, traditional detection methods of food-borne pathogenic bacteria, and the electrochemical and optical aptasensors for detection of food-borne pathogenic bacteria in recent years. The descriptions covered the detection strategy of each method and provided details such as the detection time, range and limit. Finally, the paper pointed out the flaw of aptasensor in food safety detection, and the research development tendency was prospected, which provided bases for the further related work.
food-born pathogen bacteria; aptamer; rapid detection; food safety

10.3864/j.issn.0578-1752.2021.11.014
2020-09-16;
2020-12-21
陕西省重点研发计划重点产业创新链项目(2019ZDLNY06-06)、中央高校基本科研业务费专项(GK202003084)、西安市科技计划农业技术研发项目(20NYYF0014,20NYYF0018)
惠媛媛,E-mail:570421161@qq.com。通信作者王毕妮,E-mail:biniwang@snnu.edu.cn
(责任编辑 赵伶俐)

