四倍体小麦与六倍体小麦杂种的染色体遗传特性
罗江陶 郑建敏 蒲宗君,* 范超兰 刘登才 郝 明,*
四倍体小麦与六倍体小麦杂种的染色体遗传特性
罗江陶1郑建敏1蒲宗君1,*范超兰2刘登才2郝 明2,*
1四川省农业科学院作物研究所 / 农业农村部西南地区小麦生物学与遗传育种重点实验室, 四川成都 610066;2四川农业大学小麦研究所, 四川成都 611130
四倍体栽培小麦(L., AABB)和普通小麦(L., AABBDD)是两种目前主要的小麦栽培种。通过远缘杂交转移利用四倍体小麦(或六倍体小麦)基因是六倍体小麦(或四倍体小麦)遗传改良的重要方法。然而, 两者杂种F1为基因组组成不平衡的五倍体, 其中A和B基因组染色体均为两套, 而D基因组染色体仅一套。亲本间的遗传差异, 包括核基因组和细胞质基因组, 可能影响五倍体杂种的染色体传递效率。本研究以多个不同遗传背景的四倍体小麦和六倍体小麦为亲本, 配置正反交五倍体杂种F1, 采用多色荧光原位杂交技术分析自交F2代植株的染色体组成规律。结果表明, 杂交亲本的遗传背景对杂种F1自交结实率影响显著; 不论是以四倍体小麦还是六倍体小麦做母本, AB基因组染色体在F1自交过程中相对稳定, F2后代的数目均接近28条(27.9 vs. 28.0); 以四倍体小麦为母本F2平均保留的D基因组染色体数显著多于以六倍体小麦为母本的后代(7.0 vs. 2.9)。因此, 以四倍体小麦为最终目标后代时, 应优先以六倍体小麦为母本进行杂交组合的配置; 以六倍体小麦为最终目标后代时, 应优先以四倍体小麦为母本开始最初的杂交组合配置。
四倍体小麦; 六倍体小麦; 染色体传递
小麦是重要的粮食作物, 目前主要的栽培种包括六倍体小麦(L., AABBDD, 又称普通小麦)和四倍体小麦(L., AABB), 前者约占世界小麦年产出的95%, 后者占5%[1]。培育抗病虫、优质和高产的新品种是应对气候变化、不断增长的物质需求和人口持续扩增问题的关键, 其依赖于小麦群体的遗传多样性。约八千至一万年前, 普通小麦形成于栽培四倍体小麦和二倍体节节麦(, DD)间的异源多倍化事件[2-4]。在普通小麦的起源过程中, 仅有少数四倍体小麦和节节麦居群参与, 产生了“进化瓶颈”。因此,大量四倍体小麦的遗传多样性被排除在目前的普通小麦群体之外, 例如源自野生二粒小麦的广谱抗条锈病基因[5-6]。同样, 由于物种形成后的独立进化, 额外基因组的加入(D基因组)以及近现代育种过程中有意识地渗入近缘属种的遗传物质, 一些六倍体小麦中的优异基因(等位变异或性状)也不存在于四倍体小麦中。因此, 加强四倍体小麦和六倍体小麦间基因的交流, 是促进小麦遗传育种发展的重要方式。
虽然转基因技术发展进步快速, 但是受限于目前的政策以及技术本身对特定受体的依赖性, 杂交仍然是不同物种间转移基因的主要方法。由于倍性不同, 杂交转移四倍体小麦基因至六倍体小麦背景通常有两种途径。一种途径是模拟普通小麦形成过程, 将四倍体小麦和二倍体节节麦合成双二倍体(称为人工合成小麦), 再以其为桥梁亲本进行杂交转移。通过该方法, 一批导入了四倍体小麦优异基因的六倍体小麦新品种已经被审定并应用于生产[7-8]。另外一种途径是直接杂交。无论是单个位点还是大规模的群体数据分析均表明, 普通小麦可能通过“五倍体”桥梁, 在形成初期与四倍体野生二粒小麦间发生了基因交流[9-11]。通过人工直接杂交, 然后回交或顶交, 一批源于四倍体小麦的优异基因也已成功转入普通小麦。例如, 源于四倍体小麦的白粉病抗性基因[12]、[13]、[14]、[15]、[16]、[17]以及高蛋白含量基因[18]等。六倍体小麦向四倍体小麦转移基因也是采用直接杂交法。然而, 四倍体小麦和六倍体小麦间的细胞质、基因组倍性和遗传背景差异可能影响目标杂种后代的获得效率。
四倍体小麦与六倍体小麦杂种F1为五倍体, 基因组组成为AABBD。因此, 成单的D基因组染色体在减数分裂过程中的行为和随后配子传递频率是决定后代选择效率的关键因素。利用微卫星标记(SSR)、多样性阵列标记(Dart)和单核苷酸多态性标记(SNP)对四倍体/六倍体小麦杂种后代的鉴定发现, 亲本的遗传背景和杂交过程中的母本选择(四倍体做母本或六倍体做母本)对后代的染色体组成具有重要影响[19-23]。Martin等[21]和Padmanaban等[24-25]还发现, 五倍体杂种(AABBD) D基因组染色体与A和B基因组染色体间的传递存在相互影响, 保留大量D基因组染色体的品系往往含有更高比例的普通小麦的A和B基因组。不同于分子标记, 细胞学方法能更为直观和准确地鉴定染色体的结构和数量。Padmanaban等[26]利用基因组原位杂交(GISH)技术, 鉴定了部分四倍体小麦和六倍体小麦杂种后代的D基因组染色体, 但仅用D基因组作为探针无法检测是否存在A、B基因组染色体的变异重组, 也不能有效鉴定染色体的拷贝数。用不同颜色荧光标记的混合寡序列为探针的多色荧光原位杂交技术, 能准确鉴定植株染色体数目、存在形式及结构变异[27-29]。本研究应用该技术, 对四倍体小麦和六倍体小麦杂种F2代的D染色体保留情况进行了详细分析, 探讨四倍体小麦与六倍体小麦杂交时的亲本选配原则。
1 材料与方法
1.1 试验材料
本实验利用14份六倍体普通小麦和11份四倍体小麦构建实验群体。普通小麦包括贵协011-1、贵协011-2、贵协2号、Li-50、Li-22、13L2071-2、13L2069、亲2120 (Isidora塞尔维亚小麦)、亲2122 (KM 113-13MRS捷克小麦)、亲2142 (Queroinia智利小麦)、亲2147 (Slavan乌克兰小麦)、WJN1428、川麦608和P1561, 四倍体小麦包括PI185192、PI113961、CITR14139、PI223171、PI34945、PI415152、PI352369、AS2255、PI190973、PI191808和PI94666。杂交组配参见表1。PI编号的材料由美国国家种质资源库(http://www.ars-grin.gov/)提供, AS2255、WJN1428由四川农业大学小麦研究所提供, 贵协系列由贵州大学提供, 13L2071-2、13L2069由中国农业科学院作物科学研究所提供。
2017年在四川省农业科学院新都试验基地进行杂交, 采用传统的人工去雄和授粉。次年, 杂种F1种植于相同基地, 抽穗后套袋自交, 获得的杂种F2种子用于后续实验。去除基部不发育或发育不良小穗, 统计所有套袋F1穗子的小穗总数和种子数, 计算单个小穗的自交结实率(以下简称自交结实率), 即自交结实率= (结实种子数/总小穗数)×100%。
1.2 原位杂交分析
随机选取不同杂交组合的种子, 在垫有湿润滤纸的培养皿中发芽。待根尖长至3~4 cm的时候, 剪取根尖。根尖的处理和体细胞制片的方法与Luo等[30]一致。原位杂交的探针和方法与Luo等[30]相同, 探针包括7个寡核苷酸序列探针Oligo-pSc119.2-1、Oligo-pAs1-1、Oligo-pAs1-3、Oligo-pAs1-4、Oligo-pAs1-6、Oligo-AFA-3、Oligo-AFA-4和一个简单重复序列探针(AAC)5[29,31]。探针由成都擎科生物科技有限公司合成(中国成都)。使用配备CCD镜头的奥林巴斯BX63荧光显微镜进行杂交信号检测并图像采集。
1.3 数据分析
Microsoft Excel 2007用于统计分析。R3.6.2及R包ggplot2用于画图[32]。
2 结果与分析
2.1 AABBD五倍体杂种自交结实率分析
本实验共计获得20个杂交组合, 其中六倍体/四倍体(AABBDD/AABB) 12个, 四倍体/六倍体(AABB/AABBDD) 8个(表1)。由于花期不遇, 没有获得相同四倍体与六倍体间的正反交F1。六倍体/四倍体F1总自交结实率为27.94% (817/2924), 而四倍体/六倍体为40.57% (706/1740)。从分布看, 自交结实率低于20%的四倍体/六倍体组合仅14% (1/7), 而六倍体/四倍体组合中高达70% (7/10)。由于所有组合的亲本均不相同, 细胞质来源和核遗传背景差异均可能引起两种组配类型间的自交结实率差异。核遗传背景对AABBD的自交结实率有显著影响。这是因为在相同组配类型内, 不同杂交组合间的自交结实率变异幅度大(六倍体/四倍体为1.32%~ 71.29%, 四倍体/六倍体为18.75%~147.20%)。同时, 无论做母本还是父本, 同一材料与不同亲本杂种F1间的自交结实率显著差异。例如, Li-50/PI185192组合的自交结实率1.32%显著低于Li-50/PI113961组合的19.53% (检验,=2.18>1.96); 贵协2号/PI185192组合的自交结实率14.89%则显著高于亲Li-50/ PI185192组合的1.32% (检验,=5.77>1.96)。

表1 各杂交组合F1套袋自交结实率
*自交结实率没有统计; 加粗的杂交组合用于后续的细胞学分析。
* There is no statistics on the seed setting rate of self-delivery. The hybrid combination in bold was used for subsequent cytological analysis.
2.2 AABBD杂种自交F2后代染色体组成分析
多重寡核苷酸FISH技术能够稳定且准确地鉴定四倍体小麦和六倍体小麦亲本的所有染色体。染色体核型分析结果显示: 六倍体小麦亲本贵协011-1含有1对3A/6D相互易位染色体, 亲2120含有1对5B/7B相互易位染色体(图1); 四倍体小麦亲本AS2255则含有1对1A/7A相互易位染色体。
红框指示相互易位染色体。Boxes in red indicate the reciprocal translocation chromosomes.
本实验共鉴定了59个F2单株(37个来源于AABB/AABBDD, 22个来源于AABBDD/AABB)的染色体组成。所有单株平均染色体数为33.5条, 分布在27.5~40.0条之间(不完整染色体计数为0.5) (图2)。来源于AABB/AABBDD杂交组合的F2单株的平均染色体数为34.9条, 其中含36条染色体的单株最多,占18.9% (7/37)。但是, 以六倍体小麦为母本F2单株的平均染色体数为30.8条, 极显著少于以四倍体小麦为母本的材料(-test,=0.003), 且以含30条染色体的单株最多, 所占比例为23.81% (5/22)。因此, 以四倍体小麦为母本的AABBD五倍体杂种自交能够保留更多的染色体。
根据FISH核型结果, 将所有单株的每条染色体划分至A、B和D基因组, 其中不完整的染色体(断裂染色体片段)计为0.5, 易位染色体计为0.5/0.5 (例如1AS.1DL染色体计为0.5A和0.5D)。另外, 来自于PI352369/贵协011-1的6个F2单株含有1对(或1条)不能识别的较小染色体, 这对染色体没有计入后续分析。以四倍体小麦为母本的F2单株平均含有27.9条AB基因组染色体, 以六倍体小麦为母本的F2单株平均含有28条AB基因组染色体。但是, 以四倍体小麦为母本的F2单株平均含有7.0条D基因组染色体, 极显著多于以六倍体小麦为母本F2单株的2.9条(-test,=0.002) (图2)。因此, D基因组染色体的不同保留频率导致了2种杂交方法后代的染色体数差异。
就不同D基因组染色体的保留频率而言, 以四倍体小麦作为母本的F2单株保留频率最高的是7D (1.19条/株), 其次为5D (1.11条/株); 最低的是1D (0.84条/株)和4D (0.84条/株)染色体(图3)。以六倍体为母本的F2单株D染色体保留率最高的染色体是5D (0.55条/株), 其次为2D、6D和7D, 均为0.45条/株, 保留率最低的是4D (0.27条/株), 其次为1D (0.32条/株)。综合来看, 细胞质(四倍体小麦或六倍体)是影响不同D染色体保留频率的重要影响因素。同时, 不同的染色体间具有一定的偏向性, 即5D和7D染色体保留频率较高, 而1D和4D染色体保留频率较低。
左边图中的红色、绿色和蓝色竖线分别代表以四倍体小麦为母本F2单株的平均染色体数、以六倍体小麦为母本F2单株的平均染色体数以及所有F2单株的平均染色体数。右图中的蓝色菱形点代表平均值。
Line in red, green, and blue in the left graph indicates average chromosome number per line in AABB/AABBDD population, AABBDD/ AABB population, and both of them, respectively. Diamonds in blue in the right graph represent the mean values.
右图中的每一列代表一个单株, 虚线分隔不同的杂交组合。
Each row represents an individual plant of F2population. Dotted lines are used to divide different hybrid combinations.
分析不同D基因组染色体的拷贝数(仅完整的染色体作为有效计数)发现: 以四倍体小麦为母本的后代, 1-7D染色体保留2个拷贝的平均频率为0.30, 显著高于以六倍体小麦为母本后代的0.06 (值为0.0040); 相反, 1-7D染色体完全丢失(0个拷贝)的平均频率极显著低于以六倍体小麦为母本的后代(0.27 vs. 0.64,=8.09E-06) (图4)。1-7D染色体保留1个拷贝的平均频率在AABB/AABBDD和AABBDD/ AABB间则没有差异(值为0.0958), 分别为0.30和0.44。在四倍体小麦为母本的后代中, 出现2个拷贝频率最高的是3D染色体, 其次为5D、7D和6D; 而在六倍体小麦为母本的后代中, 出现2个拷贝频率最高的是2D, 其次为5D和6D。
对每个单株的D基因组染色体覆盖度进行打分评价: 含1条完整染色体或1对或更多拷贝, 则对应染色体覆盖度打分为1; 含单个不完整染色体打分为0.5; 对应染色体缺失则打分为0。通常, 总分等于7的单株应含有一套完整的D基因组染色体; 而总分等于0的单株则缺失了全部D基因组染色体。以四倍体小麦为母本的后代平均得分为5.3分, 极显著高于以六倍体小麦为母本后代的2.4。所有59个F2中, 有16个单株覆盖度得分为7, 即含有至少1套完整D基因组染色体, 频率为27.11%; 其中来源于四倍体/六倍体组配类型的为14株(87.5%), 而来源于六倍体/四倍体组配类型的仅2株(12.5%)。这些含1套完整D基因组染色体的材料的自交后代或与六倍体小麦的杂交后代, 更容易恢复成基因组组成为AABBDD的普通小麦后代; 得分为0的植株则总共有7株(3株来源于AABB/ AABBDD, 4株来源于AABBDD/AABB)。
2.3 AB基因组染色体变异以及染色体结构变异
59个F2植株中, 仅9个单株的A或B基因组染色体数目发生了变异(表2和图4), 频率为15.25%, 包括3*1B+3*3B (M143-7)、1*1A (M145-4)、3*6B (M146-4、M158-4)、3*3B (M146-10)、1*4B (M147-1、M147-3、M147-7)、0*4B (M147-2), 表明A和B基因组染色体的传递受到了轻微的影响。
18个单株存在染色体结构变异, 涉及A、B和D基因组(表2和图5)。其中, 10个单株含有的结构变异遗传自亲本, 包括6DS.3AL (M147-1、M147-2、M147-3、M147-6、M147-9和M147-11, 均遗传自亲本贵协011-1)、5BS.7BS和5BL.7BL (M161-1、M158-2、M158-3和M158-4, 均遗传自亲本亲2120)。另外10个单株(10/59=16.95%)具有11种新形成的结构变异染色体, 包括染色体片段5DS-(M145-5, M146-5)、6DS.6DL-(M146-5)、3DS.3DL-(M147-3)、6DS (M147-3)、3DS-(M161-8), 断裂-融合形成的易位染色体3DS-5DL (M146-5)、4AL.4AS-5DL.5DS (M147-1)、2AS.2AL-6AL (M149-7, M154-2)和等臂染色体3DS.3DS (M160-1), 以及信号发生明显变化的7D (M145-10)和3D (M157-3)。这些新形成的染色体结构变异主要涉及D基因组染色体和少量A基因组染色体, 未检测到涉及B基因组的染色体结构变化。
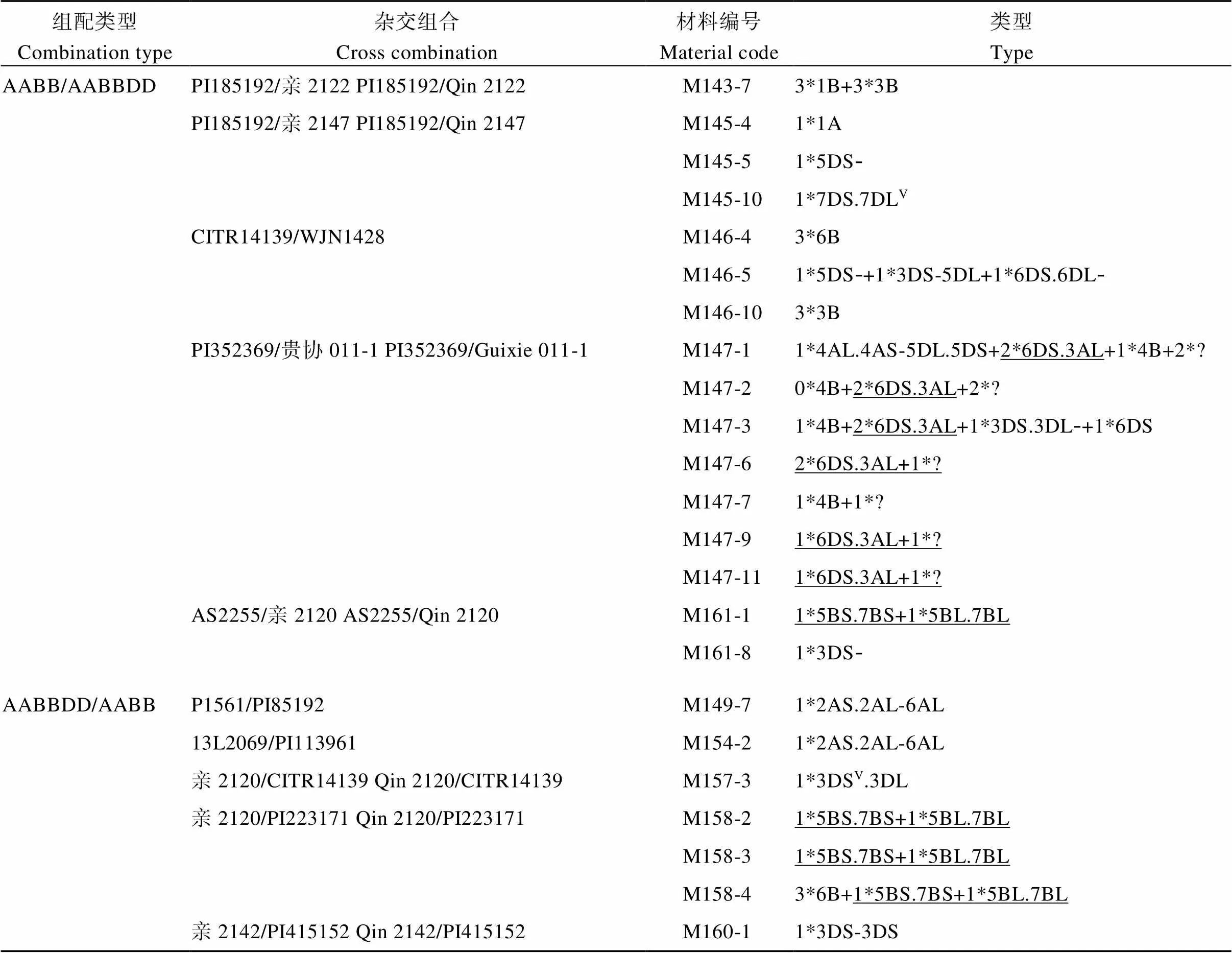
表2 F2杂种中的染色体变异
V: FISH信号变异; 下画线: 遗传自亲本的结构变异; ?: 不能识别的染色体。
V: FISH signal variation; Underline: structural variations are inherited from the parents; ?: unknown chromosome.
白色箭头指示结构变异染色体; 黄色箭头指示AB基因组染色体数目变异。A: M145-4: 仅含1条1A染色体; B: M157-3: 3DS端部红色信号缺失; C: M154-2: 含1个2AS.2AL-6AL重组染色体; D: M149-7: 含1个2AS.2AL-6AL重组染色体; E: M146-5: 含1个3DS-5DL重组染色体, 2个D片段; F: M145-10: 7DL端部红色信号缺失; G: M145-5: 含1个D片段; H: M160-1: 含1个3DS-3DS重组染色体; I: M146-10: 含3个3B染色体。
Arrows in white show the chromosomes with structural variation. Arrows in yellow show A or B chromosomes with copy number variation. A: M145-4 with single copy of 1A; B: M157-3 with a copy of 3D that showed a different 3DS karyotype; C: M154-2 with a 2AS.2AL-6AL translocation; D: M149-7 with a 2AS.2AL-6AL translocation; E: M146-5 with a copy of 3DS.5DL translocation, a copy of 6DS.6DL- and 5DS-chromosome fragment respectively; F: M145-10 with a copy of 7D that showed a different 7DL karyotype; G: M145-5 with a copy of 5DS-chromosome fragment; H: M160-1 with a copy of 3DS.3DS isochromosome; I: M146-10 with a three copy of 3B chromosomes.
3 讨论
通过种间杂交, 以同源重组(A或B基因组)或易位(通常用于将六倍体小麦D基因组染色体上的基因转移到四倍体小麦背景)的形式, 转移对方的有益基因是四倍体小麦或六倍体小麦遗传改良的一种常用方法。例如, 四倍体小麦普遍对铝离子和钠离子耐受性较差, 而位于4D染色体长臂的和基因赋予了六倍体小麦优异的抗性[33]。前人通过4DL和4BL部分同源重组, 成功将这2个基因导入到四倍体小麦品种, 提高了其对铝离子和钠离子的耐受性[34-36]。通过同源重组的方式, 从四倍体小麦(或六倍体小麦)转移优异基因到六倍体小麦(或四倍体小麦)的例子就更多[37-38]。由于倍性不同, 杂交结实率、五倍体杂种育性以及D染色体传递效率是影响四倍体小麦/六倍体小麦杂交法基因转移效率的3个关键因素。早期的研究认为, 低倍性的物种作为母本通常不太成功, 可能导致杂种种子较差, 以及随后较低的发芽率和幼苗成活率[39-40]。虽然没有详细统计, 本研究中没有观察到四倍体或六倍体小麦做母本会显著影响杂交结实率, 五倍体杂种F1发芽比较正常。同时, 亲本间的遗传背景对杂种F1的自交结实率有强烈的影响。
五倍体AABBD杂种减数分裂过程中, 成单的D基因组染色体行为是影响遗传改良效率的关键。如果最终目的是改良四倍体小麦, 那么D基因组染色体需要尽快被消除掉。反之则需要快速将D基因组染色体恢复成完整的两套。Padmanaban等[24]发现以四倍体小麦为母本的杂种F2, 含有至少1套完整D基因组染色体的频率高于以六倍体作为母本的子代。本研究利用不同四倍体和六倍体作为母本, 得到了相似的结果, 即以四倍体为母本的F2子代能保留更多的D基因组染色体。单价体在减数分裂中期I向后期I转换的过程中有两种移动方式: 一种是单极锚定的纺锤丝牵引姐妹染色单体一起移动到一极;另外一种是从两极伸出的纺锤丝分别结合在姐妹染色单体的着丝点, 牵引2个姐妹染色单体提前分开并各自移动到一极。通常两种分离移动方式的比例约各占一半, 同时还包括一定频率的染色体断裂[41]。从F2保留的D基因组染色体数目和拷贝数来看, 以四倍体小麦作为母本F1的D基因组染色体的分离模式可能更接近第2种: 在减数分裂后期I至末期I, D染色体的姐妹染色单体提前分离移动到2个二分体子细胞中, 成单的姐妹染色单体减II随机移动到两极的行为模式, 则形成的配子含更多的D基因组染色体; 这样的雌雄配子随机结合后, F2单株的总D染色体数目更多, 且部分染色体为2个拷贝(图2和图3)。而以六倍体小麦为母本的杂种F2平均每个单株仅含有2.9条D染色体。其可能对应的减数分裂行为模式是成单的D基因组染色体在F1减数分裂后期I的分离过程中, 移动分离步调严重滞后于同源配对的A和B基因组染色体, 进而形成微核被消除掉。这一推论需要详细观察2种杂种F1的减数分裂过程予以证实。然而, 无论杂种F1的减数分裂过程是否如我们推测的一样, 我们都可以得出以下建议: 当以改良四倍体小麦为目标时, 应以六倍体小麦为母本, 再以四倍体小麦亲本回交或优良品种(系)杂交; 当以改良六倍体小麦为目标时, 优先以四倍体小麦为母本, 再以六倍体小麦亲本回交或优良品种(系)杂交。
4 结论
虽然亲本间遗传背景会影响四倍体小麦和六倍体小麦杂种F1的自交结实率, 但相较于其他亲缘关系更远的远缘杂种F1(如小麦-黑麦杂种), 四倍体/六倍体和六倍体/四倍体小麦杂种F1结实率均相对较高, 因此对远缘杂交育种的影响较小。但是, 正反杂交后代的染色体传递是远缘杂交育种应该考虑的问题。根据D基因组染色体从F1代F2的传递规律, 四倍体小麦与普通小麦杂交育种时, 可以简单遵循以下原则进行杂交组合配置: 以四倍体小麦为最终目标时, 选择六倍体小麦作为母本进行杂交, 能够在杂种后代中更快地丢掉D基因组染色体; 以六倍体小麦为最终目标时, 选择四倍体小麦作为母本杂交, 回交或自交, 能够更快的恢复杂种后代为六倍体基因组组成。
[1] Shewry P R, Hey S. Do “ancient” wheat species differ from modern bread wheat in their contents of bioactive components?, 2015, 65: 236–243.
[2] Kihara H. Discovery of the DD-analyser, one of the ancestors of Triticum vulgare., 1944, 19: 889–890.
[3] McFadden E S, Sears E R. The artifcial synthesis of., 1944, 13: 26–27.
[4] Huang S X, Sirikhachornkit A, Su X J, Faris J D, Gill B, Haselkorn R, Gornicki P. Genes encoding plastid acetyl-CoA carboxylase and 3-phosphoglycerate kinase of the/complex and the evolutionary history of polyploid wheat., 2002, 99: 8133–8138.
[5] Klymiuk V, Yaniv E, Huang L, Raats D, Fatiukha A, Chen S S, Feng L H, Frenkel Z, Krugman T, Lidzbarsky G, Chang W, Jääskeläinen M J, Schudoma C, Paulin L, Laine P, Bariana H, Sela H, Saleem K, Sørensen C K, Hovmøller M S, Distelfeld A, Chalhoub B, Dubcovsky J, Korol A B, Schulman A H, Fahima T. Cloning of the wheatresistance gene sheds light on the plant tandem kinase-pseudokinase family., 2018, 9: 3735.
[6] He Y, Feng L H, Jiang Y, Zhang L Q, Yan J, Zhao G, Wang J R, Chen G Y, Wu B H, Liu D C, Huang L, Fahima T. Distribution and nucleotide diversity ofin wild emmer populations and Chinese wheat germplasm., 2020, 9: 212.
[7] Li A L, Liu D C, Yang W Y, Kishii M, Mao L. Synthetic hexaploid wheat: yesterday, today, and tomorrow., 2018, 4: 552–558.
[8] Hao M, Zhang L Q, Zhao L B, Dai S F, Li A L, Yang W Y, Xie D, Li Q C, Ning S Z, Yan Z H, Wu B H, Lan X J, Yuan Z W, Huang L, Wang J R, Zheng K, Chen W S, Yu M, Chen X J, Chen M P, Wei Y M, Zhang H G, Kishii M, Hawkesford M, Mao L, Zheng Y L and Liu D C. A breeding strategy targeting the secondary gene pool of bread wheat: introgression from a synthetic hexaploid wheat., 2019, 132: 2285–2294.
[9] Dvorak J, Akhunov E D, Akhunov A R, Deal K R, Luo M C. Molecular characterization of a diagnostic DNA marker for domesticated tetraploid wheat provides evidence for gene flow from wild tetraploid wheat to hexaploid wheat., 2006, 23: 1386–1396.
[10] Cheng H, Liu J, Wen J, Nie X J, Xu L H, Chen N B, Li Z X, Wang Q L, Zheng Z Q, Li M, Cui L C, Bian J X, Wang Z H, Xu S B, Yang Q, Appels R, Han D J, Song W N, Sun Q X, Jiang Y. Frequent intra- and inter-species introgression shapes the landscape of genetic variation in bread wheat., 2019, 20:136.
[11] He F, Pasam R, Shi F, Kant S, Keeble-Gagnere G, Kay P, Forrest K, Fritz A, Hucl P, Wiebe K, Knox R, Cuthbert R, Pozniak C, Akhunova A, Morrell P L, Davies J P, Webb S R, Spangenberg G, Hayes B, Daetwyler H, Tibbits J, Hayden M, Akhunov E. Exome sequencing highlights the role of wild-relative introgression in shaping the adaptive landscape of the wheat genome., 2019, 51: 896–904.
[12] Briggle L W. Transfer of resistance tof. sp.from Khapli emmer and Yuma durum to hexaploid wheat., 1966, 6: 459–461.
[13] Reader S M, Miller T E. The introduction into bread wheat of a major gene for resistance to powdery mildew from wild emmer wheat., 1991, 53: 57–60.
[14] Rong J K, Millet E, Manisterski J, Feldman M. A new powdery mildew resistancegene: Introgression from wild emmer into common wheat and RFLP-based mapping., 2000, 115: 121–126.
[15] Liu Z Y, Sun Q X, Ni Z F, Nevo E, Yang T. Molecular characterization of a novel powdery mildew resistance genein wheat originating from wild emmer., 2002, 123: 21–29.
[16] 张连松, 华为, 关海英, 李根桥, 张宏涛, 解超杰, 杨作民, 孙其信, 刘志勇. 野生二粒小麦导入普通小麦的抗白粉病基因分子标记定位. 作物学报, 2009, 35: 998–1005. Zhang L S, Hua W, Guan H Y, Li G Q, Xie C J, Yang Z M, Sun Q X, Liu Z Y. Molecular mapping of powdery mildew resistance genein wheat originated from wild emmer (var.)., 2009, 35: 998‒1005 (in Chinese with English abstract).
[17] 解超杰, 倪中福, 孙其信, 杨作民, 刘保申, 魏艳玲. 利用小麦微卫星标记定位一个来自野生二粒小麦的抗白粉病基因. 遗传学报, 2001, 28: 1034–1039. Xie C J, Ni Z F, Sun Q X, Yang Z M, Liu B K, Wei Y L. Molecular tagging of a major powdery mildew resistance genein wheat derived from wild emmer by using microsatellite maker., 2001, 28: 1034–1039 (in Chinese with English abstract)
[18] Mesfin A, Frohberg R C, Anderson J A. RFLP markers associated with high grain protein fromL.introgressed into hard red spring wheat., 1999, 39: 508–513.
[19] Lanning S P, Blake N K, Sherman J D, Talbert L E. Variable production of tetraploid and hexaploid progeny lines from spring wheat by durum wheat crosses., 2008, 48: 199–202.
[20] Eberhard F S, Zhang P, Lehmensiek A, Hare R A, Simpfendorfer S, Sutherland M W. Chromosome composition of an F2×spp.cross analyzed by DArT markers and MCFISH., 2010, 61: 619–624.
[21] Martin A, Simpfendorfer S, Hare R A, Eberhard F S, Sutherland M W. Retention of D genome chromosomes in pentaploid wheat crosses., 2011, 107: 315–319.
[22] Kalous J R, Martin J M, Sherman J D, Heo H Y, Blake N K, Lanning S P, Eckhoff J L A, Chao S, Akhunov E, Talbert L E. Impact of the D genome and quantitative trait loci on quantitative traits in a spring durum by spring bread wheat cross., 2015, 128: 1799–1811.
[23] Ren R S, Ray R, Li P F, Xu J H, Zhang M, Liu G, Yao X F, Kilian A, Yang X P. Construction of a high-density DArTseq SNP-based genetic map and identification of genomic regions with segregation distortion in agenetic population derived from a cross between feral and cultivated-type watermelon., 2015, 290: 1457–1470.
[24] Padmanaban S, Sutherland M W, Knight N L, Martin A. Genome inheritance in populations derived from hexaploid/tetraploid and tetraploid/hexaploid wheat crosses., 2017, 37: 48.
[25] Padmanaban S, Zhang P, Hare R A, Sutherland M W, Martin A. Pentaploid wheat hybrids: applications, characterisation, and challenges., 2017, 8: 358.
[26] Padmanaban S, Zhang P, Sutherland M W, Knight N L, Martin A. A cytological and molecular analysis of D-genome chromosome retention following F2-F6generations of hexaploid × tetraploid wheat crosses., 2018, 69: 121–130.
[27] Schwarzacher T, Leitch A R, Bennett M D, Heslop-Harrison J S.localization of parental genomes in a wide hybrid., 1989, 64: 315–324.
[28] Lim K B, Ramanna M, Jacobsen E, van Tuyl J. Evaluation of BC2progenies derived from 3-2and 3-4crosses ofhybrids: a GISH analysis., 2003, 106: 568–574.
[29] Huang X Y, Zhu M Q, Zhuang L F, Zhang S Y, Wang J J, Chen X Y, Wang D R, Chen J Y, Bao Y G, Guo J, Zhang J L, Feng Y G, Chu C G, Du P, Qi Z J, Wang H G, Chen P D. Structural chromosome rearrangements and polymorphisms identified in Chinese wheat cultivars by high-resolution multiplex oligonucleotide FISH., 2018, 131: 1967–1986.
[30] Luo J T, Zhao L B, Zheng J M, Li Y Z, Zhang L Q, Liu D C, Pu Z J, Hao M. Karyotype mosaicism in early generation synthetic hexaploid wheats., 2020, 63: 329–336.
[31] Du P, Zhuang L F, Wang Y Z, Yuan L, Wang Q, Wang D R, Dawadondup, Tan L J, Shen J, Xu H B, Zhao H, Chu C G, Qi Z J. Development of oligonucleotides and multiplex probes for quick and accurate identification of wheat andchromosomes., 2017, 60: 93–103.
[32] Wickham H. Ggplot2: Elegant Graphics for Data Analysis. New York, NY: Springer-Verlag, 2016.
[33] Delhaize E, James R A, Ryan P R. Aluminium tolerance of root hairs underlies genotypic differences in Rhizosheath size of wheat () grown on acid soil., 2012, 195: 609–619.
[34] Dvořák J, Gorham J. Methodology of gene transfer by homoeologous recombination into: transfer of K+/Na+discrimination from., 1992, 35: 639–646.
[35] Luo M C, Dubcovsky J, Goyal S, Dvořáket J. Engineering of interstitial foreign chromosome segments containing the K+/Na+selectivity geneby sequential homoeologous recombination in durum wheat., 1996, 93: 1180‒1184.
[36] Han C, Zhang P, Ryan P R, Rathjen T M, Yan Z H, Delhaize E. Introgression of genes from bread wheat enhances the Aluminium tolerance of durum wheat., 2016, 129: 729–739.
[37] Liu J, Huang L, Wang C Q, Liu Y X, Yan Z H, Wang Z Z, Xiang L, Zhong X Y, Gong F Y, Zheng Y L, Liu D C, Wu B H. Genome-wide association study reveals novel genomic regions associated with high grain protein content in wheat lines derived from wild emmer wheat.2019, 10: 464.
[38] Xiang L, Huang L, Gong F Y, Liu J, Wang Y F, Jin Y R, He Y, He J S, Jiang Q T, Zheng Y L, Liu D C, Wu B H. Enriching LMW-GS alleles and strengthening gluten properties of common wheat through wide hybridization with wild emmer.2019, 9: 355.
[39] Kihara H. Wheat Studies: Retrospect and Prospects (Developments in Crop Science). Tokyo: Kodansha Ltd, 1982.
[40] Sharma H C, Gill B S. Current status of wide hybridization in wheat., 1983, 32: 17–31.
[41] Friebe B, Zhang P, Linc G, Gill B S. Robertsonian translocations in wheat arise by centric misdivision of univalents at anaphase I and rejoining of broken centromeres during interkinesis of meiosis II.,2005, 109: 293–297.
Chromosome transmission in hybrids between tetraploid and hexaploid wheat
LUO Jiang-Tao1, ZHENG Jian-Min1, PU Zong-Jun1,*, FAN Chao-Lan2, LIU Deng-Cai2, and HAO Ming2,*
1Crop Research Institute of Sichuan Academic of Agricultural Sciences / Key Laboratory of Wheat Biology and Genetic Improvement on Southwestern China, Ministry of Agriculture and Rural Areas, Chengdu 610066, Sichuan, China;2Triticeae Research Institute of Sichuan Agricultural University, Chengdu 611130, Sichuan, China
Tetraploid wheat (L., AABB) and common wheat (L., AABBDD) are two main types of cultivated wheat. Transferring the genes from tetraploid wheat (or hexaploid wheat) into hexaploid wheat (or tetraploid wheat) by distant hybridization is an important method for wheat genetic improvement. However, the F1hybrid of tetraploid/ hexaploid wheat was pentaploid with unbalanced genome composition, containing two sets of genomes A and B, and only one set of genome D. The genetic divergences from both nuclear and cytoplasmic genomes of the two parents may affect the chromosome transmission efficiency of pentaploid hybrids. In the present study, tetraploid or hexaploid wheats with different genetic backgrounds were used as female or male parents to generate pentaploid F1s. The chromosome composition of F2s were analyzed by multicolor fluorescencehybridization. The results showed that the genetic background of parent lines has a significant effect on the self-setting rate of F1s. The A and B genome chromosomes were relatively stable during F1self-process, and the mean total number of A and B chromosomes per F2individual was close to 28 in both AABB/AABBDD and AABBDD/AABB F2s (27.9 vs. 28.0). However, the average number of D chromosomes retained in F2s with tetraploid wheat as female parent was significantly higher than that with hexaploid wheat as female parent (7.0 vs. 2.9). Therefore, when tetraploid wheat was the final target progeny, hexaploid wheat should be used as the primary female parent to generate F1hybrids; vice versa, tetraploid wheat should be used.
tetraploid wheat; hexaploid wheat; chromosome transmission
10.3724/SP.J.1006.2021.01067
本研究由四川省科技计划项目(2016NYZ0012, 2017JY0077, 2018JY0627), 四川省财政创新能力提升工程项目(2016ZYPZ-016)和四川省育种攻关项目(2021YFYZ0002)资助。
This study was supported by the Science and Technology Planning Project of Sichuan Province (2016NYZ0012, 2017JY0077, 2018JY0627), the Financial Innovation Capacity Improvement Project of Sichuan Province (2016ZYPZ-016), and the Sichuan Provincial Breeding Research Project (2021YFYZ0002).
郝明, E-mail: haomingluo@foxmail.com; 蒲宗君, E-mail: pzjun68@163.com
E-mail: jtluohao@163.com
2020-08-20;
2021-01-13;
2021-02-19.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20210219.1343.002.html

