苎麻纤维发育相关长链非编码RNA的鉴定与表达分析
饶晶,朱四元,严理,刘头明
(中国农业科学院麻类研究所,湖南 长沙 410205)
长链非编码RNA(Long non-coding RNA,lncRNA)通常是指不具有编码蛋白质的功能、长度大于200个核苷酸的非编码RNA转录本,在各种生物中广泛存在[1]。lncRNAs大致可分为5大类:基因间里lncRNA(lincRNAs);内含子的lncRNA(incRNAs);双向 lncRNAs;从相关基因的 DNA互补链转录的自然反义转录物(NATs);与同义链上另一转录物的一个或多个外显子重叠的同义lncRNAs[2]。lncRNA的作用机制和生物学功能极其多样,一些通过目标模仿、转录干扰、与多梳蛋白抑制复合体2(Polycomb repressive complex 2,PRC2)有关的组蛋白甲基化和DNA甲基化等机制介导基因表达。lncRNA生物学功能丰富,在生物体内以多种方式调节生物过程,比如在植物开花、雄性不育、营养代谢、生物和非生物胁迫等生物过程中起调节因子作用[3]。lncRNA的作用机制主要有4种:信号分子、诱饵分子、引导分子、支架分子[4]。以拟南芥、水稻、玉米和小麦等植物生物及一些成熟动物中lncRNA的分子研究作为参考,进一步研究非模式生物lncRNA的作用机制[5-6]。
苎麻(BoehmerianiveaL.)是世界上最古老的纤维作物之一,在中国已经种植了几千年[7]。作为我国南方重要的经济作物之一[8],苎麻纤维是一种纤维素含量极高的天然纤维,具有很高的弹性和耐磨性,被称为“天然纤维之王”。与棉花和杨树的纤维不同,从茎皮中提取的苎麻纤维是韧皮组织。苎麻纤维形成于茎皮中,其生长发育决定了苎麻茎皮厚度[9]。苎麻纤维具有许多优良的特性和重要的经济价值,其长度可达55 cm,在植物界是罕见的[10]。植物次生细胞壁(Secondary cellwall,SCWs)包含纤维素、半纤维素和木质素的生物合成。在本研究中,为了了解苎麻纤维形成的调控规律,测序鉴定可能参与SCWs生物合成的lncRNA,结合实时荧光定量PCR技术(Quantitative Real-time PCR,qRT-PCR)鉴定lncRNA在苎麻不同部位的表达量差异,旨在为苎麻纤维发育研究及品种改良提供参考。
1 材料与方法
1.1 试验材料
苎麻材料为栽培种苎麻中苎1号,是中国农业科学院麻类研究所培育的高产品种。所取材料经自来水和蒸馏水洗净后,将成熟期苎麻茎、顶皮、中皮、叶、根、芽用液氮迅速冷冻,保藏于-80℃超低温冰箱,用作RNA提取的材料。
1.2 主要试剂
植物RNA提取试剂盒 (Takara)、cDNA合成试剂盒(Takara)、荧光定量PCR试剂盒 (Takara),其他生化试剂均购自宝生物工程有限公司。
1.3 lncRNA的筛选
选取苎麻顶皮、中皮两个部位,3个重复,利用Illumina测序平台(HiSeq2500)对苎麻转录组进行测序,使用HISAT软件将转录组测序clean reads与苎麻基因组(accession ID:PHNS00000000)进行比对[11],使用StringTie程序[12]进行转录的组装,使用cufflinks比较程序[13]将所有转录本的基因组位置与参考基因组注释的已知基因进行比较[14]。使用4种方法检测转录本的蛋白质编码潜力,包括与PFAM数据库比对、利用CPC软件[15]、txCdsPredict和CNCI[16]进行预测。4种预测方法中至少有3种报告为lncRNA的转录本被鉴定为lncRNA,而lncRNA是通过cis-or trans-way靶向mRNA实现其功能[17]。通过估计Spearman相关系数和Pearson相关系数,仅考虑spearman_cor≥0.6和pearson_cor≥0.6进行后续分析。如果定位在一个10 kb区域,则认为lncRNA通过顺式调控靶向;如果没有则使用RNAplex软件分析反式作用靶标[18],参数为-e-30。
1.4 总RNA提取和cDNA第一条链合成
取约50 mg的组织样,置于液氮中研磨之后,用试剂盒法(Takara公司)提取总RNA。用超微量紫外分光光度计检测其浓度和OD值,根据超微量分光光度计定量,OD260 nm/OD280 nm值均在1.8~2.0,说明提取的总RNA纯度较高,然后用琼脂糖凝胶电泳检测其完整性,继而反转录和实时荧光定量PCR。根据逆转录试剂盒(Takara公司)说明书将总RNA(1μg)反转成cDNA,构建cDNA文库。20μL体系,反应条件:30°C 10 min,42°C 20 min,95°C 5min,反转录完成后再加无菌水稀释5倍成100μL,逆转录后的cDNA置于-80°C冰箱保存。
1.5 引物设计及合成
根据GenBank中中苎1号基因序列,采用Premier 5.00软件设计特异性引物,以苎麻18s基因作为内参基因,由擎科生物有限公司设计合成实时荧光定量PCR引物,引物序列见表1。

表1 RT-qPCR引物列表Table 1 List of qRT-PCR primers
1.6 实时荧光定量PCR验证
采用SYBRGreen荧光染料法在实时荧光定量PCR仪上进行定量。反应体系(25μL):TB GreenPremix Ex TaqII 12.5μL,cDNA 2μL,无菌水8.5μL,上、下游引物各 1μL。反应条件:95°C 30 s,95°C 5 s,60°C 30 s,40个循环后进行熔解曲线分析,以每5秒上升0.5°C的速率从65°C升高到95°C,荧光信号在循环结束时检测,每个样品做3个重复,扩增结束后进行溶解曲线分析。
1.7 统计分析
数据采用Excel进行统计和分析,相对表达量的计算以18s为内参基因,计算Ct值(Ct代表目标扩增产物达到设定阈值所经历的循环数),通过计算ΔC(ΔC=Ct目的基因-Ct内参),然后获得ΔΔC(ΔΔC=ΔC(实验组)-ΔC(对照组),得到RQ值(RQ=2-ΔΔC)(表达量变化倍数,RQ值)。用Ct值3次重复的平均值作为该基因在该处理组的表达量,并计算RQ的误差[19]。
2 结果与分析
2.1 lncRNA鉴定
转录本编码能力预测如图1,通过CPC、CNCI、txCdsPredict 3个软件和PFAM数据库比对,4种预测方法中至少有3种报告为lncRNA的转录本被鉴定为lncRNA,共得到4316个lncRNA。测序得到可能参与SCW生物合成的8个靶基因,进一步分析靶基因的功能注释如表2,whole_GLEAN_10005546、whole_GLEAN_10005548、whole_GLEAN_10025325、whole_GLEAN_10016451 4个基因属与拟南芥MYB(v-mybavianmyloblastosisviraloncogenehomolog)蛋白,whole_GLEAN_10012500属于拟南芥NAC(NAM,ATAF1/2和CUC2)蛋白,whole_GLEAN_10022328属于拟南芥BLH(Bel like homeodomain 1)蛋白,whole_GLEAN_10020282属于拟南芥LOB(Lateralorganboundaries)蛋白,whole_GLEAN_10008444属于拟南芥IRX(Irregularxylem)蛋白。8个靶基因均为拟南芥纤维发育同源基因,这些靶基因被lncRNA主要以cis、Lnc-AntiOverlap-mRNA、trans和Lnc-Overlap-mRNA 4种靶向方式调控。再根据8个靶基因选出靶向调控他们的10个lncRNAs做进一步结构分析。由表3可知,10个候选基因有1~2个外显子;转录本长度最短的为239 bp,最长的为1401 bp。

图1 预测结果韦恩图-lncRNAFig.1 Predicted results:Venn graph-lncRNA

表2 lncRNA靶基因的功能注释Table 2 Functional annotation of target genes of lncRNA
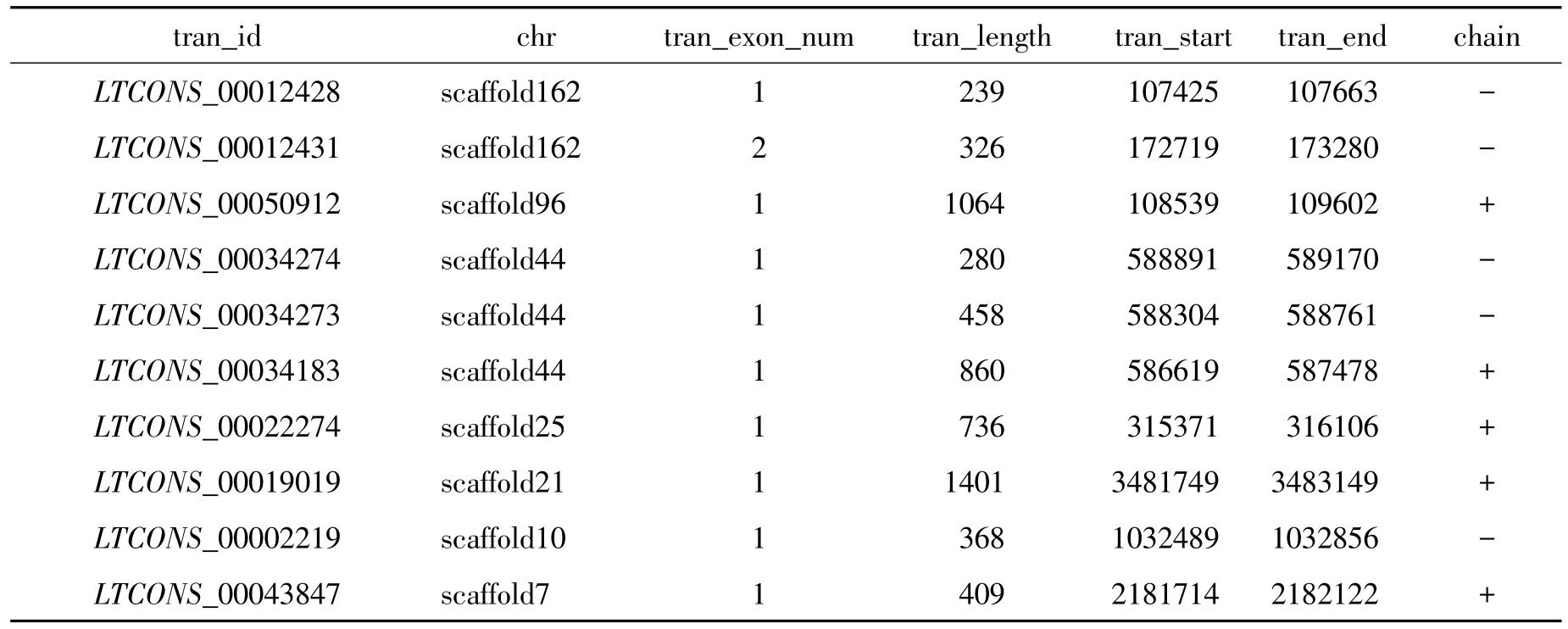
表3 lncRNA结构统计结果Table 3 Statistical results of lncRNA structure
2.2 荧光定量PCR扩增曲线和熔解曲线分析
为了检测扩增产物的特异性,避免qRT-PCR扩增过程中非特异性扩增产物和引物二聚体产生的荧光信号造成假阳性结果[20-21],本文对扩增产物进行熔解曲线分析[22]。由图2可知,18s基因和10个候选基因在中苎1号不同器官中的扩增产物熔解曲线为单一的峰,无非特异性扩增产物及引物二聚体产生,说明设计的引物符合试验要求,扩增体系、退火温度及循环程序均适合。

图2 18s和10个候选基因的溶解曲线分析Fig.2 Dissolution curve analysis of 10 candidate genes and 18s
2.3 lncRNA表达差异分析
理想的内参基因应具备下列特性:在所有组织和细胞类型中表达、在所有的环境和试验条件下稳定表达、具有与目标基因相似的稳定表达水平[23]。本研究利用苎麻18s基因作为内参,该基因在苎麻茎、顶皮、中皮、叶、根、芽6个部位都可以表达。从表4来看,该基因在不同部位标准误较小(0.109~0.537),变异率(C.V)也较小(1.056~6.254)。因此 18s基因可以作为内参进行苎麻RT-qPCR相对定量分析研究。

表4 内参基因18s在不同部位中的表达稳定性Table 4 The expression stability of reference gene 18s in different sites
采用表达量变化倍数(RQ值)对10个lncRNA基因在中苎1号苎麻品种中不同器官间的表达情况进行分析。以中苎1号顶皮的表达作为对照,设定表达量为1。由图3可知,LTCONS_00012428在茎中表达量约为顶皮部位中的42倍,LTCONS_00022274在茎中表达量约为顶皮部位中的29倍,LTCONS_00034183在茎中表达量约为顶皮部位中的7倍,这3个基因在中苎1号不同部位间的表达量不同,在茎中的表达量最高,而在其他部位的表达则较少。LTCONS_00012431在中苎1号不同部位的表达量不同,在叶中的表达量最高,约为顶皮部位中的10倍,芽次之,表达量约为顶皮部位中的9倍,在茎与中皮中也有差异表达,而在根中表达则较少。LTCONS_00034274在芽中表达量约为顶皮部位中的18倍,LTCONS_00002219在芽中表达量约为顶皮部位中的1.2倍,LTCONS_00050912在芽中表达量约为顶皮部位中的19倍,LTCONS_00019019在芽中表达量约为顶皮部位中的21倍,这4个基因在中苎1号不同部位间的表达量不同,在芽中的表达量最高,而在其他部位的表达相对较少。LTCONS_00043847在中苎1号不同部位的表达量不同,在顶皮部位表达量最高为1,芽次之,约为顶皮部位的0.7倍,而在其他部位的表达则较少。LTCONS_00034273在茎中表达量高达顶皮部位中的3100倍。总之,他们在不同部位的表达量差异明显。
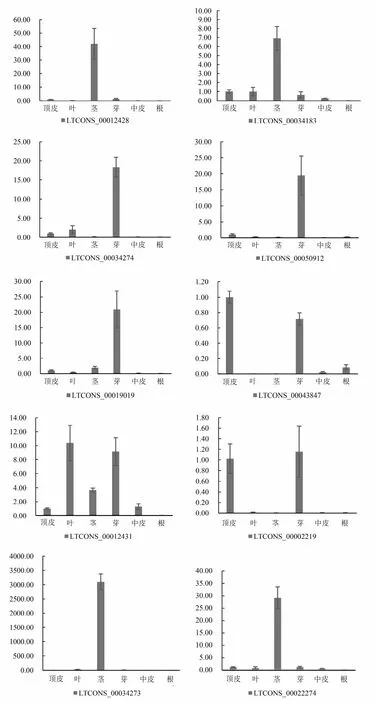
图3 10个lncRNA在不同部位的差异表达分析Fig.3 Analysis of differential expression of 10 lncRNAs in different positions
2.4 纤维发育相关lncRNA鉴定
由于顶皮与中皮纤维发育差异明显,中皮纤维处于生长期,次生壁正在加厚,而顶皮纤维尚未起始生长,若两个部位有差异表达则认为可能与纤维发育有关。根据顶皮与中皮纤维发育差异特征,将10个lncRNA在顶皮、中皮两个部位进行荧光定量分析,如图3所示,9个lncRNA在两个部位表现出明显差异,推测他们与苎麻纤维发育相关。1个lncRNA在两个部位表现差异较小,推测他们与苎麻纤维发育无关,具体表述如下:LTCONS_00012428在顶皮表达量是中皮的21.2倍;LTCONS_00022274在顶皮表达量是中皮的2.2倍;LTCONS_00034183在顶皮表达量是中皮的3.8倍;LTCONS_00034274在顶皮表达量是中皮的31.5倍;LTCONS_00043847在顶皮表达量是中皮的48.3倍;LTCONS_00050912在顶皮表达量是中皮的92.8倍;LTCONS_00034273仅在顶皮中有表达;LTCONS_00002219在顶皮表达量是中皮的172.3倍;LTCONS_00019019在顶皮表达量是中皮的8.8倍;LTCONS_00012431在顶皮表达量是中皮的0.8倍。可以看出LTCONS_00012431在这两个部位差异不明显,推测其与苎麻纤维发育不相关。
3 讨论与结论
lncRNA以多种方式参与基因的表达调控,在其效应过程中也可能涉及蛋白质、DNA或 RNA等多种相关因子[24]。现在研究广泛认为lncRNA能在转录及转录后水平调控基因表达,从而调控生物的生长发育[25]。植物lncRNA的研究目前已在拟南芥、蒺藜苜蓿、水稻和玉米中进行了全基因组lncRNA的检索及相关研究[26-27],其他物种的lncRNA研究表明其在生物体内起着重要的作用。本文首次从苎麻中测序得到4316个lncRNA,从4316个lncRNA选取10个lncRNA做表达谱分析,发现其中3个lncRNA在茎中的表达量最高,其原因可能是苎麻茎中纤维含量丰富;而1个基因在芽表达量最高,可能是该基因在该部位含量较丰富导致;5个基因在叶表达量最高,可能与苎麻叶中的纤维含量有关;1个基因在顶皮的表达量最高,在其他部位的表达则较少,试验中苎麻顶皮纤维尚未起始生长,可能由于该基因在该部位含量较丰富导致。总之,10个候选基因中大多数基因集中在叶、茎部位表达,少数基因集中在顶皮、芽部位表达。进一步研究其与纤维发育的关系发现,其中9个基因在顶皮、中皮两个部位差异表达明显,推测其可能与纤维发育相关。而靶基因测序得到10个lncRNA所对应的8个靶基因是拟南芥纤维发育基因同源基因,大部分属于MYB、NAC等蛋白,在拟南芥中,目前认为SCW主要是由一系列NAC转录因子和MYB转录因子形成分层次的网络逐级调控下游次生壁中纤维素、半纤维素和木质素的合成[28]。在拟南芥纤维发育调控中,NAC基因主要调控MYB转录因子再逐级调控完成整个SCW的生物合成过程。文献[29]报道LOB(lateralor-ganboundaries)基因通过与转录因子、激素等作用参与拟南芥SCW的形成。而拟南芥基因IRX15(IRREGULAR XYLEM 15)对于正常木聚糖在次级细胞壁中的沉积至关重要[30]。拟南芥BLH1(BEL1-like homeodomain)基因通过与蛋白质的相互作用调节拟南芥从营养期到生殖期,从而参与纤维发育[31]。故而推测差异lncRNA基因主要通过靶向他们各自的靶基因从而调控纤维的发育。

