普通小麦籽粒硬度的全基因组关联分析
胡文静,张 晓,刘 巧,方正武,高德荣,
(1. 江苏里下河地区农业科学研究所/农业部长江中下游小麦生物学与遗传育种重点实验室,江苏扬州225007; 2.扬州大学/江苏省粮食作物现代产业技术协同创新中心,江苏扬州225009; 3.长江大学农学院,湖北荆州 434025)
籽粒硬度是决定小麦市场分级、碾磨性能和最终用途的重要指标和依据[1]。根据籽粒硬度性状的不同,可以将小麦分为软麦、混合麦和硬麦。不同硬度类别的小麦具有不同的使用价值。硬质麦面粉颗粒度大,破损淀粉含量高,吸水能力较强,适合制作面包和优质面条等食品;软质麦面粉颗粒度较小,破损淀粉含量低,吸水能力较弱,适合制作饼干和糕点等食品[2]。籽粒硬度对小麦磨粉品质、淀粉品质、蛋白质品质和加工品质都有显著的影响[3-5]。赵 新等[6]发现,随着硬度指数的增加,小麦面粉的出粉率和蛋白质含量逐渐升高,淀粉含量逐渐降低,同时籽粒硬度增加对面包品质的改良效果比面条品质更为明显,原因可能与硬度较大的硬质小麦面团具有较高的弹性和较弱的延展性有关。吴宏亚等[7]研究发现,在一定范围内,出粉率和灰分含量与籽粒硬度的变化趋势成正比。姜兰芳等[8]发现,小麦籽粒硬度指数与出粉率、吸水率和最大拉伸阻力均呈极显著正相关。夏云祥等[9]发现,除蔗糖溶剂保持力(solvent retention capacity,SRC)外,籽粒硬度与小麦粉水SRC、碳酸钠SRC和乳酸SRC均呈极显著相关。多数研究结果表明,不同puroindoline基因型间籽粒硬度存在显著差异[10-12]。
籽粒硬度由1对主效基因和一些微效修饰基因控制,主效基因puroindolinea(Pina)和puroindolineb(Pinb)位于5DS染色体上,已发现的微效基因主要位于1A、2A、2D、3A、5A、5B和6D染色体上[13-17]。Wang等[18]利用“超软”小麦构建的重组自交系(recombinant inbred line,RIL)群体发现,控制软质小麦籽粒硬度和出粉率的2个主效位点Qkha.orr-4B和Qkha.orr-4D分别位于4B和4D染色体上,与小麦矮秆基因Rht-B1和Rht-D1位置接近,可解释籽粒硬度20%~34%的表型变异。
小麦双亲群体连锁分析理论上可以应用于所有性状的遗传解析,但受遗传群体亲本的差异度和群体大小等限制,定位QTL的来源和数目有限[19]。而关联分析则是以自然群体中的连锁不平衡(linkage disequilibrium,LD)为基础,鉴定目标性状与遗传标记或者候选基因之间非随机关联程度,从而确定关联位点[20]。由于关联分析研究对象为自然群体,材料相对广泛,是解析复杂性状和鉴定目标性状优异等位变异的有效方法[21-22]。该技术已在大豆、玉米、水稻等作物遗传研究应用较多[23-24],在小麦的产量、品质和抗病性等遗传研究上也被广泛采用[25-28]。
近年来,籽粒硬度这一品质特性已越来越引起小麦育种工作者和面粉加工企业的重视,张 晓等[29]认为,籽粒硬度可以作为品质育种高效实用的选择指标,优质弱筋小麦育种需加强软质基因型的筛选。而以自然群体为材料用关联分析的方法对小麦籽粒硬度基因开展的研究还未见报道。本研究以171个小麦品种组成的自然群体为材料,利用小麦90K SNP芯片对籽粒硬度进行全基因组关联分析(genome-wide association study,GWAS),以期发掘与小麦籽粒硬度显著关联的SNPs位点,为小麦硬度的分子标记辅助育种提供依据。
1 材料与方法
1.1 供试材料
供试材料来自中国农业科学院和江苏省农科院,由江苏里下河地区农业科学研究所搜集,包括国内外171个小麦品种,其中,3个分别来自意大利、墨西哥和日本,其余168个分别来自我国陕西、河北、北京、山西、山东、河南、安徽、江苏、湖北、湖南、四川和广州17个省市[31]。供试材料于2017-2018和2018-2019年度分别种植于江苏里下河地区农业科学研究所万福基地和江苏省高邮市小麦试验基地(简称2018YZ、2018GY、2019YZ和2019GY),秋播时0~20 cm耕层土壤有机质含量16.57 g·kg-1,碱解氮含量71.89 mg·kg-1,速效磷含量17.65 mg·kg-1,速效钾含量59.28 mg·kg-1,全氮含量1.23 g·kg-1,全磷含量0.59 g·kg-1,全钾含量11.98 g·kg-1。试验当年小麦播期为10月25日,全生育期施270 kg·hm-2纯氮,基肥∶壮蘖肥∶拔节肥= 5∶4∶1,基肥于播种前一天施用,壮蘖肥于4叶期施用,拔节肥于叶龄余数2.5叶期施用。P2O5、K2O肥施用量均为105 kg·hm-2,全部基施。试验采用随机区组设计,3行区,2次重复,每行60粒,行长 1.33 m,行距0.23 m。生育期田间管理同当地大田生产,及时防治病虫草害,成熟期分区收获并清理籽粒样本,无穗发芽发生。为了减少误差,将经清理的2个重复的种子等量混合作为最终试验籽粒样本。
1.2 籽粒硬度测定
采用瑞典波通(Perten)仪器公司的单粒谷物特性测定仪(SKCS-4100)测定供试材料的籽粒硬度,2次重复,取平均值。
1.3 表型数据统计
采用SPSS 22.0和Excel 2019对表型数据进行处理和统计分析。
1.4 基因型分析
运用小麦90 K SNP芯片(81 857个SNP)对171个小麦品种进行基因分型。人工对分型结果进行质量控制,剔除数据缺失率>10%和最小等位基因频率(minor allele frequency,MAF)< 0.05的SNP标记,保留高质量的SNP标记进行关联分析。参考Hu等[30]的方法利用Tassel v5.0对171个小麦品种的基因型进行LD分析,根据LD分析结果选用平均分布于21条染色体上的1 676个标记(r2<0.2),利用Structure 2.3.4进行群体结构分析,采用Tassel v5.0进行亲缘关系评估(kinship matrix,K matrix)运算。
1.5 全基因组关联分析
在考虑群体结构和亲缘关系的情况下(Q+K),应用Tassel v5.0中的混合线性模型(mixed linear model,MLM)进行籽粒硬度性状与SNP标记之间的关联分析。在全基因组范围内检测与目标性状显著关联的SNP标记位点的分布情况(如位于哪一染色体及其在对应染色体上的位置)及其对应的-log10(P)值。当标记的-log10(P)≥3,即P≤0.001时认为标记与性状存在关联。结果中多个标记是否位于同一位点由对应基因组的LD衰减距离决定。为了与前人结果比较,将连锁标记或者基因的序列与EnsemblPlants数据库(http://plants.ensembl.org/)中的中国春参考基因组序列进行比对,获得连锁标记或者基因的物理位置。
2 结果与分析
2.1 基因型分析
参考Hu等[30]的分析结果,基因型过滤后有23 556个SNPs。利用平均分布于21条染色体上的1 676个标记(r2<0.2)进行群体结构分析,结果(图1)表明,供试的171个小麦品种可分为2个亚群。亚群1包括99个小麦品种,主要来自安徽、江苏、河南、陕西和湖南;亚群2包括72个小麦品种,主要来自河南、江苏、山东和山西。LD衰减距离分析结果表明,自然群体中A、B和D基因组分别含有39.7%、50.0%和24.0%的显著性标记,LD衰减距离分别为10、9.5和12 Mb,全基因组的LD衰减距离为10.5 Mb。
2.2 小麦籽粒硬度的变异
171个小麦品种在2018YZ、2018GY、2019YZ和2019GY四个环境下的硬度平均值分别为27.41、30.73、24.15和26.88,变异系数为56.59%~66.80%,说明供试材料的籽粒硬度的变异范围广,具有较大的选择潜力。供试小麦品种的籽粒硬度在不同环境之间呈极显著正相关,相关系数范围为0.88~0.92(表1)。
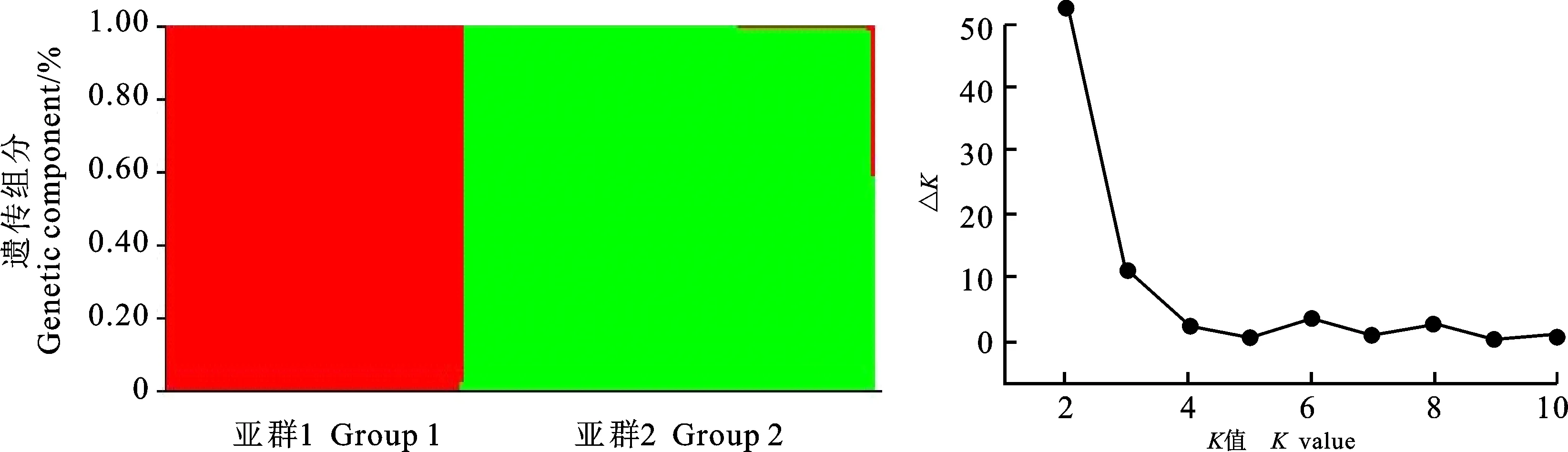
图1 171个小麦品种的群体结构分析Fig.1 Population structure analysis of 171 wheat cultivars
2.3 小麦品种籽粒硬度的全基因组关联分析(GWAS)
结果(图2,表2)表明,与小麦籽粒硬度显著关联的SNPs有20个,分别位于1A(5个)、1B(4个)、1D(1个)、2A(1个)、5A(3个)、7A(4个)和7B(1个)染色体上,可解释6.76%~ 11.79%的表型变异。其中,在2个及以上环境中检测到的SNP标记有14个,参考Hu等[30]前期LD 衰减距离分析结果,即同一条染色体上显著性标记之间物理距离小于对应基因组的LD衰减距离可以视为同一位点,将这14个SNP标记可归纳为7个位点,分别位于1A、1B、1D、2A、5A和7A染色体上,可解释 6.76%~11.79%的表型变异,其中,1A染色体上有2个位点,其他5条染色体上分别有1个位点。14个SNPs中表型变异率超过10%的有4个,其中,1D染色体上的标记 wsnp_Ku_c19622_29138795在3个环境中能被检测到,可解释9.08%~11.79%的表型变异;2A染色体上的标记Excalibur_c12675_1789在4个环境中均能被检测到,可解释9.10%~10.86%的表型变异;5A染色体上的标记wsnp_Ra_c24707_34262900和BS00041219_51在3个环境中能被检测到,可解释6.76%~10.35 %的表型变异。
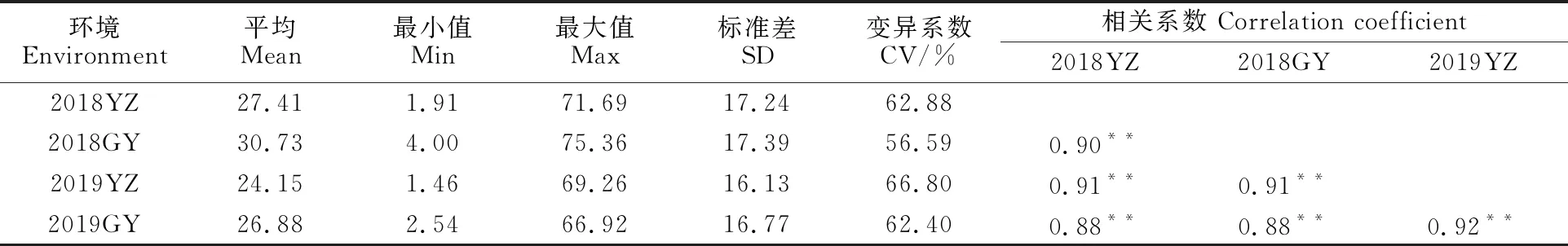
表1 171个小麦品种籽粒硬度的描述性统计分析Table 1 Descriptive statistics of grain hardness of 171 wheat varieties

图2 全基因组关联分析的曼哈顿图Fig.2 Manhattan plots for genome-wide association study
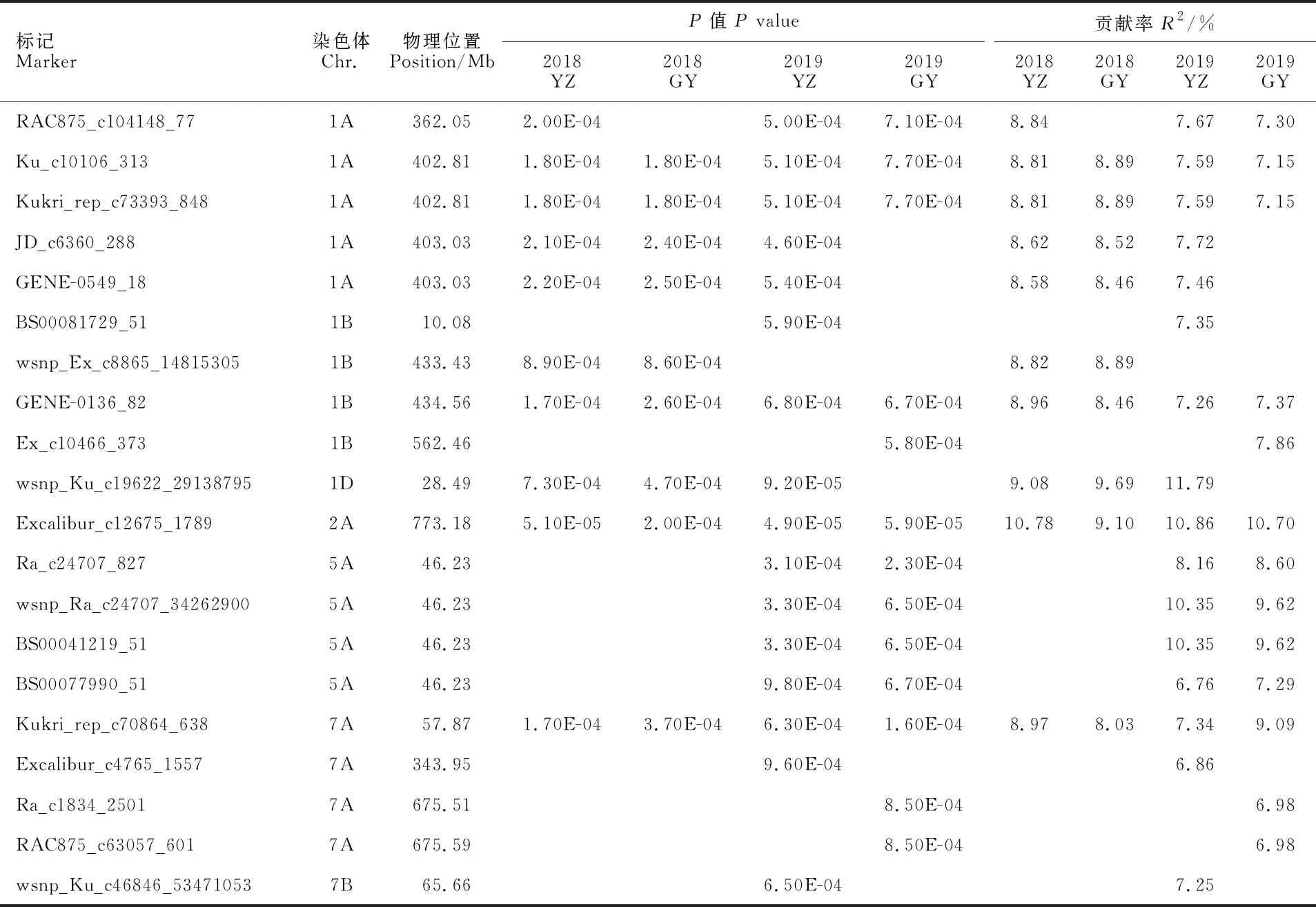
表2 与171个小麦品种籽粒硬度显著关联的SNPs标记Table 2 SNPs significantly associated with grain hardness in 171 wheat varieties
3 讨 论
4个环境下小麦籽粒硬度的统计学分析结果表明,自然群体的表型变异范围广,具有较大的选择潜力,环境对该表型影响较小,说明小麦籽粒硬度主要受遗传因素控制,应在育种的早期世代对其进行选择,这与前人研究结果一致[31]。本研究利用90K SNP芯片对171个小麦品种在四个环境下的籽粒硬度进行全基因组关联分析,共检测到20个与籽粒硬度显著相关的SNPs,其中,在2个及以上环境中检测到的SNPs有14个,共7个位点,分别位于1A(362.05 Mb、402.81~403.03 Mb)、1B(433.43~434.56 Mb)、1D(28.49 Mb)、2A(773.18 Mb)、5A(46.23 Mb)、7A(57.87 Mb)染色体上,可解释6.76%~ 11.79%的表型变异。14个SNPs中表型变异率超过10%的有4个,分别位于1D、2A和5A染色体上,可以优先考虑选择这些贡献率较大的位点进一步精细定位、图位克隆及育种标记开发,检测软质或者硬质相关分子标记来选择相应的小麦品种,进而改良小麦籽粒硬度。
前人研究结果表明,小麦籽粒硬度不仅受到5DS染色体上主效基因(Ha)控制,还受到其他染色体上微效基因的影响[13-17,32]。李文福等[33]以小麦品种花培3号、豫麦57构建的DH群体和IF2群体为材料,利用368个SSR标记定位了控制硬度的QTL位点,分别位于1B、2A、4B、5A、6A和7D染色体上,经比对,发现5A染色体上的QTL与本研究在5A染色体上定位到的显著性位点物理位置一致,推测是同一位点。王雪荣[34]利用205个小麦品种构成的自然群体,结合SNPs、DArt和SSR标记构建复合遗传图谱,在三个环境中检测到128个与籽粒硬度相关和29个与硬度指数相关的位点,其中1D染色体上的标记Kukri_c3150_1519、wsnp_Ku_c3682_6786230与本研究检测到的wsnp_Ku_c19622_29138795位置接近,推测是同一位点。杨 林等[35]利用小麦中国春(母本)和兰考大粒(父本)F2:3家系构建的含有169个多态性分子标记的遗传连锁图谱,共检测到17个与小麦籽粒硬度性状显著相关的QTL,并在1B染色体上发现存在同时控制籽粒容重、硬度、蛋白和结合水含量的QTL,本研究也在1B染色体上发现4个与硬度性状显著相关的标记,包含1个稳定表达的位点,与前人研究物理位置一致,推测是同一位点,也证实了1B染色体对小麦品质的显著影响。
本研究发现在所有环境下与小麦籽粒硬度性状均相关的位点有4个,分别位于1A(402.81~403.03 Mb)、1B(434.56 Mb)、2A(773.18 Mb)和7A(57.87 Mb)染色体上,但是只有2A染色体上的位点对小麦籽粒硬度的贡献率超过10%,值得进一步研究。经过比对,本研究在1A、2A、7A和7B染色体上检测到的与小麦籽粒硬度显著关联的位点不同于前人研究结果,推测是新的籽粒硬度基因位点。
4 结 论
本研究利用171个小麦品种组成的自然群体,结合覆盖小麦全基因组的90K SNP芯片,对小麦籽粒硬度进行全基因组关联分析,共检测到20个显著关联的SNPs,包含12个位点,在2个及以上环境中能被检测到的共有7个位点,其中,表型变异率超过10%的有3个,分别位于1D、2A和5A染色体上,在所有环境下均能被检测到的位点有4个,分别位于1A、1B、2A和7A染色 体上。

