水稻小穗簇生基因OsCL6的定位及候选基因分析
刘维 陆展华 卢东柏 王晓飞 王石光 薛皦 何秀英
水稻小穗簇生基因的定位及候选基因分析
刘维 陆展华 卢东柏 王晓飞 王石光 薛皦 何秀英*
(广东省农业科学院 水稻研究所/广东省水稻育种新技术重点实验室, 广州 510640;*通信联系人, E-mail: xyhe@163.com)
【】通过对水稻小穗簇生基因的定位与候选基因分析,为进一步克隆幼穗发育过程中缩短枝梗长度造成小穗簇生的功能基因奠定基础。以航天诱变育种技术处理常规优质稻品种粤农丝苗获得一个稳定遗传的小穗簇生突变体为试验材料,与粤金银占构建F1、F2作图群体,对簇生基因进行定位,结合转录组测序对候选基因进行预测与评价。遗传分析结果表明,突变体的簇生性状由非完全显性单基因控制,混合分组分析法将该基因定位于第6染色体上约416 kb的区间。进一步通过表达谱分析、转录组测序技术、基因序列差异性分析和qRT-PCR验证等筛选出为最佳候选基因,其5′-UTR区域发生9个碱基的插入突变,暗示该基因可能通过二级结构改变调控转录或翻译水平,参与水稻幼穗发育过程中枝梗的分化。【】本研究结果为解析水稻二次枝梗和小穗梗缩短机制奠定一定理论基础。
航天诱变;小穗簇生;基因定位;转录组测序;候选基因
水稻(L.)不仅是世界上最重要的粮食作物,还是禾本科作物分子生物学研究领域重要的模式植物[1-2]。穗是水稻产量形成的关键部位,穗型是株型的重要组成部分[3-4]。水稻穗的形成过程不仅是植株形态建成中的重要事件,也是决定水稻能否顺利完成正常生殖发育的重要阶段之一,并且穗部结构与水稻产量息息相关[5]。水稻穗部形态主要由枝梗分化和小穗的着生位置决定[6],它的形成涉及营养分生组织、花序分生组织及花分生组织之间复杂的转化过程[7]。目前,对双子叶模式植物花序发育的生物学过程研究很详细,但是对单子叶植物,特别是水稻穗发育过程的研究进展十分缓慢。
现今主要通过同源基因的克隆和不同类型的突变体来研究水稻幼穗发育调控机制[8],Kobayashi等根据控制发育的阶段将它们分为3类[9]。第1阶段的基因突变可能阻断茎端分生组织向花序分生组织转化,如基因能导致花序分生组织的不确定性,超量表达基因会出现抽穗期提前,干扰基因表达植株开花期延迟,引起短穗和穗部枝梗减少,严重时表现为不抽穗[10];基因编码F-box蛋白,超量表达植株表现出花序分生组织转变时间延长,干扰表达植株花序分生组织转变时间提前[11-13];被称为盒基因,编码类磷脂酰乙醇胺结合蛋白,在花分生组织的决定中发挥重要作用,过表达的水稻幼苗表现出节间伸长提前、地上部的冠状根发育、株高变矮以及早花等早熟表型,侧芽发育也加速,芽会频繁生长成有效分蘖,突变体在形成花的位置形成幼苗,同时影响穗轴和枝梗伸长[14-15]。第2阶段基因主要维持花序分生组织的特性并控制其向花分生组织转化,突变将影响穗的发育。(IDEAL PLANT ARCHITECTURE1)基因编码类Squamosa启动子结合蛋白,在营养生长期控制水稻分蘖,在生殖生长期高表达促进穗分枝,在稻瘟病菌侵染时受诱导磷酸化提高免疫反应,既能增产又能提高稻瘟病抗性[16-18]。表达量降低,影响OsER1-OsMKKK10-OsMKK4- OsMPK6信号通路,引起花序分裂组织中细胞分裂素积累,进而增加颖花数,提高水稻产量[19-20]。第3阶段基因主要在花器官发育过程中起作用。水稻中的A类基因、在各分生组织和各轮花器官原基中均表达,调控水稻开花启动并决定花器官原基的特性,过表达可使开花提前,并加速茎分生组织分蘖[21-24]。水稻中的B类基因、、调控浆片和雄蕊的发育[25-27],水稻中的C类基因、调控心皮和雄蕊发育[28-29]。水稻中的D类基因仅在心皮壁内部细胞和胚珠原基中表达,功能缺失突变体雄性不育,胚珠转变成心皮状结构,心皮内部出现心皮[30-31],在胚珠和心皮壁中均表达,突变体没有明显表型,不能正常控制胚珠特征的决定[32]。水稻中E类基因、、(与同源)、(与同源)、在各轮花器官中重叠表达,影响花器官的发育[33]。虽然上述大部分基因已被克隆,但是控制前两个阶段的基因数量少且很多与性状间不存在连接点即缺少相应的突变体,因此不能完全确认其在水稻发育中的功能,限制了人们对水稻幼穗分化发育分子机制的理解。近年来的研究发现植物内源激素和许多microRNA(miRNA)也在穗型发育的枝梗分化过程中影响广泛而调控机制简约。有研究者找到一条miR396控制水稻枝梗有无的通路[34],但是关于水稻枝梗缩短的机制仍然知之甚少。
航天诱变育种技术又称空间诱变育种或航天诱变育种,是指用航天器(返回式卫星、高空气球、航天飞机、宇宙飞船等)将农作物种子带到一定的空间环境,诱导作物种子发生突变和变异,再在地面选育新材料、新种质,培育新品种的作物育种新方法[35-36]。粤农丝苗由广东省农业科学院水稻研究所育成,是当前广东省丰、抗、优综合最好的水稻品种之一,分别被列为广东省和广州市优质稻主导品种[37-38],通过了海南、广西、湖北、湖南、江西、安徽、河南等多个省份品种审定或引种许可,以其为父本配组的杂交稻组合超过30个,且多个组合通过国家品种审定(数据来自国家水稻数据中心网站)。2011年通过神州八号飞船搭载粤农丝苗干种子100粒进行航天诱变育种试验,从诱变2代中获得一个花序结构异常、二次枝梗和小穗梗严重缩短,顶端小穗3粒簇生在一起的小穗簇生突变体材料(clustered spikelets 6)。前期对其开展了农艺性状调查、花粉育性观测,发现该突变体农艺性状与粤农丝苗极为相似,熟色好,产量高,但花序结构异常,一次枝梗数增加,二次枝梗和小穗梗缩短。细胞学观测发现差异主要由幼穗分化的第Ⅲ期到第Ⅳ期发育异常所致,遗传分析表明该性状由1对半显性基因控制[39-40]。在此基础上,为了深入解释小穗发育过程中枝梗缩短的分子机理,本研究利用混合群体分离分析(bulked segregate analysis, BSA)和转录组测序法对小穗簇生基因进行定位及候选基因分析,筛选造成突变体小穗簇生性状的候选基因。
1 材料与方法
1.1 试验材料
利用航天诱变育种技术处理粤农丝苗,田间筛选得到小穗簇生突变体,自交,簇生性状能多代稳定遗传,命名为,中文名为粤花占1号,已获得农业部植物新品种权。粤金银占是由广东省农业科学院水稻研究所选用的粤金油占与粤银丝苗杂交而成的感温型常规稻品种,抗倒力强,后期熟色好,高抗稻瘟病。将突变体与粤金银占杂交构建F2定位群体约1000株。所有材料种植于广东省农业科学院水稻研究所大丰试验基地,插植规格为20 cm×16.7 cm,常规肥水管理。
1.2 分子标记定位基因
以粤金银占为母本,为父本杂交得到F1代,再自交得到F2代定位群体971株。用CTAB法提取两个亲本及分离群体叶片DNA保存于−4℃冰箱中备用。选取均匀分布于水稻12条染色体上的600对SSR标记,在两个亲本间进行多态性筛选,找到具有多态性的标记。采用BSA法从粤金银占/F2分离群体中随机选取小穗簇生和正常穗型各15株单株DNA,等量混合构建突变型和野生型DNA混池。利用多态性分子标记对两个DNA混池进行基因型分析并筛选出连锁标记。最后用筛选到的连锁标记对129株隐性单株DNA进行关联分析,完成基因定位。
PCR扩增仪为C1000TM热循环仪,反应体系为15.0 µL:5 U/µLDNA聚合酶0.5 µL,10×缓冲液(含Mg2+)1.5 µL,DNA模板1.0 µL,dNTP 0.5 µL,正、反向引物各0.3 µL。扩增反应程序如下:94℃下预变性3 min;94℃下30 s,55℃下30 s,72℃下45 s,35个循环;最后72℃下延伸10 min。6%变性聚丙烯酰胺凝胶电泳(北京市六一仪器厂垂直电泳系统),银染显色,Gel DocTM XR+凝胶成像系统观察拍照。统计交换个体数,按照重组频率=(双交换个体×2+单交换个体)/(总个体数×2)×100的公式计算重组频率,用Kosanbi函数将重组频率转化成遗传距离。根据Gramene网站上提供的水稻基因组序列信息构建物理图谱。
1.3 转录组测序
由于小穗发育第Ⅰ期(第一苞形成)到第Ⅲ期(二次枝梗与小花分化)二次枝梗和小穗梗均未伸长,第Ⅲ期(二次枝梗与小花分化)到第Ⅳ期(雌雄蕊形成)二次枝梗和小穗梗基本无伸长而野生型粤农丝苗按比例伸长,差异极显著。故分别剥离野生型和突变体小穗发育第Ⅲ期和第Ⅳ期的幼穗各3份保存于液氮中,快速取出加入10倍体积的TRIZOL(Invitrogene)提取总RNA,溶于去RNA酶ddH2O,置于1.5 mL无酶离心管中,使用Parafilm封住管口。将提取的总RNA用干冰送至北京百迈客生物科技有限公司,公司使用Nanodrop 2000与Agilent 2100检测总RNA质量、纯度、完整性等,检测合格的样品使用AMARTer PCR cDNA合成试剂盒合成全长cDNA,构建质量合格的文库。基于边合成边测序(Sequencing By Synthesis,SBS)技术,采用Illumina HiSeq高通量测序平台对cDNA文库进行测序,过滤得到Clean Data,与日本晴参考基因组进行序列比对,获得差异表达基因信息,并对差异表达基因进行基因聚类、基因功能注释、基因富集及蛋白互作网络分析。
1.4 候选基因预测
参考日本晴的基因组序列,利用水稻基因组注释数据库,根据分子标记与目标基因之间的物理距离和遗传距离,在定位区段上获取目的基因信息。从公共数据库CREP中查找候选基因在籼稻品种明恢63不同发育时期的表达数据,进一步筛选出目标基因。
1.5 候选基因表达量分析
取野生型和突变体幼穗发育Ⅲ期到Ⅳ期转变时期的幼穗样品,使用RNeasy Plus Mini Kit(QIAGEN公司,德国)提取RNA,用甲醛凝胶电泳检测其完整性,使用ReverTra Ace qPCR PCR Kit(TOYOBO公司,日本)反转录试剂盒反转录合成第一链cDNA后备用。利用试剂盒SYBR qPCR Mix Kit(TOYOBO公司,日本)和实时荧光定量PCR仪(ABI 7500),以Ⅰ(GeneBank登录号:NM_001058705)基因作为内参,分析预测候选基因在野生型和突变体中的相对表达量。
反应体系为20.0 µL:10 µL 2×SYBR qPCR混合液,2 µL cDNA模板,正反引物各0.8 µL,ddH2O补至20 µL。扩增反应程序:95℃下30 s;95℃下5 s,55℃下30 s,40个循环;72℃下1 min。3次生物学和技术重复,以2–∆∆Ct计算候选基因的表达量。
1.6 候选基因序列分析
针对候选基因启动子和编码区段,利用引物设计软件Primer Premier 5.0分段重合设计引物,扩增出候选基因的基因组序列,扩增产物委托广州擎科生物技术有限公司进行测序。测序结果利用DNAstar 5.0软件拼接组合,同时利用DNAMAN 6.0.3.99软件对比基因序列。
2 结果与分析
2.1 簇生突变体cl6的表型鉴定及基因定位
农艺性状与粤农丝苗极为相似,在生育期、株高、有效穗数、穗长、一次枝梗长、二次枝梗数、每穗粒数及产量这些农艺性状上没有明显差异;而一次枝梗数、结实率及籽粒长宽比的差异均达到显著水平,特别是二次枝梗长和小穗梗长比粤农丝苗严重缩短(图1)。
以粤金银占为母本,为父本杂交得到F1代,自交得到F2代定位群体971株。选取均匀分布于水稻12条染色体上的600对SSR标记,在两个亲本间进行多态性筛选,找到具有多态性的标记136对。根据多态性结果,经3轮连锁分析,将基因定位在第6染色体分子标记RM20315和RM20341之间约3.88 cM的区域内,并将其命名为。利用定位所用的分子标记并通过BLADTN分析,构建的物理图谱(图2),侧翼标记RM20315和RM20341分别锚定物理距离约为416 kb。
2.2 Oscl6候选区间基因结构分析与基因表达分析
以日本晴的基因组序列作为参考,查询水稻基因组数据库后发现,在定位区间内共有68个开放阅读框,其中功能基因53个,转座子蛋白3个,反转座子蛋白12个,未发现已克隆的与二次枝梗和小穗梗长度发育相关的基因。
为了在目标区域内寻找最有可能的候选基因,我们从公共数据库CREP中查找了定位区间内预测基因在籼稻品种明恢63不同发育时期的表达数据,筛选出在水稻幼穗发育时期较高表达的基因11个(表1),其中功能基因10个,转座子蛋白1个。比较发现()、、、、这5个基因仅在幼穗中优势表达,即在幼穗组织中比其他组织中的表达量都高(图3),这与Wang等[41]结果一致。我们认为这些候选基因参与枝梗发育的可能性较大。
2.3 OsCL6候选基因的鉴定
为了进一步验证这一推论,我们首先采用qRT-PCR检测了基因在野生型和突变体幼穗分化Ⅲ期到Ⅳ期的表达量。结果表明,中的基因表达量极显著下调,在野生型对照基因相对表达量为1的前提下,突变体基因的相对表达量仅为原来的1/20(图4-A)。基因全长1146 bp,编码蛋白含有382个氨基酸,N端由42个氨基酸组成一个F-box基序,C端有两个与蛋白-蛋白互作相关的Kelch二级结构。根据基因序列及启动子区域,分段重合设计引物,扩增野生型和突变体的基因编码序列和启动子序列,经测序分析发现,与参考基因组日本晴和野生型粤农丝苗相比,突变体植株中基因的编码序列无差异,而其启动子在距离起始密码子185 bp的5′-UTR区域发生了9个碱基的插入突变(图4-B)。针对启动子缺失区域设计引物,对F2群体中15个隐性性状个体和15个显性性状个体进行检测,发现插入突变与簇生性状共分离(图4-C)。综合上述结果,我们推测基因为调控水稻枝梗发育的候选基因。

图1 粤农丝苗(WT)与cl6的表型
Fig. 1. Phenotypes of Yuenongsimiao(WT) and.
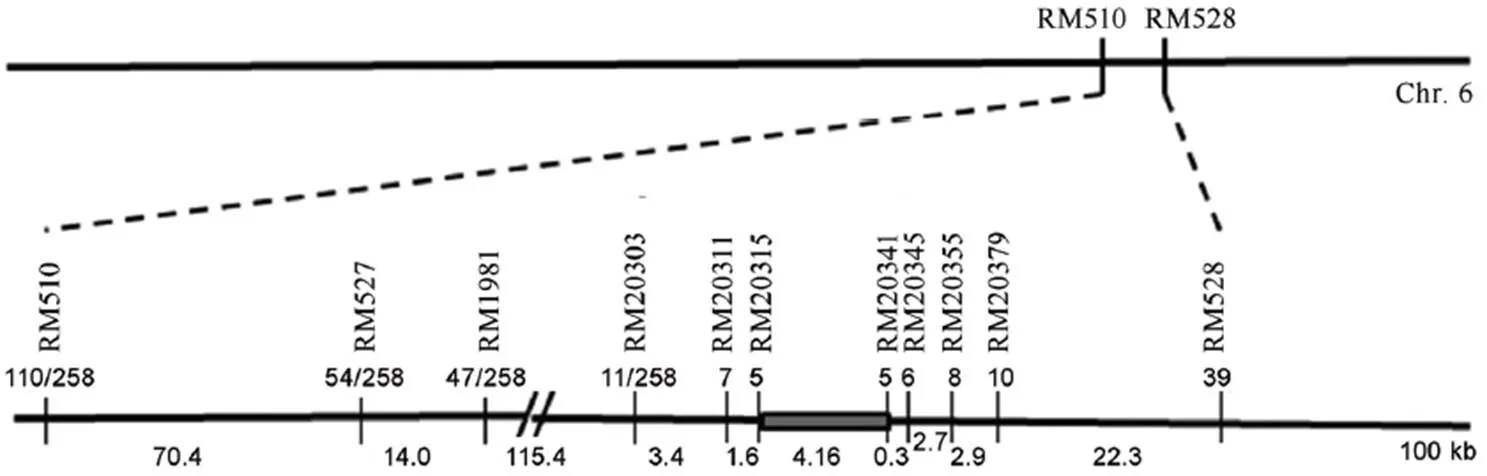
图2 OsCL6基因的定位
Fig. 2. Gene mapping of

表1 幼穗发育时期高表达的11个基因
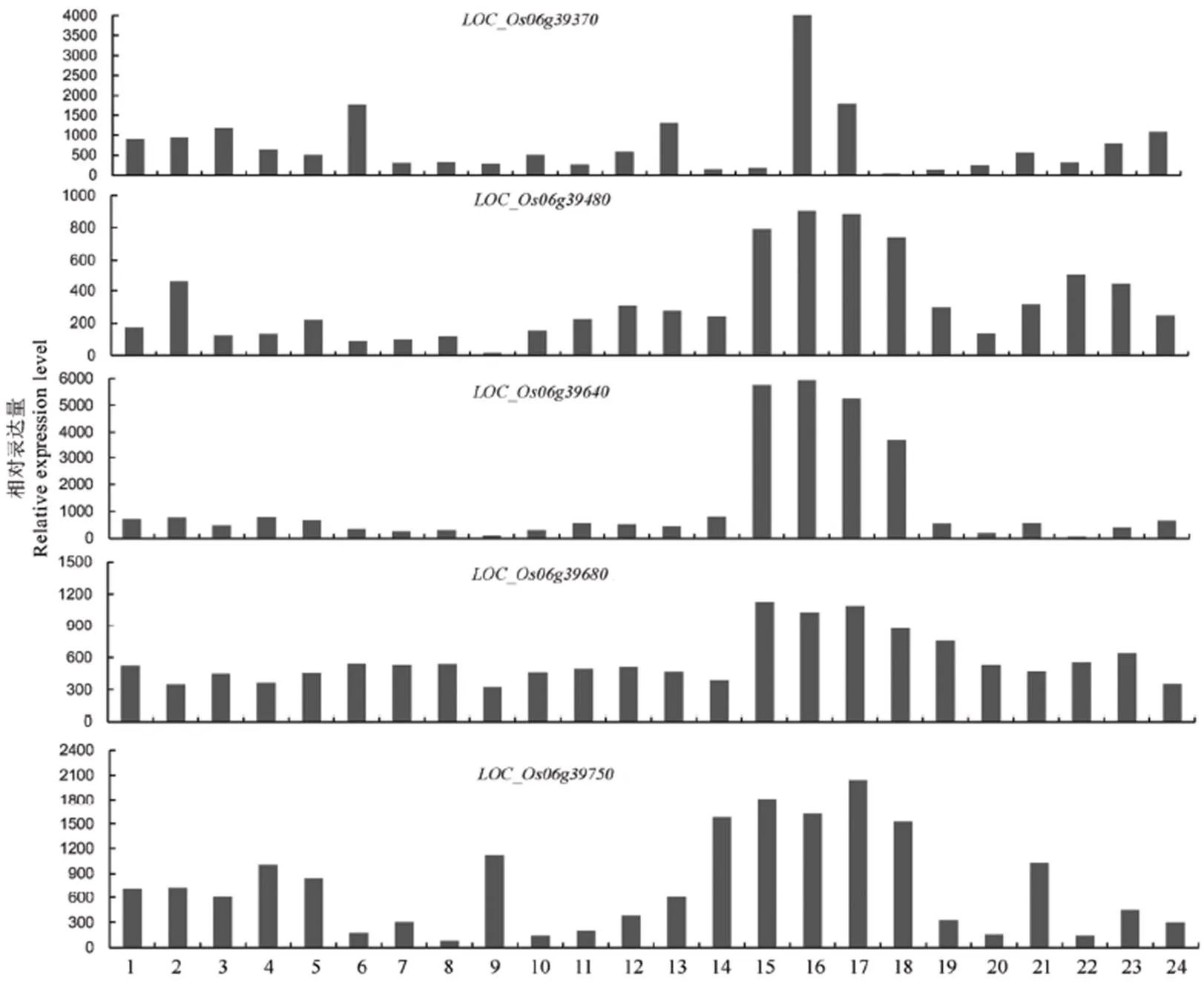
1−萌发后的胚和胚根; 2−浸泡72 h后的种子;3−二蘖苗的根;4−二蘖苗的地上部;5−3叶期的叶和根;6−幼穗分化Ⅲ期的叶片;7−幼穗长达4~5 cm时的叶片;8−抽穗前5 d的剑叶;9−抽穗前14 d的剑叶;10−幼穗分化Ⅲ期的茎鞘;11−幼穗长达4~5 cm时的茎鞘;12−抽穗前5 d的茎鞘;13−抽穗前1 d的颖壳;14−抽穗期的茎;15−处于分化Ⅲ期的幼穗;16−处于分化Ⅳ期的幼穗;17−处于分化V期的幼穗;18−4~5 cm长幼穗;19−抽穗期的穗;20−开花前1 d的雄蕊;21−授粉3 d后的小穗;22−授粉7 d后的胚乳;23−授粉14 d后的胚乳;24−授粉21 d后的胚乳。
Fig. 3. Patterns of dominant expression of candidate genes in young panicles.
2.4 转录组测序结果
为了探索基因的调控途径,收集了野生型和突变体幼穗分化Ⅲ~Ⅳ期转变时期的幼穗样品各3份,经RNA-seq测序获得了野生型和突变体植株幼穗的表达谱数据,共获得49.29 Gb Clean Data,各样品的Clean Data均达到6.99 Gb,Q30碱基百分比在91.94%及以上。分别将各样品的Clean Reads与日本晴参考基因组进行序列比对,比对效率从82.41%到83.72%不等。基于比对结果,进行可变剪接预测分析、基因结构优化分析以及差异表达基因的发掘,共获得差异表达基因1 439个,其中1 123个得到功能注释。基于比对结果,进行基因表达量分析,共获得了461个在突变体中表达上调的基因和540个表达下调的基因(Fold Change>2,FDR<0.01)(图5-A)。其中,定位区间的候选基因()在幼穗发育期优势表达,转录组测序结果也表明其表达水平存在显著差异。此外,通过KEGG通路富集分析,我们发现突变体与野生型植株相比,许多基因富集在了植物激素信号转导、植物病原菌互作、碳代谢等通路(图5-B)。通过对这些差异表达基因的功能的进一步分析,发现赤霉素(GA)信号通路的许多基因发生富集,预示着GA信号通路可能在调控水稻枝梗发育中发挥了重要的作用(图5-C)。编码包含F-BOX结构域和Kelch重复的蛋白,目前从各种植物中已鉴定出大量的F-box蛋白质,它们通过构成E3泛素连接酶复合体SCF[Skp1-Cdc53 (cullin)-F-box],参与植物激素(乙烯、生长素、赤霉素和茉莉酸)的信号传导[47-50]。该基因在拟南芥中的同源基因通过细胞分裂素信号途径调控拟南芥的生长发育[51],然而在水稻中尚未见定位、克隆或功能分析的报道。
3 讨论
稻穗为圆锥花序,由穗轴、一次枝梗、二次枝梗、小穗梗和小穗(颖花)组成,穗轴上一般有8~15个穗节,一次枝梗着生在各穗节上,二次枝梗着生在一次枝梗上,各枝梗上着生着小穗梗,其末端着生一个小穗[52]。水稻穗部形态主要由枝梗分化和小穗着生位置决定[6],枝梗分化作为穗发育的重要环节,包括一次枝梗、二次枝梗及小穗梗等的分化,直接决定水稻能否进入和完成生殖发育过程,进而影响水稻的产量和品质。簇生稻是穗型突变体的一类,在主穗轴和枝梗顶端长着2~3粒颖花,其发现与研究不但是鉴定穗型发育功能基因的重要途径之一,也是水稻新品种选育、生物技术研究以及农业生产的物质基础[53-54]。研究人员在长期的田间工作中,利用航天技术诱变广东省水稻主导品种粤农丝苗,发现一个全新的小穗簇生突变体材料,不同于以往的研究,簇生性状是由于二次枝梗和小穗梗缩短所致,是一个优良的新种质材料,定名粤花占1号[39],并于2019年获得农业农业部授予的农作物新品种权(品种权号CNA20151728.8)。该材料首先丰富了水稻突变体的多样性,适用于穗型发育过程中枝梗伸长缩短分子机理的研究;其次,通过对非簇生水稻簇生性状的转育,能提高穗着粒密度及每穗粒数、有效缩短穗长度及改良稻米品质;此外,该突变体在抽穗灌浆期具有观赏性,可满足观光农业发展的迫切需要。
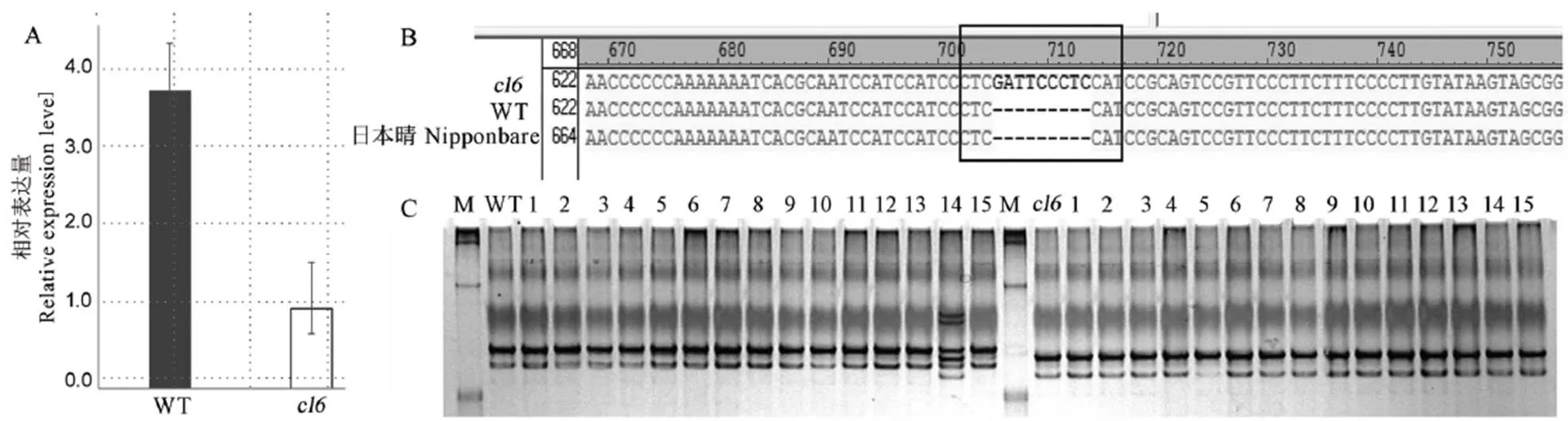
A−采用qRT-PCR验证OsFKB16基因的表达差异; B−OsFKB16的启动子测序结果; C−OsFKB16的启动子共分离验证。WT−粤农丝苗。
Fig. 4. Candidate genevalidation.
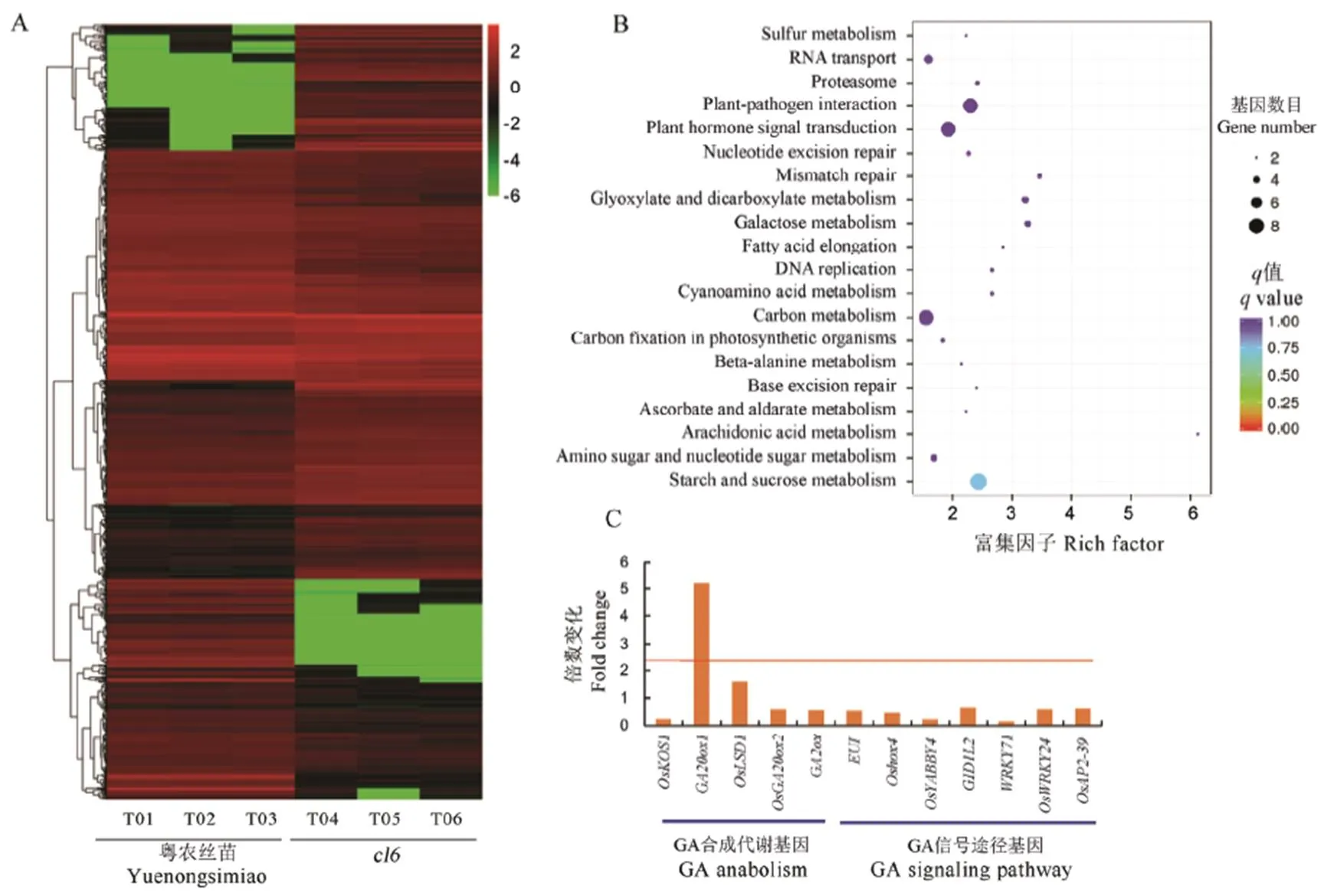
A−差异表达基因聚类分析,log2(FPKM+1); B−差异表达基因KEGG通路富集散点图;C−GA合成代谢和信号途径相关基因的表达差异。
Fig. 5. Partial analysis results of transcriptome sequencing of the wild type and its mutant.
遗传分析表明控制簇生性状的基因为半显性单基因,BSA性状定位法将该基因定位于第6染色体约416 kb的区间内,该区间共有68个开放阅读框(ORF)。公共数据库CREP中发现明恢63在该区间内有11个基因在幼穗发育时期高表达,包括10个功能基因和1个转座子蛋白,其中有5个基因只在幼穗中优势表达。野生型与突变体幼穗发育3~4期的幼穗转录组分析表明,定位区间内的68个基因有5个基因呈现显著差异表达(2倍)。而基因在幼穗中优势高表达,在突变体中的表达量显著下调,并且该基因5′-UTR区具有9个碱基的插入突变,导致植物激素相关基因差异表达,因此,我们认为该基因是候选基因。
真核生物体内,F-box蛋白在泛素-蛋白酶体途径(ubiquitin-proteasome pathway, UPP)中参与调控胞内蛋白降解、受体识别、信号传导等过程[55]。F-box蛋白作为泛素连接酶复合物(Skp1-Cullin1- F-box, SCF)的调节亚基,参与降解底物的特异性识别[56]。LOC_Os06g39370编码一个含有382个氨基酸的F-box结构蛋白,其同源基因在拟南芥中通过细胞分裂素信号途径控制拟南芥的生长发育[51],然而在水稻中尚未见定位、克隆或功能分析的报道。对该基因序列及启动子区进行测序拼接对比,发现突变体在启动子区有9个bp的插入突变,暗示其可能通过启动子二级结构等的改变来调控转录翻译过程,调控下游基因的表达,幼穗发育过程中枝梗伸长受阻,稻穗出现簇生表型。
水稻穗型发育是众多基因参与调控的一个复杂而有序的网络系统,从当前的研究来看,参与水稻穗型发育的基因可以分为5类:腋生分生组织起始相关基因、分生组织发育时间转换相关基因、调控花序结构内枝梗及穗长相关基因、调控穗中多种分生组织的细胞特性基因、穗轴和花序分生组织标记基因[57]。植物内源激素如生长素、细胞分裂素、赤霉素、独脚金内脂等,多是通过调节分枝原基的产生与伸长参与穗型的遗传调控[58-60],独脚金内酯作为新型植物激素能够抑制分枝并参与多种发育过程[61-62],油菜素内酯是否参与调控水稻穗型形成及相关机理还有待研究确认。近年来的研究发现许多microRNA(miRNA)也在穗型发育的枝梗分化过程中影响广泛而调控机制简约而备受关注。如miR172的通过靶基因决定枝梗分生组织的分枝程度[63],miR396通过调控基因的表达,从而调节水稻二次枝梗数和每穗粒数,并最终控制水稻产量[34]。据笔者统计,截至目前共有19个一次枝梗分化相关基因、15个二次枝梗分化相关基因被克隆,而它们绝大部分参与枝梗数的分化调控,极少数与枝梗分化长度相关。因此,对本研究突变体材料二次枝梗和小穗梗长度严重缩短功能基因的鉴定与挖掘就显得尤为重要。基因位于第6染色体,无可变剪接,编码F-box结构蛋白,F-box蛋白质是一类真核生物中普遍存在的蛋白质家族,对细胞内的蛋白质降解有着关键的调控作用。该基因在水稻中的功能尚不清楚,亟需进一步的工作加以验证和明确。
4 结论
本研究材料小穗簇生突变体与其野生型相比较,花序结构异常,一次枝梗数增加,二次枝梗和小穗梗显著缩短,一次枝梗顶端小穗3粒簇生在一起。遗传分析表明簇生性状由单个半显性基因控制,BAS性状定位法将该基因精细定位于第6染色体分子标记RM20315和RM20341之间约3.88 cM/416 kb的区段。参考日本晴基因组序列,在定位区间内发现开放性阅读框68个,表达谱分析发现其中11个基因在水稻幼穗发育时期高表达。转录组测序得到在突变体中的表达量显著降低。该基因编码包含F-box结构域和Kelch重复的蛋白,通过构成E3泛素连接酶复合体SCF参与植物激素的信号传导,调节植物生长发育过程。在水稻中的具体功能尚不清楚。对该基因序列差异进行分析发现其启动子在距离起始密码子185 bp的5′-UTR区域发生9个碱基的插入突变,能够通过二级结构的该表调控翻译过程,参与水稻幼穗发育枝梗分化途径。本研究结果为进一步克隆新基因并研究其功能,解析水稻穗发育过程中二次枝梗和小穗梗缩短的机理奠定一定的基础。
[1] 罗琼, 朱立煌. 水稻花发育的分子生物学研究进展[J]. 遗传, 2002(1): 87-93.
Luo Q, Zhu L H. Progress of molecular floral development research in rice[J]., 2002(1): 87-93. (in Chinese with English abstract)
[2] 肖景华, 吴昌银, 袁猛, 王妮丽, 范优荣, 杨猛, 欧阳亦聃, 阮一骏, 张启发. 中国水稻功能基因组研究进展与展望[J]. 科学通报, 2015, 60(18): 1711-1723.
Xiao J H, Wu C Y, Yuan M, Wang N N, Fan Y R, Yang M, Ouyang Y R, Ruan Y J, Zhang Q F. Advances and prospects of rice functional genomics in China[J]., 2015, 60(18): 1711-1723. (in Chinese)
[3] 淳雁, 李学勇. 水稻穗型的遗传调控研究进展[J]. 植物学报, 2017, 52(1): 19-29.
Chun Y, Li X Y. Advances in genetic regulation of panicle shape in rice[J].2017, 52(1): 19-29. (in Chinese)
[4] 陈峰, 高洁, 周继华, 朱文银, 朱其松, 孙公臣, 袁守江, 杨连群. 水稻穗型的研究进展[J]. 江苏农业学报, 2009, 25(5): 1167-1172.
Chen F, Gao J, Zhou J H, Zhu W Y, Zhu Q S, Sun G C, Yuan S J, Yang L Q. Research progress of panicle shape in rice[J].,2009, 25(5): 1167-1172. (in Chinese with English abstract)
[5] Sakamoto T, Matsuoka M. Identifying and exploiting grain yield genes in rice[J]., 2008, 11(2): 209-214.
[6] Ikeda M, Kawarabayashi T, Harigaya Y, Sasaki A, Yamada S, Matsubara E, Murakami T, Tanaka, Kurata T, Xu W H, Ueda K, Kuribara H, Ikarashi Y, Nakazato Y, Okamoto K, Abe K, Mikio S. Motor impairment and aberrant production of neurochemicals in human alpha-synuclein A30P+A53T transgenic mice with alpha-synuclein pathology[J]., 2009, 1250: 232-241.
[7] Li M, Tang D, Wang K J, Wu X R, Lu L L, Yu H X, Gu M H, Yan C J, Cheng Z K. Mutations in the F-box geneimprove the panicle architecture and enhance the grain yield in rice[J].2011, 9(9): 1002-1013.
[8] Luo Q, Zhou K, Zhao X F, Zeng Q C, Xia H G, Zhai W X, Xu J C, Wu X J, Yang L H. Identification and fine mapping of a mutant gene for palealess spikelet in rice[J]., 2005, 221(2): 222-230.
[9] Kaoru K, Masahiko M, Akio M, Hirohiko H, Junko K.(PAP2), encoding a SEPALLATA subfamily MADS-box protein, positively controls spikelet meristem identity in rice[J]., 2010, 51(1): 47-57.
[10] Rao N, Prasad K, Kumar P R, Usha V. Distinct regulatory role for RFL, the rice LFY homolog, in determining flowering time and plant architecture[J]., 2008, 105(9): 3646-3651.
[11] Ikeda K, Ito M, Nagasawa N, Kyozuka J, Nagato Y. Rice, encoding an F-box protein, regulates meristem fate[J]., 2010, 51(6): 1030-1040.
[12] Ookawa T, Hobo T, Yano M, Murata K, Ando T, Miura H, Asano K, Ochiai Y, Ikeda M, Nishitani R, Ebitani T, Ozaki H, Angeles E R, Hirasawa T, Matsuoka M. New approach for rice improvement using a pleiotropic QTL gene for lodging resistance and yield[J]., 2010, 1: 132.
[13] Terao T, Nagata K, Morino K, Hirose T. A gene controlling the number of primary rachis branches also controls the vascular bundle formation and hence is responsible to increase the harvest index and grain yield in rice[J].2010, 120(5): 875-893.
[14] Li F, Liu W, Tang J,Chen J F, Tong H N, Hu B, Li C L, Fang J, Chen M S, Chu C C. Riceis essential for determining panicle outgrowth and elongation[J]., 2010, 20(7): 838-849.
[15] Lu S J, Wei H, Wang, Y, Wang H M, Yang R F, Zhang X B, Tu J M. Overexpression of a transcription factor OsMADS15 modifies plant architecture and flowering time in rice (L.) [J]., 2012, 30(6):1461-1469.
[16] Jiao Y Q, Wang Y H, Xue D W, Wang J, Yan M X, Liu G F, Dong G J, Zeng D L, Lu Z F, Zhu X D, Qian Q , Li J Y. Regulation of OsSPL14 by OsmiR156 defines ideal plant architecture in rice[J]., 2010, 42(6): 541-544.
[17] Miura K, Ikeda M, Matsubara A, Song X J, Ito M, Asano K, Matsuoka M, Kitano H, Ashikari M. OsSPL14 promotes panicle branching and higher grain productivity in rice[J]., 2010, 42(6): 545-549.
[18] Wang J, Zhou L, Shi H, Chern M S, Yu H, Yi H, He M, Yin J J, Zhu X B, Li Y, Li W T, Liu J L, Wang J C, Chen X Q, Qing H, Wang Y P, Liu G F, Wang W M, Li P, Wu X J, Zhu L H, Zhou J M, Ronald P C, Li S G, Li J Y, Chen X W. A single transcription factor promotes both yield and immunity in rice[J]., 2018, 361(6406): 1026-1028.
[19] Ashikari M, Sakakibara H, Lin S Y,Yamamoto T, Takashi T, Nishimura A, Angeles E R, Qian Q, Kitano H , Matsuoka M. Cytokinin oxidase regulates rice grain production[J], 2005, 309(5735): 741-745.
[20] Guo T, Lu Z Q, Shan J X, Ye W W, Dong N Q, Lin H X. ERECTA1 acts upstream of the OsMKKK10-OsMKK4- OsMPK6 cascade to control spikelet number by regulating cytokinin metabolism in rice[J]., 2020, 32(9): 2763-2779. DOI: https://doi.org/10.1105/ tpc.20.00351
[21] Kobayashi K, Yasuno N, Sato Y, Yoda M, Yamazaki R, Kimizu M, Yoshida H, Nagamura Y, Kyozuka J. Inflorescence meristem identity in rice is specified by overlapping functions of three AP1/FUL-like MADS box genes and, abox gene[J]., 2012, 24(5): 1848-1859.
[22] Lee S, Kim J, Han J J, Han M J, An G. Functional analyses of the flowering time gene, the putative(SOC1/AGL20) ortholog in rice[J]., 2004, 38(5): 754-764.
[23] Masiero S, Imbriano C, Ravasio F, Rebecca F, Nilla P, Sari G M, Roberto M, Lucia C, Kater M M. Ternary complex formation between MADS-box transcription factors and the histone fold protein NF-YB[J]., 2002, 277(29): 26 429-26 435.
[24] Wang K J, Tang D, Hong L, Xu W Y, Huang J, Li M, Gu M H, Xue Y B, Cheng Z K.andregulate reproductive habit in rice[J]., 2010, 6(1): e1000818.
[25] Xiao H, Wang Y, Liu D, Wang W M, Li X B, Zhao X F, Xu J C, Zhai W X, Zhu L H. Functional analysis of the rice AP3 homologue OsMADS16 by RNA interference[J]., 2003, 52(5): 957-966.
[26] Yadav S R, Prasad K, Vijayraghavan U. Divergent regulatory OsMADS2 functions control size, shape and differentiation of the highly derived rice floret second-whorl organ[J]., 2007, 176(1): 283-294.
[27] Yao S G, Ohmori S, Kimizu M, Hitoshi Y. Unequal genetic redundancy of rice PISTILLATA orthologs, OsMADS2 and OsMADS4, in lodicule and stamen development[J]., 2008, 49(5): 853-857.
[28] Nagasawa N, Miyoshi M, Sano Y, Hikaru S, Hiroyuki H, Hajime S, Yasuo N.genes control floral organ identity in rice[J].2003, 130: 705-718.
[29] Yamaguchi T, Nagasawa N, Kawasaki S, Matsuoka M, Nagato Y, Hirano H Y. The YABBY generegulates carpel specification and midrib development in[J]., 2004, 16(2): 500-509.
[30] Li H F, Liang W Q, Yin C S, Zhu L, Zhang D B. Genetic interaction of,, andin specifying rice floral organ identities and meristem determinacy[J]., 2011, 156(1): 263-274.
[31] Lopez-Dee Z P, Wittich P, Pe M E, Gorla M S, Kater M M, Colombo L., a novel rice MADS-box gene expressed during ovule development[J]., 2015, 25(3): 237-244.
[32] Dreni L, Jacchia S, Fornara F, Fornari M, Ouwerkerk P F, An, Colombo G L, KaterM M. The D-lineage MADS-box genecontrols ovule identity in rice[J]., 2010, 52(4): 690-699.
[33] Zhang Y, Yu H, Liu J, Wang W, Sun J, Gao Q, Zhang Y, Wang J, Xu Z. Loss of function of OsMADS34 leads to large sterile lemma and low grain yield in rice (L.)[J]., 2016, 36(11): 147.
[34] Gao F, Wang K, Liu Y,Chen Y P, Chen P, Shi Z Y, Luo J, Jiang D Q, Fan F F, Zhu Y G, Li S Q. Blocking miR396 increases rice yield by shaping inflorescence architecture[J].2015, 2: 15196.
[35] 陈志强, 周丹华, 郭涛, 王慧. 水稻航天生物育种研究进展[J]. 华南农业大学学报, 2019, 40(5): 195-202.
Chen Z Q, Zhou D H, Guo T, Wang H. Research progress of rice space mutation bio-breeding[J].,2019, 40(5): 195-202. (in Chinese with English abstract)
[36] 张福彦, 张建伟, 程仲杰, 陈晓杰, 齐红志, 杨保安, 范家霖, 崔龙. 航天诱变技术在小麦育种上的应用[J]. 核农学报, 2019, 33(2): 262-269.
Zhang F Y, Zhang J W, Cheng Z J, Chen X J, Qi H Z, Yang B A, Fan J L, Cui L.Application of space mutation technology in wheat breeding[J]., 2019, 33(2): 262-269. (in Chinese with English abstract)
[37] 何秀英, 廖耀平, 陈钊明,程永盛, 陈粤汉, 刘维. 优质抗病水稻新品种粤农丝苗的选育及应用[J]. 中国稻米, 2014, 20(2): 69-70.
He X Y, Liao Y P, Chen Z M, Cheng Y S, Chen Y H, Liu W.Breeding and application of a new rice variety with high quality and disease resistance Yuenongsimiao[J]., 2014, 20(2): 69-70. (in Chinese with English abstract)
[38] 陆展华, 刘维, 卢东柏, 王晓飞, 王石光, 何秀英. 优质稻品种‘粤农丝苗’稻瘟病广谱抗性遗传及基因组成分析[J]. 植物遗传资源学报, 2020, 21(4): 827-833.
Lu Z H, Liu W, Lu D B, Wang X F, Wang S G, He X Y. Genetic analysis and gene identification of high-quality rice ‘Yuenong Simiao’ with broad spectrum resistance against rice blast[J]., 2020, 21(4): 827-833. (in Chinese with English abstract)
[39] 刘维, 何秀英, 陆展华, 卢东柏, 廖耀平, 陈钊明, 程永盛, 陈粤汉. 水稻穗型突变体的鉴定、保护与遗传分析[J]. 植物遗传资源学报, 2017, 18(6): 1210-1215.
Liu W, He X Y, Lu Z H, Lu D B, Liao Y P, Chen Z M, Cheng Y S, Chen Y H. Observation and genetic analysis on rice panicle architecture mutant[J]., 2017, 18(6): 1210-1215. (in Chinese with English abstract)
[40] 刘维, 何秀英, 陈钊明, 程永盛, 卢东柏, 廖耀平. 航天诱变水稻小穗簇生突变体的遗传分析与基因定位[C]//全国植物航天诱变育种学术研讨会2015年学术会议文集. 广州: 全国植物航天诱变育种学术研讨会, 2015: 11.
Liu W, He X Y, Chen Z M, Cheng Y S, Lu D B, Liao Y P. Genetic analysis and gene mapping of rice spikelet cluster mutant CL6 induced by spaceflight[C]// Proceedings of 2015 National Symposium on Plant Space Mutagenesis Breeding. Guangzhou: National Symposium on Plant Space Mutagenesis Breeding, 2015: 11. (in Chinese)
[41] Wang L, Xie W, Chen Yang Q, Tang W J, Yang J Q, Ye R Q, Liu L, Lin Y Q, Xu C Q, Xiao J H, Zhang Q F. A dynamic gene expression atlas covering the entire life cycle of rice[J]., 2010, 61(5): 752-766.
[42] Junichi S, Kumiko I, Takeshi N, Nobuhiro I, Masafumi T, Yasuhiro H, Shou T. Characterization of rice nucleotide sugar transporters capable of transporting UDP-galactose and UDP-glucose[J]., 2010, 148(1): 35-46.
[43] Asuka N, Momoyo I, Noriko K, Sato Y, Matsuoka M.regulates leaf development and maintenance of the shoot apical meristem in rice[J]., 2002, 30(2): 189-201.
[44] Yu D, Ranathunge K, Huang H, Pei Z, Franke R, Schreiber L, He C.encodes a β-ketoacyl CoA synthase involved in biosynthesis of cuticular waxes on rice leaf[J], 2008, 228(4): 675-685.
[45] Tomoaki S, Ayami K, Asako T S, Bun-Ichi S, Suguru T, Y Shimada, Shozo F, Mizutani M. Rice CYP734As function as multisubstrate and multifunctional enzymes in brassinosteroid catabolism[J]., 2011, 67(1): 1-12.
[46] Wen J Q, Chao W, Ya P F, Hu G C, He Z Q, Liu W Z. Novel rice mutants overexpressing the brassinosteroid catabolic gene[J]., 2017, 93(1): 197-208.
[47] 吴丹, 唐冬英, 李新梅, 李丽, 赵小英, 刘选明. F-box蛋白在植物生长发育中的功能研究进展[J]. 生命科学研究. 2015, 19(4): 362-367.
Wu D, Tang D Y, Li X M, Li L, Zhao X Y, Liu X M. Progresses on F-box protein function in plant growth and development[J]., 2015, 19(4): 362-367. (in Chinese with English abstract)
[48] 贾琪, 孙松, 孙天昊, 林文雄. F-box蛋白家族在植物抗逆响应中的作用机制[J]. 中国生态农业学报, 2018, 26(8): 1125-1136.
Ja Q, Sun S, Sun T H, Lin W X. Mechanism of F-box protein family in plant resistance response to environmental stress[J]., 2018, 26(8): 1125-1136. (in Chinese with English abstract)
[49] 秘彩莉, 刘旭, 张学勇. F-box蛋白质在植物生长发育中的功能[J]. 遗传, 2006(10): 1337-1342.
Mi C L, Liu X, Zhang X Y.The function of F-box protein in plant growth and development[J]., 2006(10): 1337-1342. (in Chinese with English abstract)
[50] 许媛, 李铃仙, 于秀梅, 刘大群. F-box蛋白在植物抗逆境胁迫中的功能[J]. 植物生理学报, 2015, 51(7): 1003-1008.
Xu Y, Li L X, Yu X M, Liu D Q. The functions of F-box protein in plant resistance to stress[J]., 2015, 51(7): 1003-1008. (in Chinese with English abstract)
[51] Kim H J, Chiang Y H, Kieber J J, Schaller G E. SCF(KMD) controls cytokinin signaling by regulating the degradation of type-B response regulators[J]., 2013, 110(24): 10 028-10 033.
[52] 伍艳莲, 汤亮, 刘小军, 张文宇, 曹卫星, 朱艳. 基于形态特征参数的稻穗几何建模及可视化研究[J]. 中国农业科学, 2009, 42(4): 1190-1196.
Wu Y L, Tang L, Liu X J, Zhang W Y, Cao W X, Zhu Y. Architectural parameter-based geometric modeling and visualization of rice panicle[J]., 2009, 42(4): 1190-1196. (in Chinese with English abstract)
[53] 叶卫军, 胡时开, 李媛媛, 马伯军, 郭龙彪. 水稻种质资源的分子鉴定和育种利用[J]. 分子植物育种, 2013, 11(4): 625-633.
Ye W J, Hu S K, Li Y Y, Ma B J, Guo L B. Molecular identification and breeding application of rice germplasm[J]., 2013, 2013, 11(4): 625-633. (in Chinese with English abstract)
[54] 苏德文, 郑燕梅, 何炜, 张建福, 谢华安. 水稻簇生穗突变体基因研究进展[J]. 福建农业学报, 2013, 28(9): 931-937.
Su D W, Zheng Y H, He W, Zhang J F, Xie H A. Progress on the genes of rice clustered spikelets() mutant[J]., 2013, 28(9): 931-937. (in Chinese with English abstract)
[55] Matsui M. F-box proteins of plants and their various roles[J]., 2012, 84(6): 432-439.
[56] 贾凤娟. 拟南芥F-box基因调节盐胁迫抗性的分子机理[D]. 山东农业大学, 2015.
Ja F J. Molecular mechanism of the regulation of salt stress resistance by F-box genein[D]. Shandong Agricultural University, 2015. (in Chinese with English abstract)
[57] 卢寰, 时振英. 水稻穗发育的分子生物学研究进展[J]. 植物生理学报, 2013, 49(2): 111-121.
Lu H, Shi Z Y.Molecular research progress of rice panicle development[J]., 2013, 49(2): 111-121. (in Chinese with English abstract)
[58] You X, Zhu S, Zhang W, Zhang J, Wang C M, Jing R N, Chen W W, Wu H Q, Cai Y, Feng Z M, Hu J L, Yan H Q, Kong F, Zhang H, Zheng M, Ren Y L, Lin Q B, Cheng Z J, Zhang X, Lei C L, Jiang L, Wang H Y, Wan J M.regulates rice spikelet development through modulating jasmonic acid biosynthesis[J]., 2019, 224(2): 712-724.
[59] Zhang W, Sheng J, Xu Y, Xiong F, Wu Y F, Wang W L, Wang Z Q, Yang J C, Zhang J H. Role of brassinosteroids in rice spikelet differentiation and degeneration under soil-drying during panicle development[J]., 2019, 19(1): 409.
[60] 刘拥海, 俞乐, 丁君辉, 王若仲, 黄志刚, 萧浪涛. 植物激素对分枝发育的协同调控作用研究进展[J].植物生理学报, 2012, 48(10): 941-948.
Liu Y H, Yu L, Ding J H, Wang R Z, Huang Z G, Xiao L T. Research progress in synergistic regulatory roles of phytohormones in shoot branching[J]., 2012, 48(10): 941-948. (in Chinese with English abstract)
[61] Wang Y, Shang L, Yu H, Zeng L Q, Hu J, Ni S, Rao Y C, Li S F, Chu J F, Meng X B, Wang L, Hu P, Yan J J, Kang S J, Qu M H, Lin H, Wang T, Wang Q, Hu X M, Chen H Q, Wang B, Gao Z Y, Guo L B, Zeng D L, Zhu X D, Xiong G S, Li J Y, Qian Q. A strigolactone biosynthesis gene contributed to the green revolution in rice[J].2020, 13(6): 923-932
[62] 王玫, 陈洪伟, 王红利, 刘克锋. 独脚金内酯调控植物分枝的研究进展[J]. 园艺学报, 2014, 41(9): 1924-1934.
Wang M, Chen H W, Wang H L, Liu K F.Research progress in regulatory role of strigolactones in shoot branching[J].2014, 41(9): 1924-1934. (in Chinese with English abstract)
[63] Wang L, Sun S, Jin J,Fu D B, Yang X F, Weng X Y, Xu C G, Li X H, Xiao J H, Zhang Q F. Coordinated regulation of vegetative and reproductive branching in rice[J]., 2015, 112(50): 15504-15509.
Location and Candidate Gene Analysis of Rice Clustered Spikelets Gene
LIU Wei, LU Zhanhua, LU Dongbai, WANG Xiaofei,WANG Shiguang, XUE Jia, HE Xiuying*
(Rice Research Institute, Guangdong Academy of Agricultural Sciences/Guangdong Provincial Key Laboratory of New Technology in Rice Breeding, Guangzhou 510640, China;*Corresponding author, E-mail: xyhe@163.com)
【】The mapping and candidate gene analysis of rice clustered spikelet genes helpclone the functional genes for clustered spikelet caused by shortening of the branch length in the process of panicle development. 【】A stable clustered spikelet mutantwas obtained through the spacemutationbreedingtechnique with Yuenongsimiao, a good quality rice cultivar in Guangdong Province as material. F1and F2mapping populations were constructed by crossingwith Yuejinyinzhan. The clustered spikelet genewas mapped and candidate genes were predicted and evaluated by combining with transcriptome sequencing. 【】Genetic analysis showed that the trait was controlled by a pair of semi-dominant gene. The gene was mapped to an interval about 416kb on chromosome 6 by Bulked Segregant Analysis(BSA). Furthermore,was selected as the best candidate gene through expression profiling analysis, RNA-seq, gene sequence differences analysis and qRT-PCR verification. A 9 bp insertion mutation in the 5'-UTR region of this gene suggests that it may regulate transcription or translation process through secondary structural changesand participating in the differentiation of stems during the development of young panicle of rice. 【】It will lay a theoretical foundation for analyzing the shortening mechanism of secondarybranches and spikelet peduncle length.
spacemutation; spikelet cluster; gene mapping; transcriptome sequencing; candidate gene
10.16819/j.1001-7216.2021.0604
2020-06-10;
2020-08-06。
广东省现代农业产业技术体系项目(2019KJ105);国家重点研发计划资助项目(2017YFD0100102);广东省自然科学基金资助项目(2017A030313183);科技创新战略专项资金资助项目(高水平农科院建设);优质稻良种重大科研联合攻关项目(粤财农[2019]73号);广东省重点领域项目(2018B020206002)。

