水稻光周期调控开花的研究进展
王玉博 王悦 刘雄 唐文帮
水稻光周期调控开花的研究进展
王玉博 王悦 刘雄 唐文帮*
(湖南农业大学 农学院,长沙 410128;*通信联系人,E-mail: tangwenbang@163.com)
水稻抽穗期作为重要的农艺性状,由自身遗传因素和环境因素共同决定,对品种生态适应区域和产量因子均有较大影响。过去的二十年里,从叶片的日长识别到茎尖分生组织的成花激活,水稻光周期诱导抽穗开花分子调控机理已取得较大进展,分离并克隆大量与成花相关的调控基因,并整合到光周期调控分子网络中。当植物处于有利条件时,该网络激活成花调控基因,促进成花素表达,将成花素运输至顶端分生组织,从而驱动分生组织细胞发育,最终成花。本文以拟南芥为对照参考,对水稻光周期调控网络及由低纬度地区向高纬度地区扩展遗传变异进行讨论,以期为生态型品种培育和光周期调控成花分子机理研究提供参考。
水稻;开花;光周期;光周期调控;抽穗期
植物开花是一系列成花基因在时间和空间上按照特定顺序特异表达,由自身遗传因素和环境因素共同决定的复杂过程,代表着植物由营养生长向生殖生长的转变,对完成后代繁衍和适应特定生态环境发挥着重要作用[1-3]。为最大限度提高生殖生长转变成功率和繁衍足量的种子,各种环境因子(光照、温度、营养成分、水分等)和自身因素(遗传因子、自身激素含量变化等)相互作用,整合之后精准调控植物开花时间。植物响应季节变化而表现出的开花时间差异由光周期感知的不同造成,根据开花对光周期响应的不同,将植物分为长日照植物(拟南芥、小麦等)、短日照植物(水稻、玉米等)、日中性植物(黄瓜、番茄等)、长-短日照植物(芦荟、大叶落地生根等)、短-长日照植物(鸭茅、白三叶草等)和中日照植物(甘蔗等)。同一物种的开花诱导临界日照长度基本固定,但种质资源间存在细微差异。对模式植物拟南芥()和水稻()的分子遗传学研究,已鉴别出大量与光周期相关的成花基因(表1),尽管两物种在1.5亿年前已开始分离进化[4],但许多成花基因存在序列和功能的高度保守,如水稻基因()()()与拟南芥()()()基因分别同源,能恢复拟南芥突变体晚花表型,两者的表达产物均为成花素,可从叶片移动至顶端分生组织,促进开花;()()()为水稻和拟南芥共有保守途径;但在长期进化过程中,部分基因不断进化出新的功能,新的成花途径逐渐形成和完善,如水稻基因在短日照下促进开花,在长日照下抑制开花,转录水平不因日照长度变化而发生较大波动;而拟南芥基因在长日照条件下诱导表达,启动开花,短日照条件下表达受到抑制,但不影响成花;在开花途径上,途径为水稻特有途径[5-6],通过调节成花素基因和的表达而调控水稻开花。
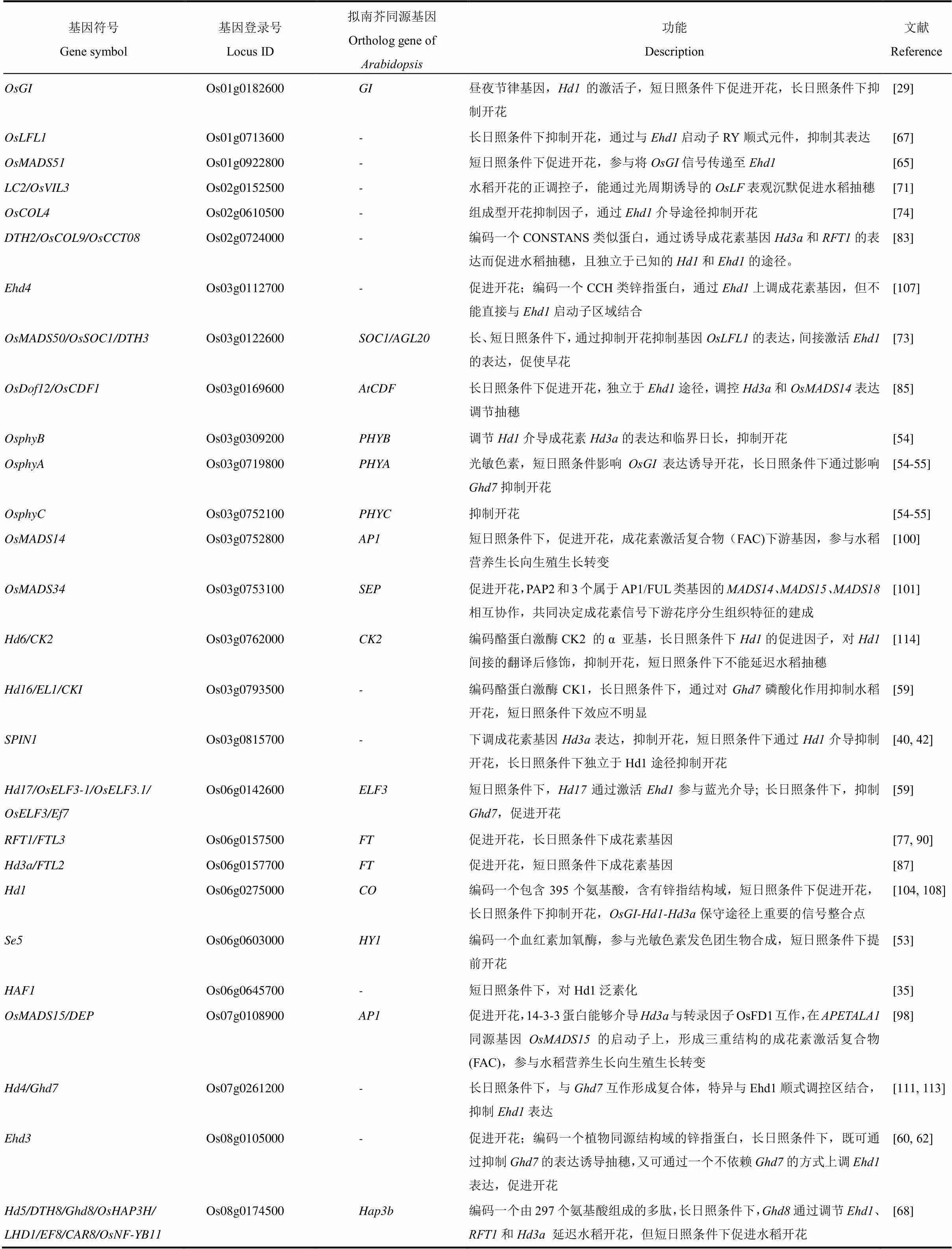
表1 水稻光周期调控相关基因
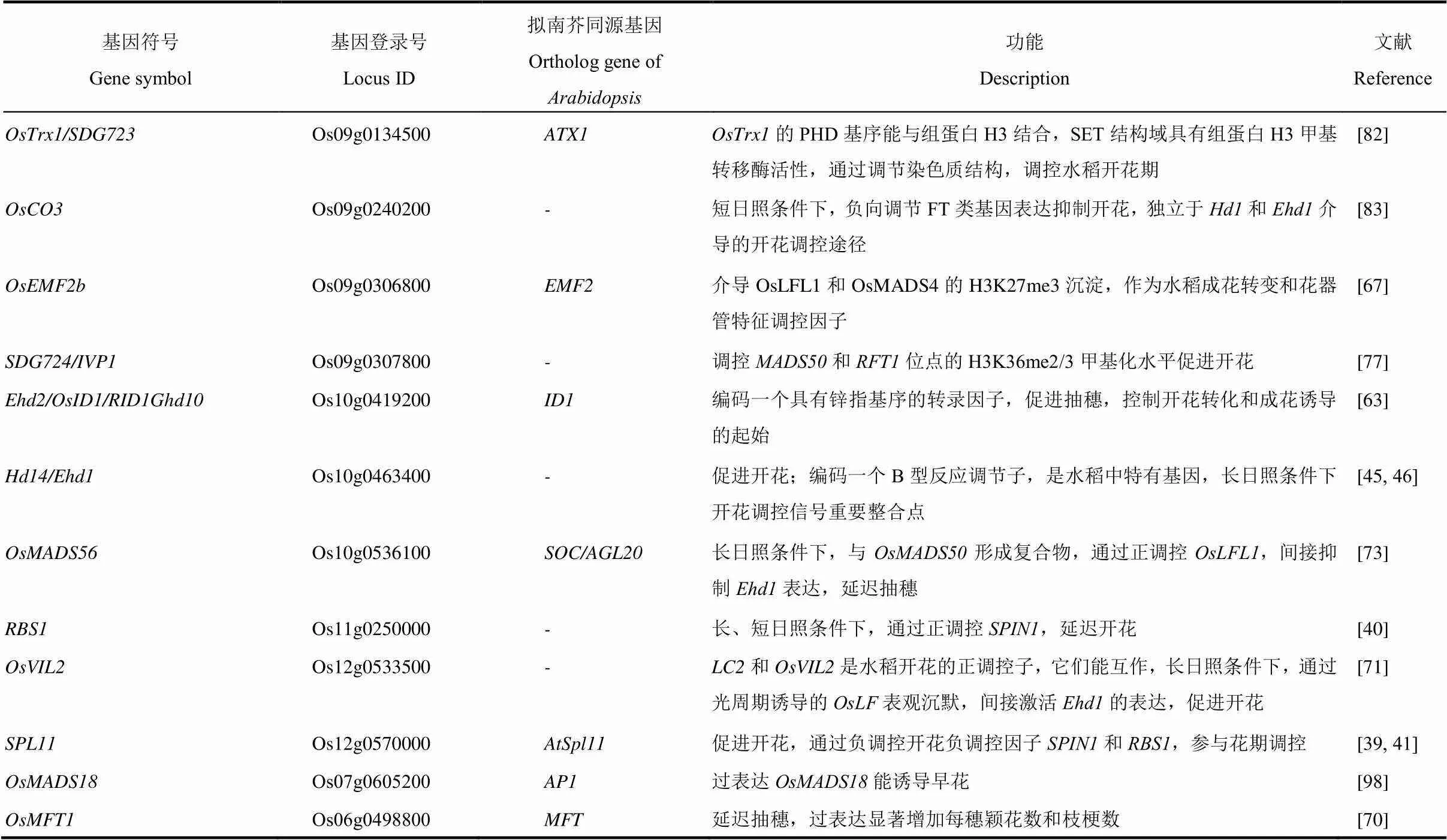
续表1
-表示未发现同源基因。
-, No ortholog gene in.
水稻最初在中国南方驯化栽培,经长时间的不断进化和改良扩展,从南纬36°至北纬53°均存在水稻种植区域,开花时间多样性是实现广泛种植的主要因素[7-8]。水稻作为短日照植物,短日照条件下,加速开花;但在长日照条件下,经过长期人工选择,部分种质资源光敏感度不断降低,使得在中国北方、日本等高纬度区域成功实现大面积种植。同时,对光周期反应的差异导致开花时间的多样性,为育种家种质资源的筛选利用提供了多种选择。研究水稻光周期调控开花的分子机理,对揭示栽培稻区域适应性、扩大种植范围具有重要的指导意义。本文以拟南芥为对照,围绕核心开花基因()和(),对水稻开花途径及向北扩展的分子机理进行综述。
1 OsGI(GI)-Hd1(CO)-Hd3a(FT)途径
Pittendrigh和Minis[18]于1964年提出外部一致模型,该模型认为,目标基因的转录表达在内源中心振荡器调控下产生接近24 h周期性节律,在特定阶段与外部环境因素(如光照、温度等)达到时间和空间同步时,才能够引发植物特定的生长发育。如图1所示,拟南芥成花信号传递途径在昼夜节律循环的特定时间均需要光照的参与。在长日照条件下,与的mRNA转录表达同时达到峰值,此时若有光照,GI与FKF1实现互作表达,且形成稳定蛋白复合物。与此相似,CO蛋白仅在有光照参与的长日照条件下表现稳定,在黄昏实现累积,进而激活表达,诱导开花;而在短日照条件下,GI与FKF1蛋白缺乏互作,CO蛋白维持在较低水平,难以激活表达[19]。因此,在特定的时间里,外部环境因素(光照)的参与对基因途径激活诱导是必要的,在与内部遗传因素,如GI、FKF1、CO蛋白积累量及的转录表达达到时间和空间的一致时,将光信号通过韧皮部由叶片进一步传达,最终引发开花。拟南芥开花是在光照参与下,通过之间互作形成的蛋白复合物调控基因转录表达,完成CO蛋白一定量的积累和基因诱导实现,该途径符合外部一致模型特征。
水稻具有类似的光周期途径。是基于图位克隆方法克隆的第一个调控开花的主效QTL,属于水稻家族基因(,,)亚类[20]。与拟南芥基因同源,两者序列相似度达到70%,编码的蛋白质在N端类锌指B-box结构域和C端CCT结构域高度同源,在进化上为最相近基因[21]。与相似,表达受节律钟调控。生物钟基因高度保守,水稻的与拟南芥的、同源,、和对应拟南芥的基因家族,并由依次传递节律变化,进而调控下游基因[22-23]。但对成花素基因的调控与拟南芥不同,在短日照条件下,为的促进因子,而在长日照条件下,为的抑制因子。突变体光周期敏感性缺失,在短日照条件下转录水平下降,表现为开花延迟;在长日照条件下转录水平上升,表现为开花提前[24]。拟南芥基因仅作为长日照条件下的促进因子,而具有依赖于光周期的独特双功能性。因此,对成花素基因的转录表达和开花转换的双重功能取决于光周期。转录表达受光敏色素的介导,能与光敏色素形成转录复合物或者受光敏色素的磷酸化[25]。在短日照条件下,转录表达于夜间达到峰值,Hd1蛋白在夜间作为激活子诱导表达;在长日照条件下,转录峰值正处于光敏期,Hd1与光敏色素相互作用,Hd1蛋白作为的抑制子抑制开花。在夜间短暂的光照处理能够改变Hd1蛋白活性,对转录表达由促进转化为抑制[26]。在光照存在的条件下,Hd1蛋白水平在Hd1过表达植株中并没有发生变化,而光敏色素对Hd1或Hd1复合物蛋白的修饰,使得由增强子转变为抑制子,进而调控的转录表达[27]。光周期介导的Hd1蛋白稳定性调节并非关键,Hd1蛋白复合物活性可能是水稻光周期开花的主要决定因子[28]。夜间为成花素蛋白表达起始,此时会充当夜间长度传感器,当夜间阶段足够长时,转录水平达到峰值,在无介导时,激活表达,促使足量Hd3a蛋白积累,并启动开花。外部一致模型同样应用于水稻,黑暗与表达峰值的重合对于触发开花反应至关重要。
1.1 Hd1表达的促进因子
不论在长日照还是短日照条件下,均为主要的正向调节因子,影响水稻对光周期的敏感性。位于节律钟下游,转录表达受昼夜节律钟控制[29]。在长日照和短日照条件下,的时间表达模式相似,均在第30天时其转录水平达到峰值,而后至第55天降至最低水平,后于开花前保持不变。在短日照条件下,突变株中表现出昼夜节律性表达的开花调节因子转录表达水平显著降低,表现出光周期的强烈敏感性;在长日照条件下,这些成花调节因子的转录表达没有显著改变[30]。乙烯应答因子()通过调控表达,影响茎中淀粉积累,造成晚花表型[31]。
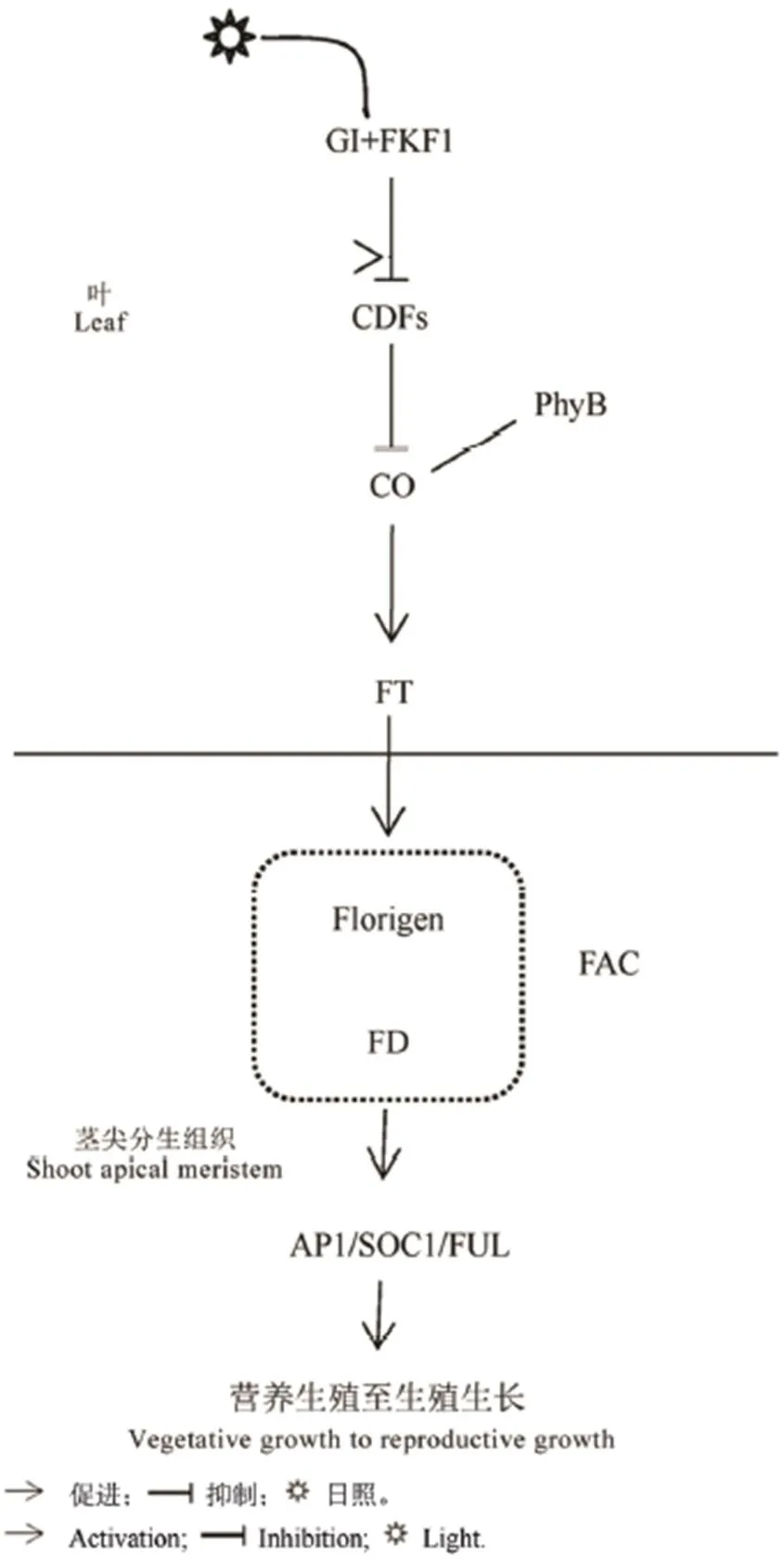
图1 拟南芥开花光周期调控途径
Fig. 1. Photoperiodic regulatory pathway offlowering.
同时,影响()和几个()基因的表达,包括和。表达未受影响,表明影响水稻开花,但独立于途径[32]。转录因子也存在于水稻基因组中,其中的与光周期开花相关。在长日照条件下,过表达的转基因植株表达水平上升,开花较野生型植株提前;但在短日照条件下,表达无显著变化。过表达不能引起mRNA水平的改变,表明是独立于开花的遗传途径[33]。
位于第3染色体,在长日照条件下,延迟抽穗。编码酪蛋白激酶CK2 的α亚基,不同于拟南芥,该蛋白不依赖于昼夜节律机制,对开花调控需要功能性基因。长日照条件下,非直接调控表达,而是间接通过磷酸化相关复合物促进对的抑制作用[34]。
1.2 Hd1表达的抑制因子
()编码一个C3H4 环结构域的E3泛素连接酶,具有E3泛素连接酶活性。能与Hd1蛋白互作,并能泛素化及介导Hd1蛋白的降解,在短日照和长日照条件下,突变体均表现为晚花表型[35]。此外,在短日照条件下,双突变体的开花较突变体迟,但在长日照条件下表现出与相似的抽穗期,对Hd1蛋白积累精准调控以确保适时的光周期反应[36];在长日照下,介导HIF1(HAF1 INTERACTING FACTOR 1)蛋白泛素化,并通过26S蛋白酶体复合物对HIF1蛋白进行降解完成开花调控[37]。最新的研究表明,为体内外泛素化的直接底物,可精确调节积累的昼夜节律,以确保适时的开花。与互作域内的一个氨基酸变异(L558S)对粳稻品种抽穗期的变异有很大的贡献,携带()型等位基因的粳稻品种分布在高纬度地区,携带()型等位基因的粳稻品种分布在低纬度地区[38]。
()编码一个RNA/DNA结合蛋白,能够与SPL11在细胞核内互作,在短日照和长日照条件下,过表达均导致晚花表型,是一个开花负调控因子[39],但具体调控机制不同,在短日照条件下通过介导实现,而在长日照条件下则通过独立于途径调控表达[40]。SPL11为水稻U-box/ARM类型的E3泛素连接酶,通过与SPIN1蛋白互作,负调控细胞程序性死亡(PCD)和抗病性,进而控制开花时间[41]。()为开花时间负调节因子,它与存在正向调控关系,与存在负向调控关系[42]。在细胞核中与互作,同时也是控制开花和PCD途径之间的联系桥梁[43],在短日照和长日照条件下,其过表达均导致水稻开花延迟[44]。
2 Ghd7-Ehd1途径
水稻成花除保守的信号传导途径,还有特有的途径,该途径包含了()()()()等水稻特有基因(拟南芥中无对应同源基因)参与调控。
(1)在现代机械制造工艺的发展中,市场经济发挥着至关重要的作用。现代机械制造工艺更具有市场化,其所包含的包装以及运输等等都是为了满足市场经济的需求。
()基因是水稻中另一个重要的光信号整合点,为来自非洲栽培稻(Steud.)的早抽穗数量性状基因,位于第10染色体,编码一个含341个氨基酸的B型RR蛋白(B2 type response regulator protein),通过与DNA结合的GARP域()调控靶基因表达[45]。短日照条件下,GARP域的突变或RNA干扰均造成基因表达水平降低。与调控途径为平行通路,与基因在水稻开花调控途径中有双重功能不同,无论在短日照条件下还是长日照条件下,在水稻开花途径中都起促进作用[46]。
的调控对于正确的开花时间选择至关重要,大量对其促进和抑制的调控表达的基因已完成克隆(图2)。的抑制因子在光周期网络中起着核心作用,其中,对的昼夜节律和季节性转录表达最为重要[47]。位于第7染色体,编码一个含有CCT(CONSTANS, CO-like, and TOC1)结构的核蛋白,是水稻产量和生态适应性的重要基因[48]。长日照条件下是的主要抑制因子,进一步发现,和可分别控制表达的昼长阈值,该发现极大丰富了成花素随光周期变化调控水稻开花的机制[49]。除了对开花的影响外,表达量与穗粒数和株高表型正相关,表明存在功能多效性和剂量效应。
与拟南芥成花素基因不同,日长由13.0 h到13.5 h的30 min变化能够显著影响表达。当日长短于13 h时,和表达被诱导,表达被抑制。这种日长与内源基因的相互作用通过和光敏色素的双重调节机制实现。和光响应表达分析定义了一种新型的双阀门系统,该系统设置了水稻成花素基因表达的临界日长,解释了水稻长日下抑制开花,短日下促进开花的现象[50]。所谓阀门系统是指与的表达分别受蓝光和红光的诱导,但蓝光与红光不是任何时候都能诱导和的表达,而是由生物节律钟控制的阀门所调控。清晨为昼夜节律钟设置的光响应敏感时期阀门不受日长影响,在短日照和长日照下,均在清晨开启,在有蓝光射入和功能性参与下,基因得以诱导表达,进而激活基因。OsGI蛋白累积在黎明时达到谷底,因此其对阀门的影响可能是间接的。在长日照条件下,阀门于清晨打开,在红光参与下,基因表达,其对基因的诱导与和蓝光依赖性阀门同时被管控,高水平转录表达足以抑制转录并延迟开花[51]。此时,夜间10 min红光中断处理,可将光敏色素由生理活跃型Pfr转化为生理失活型Pr,表达大幅降低,表明功能性光敏色素是正确表达和开花抑制所必需,且红光信号通过整合在光周期开花网络中[52]。在临界阈值以下,随着日照长度的变短,的最大诱导能力不断受到限制,导致第二天清晨表达量降低。在短日照条件下,阀门于午夜打开,此时缺少红光诱导,导致基因不表达。此时若夜间中断处理,表达则被诱导,从而抑制表达。光周期敏感基因()编码一个血红素加氧酶,参与光敏色素发色团生物合成,功能缺失突变体表现为早花和对光周期不敏感[53]。对水稻三个光敏色素基因()单突变体和双突变体研究发现,同源二聚体和异源二聚体均可诱导转录表达,而可单独抑制表达[54-55],三个光敏色素通过基因网络协调控制水稻成花素表达的临界日长。
在拟南芥中未发现和的同源基因[56],也未发现类似水稻的双阀门系统。与表达不同,表达也无明确的临界日长阈值。对不同纬度地区收集的72个拟南芥种质资源研究发现,大多数种质资源在短日照和长日照条件下可以区分日长差异2 h变化,但不能明确确定光周期临界日长[57]。水稻种质资源在不同纬度、不同地域间存在广泛的遗传变异,长日照和短日照下促进开花的光周期感知方式存在一定差异,这一现象是不同类型种质资源对特定生态环境适应的结果。
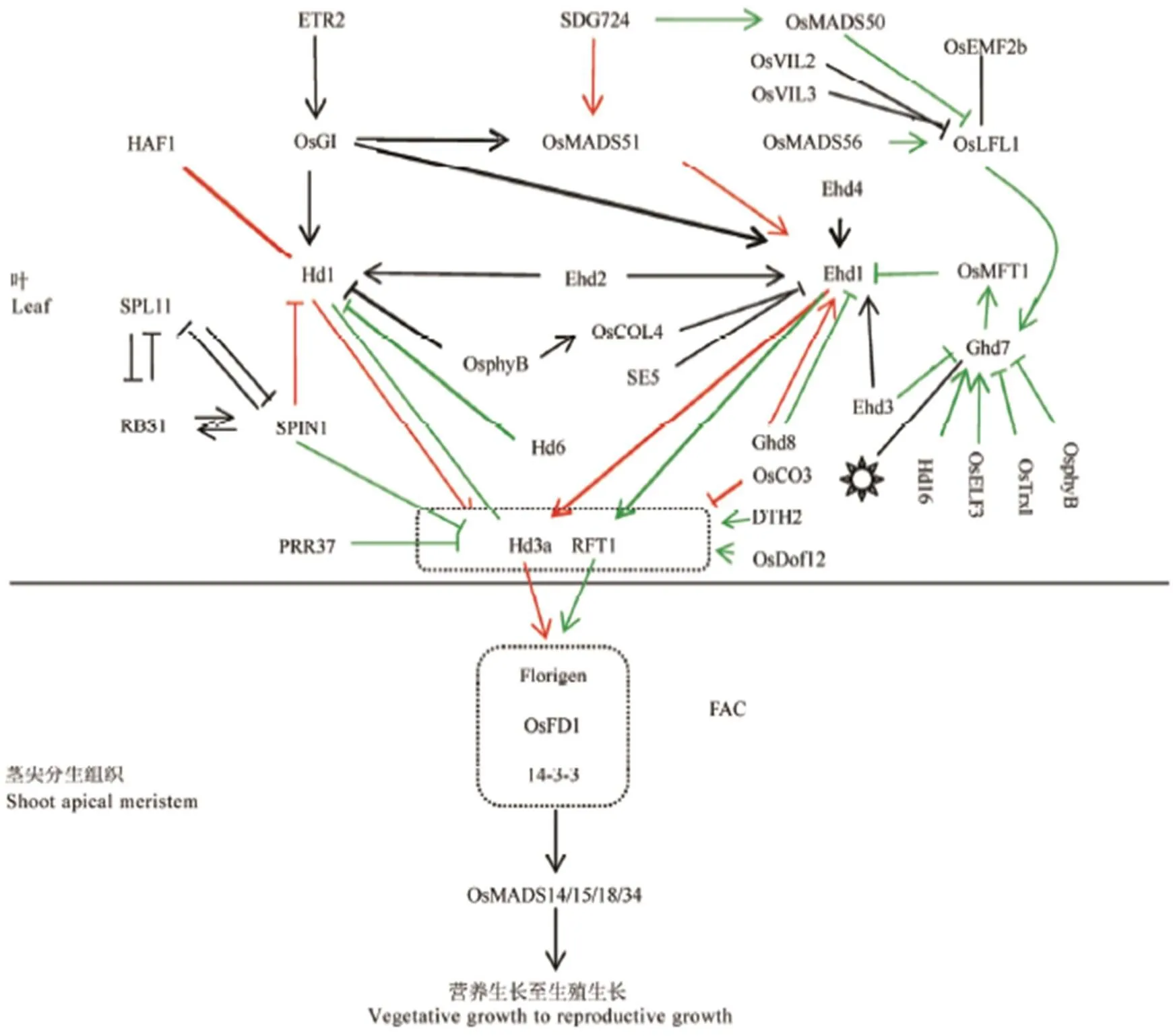
图2 水稻开花的光周期调控途径
Fig. 2. Photoperiodic regulatory pathway of rice flowering.
2.1 Ghd7调控
和之间互作的双阀门系统对日长测量至关重要,而转录表达和转录后调控决定了测量的精度[58]。()()和()通过水平变化直接或间接参与途径。位于第3染色体,编码一个酪蛋白激酶,参与赤霉素介导的开花途径;长日照条件下,通过对磷酸化作用,从而增强其功能,下调基因和的转录水平,最终延迟开花[59]。编码一个植物同源结构域的锌指蛋白,位于细胞核,是水稻开花的关键促进因子,长日照条件下,通过抑制转录表达,间接诱导表达[60],同时,又能通过一个不依赖的方式直接上调的表达[61]。是拟南芥同源基因,通过正调控的表达,负调控基因,影响水稻昼夜节律;在长日照下,通过抑制间接充当开花促进因子,与生物钟基因互作促进水稻开花[62]。
2.2 Ehd1表达的促进因子
位于第10染色体,编码一个型锌指转录因子,与玉米()基因同源。在长日照和短日照条件下,突变体的和成花素基因水平降低,导致极端晚花表型,长时间停留在营养生长阶段[63]。不依赖于昼夜节律,控制开花转换和成花诱导的起始,是水稻从营养生长向生殖生长转变的“总开关”。
()编码一个新型的CCCH类锌指蛋白,该蛋白位于细胞核,在体外可与核酸结合。通过上调成花素基因和表达水平促进开花,与无关。进一步发现,不能直接与启动子区域结合,表明可能是的间接靶标基因[64]。
OsMADS51为I型MADS-box转录因子,在短日照条件下,直接作用于,促进开花。过表达或抑制表达在短日照条件下对开花均有影响,但在长日照条件下对开花无影响。位于和下游,编码一个SET结构域的组蛋白甲基转移酶,通过调控和位点H3K36,影响途径促进水稻开花[65]。和与功能差异,部分是由于H3K36me2/3甲基化水平积累差异造成。通过和蓝光信号参与两种途径促进的表达,后者对水稻临界日长反应的形成至关重要[66]。参与将和信号传递至,两者功能的缺失,均导致和表达量降低,在长、短日照条件下均延迟开花[67]。
2.3 Ehd1表达的抑制因子
()编码一个CCAAT元件结合蛋白,是异源三聚亚铁血红素激活蛋白(HAP)家族的成员。定位于核内,但不具有转录活性。过表达并不造成开花时间的改变,而抑制的表达可以使得水稻开花提前2~3 d。在长日照条件下,通过抑制进而抑制水稻成花素基因及的表达而调控开花。在短日照条件下,可诱导和表达,促进开花,表现为类似的双功能性[68]。与之间存在互作,可以导致极端晚花表型。不能调控的表达。与拟南芥基因高度同源,在拟南芥中过表达可造成拟南芥在长日照条件下提前开花,这与在拟南芥中的功能类似,表明与存在功能保守[69]。
为调控水稻开花和穗型的基因,过表达抑制的转录表达表现为开花延迟,但显著增加每穗颖花数和分蘖数;敲除则导致开花提前,每穗颖花数减少[70]。位于上游的基因还包括())()。编码含有B3 DNA结合结构域的转录因子,通过RY基序与的启动子直接结合,可在体内和体外激活的表达。为VEL家族基因,编码蛋白均具有PHD结构域保守的C4HC3的基序特性[71]。在长、短日照条件下,突变体上调基因,下调和基因,表现为晚花表型。为拟南芥的同源基因,其T-DNA插入突变体和RNAi转基因植株均导致晚花表型,而过表达则导致早花表型[72]。结合至的启动子区,并通过修饰组蛋白H3K27的三甲基化水平来抑制的表达。和均属于MIKC型MADS -box蛋白,长日照条件下,二者形成功能拮抗的复合异质二聚体,参与途径,其中过表达提高表达水平,呈现晚花表型[73]。
()是水稻()家族成员,是组成型开花抑制因子。作用于上游和下游。在长日照和短日照条件下,均作为抑制因子作用于,且独立于等开花途径[74]。
3 OsGI-Hd1-Hd3a和Ghd7-Ehd1途径的表观遗传调控因子
植物成花调控经历着复杂的染色质修饰。目前,已发现、、、()和()参与拟南芥成花调控,参与水稻和途径成花调控。拟南芥的与水稻分别同源,且均为SET[Su(var)3-9, Enhancer-of-zeste, Trithorax]结构域KMT3亚家族成员[75]。拟南芥的和与水稻均是通过H3K36甲基化酶的活性影响植物开花,而拟南芥的与水稻同源基因具有不同的甲基化酶活性,分别通过改变H3K27和H3K36甲基化水平调控开花[76]。通过调控和染色质区域的H3K36甲基化水平,影响途径促进水稻开花,和、与功能差异,部分原因是由于H3K36me2/3甲基化水平积累差异造成[77]。功能缺失后,在长、短日照条件下均延迟抽穗,和表达量下降。与相同,也通过H3K36 me2/3甲基化介导开花。具有组蛋白H3K36专一性的甲基转移酶活性,表达量下调突变体在长、短日照条件下均为晚花表型,等基因表达水平大幅下调,而表达水平上调[78]。染色质免疫共沉淀发现与SDG725蛋白以相互促进的方式结合在与基因的染色质区域,并通过建立H3K36me3促进基因转录,表明催化的H3K36甲基化修饰对水稻具有促进作用。与类似,拟南芥EMF2在EMF1的协助下,通过介导H3K27me3甲基化方式作用于靶基因染色质,影响开花[79-80]。与拟南芥机制不同,水稻()同源基因通过抑制表达来促进开花,同时也参与H3K27me3甲基化方式调控开花[81]。编码一个SET结构域包含蛋白SDG723,长日照条件下,突变导致晚花表型。的PHD基序能与组蛋白H3结合,SET结构域C端具有组蛋白H3甲基转移酶活性,使得与形成复合体甲基化下游基因[82]。位于上游,作为的负调控因子,通过途径调节染色质结构调控水稻开花。
4 其他独立途径和相关调控基因
水稻成花素调控途径中,存在独立于和两途径之外的其他调节因子,如和。和编码一个含有两个B-box和一个CCT结构域蛋白,与Hd1一样,均属于COL蛋白家族成员[83]。在短日照条件下,过表达植株的、FTL表达量降低,抽穗延迟,而成花素上游基因表达量无显著变化,表明在短日照条件下,独立于已知短日照调控途径,作为抑制子负调控成花素基因;为位于第2染色体,在长日照条件下促进水稻抽穗的微效QTL。在第25位的S1(A/G)和第1721位的S4(T/G)存在两个功能核苷酸多态位点,在亚洲北部自然长日照生态环境下,与长期人工选择早抽穗和提升繁殖有关[84]。是位于水稻第3染色体上的单拷贝基因,编码一个440个氨基酸组成的Dof蛋白。表达无组织特异性,但受暗处理强烈抑制。长日照条件下,过表达使得水稻抽穗提前,成花素表达量上升;短日照条件下,过表达对抽穗期无影响;在不同的日照条件下,成花素上游基因表达量无显著性变化,表明是独立于的长日照抽穗促进因子[85]。
5 成花素基因及顶端分生组织内成花素受体及功能
植物由叶片感知光周期变化,产生可移动信号从叶片经长距离运输至顶端分生组织诱导开花,此信号即为成花素。Laurent等[86]通过GFP(green fluorescence protein)荧光蛋白追踪了FT蛋白从叶片移动到顶端分生组织全过程,并通过嫁接实验,进一步证明FT蛋白是拟南芥成花素。水稻与拟南芥高度同源,在叶片维管组织中表达,产生成花素蛋白,经由维管组织运输至顶端分生组织与下游蛋白互作,启动茎尖分生组织向花序分生组织转变。是水稻短日照下成花素基因,在短日照下高表达,在长日照下几乎不表达,临界日长13 h。隶属于类基因家族,是磷脂酰乙醇胺()基因家族中的一个亚族。家族基因在植物中普遍存在,具有高度保守性,编码一个小分子球状蛋白,该类蛋白由一个中央较大、两侧较小的β-折叠和α-螺旋形成的阴离子结合袋,能够识别并结合氨基酸磷酸化残基[87]。对从拟南芥、番茄、水稻等植物分离出的类基因研究发现,类基因家族编码的蛋白质均具有成花素活性,控制植物由营养生长向生殖生长转变和开花时间[88]。水稻类基因家族共有13个成员(至),其中至与水稻开花时间无关,()与同源性最高,均位于第6染色体短臂,相距11.5 kb[89]。是水稻长日照下成花素,长日照下表达受到抑制时,表达量上调,其蛋白通过韧皮部移动至顶端分生组织,促进水稻开花。和基因在叶片中同样是有活力的,可在叶片中形成转录激活或者抑制复合物,并且可以反馈调节自身转录[90]。
顶端分生组织位于茎与根的主轴和侧枝的顶端,按特定的方式不断分裂分化,形成植物各种器官并发育成特定的形态[91]。在顶端分生组织细胞内,成花素不能直接充当转录调节因子调控下游成花基因,需与()转录因子()一同形成转录激活复合物,起始下游开花基因。水稻的与拟南芥的基因同源,功能高度保守,形成的二聚体与水稻功能类似。Hd3a蛋白位于细胞膜和细胞核,OsFD1蛋白位于细胞核内,两者互作需由14-3-3蛋白(GF14c)介导,该蛋白定位于细胞质内。14-3-3蛋白家族成员高度保守,是一类重要的调节蛋白,通过与蛋白激酶、转录因子及其他信号分子结合参与体内多种信号转导途径的调节[92]。水稻中过表达14-3-3蛋白可延迟开花,其T-DNA插入功能缺失突变体则表现早花,14-3-3蛋白为开花抑制因子。水稻成花素运输至顶端分生组织,与细胞质内的14-3-3蛋白形成Hd3a-GF14c二级复合体,随后该复合体移动至细胞核,与OsFD1蛋白形成异源六聚三级复合体,即成花素激活复合体(florigen activation complex FAC),该结构需要OsFD1的磷酸化作用[93]。
花的形成经历一系列组织的分化调控过程,FAC诱导花分生组织特征基因,进而激活花器管特征基因和下游决定各组织和细胞类型的基因,产生花分生组织,随后发育成花和各类型器官[94]。()为拟南芥花分生组织决定基因,又是花器管发育特征基因,既在花原基发育早期阶段表达,又决定花器官起始和发育[95],在成花中起关键调节作用。花分生组织起始和花分生组织特性维持的决定因子还包括()、()、()和(),其中、和均为MADS-box基因。与高度同源,在花发育早期的花分生组织中均表达,参与决定花分生组织属性,存在功能冗余,可维持在花分生组织中表达;受负调控,在花序分生组织中表达,受介导影响,在花分生组织中不表达,在决定花分生组织属性方面,与、存在功能冗余;通过精准调控B、C类基因的表达而阻止花分生组织的早熟,决定花器官适时的形成,为控制开花时间基因;为基因功能抑制因子,与功能相反;在叶片表达,编码的成花素蛋白运转至茎尖分生组织处,与共同作用,上调和基因的表达,促进花分生组织的形成;在茎尖分生组织处延迟的上调或阻止信号转导,从而抑制AP1活性;TFL1与AP1间的平衡调控着茎尖分生组织样式,确保植物在合适的时间和位置开花[96]。总之,是花序分生组织向花分生组织转换的核心因子;植物体内外信号汇集于开花整合基因和,在分生组织侧翼激活的表达,进而抑制,在分生组织侧翼形成花分生组织;反之,抑制在花序分生组织处表达,在分生组织中央维持花序的无限性[97]。
在水稻中,受FAC调控,与成花转变相关的基因有/和三个AP1/FUL类基因的、和[98],四个基因均为MADS-box家族成员,是成花的正向调节因子。的表达受Hd3a和14-3-3蛋白形成的二级复合体调控,单独的或均不能调控的表达;不同位置的Hd3a蛋白突变体,在不与14-3-3结合情况下,即使与共表达,也不能诱导的表达。表达模式与拟南芥的类似,在颖花发育阶段,其转录表达主要集中在分生组织中,随后在颖片、内稃、外稃和浆片原基中表达对水稻花序和花器官发育、不定根和茎等器官的发育和生长均起着重要作用[99]。MADS-box基因的功能主要通过形成同源或异源二聚体或四聚体来实现。在烟草 () 细胞 BIFC 实验发现,在细胞核内,相连的nYFP可与相连的cYFP分别形成互补而产生荧光,证实两基因间存在互作,但其机理和功能还有待研究[100]。当和三个基因同时被抑制时,花序的发育并未受到严重影响;而同时基因敲除时,即四个基因表达同时受到抑制时,则严重阻碍植物从营养生长向生殖生长转变。同时,单基因突变体的生殖生长转变为正常。这表明三个基因与基因在决定花序分生组织特征建成时,存在部分冗余和功能互作[101]。与拟南芥高度同源,在长日照下,通过途径正向调节成花,在茎尖分生组织可检测到转录表达,但尚不清楚是否参与茎尖分生组织成花网络[102]。
6 由低纬度至高纬度地区扩展的遗传变异
野生稻(为栽培稻()的原始祖先,主要分布在低纬度地区,对短日敏感,主要分布于热带和亚热带地区。而栽培稻在中国和朝鲜的北部省份,自然日长接近15 h,最高纬度至53°N。在水稻驯化过程中,由热带和亚热带地区北移至温带地区伴随着开花性状的人工选择,以确保在寒冷的天气到来之前完成收获,其中光周期敏感性的降低、非诱导条件下开花是水稻由低纬度至高纬度地区适应性成功的关键。光周期敏感性受之前所述大量QTL的影响,如和为主要关键因子,成花素基因为其靶向调控基因。
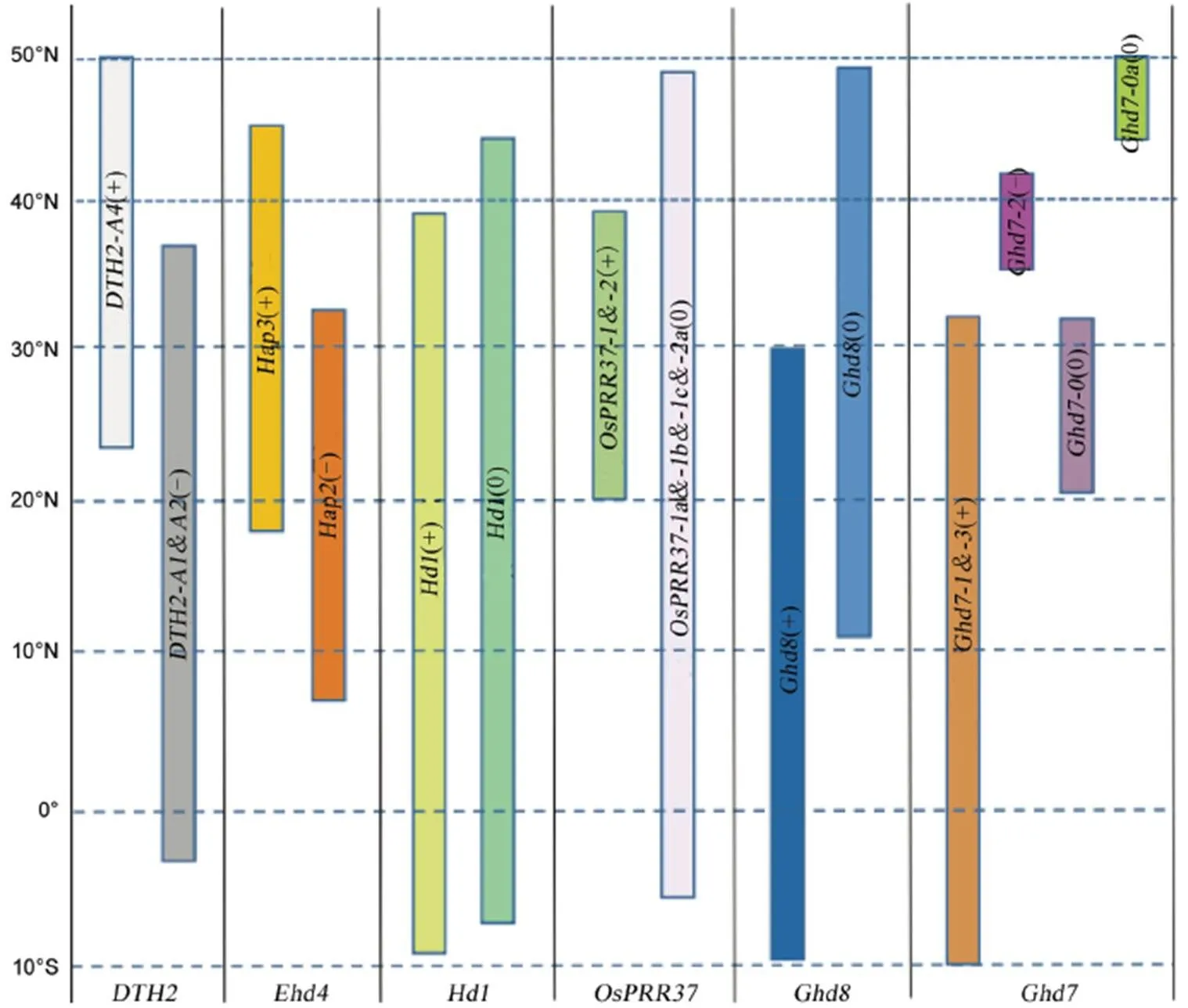
竖轴代表纬度;横轴代表基因;+、−、0分别代表等位基因的强效应、弱效应和无效应。
Fig. 3. Distribution of alleles influencing flowering of rice.
水稻开花时间多样性受成花素基因转录表达水平调节,与成花素基因和成花素调控基因的自然变异息息相关。水稻核心种质资源中启动子区域变异和基因组编码区单核苷酸突变、蛋白质序列的氨基酸替换均导致成花时间的多样性[103-104]。在长日照地区(日长大于13 h,纬度为23.6°N-53°N),水稻品种利用和途径参与成花,在低纬度地区(10°S-23.6°N),大部分品种为途径参与成花,表明较,可能是水稻长日照条件下控制开花的关键基因[105]。鉴于成花素基因物种间的高度保守,开花时间的多样性主要受成花素调控基因的影响。
成花素基因促进因子的自然变异对水稻栽培向北扩增具有重要作用(图3)。为效应较小的水稻抽穗数量性状位点,存在两个功能核苷酸多态S1和S4,引起抽穗期的变化较小(1周内),但对水稻产量和生殖适应具有较大差异;通过人工不断驯化选择,向北扩展的粳稻品种不断降低核苷酸多态性,在短生育期的粳稻地方品种中广泛存在的A4等位基因,主要分布于日本、韩国和中国北方高纬度地区。两个功能核苷酸多态(S1和S4)可能在很大程度上促进了水稻品种的分布和适应不同生态的当地气候,并且在水稻向北扩展期间选择了的A4等位基因[106]。有两个主要单体型,籼稻品系的主要单体型为,主要分布在低纬度和低海拔地区;粳稻品系主要单体型为,主要分布在高纬度和高海拔地区;遗传分析表明,在自然长日照条件下促进开花的作用更强,是促进栽培稻向高纬度地区北移和区域适应的主要原因[107]。
长日照条件下,成花抑制因子在抑制成花素基因表达,导致抽穗延迟方面起着关键性作用(图3),其非功能性等位基因与光周期敏感性降低相关。和基因的非功能性等位基因由碱基的插入、缺失或替换产生,导致成花素表达和开花发生异常。基因具有自发的高度多态性,在不同纬度地区均发现了携带突变体的水稻品种,证实高度多态性与转录水平变化和开花时间相关。功能性和非功能性的等位基因分别影响开花的早迟,表明是水稻品种中决定开花时间变异的重要因子[108]。在水稻生长的短日照地域,携带非功能性等位基因品种延迟开花,这对于延长营养生长期以增加稻谷产量非常重要;相反,在长日照地域,携带非功能性等位基因品种有望抽穗,有助于进一步向高纬度地区延伸种植。长日照条件下,抑制的表达,在温带地区及水稻栽培的最北地区,均存在无功能的等位基因品种。在长日照条件下下调的表达,进而抑制和的转录;根据GHD7蛋白序列可将分为5个等位基因,和效应较大,长日照条件下延迟开花,主要分布在热带、亚热带地区栽培品种;存在于温带地区的粳稻品种中;和翻译提前终止的均为无功能等位基因,在长日照条件下,没有或减少延迟抽穗的作用,分布在华中和华南双季稻早稻品种中,而则分布在黑龙江地区品种中[5]。的同源基因使得玉米从热带向温带地区扩展[109]。栽培品种中不同成花相关QTL的不同等位基因组合有助于栽培面积和地区的扩展,并且当靶向独立抑制因子参与时,在长日照地区可出现早花表型[110]。遗传分析表明,和等位基因对抽穗期的影响是加性的,携带和的两个非功能等位基因的粳稻品种极早开花,并能够适应北纬53°的水稻栽培最北部地区,表明和的自然变异有助于水稻向温带和寒冷地区扩展[111]。但和对成花素的调控途径不同,作为独立途径,不参与对的调控。在中国、法国、菲律宾和日本最北端的北海道地区品种存在广泛的变异,携带非功能等位基因表现为早抽穗,该非功能基因来自于20世纪90年代北海道水稻育种项目选育的早抽穗品种,在长日照地区表现出比功能等位基因表型更好的农艺优势[112];独立于与调控途径,通过、突变等位基因的累积有可能进一步降低光周期敏感性和作物周期。、、与基因大部分变异位点间存在一定的连锁关系,而和位点间连锁更为显著,在进化过程中可能是协同进化影响开花时间[113]。
除了和外,其他的成花素调控基因的DNA序列多态性也促进了水稻向北扩展。在长日照下介导途径,携带非功能等位基因降低了(非完全)对的抑制作用,促进了开花时间的多样性,在部分粳稻品种中均存在非功能等位基因。为长日照依赖性抑制因子,在长日照地区,通过对的磷酸化抑制的表达。在高纬度地区栽培的粳稻品种中发现了两种类型的非功能性变异,而携带功能性品种分布与纬度无关,表明的自然变异有助于温带和寒冷地区水稻对长日照提早抽穗的适应。和突变不仅是控制开花时间的有益突变,而且可用于提高日本品种越光的产量[114]。
7 展望
水稻成花调控的研究极大扩展了对成花诱导和调控分子机制的理解,随着新基因挖掘和基因间互作机制的深入理解,分子调控网络愈加复杂。成花调控分子网络还涉及了激素途径、自主途径、温度途径,与光周期途径之间既相对独立又相互配合,构成植物自身内源信号和外界环境信号对开花时间的精准调控,彰显了植物成花机制的复杂性。尽管依赖于和调控途径的研究取得了巨大的进展,克隆了大量相关基因,但基因间的作用方式、各功能基因和非功能等位基因组合对外界环境信号的响应以及和基因表达的转录和转录后调控并不完全清晰,同时涉及表观遗传调控开花时间的组蛋白甲基化相关蛋白(如SDG708、SDG724、SDG725、OsTrx1、OsVIL2等)引起的主要开花基因的转录沉默或激活仍需深入探讨。但随着基因组测序、转录组分析、基于CRISPR(clustered regularly interspaced short palindromic repeats)基因编辑以及互作蛋白的大规模筛选等探索手段的多样化,新的开花节点基因和遗传调控网络会不断拓展,对不同物种间生态环境生存策略的差异,千差万别的遗传调控网络将具有很好的解析。
水稻开花调控机制的研究不仅仅停留于基础科研,在科研转化应用上具有广阔的前景。对重要的成花基因(如、和)的研究发现,成花基因也存在杂种优势,不同等位形式组合对单穗粒数、结实率以及单株籽粒产量等方面均优于亲本,在杂交水稻三系和两系品种中均有大面积应用。结合低纬度至高纬度特定地域的最优成花基因等位形式及组合,将为育种家生态型品种的选育提供更加针对性、有效性的选择,为未来分子设计育种奠定基础。
[1] 徐铨, 奥本裕, 王晓雪. 水稻开花期调控分子机理研究进展[J]. 植物遗传资源学报, 2014, 15(1): 134-140.
Xu Q, Ao B Y, Wang X X. Research progress on regulatory molecular mechanisms of flowering time in rice[J]., 2014, 15(1): 134-140. (in Chinese with English abstract)
[2] Hayama R, Yokoi S, Tamaki S, Yano M, Shimamoto K. Adaptation of photoperiodic control pathways produces short-day flowering in rice[J]., 2003, 422(6933): 719-722.
[3] 魏鑫, 曹立荣, 杨庆文, 曾汉来. 水稻抽穗期的光周期调控分子基础研究进展[J]. 生命科学研究, 2010, 14(5): 456-463.
Wei X, Cao L R, Yang Q W, Zeng H L. Research progress of photoperiodic response controlling heading in rice[J]., 2010, 14(5): 456-463. (in Chinese with English abstract)
[4] Chaw S M, Chang C C, Chen H L, Li W H. Dating the monocot–dicot divergence and the origin of core eudicots using whole chloroplast genomes[J]., 2004, 58(4): 424-441.
[5] Xue W Y, Xing Y Z, Weng X Y, Zhao Y, Tang W J, Wang L, Zhou H J, Yu S B, Xu C G, Li X H, Zhang Q F. Natural variation inis an important regulator of heading date and yield potential in rice[J]., 2008, 40(6): 761-767.
[6] Nakamichi N, Tudo T, Makita N, Kiba T, Kinoshita T, Sakakibara H. Flowering time control in rice by introducingis clock-associated PSEUDO- RESPONSE REGULATOR 5[J]., 2020, 3(27): 970-979.
[7] Zhang W F, Tan L B, Sun H Y, Zhao X H, Liu F X, Cai H W, Fu Y C Sun X Y, Gu P, Zhu Z P, Sun C Q. Natural variations atencoding a TCP transcription factor contribute to plant architecture domestication in rice[J]., 2019, 12 (8): 1075-1089.
[8] Chen E W, Huang X H, Tian Z X, Wing R A, Han B. The genomics ofspecies provides insights into rice domestication and heterosis[J]., 2019, 70: 639-665.
[9] Liu Y, Yang J, Yang M. Pathways of flowering regulation in plants[J]., 2015, 31(11): 1553-1566.
[10] 彭凌涛. 控制拟南芥和水稻开花时间光周期途径的分子机制[J]. 植物生理学通讯, 2006, 42(6): 1021-1030.
Peng L T. Molecular mechanism of flowering time controlling photoperiod pathway inand rice[J]., 2006, 42(6): 1021-1030. (in Chinese)
[11] Sawa M, Nusinow D A, Kay S A, Imaizumi T. FKF1 and GIGANTEA complex formation is required for day-length measurement in[J]., 2007, 318(5848): 261-265.
[12] Imaizumi T, Schultz T F, Harmon F G, Ho L A, Kay S A. FKF1 F-box protein mediates cyclic degradation of a repressor of CONSTANS in[J]., 2005, 309(5732): 293-297.
[13] Niwa Y, Ito S, Nakamichi N, Mizoguchi N, Niinuma T, Yamashino K, Mizuno T. Genetic linkages of the circadian clock-associated genes,,and, in the photoperiodic control of flowering time in[J]., 2007, 48(7): 925-937.
[14] Chang G X, Yang W J, Zhang Q L, Huang J L, Yang Y P, Hu X Y. ABI5-BINDING PROTEIN2 coordinatesto delay flowering by recruiting the transcriptional corepressor TPR2[J]., 2019, 179(2): 477-490.
[15] Kozarewa I, Ibáñez C, Johansson M, Ogren E, Mozley D, Nylander E, Chono M, Moritz T, Eriksson M E. Alteration of PHYA expression change circadian rhythms and timing of bud set in[J]., 2010, 73(12): 143-156.
[16] Liu L J, Zhang Y C, Li Q H, Sang Y, Mao J, Lian H L, Wang L, Yang H Q. COP1-mediated ubiquitination ofis implicated in cryptochrome regulation of flowering in[J]., 2008, 20(2): 292-306.
[17] 骆倩. 拟南芥PIF3-LIKE1与phyB和COP1互作调控光形态建成的分子机制研究[D]. 上海: 上海交通大学, 2014.
Luo Q. COP1 and phyB physically interact with PIL1 to regulate its stability and photomorphogenic development in[D]. Shanghai: Shanghai Jiaotong University, 2014. (in Chinese with English abstract)
[18] Pittendrigh C S, Minis D H. The entrainment of circadian oscillations by light and their role as photoperiodic clocks[J]., 1964, 98(902): 261-294.
[19] 陈福禄, 傅永福, 林辰涛./调节元件与植物开花时间调节研究进展[J]. 中国农业科技导报, 2009, 11(2): 17-22.
Chen F L, Fu Y F, Lin C T. Research progress onregulon and its role in adjusting plant flowering[J]., 2009, 11(2): 17-22. (in Chinese with English abstract)
[20] Yano M, Katayose Y, Ashikari M, Yamanouchi U, Monna L, Fuse T, Baba T, Yamamoto K, Umehara Y, Nagamura Y, Sasaki T., a major photoperiod sensitivity quantitative trait locus in rice, is closely related to theflowering time gene[J]., 2000, 12(12): 2473-2483.
[21] Izawa T, Takahashi Y, Yano M. Comparative biology comes into bloom: Genomic and genetic comparison of flowering pathways in rice and[J]., 2003, 6(2): 113-120.
[22] Ogiso E, Takahashi Y, Sasaki T, Yano M, Izawa T. The role of casein kinaseⅡin flowering time regulation has diversified during evolution[J]., 2010, 152(2): 808-820.
[23] Murakami M, Tago Y, Yamashino T, Mizuno T. Characterization of the rice circadian clock-associated pseudo-response regulators in[J].2007, 71(4): 70048-70052.
[24] Kojima S, Takahashi Y, Kobayashi Y, Monna L, Sasaki T, Araki T, Yano M., a rice ortholog of thegene, promotes transition to flowering downstream ofunder short-day conditions[J]., 2002, 43(10): 1096-1105.
[25] Ishikawa R, Aoki M, Kurotani K, Yokoi S, Shinomura T, Takano M, Shimamoto K. Phytochrome B regulates()-mediated expression of rice florigenand critical day length in rice[J]., 2011, 285(6): 461-470.
[26] Tsuji H, Taoka K, Shimamoto K. Florigen in rice: Complex gene network for florigen transcription, florigen activation complex, and multiple functions[J]., 2013, 16(2): 228-235.
[27] Goretti D, Martignago D, Landini M, Brambilla V, Ariza J G, Gnesutta N, Galbiati F, Collani S, Takagi H, Terauchi R, Mantovani R, Fornara F. Transcriptional and post-transcriptional mechanisms limit() function to adapt rice to high latitudes[J]., 2017, 13(1): e1006530.
[28] Nemoto Y, Nonoue Y, Yano M, Izawa T., a CONSTANS ortholog in rice, functions as anrepressor through interaction with monocot-specific CCT-domain protein Ghd7[J]., 2016, 86(3): 221-233.
[29] Lee Y S, An G.controls flowering time by modulating rhythmic flowering time regulators preferentially under short day in rice[J]., 2015, 58(2): 137-145.
[30] Nguyen Q N, Lee Y S, Cho L H, Jeong H J, An G, Jung K H. Genome-wide identification and analysis ofRLK1-like kinases in rice[J]., 2015, 241(3): 603-613.
[31] Wuriyanghan H, Zhang B, Cao W H, Ma B, Lei G, Liu Y F, Wei W, Wu H J, Chen L J, Chen H W, Cao Y R, He S J, Zhang W K, Wang X J, Chen S Y, Zhang J S. The ethylene receptor ETR2 delays floral transition and affects starch accumulation in rice[J]., 2009, 21(5): 1473-1494.
[32] Koo B H, Yoo S C, Park J W, Kwon C T, Lee B D, An G, Zhang Z Y, Li Z C, Paek N C. Natural variation inregulates heading date and contributes to rice cultivation at a wide range of latitudes[J]., 2013, 6(6): 1877-1888.
[33] Li D J, Yang C H, Li X B, Gan Q, Zhao X F, Zhu L H. Functional characterization of rice[J]., 2009, 229(6): 1159-1169.
[34] Takahashi Y, Shomura A, Sasaki T, Yano M., a rice quantitative trait locus involved in photoperiod sensitivity, encodes the α subunit of protein kinase CK2[J]., 2001, 98(14): 7922-7927.
[35] Yang Y, Fu D, Zhu C, He Y, Zhang H, Liu T, Wu C. The RING-finger ubiquitin ligase HAF1 mediates heading date 1 degradation during photoperiodic flowering in rice[J]., 2015, 27(9): 2455-2468.
[36] Subudhi P K, De Leon T B, Tapia R, Chai C, Karan R, Ontoy J, Singh P K. Genetic interaction involving photoperiod-responsivepromotes early flowering under long-day conditions in rice[J]., 2018, 8(1): 1-11.
[37] Zhu C, Peng Q, Fu D, Zhuang D X, Yu Y M, Duan M, Xie W B, Cai Y H. The E3 ubiquitin ligase HAF1 modulates circadian accumulation of EARLY FLOWERING3 to control heading date in rice under long-day conditions[J]., 2018, 30(10): 2352-2367.
[38] Yang Y, Peng Q, Chen G X, Li X H, Wu C Y.is involved in circadian clock regulation for promoting flowering under long-day conditions in rice[J]., 2013, 6(1): 202-215.
[39] Vega-Sánchez M E, Zeng L R, Chen S B, Leung H, Wang G L. SPIN1, a K homology domain protein negatively regulated and ubiquitinated by the E3 ubiquitin ligase SPL11, is involved in flowering time control in rice[J]., 2008, 20(6): 1456-1469.
[40] Cai Y, Vega-Sánchez M E, Park C H, Bellizzi M, Guo Z, Wang GL. RBS1, an RNA binding protein, interacts with SPIN1 and is involved in flowering time control in rice[J]., 2014, 9(1): e87258.
[41] Liu J, Park C H, He F, Nagano M, Wang M, Bellizzi M, Zhang K, Zeng X S, Liu W D, Ning Y, Kawano Y, Wang G L. The RhoGAP SPIN6 associates with SPL11 and OsRac1 and negatively regulates programmed cell death and innate immunity in rice[J]., 2015, 11(2): e1004629.
[42] Yamashita H, Komeda Y. Control of flower development[J].2010, 15(1): 195-208.
[43] Vega-Sanchez M E. The E3 ubiquitin ligase SPL11 regulates both programmed cell death and flowering time in rice[D]. Ohio: The Ohio State University, 2008.
[44] Shirsekar G S, Vega-sanchez M, Bordeos A, Baraoidan M, Swisshelm A, Fan J B, Park C H, Leung H, Wang G L. Identification and characterization of suppressor mutants of-mediated cell death in rice[J].2014, 27(6): 528-536.
[45] Doi K, Izawa T, Fuse T, Yamanouchi U, Kubo T, Shimatani Z, Yano M, Yoshimura A., a B-type response regulator in rice, confers short-day promotion of flowering and controls FT-like gene expression independently of[J]., 2004, 18(8): 926-936.
[46] 宋远丽, 栾维江. 水稻开花的光温调控分子机理[J]. 中国水稻科学, 2012, 26(4): 383-392.
Song Y L, Luan W J. The regulatory pathways of rice flowering in different light and temperature conditions[J]., 2012, 26(4): 383-392. (in Chinese with English abstract)
[47] Lu L, Yan W, Xue W Y, Shao D, Xing Y Z. Evolution and association analysis ofin rice[J]., 2012, 7(5): e34021.
[48] 崔志文, 夏烨, 孙小娟, 蔡志明. 温度和光周期对水稻抽穗期调控的交互作用[J]. 生命科学, 2012, 24(4): 316-320.
Cui Z W, Xia Y, Sun X J, Cai Z M. Interaction between temperature and photoperiod in regulation of flowering time in rice[J]., 2012, 24(4): 316-320. (in Chinese)
[49] Itoh H, Nonoue Y, Yano M, Izawa T. A pair of floral regulators sets critical day length forflorigen expression in rice[J]., 2010, 42(7): 635-638.
[50] Xu Q, Ao B, Wang X X. Review of mechanism of rice flowering in response to photoperiod[J]., 2014, 15(1): 129-136.
[51] Lee Y S, Yi J, An G. OsPhyA modulates rice flowering time mainly throughunder short days andunder long days in the absence of phytochrome B[J]., 2016, 91(4): 413-427.
[52] Xu Q, Saito H, Hirose I, Katsura K, Yoshitake Y, Yokoo T, Tsukiyama T, Teraishi M, Tanisaka T, Okumoto Y. The effects of the photoperiod-insensitive alleles,,and, on yield components in rice[J]., 2014, 33(4): 813-819.
[53] 胡时开, 苏岩, 叶卫军, 郭龙彪. 水稻抽穗期遗传与分子调控机理研究进展[J]. 中国水稻科学, 2012, 26(3): 373-382.
Hu S K, Su Y, Ye W J, Guo L B. Advances in genetic analysis and molecular regulation mechanism of heading date in rice (L.)[J]., 2012, 26(3): 373-382. (in Chinese with English abstract)
[54] Zheng T, Sun J, Zhou S, Chen S, Lu J, Cui S, Tian Y, Zhang H, Cai M, Zhu S, Wu M, Wang Y, Jiang L, Zhai H, Wang H, Wan J. Post-transcriptional regulation ofprotein stability by phytochrome andin photoperiodic control of flowering in rice[J]., 2019, 224(1): 306-320.
[55] Lee Y S, Yi J, Jung K H, An G. Comparison of rice flowering-time genes under paddy conditions[J]., 2016, 59(3): 238-246.
[56] Matsubara K, Hori K, Ogiso-Tanaka E, Yano M. Cloning of quantitative trait genes from rice reveals conservation and divergence of photoperiod flowering pathways inand rice[J]., 2014, 5(1): 193-200.
[57] Giakountis A, Cremer F, Sim S, Reymond M, Schimitt J, Coupland G. Distinct patterns of genetic variation alter flowering responses ofaccessions to different daylength[J]., 2010, 152(1): 177-191.
[58] Hu Y, Li S, Xing Y. Lessons from natural variations: Artificially induced heading date variations for improvement of regional adaptation in rice[J]., 2019, 132(2): 383-394.
[59] Hori K, Ogiso-Tanaka E, Matsubara K, Yamanouchi U, Ebana K, Yano M., a gene for casein kinase I, is involved in the control of rice flowering time by modulating the day-length response[J]., 2013, 76(1): 36-46.
[60] Xiang C, Qu L J, Gao Y M, Shi Y Y. Flower development and photoperiodic control of flowering in rice[J]., 2013, 20(2): 79-87.
[61] Matsubara K, Yamanouchi U, Nonoue Y, Sugimoto K, Wang Z X, Minobe Y, Yano M., encoding a plant homeodomain finger-containing protein, is a critical promoter of rice flowering[J]., 2011, 66(4): 603-612.
[62] Zhao J M, Huang X, Ouyang X H, Chen W L, Du A P, Zhu S G, Deng X W, Li S G., an ortholog of, regulates rice circadian rhythm and photoperiodic flowering[J]., 2012, 7(8): e43705.
[63] Matsubara K, Yamanouchi U, Wang Z X, Minobe Y, Izawa T, Yano M., a rice ortholog of the maizegene, promotes flowering by up-regulating[J]., 2008, 148(3): 1425-1435.
[64] Gao H, Zheng X M, Fei G L, Chen J, Jin M N, Ren Y L, Wu W X, Zhou K N, Sheng P K, Zhou F, Jiang L, Wang J, Zhang X, Guo X P, Wang J L, Cheng Z J, Wu C Y, Wang H Y, Wan J M.encodes a novel and Oryza-genus-specific regulator of photoperiodic flowering in rice[J]., 2013, 9(2): e1003281.
[65] Sui P F, Shi J L, Gao X Y, Shen W H, Dong A W. H3K36 methylation is involved in promoting rice flowering[J]., 2013, 6(3): 975-977.
[66] 孔德艳, 陈守俊, 周立国, 高欢, 罗利军, 刘灶长. 水稻开花光周期调控相关基因研究进展[J]. 遗传, 2016, 38(6): 532-542.
Kong D Y, Chen S J, Zhou L G, Gao H, Luo L J, Liu Z C. Research progress of photoperiod regulated genes on flowering time in rice[J]., 2016, 38(6): 532-542. (in Chinese with English abstract)
[67] Xie S Y, Chen M, Pei R, Ouyang Y D, Yao J L,.acts as a regulator of flowering transition and floral organ identity by mediating H3K27me3 deposition atandin rice[J]., 2015, 33(1): 121-132.
[68] Wu M J, Liu H Q, Lin Y, Chen J M, Fu Y P, Luo J M, Zhang Z J, Liang K J, Chen S B, Wang F. In-frame and frame-shift editing of thegene to developrice with prolonged basic vegetative growth periods[J]., 2020, 11(4): 307-321.
[69] Dai X D, Ding Y N, Tan L B, Fu Y C, Liu F X, Zhu Z F, Sun X Y, Sun X W, Gu P, Cai H W, Sun C Q., an allele of, controls late heading date in common wild rice ()[J]., 2012, 54(10): 790-799.
[70] Song S, Wang G F, Hu Y, Liu H Y, Bai X F, Qin R, Xing Y Z.increases spikelets per panicle and delays heading date in rice by suppressing,andgenes[J]., 2018, 69(18): 4283-4293.
[71] Wang J, Hu J, Qian Q, Xue H W.andpromote rice flowering by photoperoid-induced epigenetic silencing of[J]., 2013, 6(2): 514-527.
[72] Chen M, Xie S Y, Ouyang Y D, Yao J L. Rice PcG genecontrols seed dormancy and seedling growth by regulating the expression of[J]., 2017, 260(7): 80-89.
[73] Ryu C H, Lee S, Cho L H, Kim S L, Lee Y S, Choi S C, Jeong H J, Yi J, Park S J, Han C D, An G.andfunction antagonistically in regulating long day (LD)-dependent flowering in rice[J]., 2009, 32(10): 1412-1427.
[74] Sheng P K, Wu F Q, Tan J J, Zhang H, Ma W W, Chen L P, Wang J C, Wang J, Zhu S S, Guo X P, Wang J L. Zhang X, Cheng Z J, Bao Y Q, Wu C Y, Liu X M, Wan J M. A CONSTANS-like transcriptional activator,, functions as a negative regulator of flowering downstream ofand upstream ofin rice[J]., 2016, 92(2): 209-222.
[75] Liu B, Wei G, Shi J L, Jin J, Shen T, Ni T, Shen W H, Yu Y, Dong A W. SET DOMAIN GROUP 708, a histone H3 lysine 36-specific methyltransferase, controls flowering time in rice ()[J]., 2016, 210(2): 577-588.
[76] 何星辉, 王金彪, 阮颖, 黄勇. 水稻抑制拟南芥表达调控开花时间[J]. 基因组学与应用生物学, 2020, 39(5): 2118-2126.
He X H, Wang J B, Yuan Y, Huang Y.regulating flowering time by reducing expressing of FLC in[J]., 2020, 39(5): 2118-2126. (in Chinese with English abstract)
[77] Sun C H, Fang J, Zhao T L, Xu B, Zhang F T, Liu L C, Tang J Y, Zhang C F, Deng X J, Chen F, Qian Q, Cao X F, Chu C C. The histone methyltransferasemediates H3K36me2/3 deposition atandand promotes flowering in rice[J]., 2012, 24(8): 3235-3247.
[78] 隋鹏飞. 水稻组蛋白H3K36甲基转移酶SDG725调控植物生长发育的功能研究[D]. 上海: 复旦大学, 2013.
Sui P F. Functional study of rice H3K36 methyltransferase SDG725 in rice growth and development[D]. Shanghai: Fudan University, 2013. (in Chinese with English abstract)
[79] Sang Y K, Zhu T, Sung Z R. Epigenetic regulation of gene programs byandin[J]., 2010, 152 (2): 516-528.
[80] Sánchez R, Kim M Y, Calonje M, Moon Y H, Sung Z R. Temporal and spatial requirement ofactivity forvegetative and reproductive development[J]., 2009, 2(4): 643-653.
[81] 蒋彬. 水稻OsEMF2b介导的花器官发育研究[D]. 雅安: 四川农业大学, 2013.
Jiang B. OsEM3b mediated floral organ development research in rice[D]. Ya’an: Sichuan Agricultural University, 2013.
[82] Jiang P F, Wang S L, Zheng H, Li H, Zhang F, Su Y H, Xu Z T, Lin H Y, Qian Q, Ding Y. SIP1 participates in regulation of flowering time in rice by recruitingto[J]., 2018, 219(1): 422-435.
[83] Sun C H, Chen D, Fang J, Wang P R, Deng X J, Chu C C. Understanding the genetic and epigenetic architecture in complex network of rice flowering pathways[J]., 2014, 5(12): 889-898.
[84] 张林, 何祖华. 水稻重要农艺性状自然变异研究进展及其应用策略[J]. 科学通报, 2015, 60(12): 1066-1078.
Zhang L, He Z H. Understanding natural variations: The source of elite agronomic traits for rice breeding [J]., 2015, 60(12): 1066-1078. (in Chinese with English abstract)
[85] Wu Q, Li D Y, Li D J, Liu X, Zhao X F, Li X B, Li S G, Zhu L H. Overexpression ofaffects plant architecture in rice (L.)[J]., 2015, 6(8): 833-844.
[86] Corbesier L, Vincent C, Jang S, Fornara F, Fan Q Z, Searle L, Giakountis A, Farrona S, Gissot L, Turnbull C, Coupland G. FT protein movement contributes to long-distance signaling in floral induction of[J]., 2007, 316(5827): 1030-1033.
[87] 朱岩, 彭振英, 张斌, 毕玉平.家族基因在植物中功能的研究进展[J]. 山东农业科学, 2013, 45(2): 139-145.
Zhu Y, Peng Z Y, Zhang B, Bi Y P. Advances in function research offamily genes in plants[J]., 2013, 45(2): 139-145. (in Chinese)
[88] Klintenäs M, Pin P A, Benlloch R, Ingvarsson P K, Nilsson O. Analysis of conifer FLOWERING LOCUS T/TERMINAL FLOWER1-like genes provides evidence for dramatic biochemical evolution in the angiosperm FT lineage[J]., 2012, 196(4): 1260-1273.
[89] Tamaki S, Tsuji H, Matsumoto A, Fujita A, Shimatani Z, Terada R, Sakamoto T, Kurata T, Shimamoto K. FT-like proteins induce transposon silencing in the shoot apex during floral induction in rice[J]., 2015, 112(8): 901-910.
[90] Brambilla V, Martignago D, Goretti D, Cerise M, Somssich M, De R M, Galbiati F, Shrestha R, Lazzaro F, Simon R, Fornara F. Antagonistic transcription factor complexes modulate the floral transition in rice[J]., 2017, 29(11): 2801-2816.
[91] 孙昌辉, 邓晓建, 方军, 储成才. 高等植物开花诱导研究进展[J]. 遗传, 2007, 29(10): 1182-1190.
Sun C H, Deng X J, Fang J, Chu C C. An overview of flowering transition in higher plants[J]., 2007, 29(10): 1182-1190. (in Chinese with English abstract)
[92] Yang X W, Lee W H, Sobott F. Structural basis for protein-protein interactions in the 14-3-3 protein family[J]., 2006, 103(46): 17237-17242.
[93] Jaspert N, Throm C, Oecking C.14-3-3 proteins: Fascinating and less fascinating aspects[J]., 2011, 10(21): 1-8.
[94] 丛楠, 程治军, 万建民. 控制花器官发育的ABCDE模型[J]. 中国农学通报, 2007, 123(7): 124-129.
Cong N, Cheng Z J, Wan J M. The ABCDE model of floral organ development[J]., 2007, 123(7): 124-129. (in Chinese with English abstract)
[95] Hempel F D, Weigel D, Mandel M A, Ditta G, Zambryski P C, Feldman L J, Yanofsky M F. Floral determination and expression of floral regulatory genes in[J]., 1997, 124(19): 3845-3853.
[96] Kotoda N, Wada M., a TFL1-like gene of apple, retards the transition from the vegetative to reproductive phase in transgenic[J]., 2005, 168(1): 95-104.
[97] 戚晓利, 卢孟柱. 拟南芥基因在花发育中的网络调控及其生物学功能[J]. 中国农学通报, 2011, 27(8): 103-107.
Qi X L, Lu M Z. Regulation network and biological roles ofofin flower development[J]., 2011, 27(8): 103-107. (in Chinese with English abstract)
[98] Yin X M, Liu X, Xu P Y, Dong T, Yang T, Ye T T, Feng Y Q, Wu Y. OsMADS18, a membrane-bound MADS-box transcription factor, modulates plant architecture and the abscisic acid response in rice[J]., 2019, 70(15): 3895-3909.
[99] Lu S J, He W, Wang Y, Wang H M, Yang R F, Zhang X B, Tu J M. Overexpression of a transcription factor OsMADS15 modifies plant architecture and flowering time in rice (L.)[J]., 2012, 30(6): 1461-1469.
[100]Lu S J, Wang H M, Yang R F, Zhang X B, Tu J M. Screening of rice OsMADS15 interacting proteins by yeast two hybrid system[J]., 2013 (2): 127-136.
[101]Zhang Y, Yu H P, Liu J, Wang W, Sun J, Gao Q, Zhang Y H, Ma D R, Wang J Y, Xu Z J, Chen W F. Loss of function ofleads to large sterile lemma and low grain yield in rice (L.)[J]., 2016, 36(11): 147-152.
[102]Naranjo L, Talón M, Domingo C. Diversity of floral regulatory genes ofrice cultivated at northern latitudes[J]., 2014, 15(1): 1471-2164.
[103]Komiya R, Ikegami A, Tamaki S, Yokoi S, Shimamoto K.andare essential for flowering in rice[J]., 2008, 135(4): 767-774.
[104]Takahashi Y, Teshima K M, Yokoi S, Innan H, Shimamoto K. Variations in Hd1 proteins,promoters, andexpression levels contribute to diversity of flowering time in cultivated rice[J]., 2009, 106(11): 4555-4560.
[105]Ogiso-Tanaka E, Matsubara K, Yamamoto S I, Nonoue Y, Wu J Z, Fujisawa H, Ishikubo H, Tanaka T, Ando T, Matsumoto T, Yano M. Natural variation of thecontributes to flowering time divergence in rice[J]., 2013, 8(10): e75959.
[106]Wu W X, Zheng X M, Lu G W, Zhong Z Z, Gao H, Chen L P, Wu C Y, Wang H J, Wang Q, Zhou K N, Wang J L, Wu F Q, Zhang X, Guo X P, Cheng Z J, Lei C L, Lin Q B, Jiang L, Wang H Y, Ge S, Wan J M. Association of functional nucleotide polymorphisms atwith the northward expansion of rice cultivation in Asia[J]., 2013, 110(8): 2775-2780.
[107]Matsubara K, Yano M. Genetic and molecular dissection of flowering time control in rice//Rice Genomics, Genetics and Breeding[M]. Singapore: Springer, 2018: 177-190.
[108]Takahashi Y, Shimamoto K.(), an ortholog of, is a possible target of human selection during domestication to diversify flowering times of cultivated rice[J]., 2011, 86 (3): 175-182.
[109]Yang Q, Li Z, Li W Q, Ku L X, Wang C, Ye J R, Li K, Yang N, Li Y P, Zhong T, Li J S, Chen Y H, Yan J B, Yang X H, Xu M L. CACTA-like transposable element in ZmCCT attenuated photoperiod sensitivity and accelerated the postdomestication spread of maize[J]., 2013, 110(42): 16969-16974.
[110]Ebana K, Shibaya T, Wu J Z, Matsubara K, Kanamori H, Yamane H, Yamanouchi U, Mizubayashi T, Kono I, Shomura A, Ito S, Ando T, Hori K, Matsumoto T, Yano M. Uncovering of major genetic factors generating naturally occurring variation in heading date among Asian rice cultivars[J]., 2011, 122(6): 1199-1210.
[111]Liu C, Song G Y, Zhou Y H, Qu X F, Guo Z B, Liu Z W, Jiang D M, Yang D C.andare the major genes for general combining ability of DTH, PH and SPP in rice[J]., 2015, 5(1): 1-11.
[112]Fujino K, Yamanouchi U, Yano M. Roles of thegene controlling heading date for adaptation to the northern limits of rice cultivation[J]., 2013, 126(3): 611-618.
[113]Zhang J, Zhou X C, Yan W H, Zhang Z Y, Lu L, Han Z M, Zhao H, Liu H Y, Song P, Hu Y, Shen G J, He Q, Guo S B, Gao G Q, Wang G W, Xing Y Z. Combinations of the,andgenes largely define the ecogeographical adaptation and yield potential of cultivated rice[J]., 2015, 208(4): 1056-1066.
[114]Kwon C T, Yoo S C, Koo B H, Cho S H, Park J W, Zhang Z J, Li J J, Li Z C, Paek N C. Natural variation incontributes to early flowering in japonica rice under long days[J]., 2014, 37(1): 101-112.
Research Progress of Photoperiod Regulation in Rice Flowering
WANG Yubo, WANG Yue, LIU Xiong, TANG Wenbang*
(,,,;Corresponding author,:)
As an important agronomic trait, heading date is determined by its own genetic factors and environmental factors, which has a great impact on the ecological adaptation area and yield factors of rice variety. In the past two decades, from the recognition of day length to the activation of flowering in the stem apex meristem, great progress has been made in the molecular regulation mechanism of rice photoperiod-induced flowering. A large number of regulatory genes related to flowering have been isolated and cloned, and integrated into the molecular network of photoperiod regulation. When plants are in favorable conditions, this network activates floral regulatory genes, promotes the expression of florigen gene, and transports florigen to the apical meristem, thus driving the development of meristem cells and forming flowers finally. In this paper,is used as a reference to discuss the genetic variation of rice photoperiod regulation network and its extension from low latitude to high latitude so as to provide valuable information for the cultivation of ecological varieties and the molecular mechanism of photoperiod regulation.
rice; flowering; photoperiod; photoperiod regulation; heading date
10.16819/j.1001-7216.2021.0514
2020-05-20;
2021-03-03。
国家重点研发计划资助项目(2017YFD0100303);湖南省科技重大专项(2018NK1020)。

