水稻粒宽突变体gw4的鉴定与基因定位
邓雪梅 胡鹏 王月影 文艺 谭义青 伍豪 吴凯雄 王俊格 侯琳琳 朱黎欣 朱丽 陈光 曾大力 张光恒 郭龙彪 高振宇 任德勇 钱前 胡江
水稻粒宽突变体的鉴定与基因定位
邓雪梅1,#胡鹏1,#王月影1文艺1谭义青1伍豪2吴凯雄1王俊格1侯琳琳1朱黎欣1朱丽1陈光1曾大力1张光恒1郭龙彪1高振宇1任德勇1钱前1,*胡江1,*
(1中国水稻研究所 水稻生物学国家重点实验室, 杭州 310006;2广西作物遗传改良生物技术重点开放实验室, 南宁 530007;#共同第一作者;*通信联系人, E-mail: qianqian188@hotmail.com; hujiang588@163.com)
【】水稻粒形是影响水稻产量和决定稻米外观品质的主要性状之一。筛选和鉴定新的粒形突变材料,可为研究水稻籽粒发育的调控机制奠定基础。粳稻品种中花11经1%的EMS处理,在诱变群体中获得一份窄粒突变体();分析粒形和其他主要农艺性状,在扫描电镜下观察颖壳细胞变化;利用突变体与籼稻品种台中本地1号配组的F2分离群体,选择隐性个体完成基因的精细定位;开展生物信息和测序分析,确定定位区间的候选基因;采用RT-PCR分析该基因在根、茎、叶、鞘、穗等组织中的表达模式及其他粒形相关基因的表达水平。与野生型相比,除了表现窄粒外,的粒长、千粒重、每穗粒数、一次枝梗数和二次枝梗数等显著下降;扫描电镜发现的颖壳内外表皮细胞均小于野生型;遗传分析表明该窄粒表型受一对单隐性核基因控制;通过开发的新标记最终将该基因定位在第4染色体BS6与EX49两个标记之间约31.74 kb的范围内;测序结果发现在基因编码区发生了一个由G至A的单碱基突变,导致原来编码的甘氨酸变成了天冬氨酸;qRT-PCR结果表明,主要在幼穗中表达,且在突变体中表达显著下降。主要调控水稻粒宽的发育,预测为其候选基因。这为进一步丰富粒形的遗传调控网络打下了基础。
水稻;粒形;粒宽;基因定位;外观品质
水稻是全世界主要的粮食作物之一。保证水稻高产、稳产、优质一直是水稻育种学家的研究重点。水稻的产量主要由有效穗数、每穗粒数以及千粒重三个要素共同决定。其中,籽粒的大小和形态既决定着千粒重的大小,也决定着稻米的外观品质[1]。
水稻是禾本科的模式作物,而作为产量性状的粒形也越来越受到分子生物学家的重视。到目前为止,已经定位了多个与粒形相关的QTL/基因,在水稻的12条染色体上均有分布,其中已克隆的基因超过70个。这些基因大多表现为一因多效,并与其他粒形相关基因协同表达,进而构成遗传调控网络[2]。其中,涉及泛素介导的蛋白酶体降解途径的基因有、/、/、和等[3-7];参与G蛋白信号传导途径的有、、和等[8-11];与促分裂原活化蛋白激酶级联MAPK途径有关的有、、和等[12-14];转录因子也是一类调控粒形发育的重要作用因子,相关的基因包括、/、、/、、、、和等[15-23];而小分子RNA则通过对靶基因的表达调控参与粒形的发育[24-27],如、、和等。此外,、/[1]、/、、/和/等基因也都参与调控粒形[28-32]。
颖壳细胞的形态、大小和数量是决定水稻籽粒形态的重要决定因子,其中MAPK、G蛋白、泛素蛋白酶体以及植物激素途径都是通过调控细胞的扩增来调控粒形,而MAPK、G蛋白和泛素蛋白酶体途径还可通过促进细胞增殖来调控籽粒大小。其中,控制粒长的基因主要有[8]、[16]、[23]、/和等[33-36];控制粒宽的基因主要有[3]、[18]、[1]/等[37, 38];而控制粒厚的基因有[3]。从对粒形的效应看,[8]、[35]、[3]、/[38]、[36]等基因对粒型具有负调控效应,而[17]、[36]、[18]、[1]、[16]、[34]、[23]等基因则是正调控籽粒的大小。这些为构建水稻种子的理想形态打下了重要的理论和材料基础。
本研究在粳稻品种中花11的EMS诱变后代中鉴定出一个窄粒突变体,其主要表现为籽粒宽度下降。利用构建的分离群体和开发的分子标记,经连锁分析和染色体步移,最终将该基因精细定位在第4染色体上一个31.74 kb的区域内,并通过测序找到了差异位点,确定了候选基因。这为丰富粒形的种质资源和进一步解析粒形的遗传网络打下了基础。
1 材料与方法
1.1 研究材料
利用EMS对粳稻品种中花11进行诱变,从后代分离群体中筛选到一份粒宽变窄的突变材料,经多代连续种植,发现该性状遗传稳定,我们将其命名为()。利用突变体与籼稻台中本地1号(TN1)杂交配组,收获的F1种子自交获得F2分离群体,再鉴定取样。所有材料均种植于中国水稻研究所浙江富阳基地和海南陵水南繁基地。
1.2 相关农艺性状调查与观察
抽穗后分别调查并统计野生型与的株高、节间长、分蘖数、穗长、每穗粒数、一次枝梗数、二次枝梗数、粒长、粒宽和千粒重。用作电镜扫描的籽粒用戊二醇固定再经过磷酸缓冲液充分清洗,然后用1%的锇酸溶液固定1 h左右,接着用磷酸缓冲液清洗之后用乙醇进行梯度脱水,最后用丙酮处理后进行扫描电镜观察(Hitachi H-7650)。
1.3 水稻DNA提取及PCR
野生型和两个亲本、F2群体及混池的DNA提取采用CTAB法。剪取叶片约0.2 g置于2 mL离心管,加入2颗钢珠和700 μL CTAB,用高通量组织研磨仪破碎组织。65℃下温育45~60 min,期间震荡混匀1~2次。取出后在通风橱中加入500 μL氯仿,盖上盖子并上下振荡摇匀使DNA被充分抽提,12 000下离心8 min,吸取上清液于提前加入500 μL乙醇的1.5 mL离心管中,置于−20℃冷藏2 h以上。最后12 000下离心8 min,弃上清液,晾干沉淀后加入300 μL ddH2O充分溶解DNA备用。引物设计使用Primer Premier 3.0软件,由尚亚生物技术公司合成。本研究中使用10 μL的PCR扩增体系:2 μL模板DNA,5 μL Mix(2×),正反向引物各1 μL,1 μL ddH2O。PCR扩增程序:94℃下高温变性4 min;94℃下预变性30 s,55℃下退火30 s,72℃下延伸30 s,72℃下重复延伸10 min,15℃下保存。共计40个循环。PCR产物加入溴酚蓝核酸染料2.5 μL,1500下离心混匀。琼脂糖凝胶电泳后置于凝胶成像仪上紫外扫描,保存胶图并统计分析。
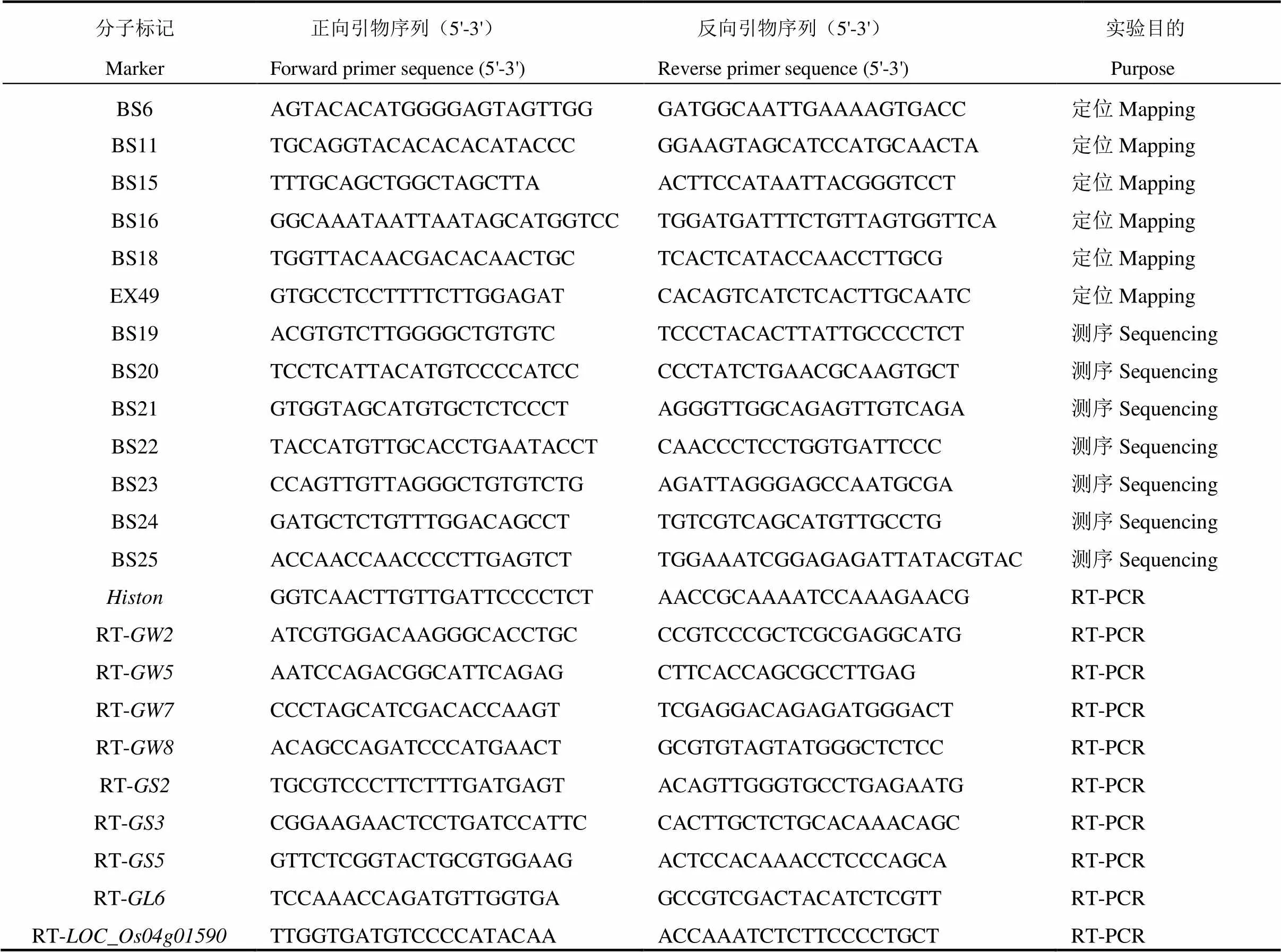
表1 用于基因定位和定量分析的分子标记
1.4 gw4的精细定位
利用与TN1构建的F2分离群体,我们构建了双亲、TN1及其F1和突变型(F2群体中随机选取30单株)的DNA混池,利用实验室自有的一套均匀分布于12条染色体的SSR和STS标记,进行多态性筛选和BSA分析,找出与目的基因连锁的分子标记。然后扩大群体验证连锁标记,确定初定位区间。基于初定位结果,对比目标区间内籼粳基因组序列,以粳稻日本晴与籼稻9311为参考,找出序列差异后用Primer Premier 3.0软件设计InDel标记引物(表1)。PCR扩增双亲及F1,用琼脂糖凝胶电泳检测,筛选出有多态性引物对F2群体进行检测。分析胶图,将具有TN1带型的单株记为A,具有带型的单株记为a,具有F1带型即交换单株记为H,统计交换单株的减少趋势及走向,以明确目标基因的区间。利用NCBI网站(https://www.ncbi.nlm.nih.gov/) 和水稻基因组注释数据库(http://rice.plantbiology.msu.edu/)预测定位区间内的候选基因,并下载其DNA序列。以候选基因序列为参考,用Primer Premier 3.0软件设计测序引物,PCR扩增野生型和突变体,并送往尚亚生物技术公司进行测序,最后用软件Seqman比对分析测序结果。
1.5 实时荧光定量PCR
取野生型与突变体植株的幼穗,使用Axygen公司的Miniprep提取试剂盒提取RNA。随后用日本TOYOBO公司生产的反转录试剂盒(ReverTra Ace quantitative PCR RT Master Mix)反转合成第1链cDNA,实验操作均按照说明书进行。反转获得cDNA后,稀释4倍用于实时荧光定量PCR。利用(SYBR Green)荧光定量PCR的预混体系和Bio-Rad CFX96 PCR仪,以作为内参,PCR扩增程序如下:95℃下预变性60 s;95℃下变性15 s,60℃下退火15 s,70℃下延伸60 s,共计40个循环。
2 结果与分析
2.1 gw4的籽粒大小
突变体的籽粒显著小于野生型中花11(图1-A, B),其中粒长由7.15 mm变为6.83 mm,减少了4.5%(图1-D);粒宽则由3.25 mm减为2.60 mm,减少20%(图1-E);长宽比由2.21变为2.64,增加19.5%(图1-F)。此外千粒重由34.62 g变为17.04 g,减少50.78%(图1-C)。因此,突变体籽粒主要是粒宽变窄,长宽比增加,使籽粒看上更为细长。
2.2 gw4的植株形态
除了显著减少粒宽外,两者在株高、分蘖、穗长和穗枝梗数上也存在显著差异(图2-A~F)。其中,株高由106.5 cm减少到83.5 cm,植株矮化了21.6%(图2-G);分蘖数由7.3变为18.9,与野生型相比增加了158.9%(图2-H);倒Ⅰ节至倒Ⅲ节的节间长度分别较野生型减少30.9%、20.5%、29.5%(图2-C、L);穗长则由19.1 cm下降到16.32 cm,减少14.55%(图2-L);每穗粒数由198.1降至137.3,下降30.69%(图2-I);一次枝梗和二次枝梗数分别减少28.5%和20.1%(图2-J、K)。
2.3 gw4籽粒颖壳细胞学观察
我们对的颖壳进行扫描电镜观察,发现的内颖壳和外颖壳细胞都显著变小(图3-B~E)。其中,中花11和外表皮细胞长分别为98.3 μm和60.4 μm(图3-F),外表皮细胞宽分别为70.2 μm和46.9 μm(图3-G),的外表皮细胞长和宽较野生型分别下降38.6%和33.2%,而中花11和内表皮细胞长分别为137.9 μm和114.6 μm(图3-H),内表皮细胞宽分别为31.5 μm和24.3 μm(图3-I),其内表皮细胞长和细胞宽分别减少16.9%和22.9%。因此,主要是通过调整细胞大小来调控籽粒发育的。
2.4 gw4的遗传分析与精细定位
利用突变体和TN1的配组,F1表现正常,在F2分离群体中发现野生型与突变型的分离比为3∶1(野生型∶突变型=2181∶969,χ2= 0.96 < χ20.05= 3.84),表明该突变性状受一对隐性单基因控制。我们利用本实验室自有的一套SSR和STS标记,通过BSA法对构建的混合池进行连锁分析,发现在第4染色体上的分子标记EX49的带型在混合池中表现为与带型相似的单带,而同时F1的双带则非常显著,因此判断其应该与基因连锁。我们通过F2群体中51株隐性纯合个体确定EX49与紧密连锁,并将其初步定位在BS6和EX49之间(图4-A)。通过进一步扩大群体和新发展分子标记,最终该基因定位在BS18和BS2之间31.74 kb的范围内(图4-B),该区间预测共有6个开放阅读框(图4-C)。
A, B−野生型和成熟籽粒,标尺=0.25 cm;C~F图中误差值代表标准误(=13),**表示0.01显著水平。ZH11−中花11。
A and B, Mature grain of WT and,bar0.25 cm; C-F, Bars represent standard error(=13). **indicate significant difference between WT andby-test(<0.01). ZH11, Zhonghua 11.
图1 野生型中花11与突变体的粒形
Fig. 1. Grains size of the wild type Zhonghua 11(ZH11) and its mutant
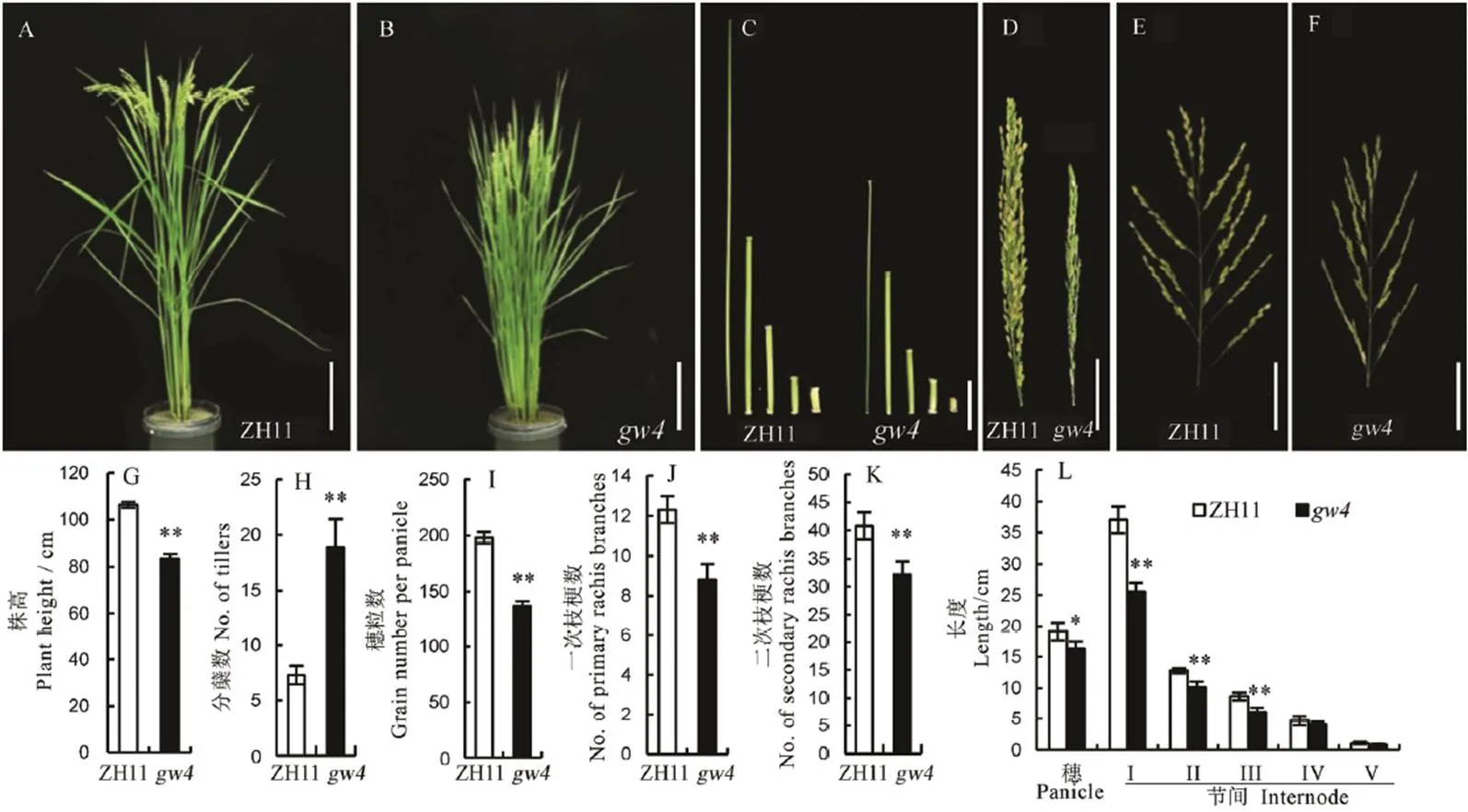
A, B−植株表型,标尺=20 cm;C−茎秆节间,标尺=5 cm;D, E, F−穗,标尺=5 cm。G~L中Bar值代表标准误(n=10),**表示0.01极显著水平,*表示0.05显著差异。
Fig. 2. Phenotype of the wild type Zhonghua 11(ZH11) and its mutant
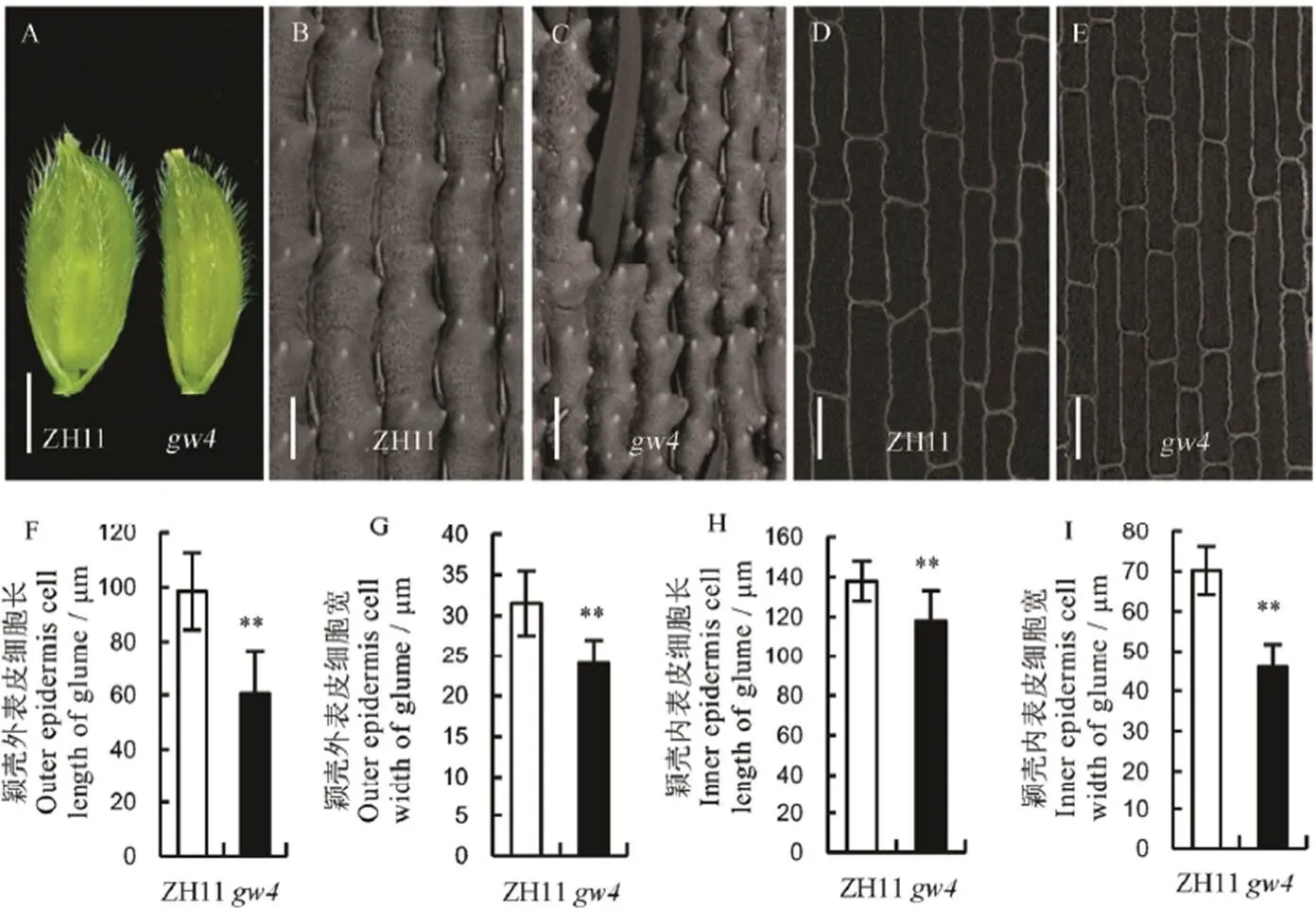
A−颖壳, 标尺=0.25 cm; B和C−颖壳外表皮, 标尺=50 μm; D和E−颖壳内表皮, 标尺=50 μm; Bar值代表标准误(n=10); **表示0.01显著水平。
Fig. 3. Glumes of the wild type(ZH11) and its mutant
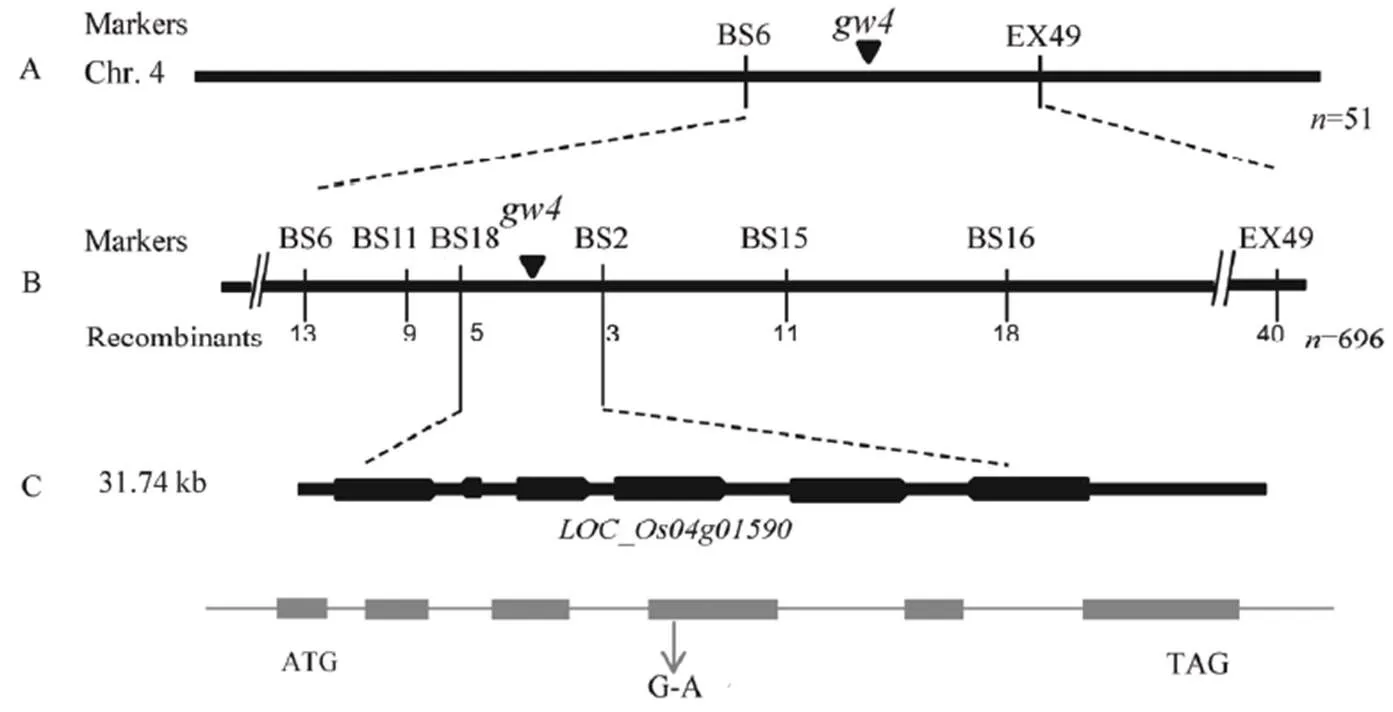
A−初定位在第4染色体;B−精细定位在31.74 kb的范围内;C−定位区间的6个预测基因。
A,was primarily mapped on chromosome 4; B,was narrowed to a 31.74 kb genomic region; C, Total six genes was predicted in this region.
图4 突变体的精细定位
Fig. 4. Fine mapping of
表2 候选基因分析
2.5 候选基因分析与确定
通过生物学数据库(http://rice.plantbiology.msu. edu/cgi-bin/gbrowse/rice/#search),我们发现在精细定位31.74 kb的区间内共含有6个预测基因(图4-C)。其中,、和均编码表达蛋白;而编码蔗糖酶/果胶甲基酯酶抑制剂家族蛋白,其主要在抽穗前和抽穗后的穗、花药和雌蕊中高表达,而在叶片和籽粒中表达量较低;编码精氨酸酶,其在叶片、穗、花药、雌蕊和籽粒中均高表达;则编码含有赤藓酸-4-磷酸脱氢酶结构域的蛋白,其主要在幼叶和幼穗中表达,而在花药、雌蕊及籽粒中表达量极低。为进一步确定候选基因,我们设计引物对精细定位区间进行扩增和测序,结果发现突变体在基因编码区第4外显子处发生了1个单碱基的突变(图4-C),由G变成A,该碱基突变导致原来编码的甘氨酸变成天冬氨酸。通过Swiss Model(http://swissmodel.expsay.org)预测,发现突变后的蛋白结构较野生型蛋白发生了明显的改变(图5)。
2.6 LOC_Os04g01590基因表达分析
为了进一步验证是否为候选基因,我们对进行了RT-PCR定量表达分析,结果发现基因在穗中的表达水平显著下调(图6-A)。同时开展基因在幼根、叶鞘、剑叶、幼穗和茎秆组织中的表达特异性分析,发现其主要也在穗中表达(图6-B),因此推测应为的候选基因。此外,我们还挑选了部分已报道涉及粒宽的基因进行了RT-PCR分析,发现突变体中、显著下调和显著上调表达外,其他粒形相关基因、、、和均未有显著性差异(图6-C)。
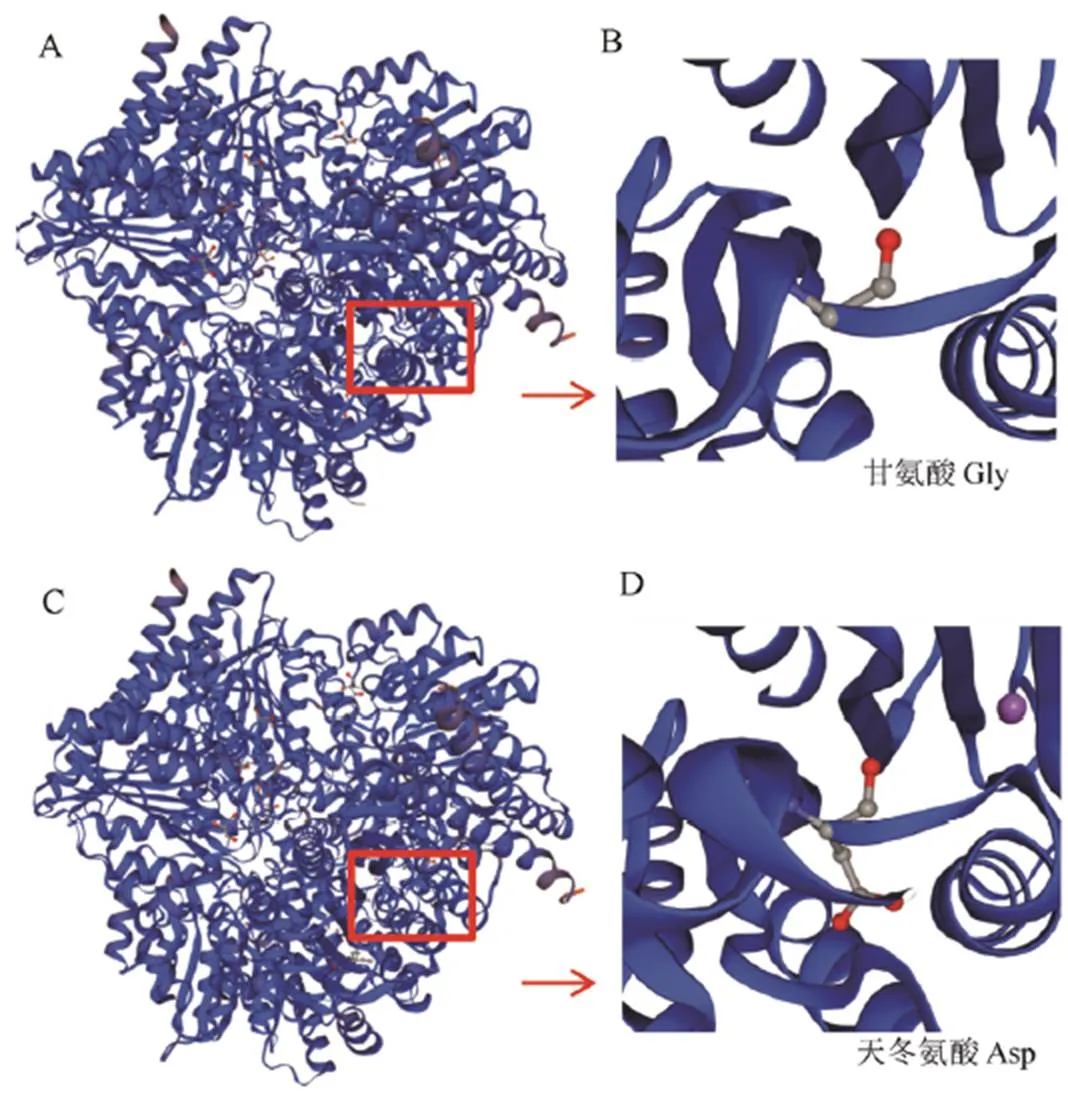
A, B−GW4蛋白的三维结构;C, D−gw4蛋白的三维结构;B和D为A和C的放大图;红框表示蛋白折叠差异位置。
Fig. 5. Three-dimensional structures of proteins GW4 and gw4.
3 讨论
籽粒形态是影响水稻产量和决定稻米外观品质的主要性状之一。由于细长形籽粒有利于后期的灌浆,从而更容易产生优质的稻米,据此育种家已经培育出了拥有籼稻外观的优质长粒粳水稻。本研究从中花11的EMS诱变群体中鉴定出了一个可以显著减少粒宽、增加长宽比的突变体,并通过图位克隆法将其编码基因精细定位在31.74 kb的范围内。测序发现定位区间内的基因在第4外显子处发生了一个G至A的单碱基替换,该碱基突变导致由原来编码的甘氨酸变成天冬氨酸(图4-C),其对应的蛋白三维结构也发生了明显变化(图5),因此该突变很可能是通过影响蛋白折叠而影响了酶的活性。Ma等报道了一个精氨酸酶编码基因/,其主要作用于尿素循环催化精氨酸水解成尿素和鸟氨酸,该基因功能缺失突变体则表现为株高降低、籽粒变小和育性下降等,并且结实率和籽粒大小会随着氮肥的增加而有所恢复[39]。的基因突变使编码蛋白提前终止,而本研究中蛋白只发生了一个氨基酸的改变,且在中的表达水平也显著下降,因而我们推测应为的弱等位变异。
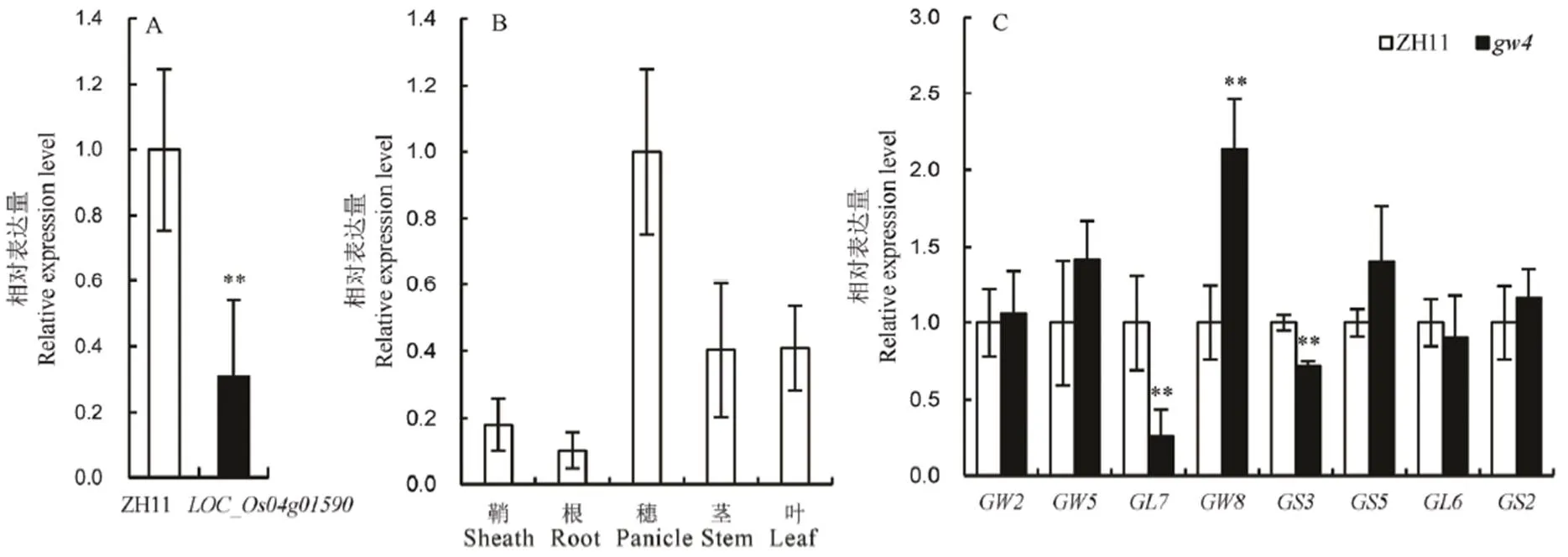
A−LOC_Os04g01590在中花11(ZH11)和gw4中的表达分析;B−LOC_Os04g01590在不同组织中的相对转录水平;C−粒形相关基因的表达分析。将ZH11的穗中的转录水平设置为1.0,Bar值表示3个生物学重复的平均值±SD;**表示0.01极显著水平。
Fig. 6. Expression analysis ofandgrain shape genes.
目前,已有多个水稻粒形相关基因的报道,他们的籽粒形态发育大多都是通过调控细胞生长完成的。针对颖壳的扫描电镜也发现,其外表皮和内表皮的细胞长较野生型分别下降了38.6%和16.9%,细胞宽则下降了33.2%和22.9%,因此,主要是通过控制细胞的大小来调控粒宽的。在的背景下,我们进一步对基因和其他粒形相关基因进行了RT-PCR分析,结果发现、、和的表达水平发生了显著性的变化。其中[1]正调控籽粒长度、[18]则负调控粒长和粒宽,而[8]也是负调控粒长的,但这些结果并不能很好地解释降低粒宽的表型,因此推测应该还介导了其他基因来调控细胞的发育。此外,/的表达水平在突变体背景下发生了显著下降,结合的窄粒表型,我们推测应该是粒宽的负调控因子。
水稻是单子叶的模式作物,近20年来以水稻为材料的分子生物学研究得到了长足的发展。其中通过各种诱变方法构建了多个突变体库,获得了大量的突变材料。尽管这些突变体在农业生产上往往存在着缺陷而无法直接使用,然而通过基因功能操作,如敲除或过表达却能够增加目标性状值,特别是近年来发现许多无法利用的突变基因,其弱等位却在生产上大范围使用,如甬优籼粳杂交稻中的粗秆大穗基因[40]和华占中的多蘖矮秆基因/[41]等。因此,开展水稻粒形相关基因的定位与克隆,对丰富籽粒发育的分子调控网络,开展分子选择育种具有重要的意义。
[1] Wang Y, Xiong G, Hu J, Jiang L, Yu H, Xu J, Fang Y, Zeng L, Xu E, Xu J, Ye W, Meng X, Liu R, Chen H, Jing Y, Wang Y, Zhu X, Li J, Qian Q. Copy number variation at thelocus contributes to grain size diversity in rice[J]., 2015, 47(8): 944-948.
[2] 尉鑫, 曾智锋, 杨维丰, 韩婧, 柯善文.水稻粒形遗传调控研究进展[J]. 安徽农业科学, 2019, 47(5): 21-28.
Wei X, Zeng Z F, Yang Wei F, Han J, Ke S W. Research progress on genetic regulation of rice grain shape[J]., 2019, 47(5): 21-28. (in Chinese with English abstract)
[3] Yan S, Zou G, Li S, Wang H, Liu H, Zhai G, Guo P, Song H, Yan C, Tao Y. Seed size is determined by the combinations of the genes controlling different seed characteristics in rice[J]., 2011, 123: 1173-1181.
[4] Huang K, Wang D, Duan P, Zhang B, Xu R, Li N, Li Y., which encodes an otubain-like protease with deubiquitination activity, influences grain size and shape in rice[J]., 2017, 91(5): 849-860.
[5] Shi C, Ren Y, Liu L, Wang F, Zhang H, Tian P, Pan T, Wang Y, Jing R, Liu T, Wu F, Lin Q, Lei C, Zhang X, Zhu S, Guo X, Wang J, Zhao Z, Wang J, Zhai H, Cheng Z, Wan J.has an important role in regulating grain width and size in rice[J]., 2019, 180(1): 381-391.
[6] Chen Y, Xu Y, Luo W, Li W, Chen N, Zhang D, Chong K. The F-box proteintargetsfor degradation and affects pleiotropic phenotypes, including leaf senescence, in rice[J]., 2013, 163(4): 1673-1685.
[7] Hu X, Qian Q, Xu T, Zhang Y, Dong G, Gao T, Xie Q, Xue Y. The U-box E3 ubiquitin ligasefunctions with a heterotrimeric Gα subunit to regulate Brassinosteroid-mediated growth in rice[J]., 2013, 9(3): e1003391.
[8] Liu Q, Han R, Wu K, Zhang J, Ye Y, Wang S, Chen J, Pan Y, Li Q, Xu X, Zhou J, Tao D, Wu Y, Fu X. G-protein βγ subunits determine grain size through interaction with MADS-domain transcription factors in rice[J]., 2018, 9(1): 852.
[9] Zhang D P, Zhou Y, Yin J F, Yan X J, Lin S, Xu W F, Baluška F, Wang Y P, Xia Y J, Liang G H, Liang J S. Rice G-protein subunitsandplay distinct roles in abscisic acid responses and drought adaptation[J]., 2015(20): 6371.
[10] Swain D M, Sahoo R K, Srivastava V K, Tripathy B C, Tuteja R, Tuteja N. Function of heterotrimeric G-protein γ subunitin providing salinity stress tolerance in rice by elevating detoxification of ROS[J]., 2017, 245(2): 367-383.
[11] Yadav D K, Islam S M, Tuteja N. Rice heterotrimeric G-protein gamma subunits (and) are differentially regulated under abiotic stress[J]., 2012, 7(7): 733-740.
[12] Xu R, Duan P, Yu H, Zhou Z, Zhang B, Wang R, Li J, Zhang G, Zhuang S, Lü J, Li N, Chai T, Tian Z, Yao S, Li Y. Control of grain size and weight by thesignaling pathway in rice[J]., 2018, 11(6): 860-873.
[13] Guo T, Chen K, Dong N Q, Shi C L, Ye W W, Gao J P, Shan J X, Lin H X.negatively regulates thecascade to coordinate the trade-off between grain number per panicle and grain size in rice[J]., 2018, 30(4): 871-888.
[14] Yi J, Lee Y S, Lee D Y, Cho M H, Jeon J S, An G.plays a critical role in cell differentiation during early embryogenesis in[J]., 2016(8): 2425-2437.
[15] Xia D, Zhou H, Liu R, Dan W, Li P, Wu B, Chen J, Wang L, Gao G, Zhang Q, He Y., a novel QTL encoding a-like kinase, epistatically interacts withto produce extra-long grains in rice[J]., 2018, 11(5): 754-756.
[16] 梁文化, 赵春芳, 张善磊, 张亚东, 朱镇, 赵庆勇, 陈涛, 王才林. 水稻粒型基因的功能标记开发与鉴定//江苏省遗传学会. 2017年学术研讨会—“技术创新与遗传学发展”论文摘要集[C]. 南京: 江苏省遗传学会, 2017: 1.
Liang W H, Zhao C F, Zhang S L, Zhang Y D, Zhu Z, Zhao Q Y, Chen T, Wang C L. Development and identification of functional markers of rice grain type gene//Jiangsu Society of Genetics. Academic Seminar of Jiangsu Society of Genetics in 2017 Meeting—“Technology Innovation and Genetics Development” Abstracts Collection[C]. Nanjing: Jiangsu Genetics Society, 2017: 1. (in Chinese)
[17] Hu J, Wang Y, Fang Y, Zeng L, Xu J, Yu H, Shi Z, Pan J, Zhang D, Kang S, Zhu L, Dong G, Guo L, Zeng D, Zhang G, Xie L, Xiong G, Li J, Qian Q. A Rare allele ofenhances grain size and grain yield in rice[J]., 2015: 1455-1465.
[18] Wang S, Li S, Liu Q, Wu K, Zhang J, Wang S, Wang Y, Chen X, Zhang Y, Gao C, Wang F, Huang H, Fu X. Theregulatory module determines grain shape and simultaneously improves rice yield and grain quality[J]., 2015, 47(8): 949.
[19] 徐乾坤, 余海平, 夏赛赛, 崔元江, 俞晓琦, 刘贺,曾大力, 胡江, 张强, 高振宇, 张光恒, 朱丽, 沈兰, 郭龙彪, 饶玉春, 钱前, 任德勇. C2H2锌指蛋白调控水稻小穗的发育[J]. 科学通报, 2020, 65(9): 753-764.
Xu Q K, Yu H P, Xia S S, Cui Y J, Yu X Q, Liu H, Zeng D L, Hu J, Zhang Q, Gao Z Y, Zhang G H, Zhu L, Shen L, Guo L B, Rao Y C, Qian Q, Ren D Y.22zinc finger proteinregulates the development of rice spikelets[J]., 2020, 65(9): 753-764. (in Chinese with English abstract)
[20] Mori M, Tomita C, Sugimoto K, Hasegawa M, Hayashi N, Dubouzet JG, Ochiai H, Sekimoto H, Hirochika H, Kikuchi S. Isolation and molecular characterization of amutant by modified activation-tagging in rice[J]., 2007, 63(6): 847-860.
[21] Yan S, Zou G, Li S, Wang H, Liu H, Zhai G, Guo P, Song H, Yan C, Tao Y. Seed size is determined by the combinations of the genes controlling different seed characteristics in rice[J]., 2011, 123(7): 1173-1181.
[22] Jia S, Xiong Y, Xiao P, Wang X, Yao J., a seed preferentially expressed gene regulates grain width by affecting cell proliferation in rice[J].2019, 280: 219-227.
[23] Prasad K, Parameswaran S, Vijayraghavan U., a rice MADS-box factor, controls differentiation of specific cell types in the lemma and palea and is an early-acting regulator of inner floral organs[J].2010, 43(6): 915-928.
[24] Zhao M, Liu B, Wu K, Ye Y, Huang S, Wang S, Wang Y, Han R, Liu Q, Fu X, Wu Y. Regulation ofthrough alternative polyadenylation improves grain yield in rice[J].2015, 10(5): e0126154.
[25] Miao C, Wang D, He R, Liu S, Zhu J K. Mutations inandincrease grain size and modulate shoot architecture in rice[J]., 2020, 18(2): 491-501.
[26] Zhang Y C, Yu Y, Wang C Y, Li Z Y, Liu Q, Xu J, Liao J Y, Wang X J, Qu L H, Chen F, Xin P, Yan C, Chu J, Li H Q, Chen Y Q. Overexpression of microRNAimproves rice yield by increasing grain size and promoting panicle branching[J]., 2013, 31(9): 848-852.
[27] Sun W, Xu X H, Li Y, Xie L, He Y, Li W, Lu X, Sun H, Xie X.acts downstream ofto regulate grain yield in rice[J].,2019, 226(3): 823-837.
[28] Chen J, Gao H, Zheng X M, Jin M, Weng J F, Ma J, Ren Y, Zhou K, Wang Q, Wang J, Wang J L, Zhang X, Cheng Z, Wu C, Wang H, Wan J M. An evolutionarily conserved gene,, plays a role in determining panicle architecture, grain shape and grain weight in rice[J]., 2015, 83(3): 427-438.
[29] Abe Y, Mieda K, Ando T, Kono I, Yano M, Kitano H, Iwasaki Y. The() gene is involved in the regulation of seed size in rice[J]., 2011, 85(5): 327-339.
[30] Wu T, Shen Y, Zheng M, Yang C, Chen Y, Feng Z, Liu X, Liu S, Chen Z, Lei C, Wang J, Jiang L, Wan J. Gene, encoding a kinesin-like protein with transactivation activity, is involved in grain length and plant height in rice[J].,2014, 33(2): 235-244.
[31] Segami S, Kono I, Ando T, Yano M, Kitano H, Miura K, Iwasaki Y.gene encodes alpha-tubulin regulating seed cell elongation in rice[J]., 2011, 5(1): 4.
[32] Wu L, Ren D, Hu S, Li G, Dong G, Jiang L, Hu X, Ye W, Cui Y, Zhu L, Hu J, Zhang G, Gao Z, Zeng D, Qian Q, Guo L. Down-regulation of a nicotinate phosphoribosyltransferase gene,, leads to withered leaf tips[J]., 2016, 171(2): 1085.
[33] Hu Z, Lu S J, Wang M J, He H, Sun L, Wang H, Liu X H, Jiang L, Sun J L, Xin X, Kong W, Chu C, Xue H W, Yang J, Luo X, Liu J X. A novel QTLencodes the-like kinasethat interacts withto negatively regulate grain size and weight in rice[J].,2018(5): 736-749.
[34] Xiong H, Yu J, Miao J, Li J, Zhang H, Wang X, Liu P, Zhao Y, Jiang C, Yin Z, Li Y, Guo Y, Fu B, Wang W, Li Z, Ali J, Li Z. Natural variation inincreases drought tolerance in rice by inducing ROS scavenging[J].,018, 178(1): 451-467.
[35] Gao X, Zhang J Q, Zhang X, Zhou J, Jiang Z, Huang P, Tang Z, Bao Y, Cheng J, Tang H, Zhang W, Zhang H, Huang J. Ricefunctions with the-like kinaseto modulate brassinosteroid signaling[J].2019, 31(5): 1077-1093.
[36] Zhao D S, Li Q F, Zhang C Q, Zhang C, Yang Q Q, Pan L X, Ren X Y, Lu J, Gu M H, Liu Q Q.acts as a transcriptional activator to regulate rice grain shape and appearance quality[J].2018, 9(1): 1240.
[37] Xu C, Liu Y, Li Y, Xu X, Xu C, Li X, Xiao J, Zhang Q. Differential expression ofregulates grain size in rice[J]., 2015, 66(9): 2611-2623.
[38] Weng J, Gu S, Wan X, Gao H, Guo T, Su N, Lei C, Zhang X, Cheng Z, Guo X, Wang J, Jiang L, Zhai H, Wan J. Isolation and initial characterization of, a major QTL associated with rice grain width and weight[J]., 2008, 18(12): 1199-1209.
[39] Ma X, Cheng Z, Qin R, Qiu Y, Heng Y, Yang H, Ren Y, Wang X, Bi J, Ma X, Zhang X, Wang J, Lei C, Guo X, Wang J, Wu F, Jiang L, Wang H, Wan J.encodes an arginase that plays critical roles in panicle development and grain production in rice[J]., 2013, 73(2): 190-200.
[40] Hang L, Yu H, Ma B, Liu G, Wang J, Wang J, Gao R, Li J, Liu J, Xu J, Zhang Y, Li Q, Huang X, Xu J, Li J, Qian Q, Han B, He Z, Li J. A natural tandem array alleviates epigenetic repression ofand leads to superior yielding rice[J]., 2017, 8: 14789.
[41] Wang Y, Shang L, Yu H, Zeng L, Hu J, Ni S, Rao Y, Li S, Chu J, Meng X, Wang L, Hu P, Yan J, Kang S, Qu M, Lin H, Wang T, Wang Q, Hu X, Chen H, Wang B, Gao Z, Guo L, Zeng D, Zhu X, Xiong G, Li J, Qian Q. A strigolactone biosynthesis gene contributed to the green revolution in rice[J]., 2020, 13(6): 923-932.
Identification and Fine Mapping of a Grain Width Mutantin Rice
DENG Xuemei1, #, HU Peng1, #, WANG Yueying1, WEN Yi1, TAN Yiqing1, WU Hao2, WU Kaixiong1, WANG Junge1, HOU Linlin1, ZHU Lixin1, ZHU Li1, CHEN Guang1, ZENG Dali1, ZHANG Guangheng1, GUO Longbiao1, GAO Zhenyu1, REN Deyong1, QIAN Qian1,*, HU Jiang1, *
(1State Key Laboratory of Rice Biology, China Rice Research Institute, Hangzhou 310006, China;2Guangxi Crop Genetic Improvement and Biotechnology Key Laboratory, Nanning 530007, China;#These authors contributed equally to this work;*Corresponding author, E-mail: qianqian188@hotmail.com; hujiang588@163.com)
【】Grain size is an important agronomic trait that determines yield and appearance quality in rice.Screening and identification of new grain size mutants will lay a foundation for further study of the regulation mechanism of grain development. 【】A grain width mutant()was obtained from 1% EMS mutagenized population of Zhonghua 11. The scanning electron microscope analysis of the outer and inner surfaces cells of glumes was carried out, and grain size and other agronomic traits were measured at the heading and maturity stages, respectively. With the map-based cloning, the mutant individuals derived from F2population ofand TN1 were used for fine mapping. The candidate gene was determined by gene sequencing based on bioinformatics analysis, and expression analysis of grain size-related genes was carried out by the RT-PCR.】Besides narrow grains,also showed decreased phenotype values in plant height, grain length, thousand grain weight, grain number, number of primary and secondary rachis branches. Scanning electron microscope observation presented that both cell length and width of inner and outer surfaces cells of glumes were smaller than the wild type. Genetic analysis showed that the narrow grain phenotype was regulated by a single recessive nuclear gene, which was fine mapped to a region of 31.74 kb between the two markers BS6 and EX49 on chromosome 4. Sequence analyses revealed a single base substitution of G to A was found inwhich resulted in a substitution of glycine to aspartic acid, and RT-PCR analysis indicated that it was mainly expressed in young panicles with declined expression level in mutant as compared with the wild type.【】is mainly involved in grain width development, and its candidate gene should be, which lays a foundation for further perfecting the genetic regulatory network of grain size.
rice; grain size; grain width; gene mapping; appearance quality
10.16819/j.1001-7216.2021.01204
2020-06-10;
2020-08-06。
浙江省自然科学基金杰出青年项目(LR19C130001); 国家自然科学基金资助项目(31871594, 91335105); 广西省自然科学基金青年科学基金资助项目(2018GXNSFBA050058)。

