中华按蚊OCT家族基因的多样性及序列特征
雷 丹, 闫振天, 张肖肖, 陈 斌
(重庆师范大学昆虫与分子生物学研究所, 媒介昆虫重庆市重点实验室, 重庆 401331)
有机阳离子转运体(organic cation transporter, OCT)家族是溶质载体超家族(solute carrier surfamily 22A, SLC22A)成员,具有OCT1, OCT2和OCT3 3个亚家族(Koepsell, 2013)。OCT利用有机阳离子迁移时所产生的电化学梯度差提供的驱动力,对有机阳离子介导的各种带正电荷的内源性代谢物(氨基酸、脂肪酸、神经递质、前列腺素、二羧酸盐和肉碱等)和异生素(药物、杀虫剂和环境毒素)进行排泄,与有机阴离子转运体(organic anion transporter, OAT)和肉碱转运蛋白(carnitine transporter, OCTN)一样被认为是一类重要药物传递蛋白(Koepsell and Endou, 2004)。过去的数十年里,人们利用动物模型、离体器官灌注或从体外组织和细胞水平对有机阳离子转运的机制进行了一些研究(舒焱, 2011)。目前,已在人及大鼠、小鼠和秀丽隐杆线虫Caenorhabditiselegans等动物中开展了OCT基因鉴定、表达和功能研究(Gründemannetal., 1994; Burckhardt and Wolff, 2000; Eralyetal., 2004)。随后,人类的一些OCT基因陆续被鉴定出来,它们间的氨基酸序列相似性在70%左右(Zhangetal., 1997)。研究表明,OCT1, OCT2和OCT3在脊椎动物一些组织中共表达,但它们在表达模式和功能上有较大差异(Motohashietal., 2002; Koepsell and Endou, 2004; Giacominietal., 2010)。这些转运蛋白尤其在脊椎动物肾脏等排泄器官中表达,提示它们可能在药物排泄中发挥作用(van Montfoortetal., 2003; Baganzetal., 2008)。在无脊椎动物中也有关于OCT家族基因的分子克隆和转运功能研究(Tayloretal., 1997; Rheault and O′Donnell, 2004)。秀丽隐杆线虫具有底物特异性的多种有机阳离子转运体,它们共同作用以排除广谱的、结构多样的异生素和环境毒素(TheC.elegansSequencing Consortium, 1998; Eralyetal., 2004)。在FlyAtlas(www.flyatlas.org)中发现CG8654在马氏小管中高度表达,且发现其具有与人类OCT2直系同源基因相似的功能,并将CG8654作为OCT直系同源(Wangetal., 2018)。目前,已有有机阳离子蛋白在调节药物相关副作用和毒性机制的综述,但在昆虫中鲜有研究,尚未有OCT家族基因在全基因组水平的系统鉴定、序列特征和系统发育分析。
蚊虫蚊媒病的控制主要依赖于对媒介蚊虫的控制,截止目前,杀虫剂仍是媒介蚊虫控制的主要措施(Fernandesetal., 2018)。但蚊虫对杀虫剂已产生了抗药性。拟除虫菊酯是目前蚊虫控制的主要杀虫剂,也是WHO推荐的唯一蚊帐药浸和主要室内喷洒杀虫剂(Fernandesetal., 2018)。中华按蚊Anophelessinensis是我国及东南亚的主要传疟媒介,近年来中华按蚊对拟除虫菊酯杀虫剂产生了广泛抗药性,严重阻碍了中国的疟疾消除计划(Chenetal., 2019)。近年来,我们以中华按蚊为模式种,以拟除虫菊酯为代表性杀虫剂在基因组水平系统地开展了中华按蚊杀虫剂抗性分子机理研究,已深度测序了中华按蚊基因组和转录组,系统地开展了P450, CCE, OBP, UGT和ABC等家族基因的多样性、序列特征及与拟除虫菊酯抗性的关系研究(Heetal., 2016; Yanetal., 2018; Heetal., 2019; Zhouetal., 2019)。然而,对于药物进入蚊虫体内后如何被代谢转运出体外,以及对于重要转运蛋白OCT家族的基因知之甚少。
本研究基于重庆师范大学中华按蚊基因组与转录组测序数据,在全基因组水平鉴定和命名了中华按蚊OCT家族的基因,分析了该家族基因的保守基序和结构域等序列特征,在基因组上定位了所有OCT基因,并系统地研究了OCT基因的系统发育关系。本研究是OCT家族基因在昆虫中的首个全基因组多样性和序列特征研究,为昆虫OCT基因提供了信息框架,也为中华按蚊OCT基因的进一步研究,特别是OCT基因在杀虫剂抗性机制方面的功能研究奠定了基础。
1 材料与方法
1.1 中华按蚊OCT家族基因的全基因组鉴定
本研究所使用的中华按蚊基因组和转录组数据来自重庆师范大学昆虫与分子生物学研究所,冈比亚按蚊Anophelesgambiae、黑腹果蝇Drosophilamelanogaster和秀丽隐杆线虫Caenorhabditiselegans等已确定的OCT氨基酸序列下载自NCBI(https:∥www.ncbi.nlm.nih.gov/)、VectorBase(https:∥www.vectorbase.org/)和EMBL(https:∥www.ebi.ac.uk/)数据库。把这些下载的序列作为询问序列(阈值设为 1e-5),使用BLASTP和TBLASTN分别搜索中华按蚊基因组及其氨基酸数据库,鉴定和提取中华按蚊OCT序列。从Pfam(https:∥pfam.xfam.org/)数据库中下载OCT保守结构域的隐马尔科夫模型文件(PF00083,PF07690),使用HMMER软件包中hmmsearch再次搜索中华按蚊基因组氨基酸数据库以获得OCT序列。将上述2种方法获得的序列整合,在对应的基因组序列左右侧翼区各扩1 500 bp以获得完整的CDs,并进行FGENESH+(https:∥www.softberry.com/berry)预测CDs及氨基酸。再将序列提交至NCBI,在线BLASTP比对进行验证,同时使用SMART(http:∥smart.embl-heidelberg.de/)在线分析其氨基酸结构域。将上述所获得的中华按蚊OCT基因作为询问序列,基于本实验室中华按蚊基因组框架图再次进行TBLASTN搜索,阈值设为1e-5,进行第二次预测及手工校对,去重复及整合以上鉴定结果,并将鉴定的氨基酸提交至Pfam(https:∥pfam.xfam.org/)与CDD(https:∥www.ncbi.nlm.nih.gov/Structure/cdd/)在线网站进行序列完整性检测以确定最终的中华按蚊OCT家族基因。基于同样的方法,在全基因组水平鉴定冈比亚按蚊OCT家族基因。
1.2 中华按蚊OCT家族基因氨基酸的理化特性及基因组定位分析
将所鉴定的OCT序列提交至ProtParam, TMHMM和SignalP在线网站(https:∥www.expasy.org/resources),分别对其分子量、等电点、亲水性系数和跨膜区等理化性质进行预测。通过本地Blastp作两两比对,以确定中华按蚊54个OCT氨基酸序列彼此之间的一致性。在线软件Muscle(https:∥www.ebi.ac.uk/tools/msa/muscle/)对中华按蚊OCT氨基酸序列进行多重比对,然后导入GeneDoc2.7.0与ScanProsit软件,分别预测保守区域及保守位点。通过在线软件MEME(http:∥meme-suite.org/tools/meme)对中华按蚊OCT氨基酸序列进行16个motif预测(长度设为6~18),以发现各个亚家族特异性保守基序,最后在多重比对图中标注出来。
通过在线软件GSDS(http:∥gsds.cbi.pku.edu.cn/)预测中华按蚊OCT基因外显子,分析内含子相位,并绘制其基因结构图。将所鉴定的中华按蚊OCT家族氨基酸序列提交到Pfam(https:∥pfam.xfam.org/)中进行结构域预测。使用中华按蚊OCT基因CDs序列搜索中华按蚊基因组序列,鉴定中华按蚊OCT家族基因在Scaffold上的位置,定位冈比亚按蚊OCT基因在染色体上的位置,并做中华按蚊和冈比亚按蚊OCT基因的共线性分析,用Mapchart, Illustrator和Adobe Photoshop CS6绘制中华按蚊OCT基因在Scaffold上的定位图。
1.3 中华按蚊OCT基因的系统发育分析及命名
使用最大似然法(maximum likelihood),通过PAUP4.0和MEGA7.0软件,以黑腹果蝇为外群,基于中华按蚊OCT家族的氨基酸序列构建系统发育树。基于ModelTest软件优化选择OCT氨基酸序列的最佳进化模型,通过1 000次重复,计算系统发育树上各分支bootstrap值,并讨论各分支的统计可信度。根据Orthomcl v.2.0.9在线软件采用内置算法BLASTP, E值设为1e-5,对中华按蚊OCT基因进行亚家族初分类,并进一步进行序列比对、结构特征及OCT基因间的系统发育关系的分析,参考已报道的OCT基因的分类系统、Cluster聚类结果、结构域和Scaffold上的位置分布对中华按蚊OCT家族基因进行分类和命名。
2 结果
2.1 中华按蚊OCT家族基因的多样性及分类
在中华按蚊和冈比亚按蚊全基因组水平均鉴定获得54个OCT家族基因,两者间同源基因氨基酸序列一致性达到55%~95%。这54个中华按蚊OCT家族基因分属于OCTA, OCTB和OCTC 3个亚家族,参考冈比亚按蚊直系同源基因的位置信息对中华按蚊33个OCTA亚家族成员、15个OCTB亚家族成员和6个OCTC亚家族成员分别命名为AsOCTA1-30,AsOCTB1-14和AsOCTC1-6。其中位于Scaffold55上的基因AsOCTA17在本实验室注释的中华按蚊数据库中未找到对应的ID号信息,是本研究新注释的基因;仅AsOCTB12不具有完整的开放阅读框,氨基酸数目大多为450~540,亲水系数在0.500左右,表明中华按蚊OCT为疏水性蛋白(表1)。肽链拓扑结构预测显示,约2/3的OCT的氨基和羧基末端都位于细胞内且有11个左右跨膜结构,每个跨膜区约有21~25个氨基酸残基。当跨膜时,在第2与第3跨膜结构之间形成一个大的细胞外环,富含糖基化位点,在第6与第7跨膜区之间有一个较大的细胞内环,结构类似左右对称,符合典型的跨膜转运蛋白。
2.2 中华按蚊OCT家族基因的结构及序列特征
中华按蚊这54个OCT基因中部分能预测完整序列的基因之间的内含子与外显子分布式样差异较大(图1),共有196个外显子,长102~998 bp,每个基因有1~9个外显子;AsOCTA5,AsOCTB13,AsOCTA16和AsOCTB12仅有1个外显子;AsOCTB6和AsOCTB7外显子数目最多,达到9个,但长度较短; 16个基因有4个外显子,7个基因有3个外显子。对密码子起始核苷酸分析,共有Phase 0(54个),Phase 1(42)和Phase 2(51)型3种相位,分别占内含子总数的36%, 30%和34%。OCTC亚家族基因之间内含子相位分布较其他两个亚家族稳定,内含122内含子相位组合。
除AsOCTA20和AsOCTB2外,其余OCT基因编码的氨基酸序列均含有MFS_1与Sugar_tr跨膜结构域,此为我们鉴定OCT基因的关键特征;这2个结构域序列中富含甘氨酸残基,有助于OCT作为膜蛋白与特异性底物结合。各亚家族基因编码的氨基酸序列中具有数目不等的跨膜结构域(transmembrane domain, TMD),其中AsOCTA有6~22个,AsOCTB有6~13个,AsOCTC有12个TMD。2/3的AsOCT与AsOCTC一样,具有12个TMD,但其中位置在前的6个TMD的氨基酸序列差异较大,亚家族之间较各个亚家族内成员的TMD氨基酸序列相似性也较低。有3个在中华按蚊各亚家族OCT氨基酸序列中均保守的序列,分别是位于TMD2与TMD3之间的GRK-(PT)-VL,TMD6后的PES-(APVS),以及TMD10和TMD11之间EQFPT-(VI)-RN(图2,红色背景);其中第1和第3种序列与脊椎动物中的OCT保守序列特征一致(Zhuetal., 2015)。在各亚家族氨基酸序列中也分别鉴定了2~3个亚家族特异保守序列(图2,黄色背景),在AsOCTA中有2个特异性保守序列,分别是TMD4后的(IL)-RG和TMD12后的LPETK;AsOCTB中在连接TMD2与TMD3之间的第一个外环中存在WBLVCD,位于TMD8内的VELP及TMD8后的(ND)R-X-GR(KR);AsOCTC亚家族氨基酸序列中2个特有的保守序列分别是TMD2前的6个氨基酸序列(MI)S-X-VLP和TMD7后的LMMWFP。此外,我们还发现在AsOCTA和AsOCTB中TMD12后具有5个LPETK保守序列。这些保守区域对于OCT及各亚家族基因的鉴定及功能认知具有重要价值。
2.3 中华按蚊OCT家族基因的定位
中华按蚊54个OCT基因被定位到26条Scaffold上(图3),近一半的基因分布在Scaffold14, Scaffold49和Scaffold116上,分别有13, 8和5个基因,其他的Scaffold上仅有1~3个基因。分布在Scaffold14上的大部分OCT基因聚集成簇(间距小于20 kb),分布在Scaffold116上的AsOCTA10a和AsOCTA10b聚集成簇,分布在Scaffold49上的AsOCTA19a和AsOCTA19b聚集成簇,这些成簇的基因在系统发育关系中紧邻,它们可能来源于基因重复事件。虽然AsOCTA23a和AsOCTA23b分别分布在Scaffold84和Scaffold150上,但基因相似度高,系统发育关系近,也可能源于基因重复事件。
冈比亚按蚊OCT家族基因被定位到5条染色体上(2L, 2R, 3L, 3R和X),共线性分析表明中华按蚊OCT基因大部分对应到冈比亚按蚊2R和3R染色体上(图3)。中华按蚊与冈比亚按蚊并非所有的OCT基因都为一对一的直系同源关系,在AsOCTA和AsOCTB中共有4对基因(AsOCTB4a和AsOCTB4b,AsOCTA10a和AsOCTA10b,AsOCTA19a和AsOCTA19b,以及AsOCTA23a和AsOCTA23b)仅分别对应到冈比亚按蚊的一个基因(表1),表明这4对基因发生了基因重复事件。

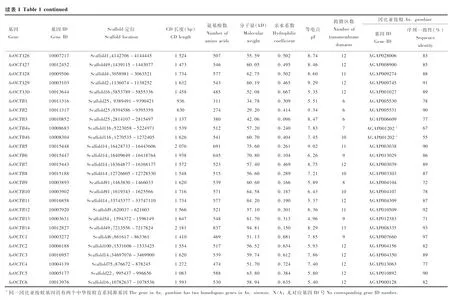
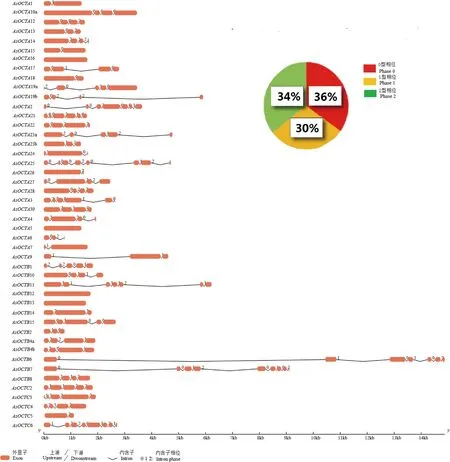
图1 中华按蚊OCT基因的结构及内含子相位统计Fig. 1 The structure and intron phase statistics of the OCT genes of Anopheles sinensis
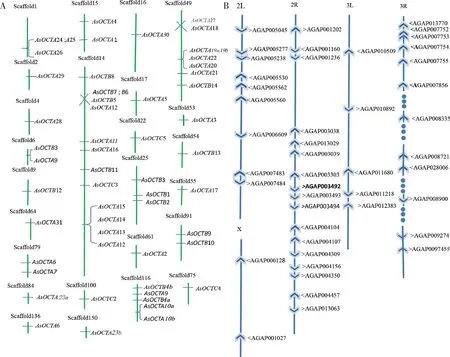
图3 中华按蚊OCT基因在Scaffold上的分布(A)及参考冈比亚按蚊在染色体上的定位(B)Fig. 3 Distribution on scaffolds of OCT genes of Anopheles sinensis (A) and their locationon chromosome in reference of An. gambiae (B)蓝色箭头标示OCT基因的转录方向。Blue arrows indicate the transcription direction of OCT genes.
2.4 中华按蚊OCT家族基因的系统发育
基于OCT氨基酸序列及其计算出最佳进化模型为LG+G+F构建了中华按蚊54个OCT基因的系统发育树(图4)。在系统发育树的分枝末端具有较好的自展值,bootstrap值在65%~100%之间(AsOCTB2与AsOCTB14除外)。这54个OCT基因明显按3个亚家族形成了3个组,OCTC基因较为原始,是一个明显的单系群,其bootstrap值为99%;OCTB基因较为进化,也是一个明显的单系群,其bootstrap值为100%;OCTA基因介于前两者之间,进化关系较为复杂。
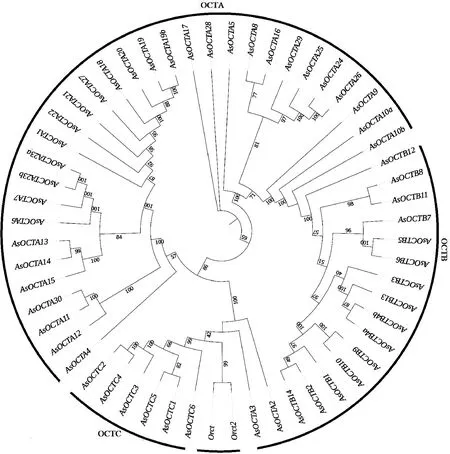
图4 基于核苷酸序列用最大似然法构建的中华按蚊OCT基因的系统发育树(1 000次重复)Fig. 4 Phylogenetic tree of the OCT genes of Anopheles sinensis based on amino acide sequencesusing maximum likelihood method (1 000 replicates)以黑腹果蝇的Orct(GenBank登录号: AFY98627.1)和Orct2(GenBank登录号: AFY98628.1)为外群。Orct (GenBank accession no.: AFY98627.1) and Orct2 (GenBank accession no.: AFY98628.1) from Drosophila melanogaster as the outgroup.
3 讨论
本研究首次在中华按蚊全基因组水平鉴定了OCT家族基因,共54个。参考已有OCT分类系统、序列相似性、基因结构特征及系统发育关系(图1和2),将这些基因分为OCTA, OCTB和OCTC 3个亚家族,分别有33, 15和6个基因。冈比亚按蚊OCT家族也有54个基因,但在中华按蚊OCTA和OCTB中共有4对基因(AsOCTB4a和AsOCTB4b,AsOCTA10a和AsOCTA10b,AsOCTA19a和AsOCTA19b,以及AsOCTA23a和AsOCTA23b)仅分别对应到冈比亚按蚊的一个基因(表1),而且这些基因对在Scaffold成簇排列(AsOCTA23a和AsOCTA23b分布在不同的Scaffold)(图3),推测这4对基因发生了基因重复事件。相应地,冈比亚按蚊也有4对基因发生了基因重复事件,这些预示了这两个种不完全相同的转运机制或者进化。冈比亚按蚊OCT家族基因被定位到2L, 2R, 3L, 3R和X,共5条染色体上,而中华按蚊OCT基因大部分对应到冈比亚按蚊2R和3R染色体上(图3)。
中华按蚊OCT家族氨基酸序列具有较高的保守性,所有OCT均具有MFS_1与Sugar_tr跨膜结构域(AsOCTA20和AsOCTB2除外),2/3的OCT基因具有OCTC的12个TMD,在空间上形成疏水性口袋,这与其他物种OCT的结构特征一致,符合该家族的细胞内和细胞外特征。中华按蚊OCT家族几乎所有的序列都存在早期脊椎动物中GRK-(PT)-VL和EQFPTVIR这2个保守基序,同时,中华按蚊OCT还具有一个共同的保守基序PES-(APVS),各OCT亚家族也存在各自的保守基序,而这些保守的基序可能与特殊的功能相关,还需要我们进一步研究探索。本研究是对OCT序列保守性的首次系统研究,对于OCT及各亚家族基因的鉴定及功能具有重要价值。中华按蚊54个OCT基因明显按3个亚家族形成了3个系统发育组(图4),OCTC基因较为原始,是一个明显的单系群;OCTB基因较为进化,也是一个明显的单系群; OCTA基因介于前两者之间,进化关系较为复杂。由此进一步提示,OCT基因的系统发育关系和分类需要在全基因组水平研究更多的物种而得以改进和完善。
本研究首次在全基因组水平上鉴定和命名了中华按蚊OCT家族基因,预测了该家族基因的理化特性,分析了OCT的结构域和保守基序,在基因组上定位了所有的OCT基因,并探究了OCT基因的系统发育关系,不仅为昆虫的OCT基因提供了信息框架,也进一步丰富了中华按蚊基因组数据。同时,对于后续中华按蚊OCT基因功能的进一步研究,特别是对该类基因在杀虫剂抗性机制方面的功能研究奠定了基础,具有一定的意义。

