环状RNA在非瓣膜性心房颤动患者发病中的作用及机制*
王君 王英 张琪 董海翠 杨吉猛 方旭 史培青 张郁青
心房颤动(简称房颤)发生的电生理机制为折返和异位激动的形成,但潜在的分子机制尚不清楚。在以往的研究中,通常采用心肌组织进行环状RNA(circ RNAs)分析,但是心肌组织的获取不简单易得。本研究通过采取患者外周血作为样本,经过差异表达分析及定量验证等过程,来明确circRNAs是否可以作为非瓣膜性房颤的生物标志物,结合其他纤维化生物标志物的组合分析来预测心肌纤维化的程度。
circ RNAs是非编码RNA 的其中一个类型,具有封闭的环状结构,不具有5′和3′端,这使得它们结构稳定,不容易受到核糖核酸酶的降解。circ RNAs在表观遗传调控和疾病病原体中发挥重要作用[1-2]。既往的一些研究利用心脏组织提取circ RNAs,因为心脏组织不简单易得,为了明确circRNAs是否可以作为房颤的一种新的生物标志物,本实验利用外周血提取circ RNAs,通过差异表达circ RNAs在内源性竞争性RNA(CeRNA)网络中与相关基因的关系,明确circRNAs在非瓣膜性房颤中的作用。
1 资料与方法
1.1 入选人群 从2017年1月到2018年12月,南京市江宁医院共有46例患者被纳入本实验。他们被分成两组:房颤组22例(14例阵发性房颤,8例持续性房颤),对照组24 例人群无任何心律失常。所有患者经过心电图(ECG),动态心电图(DCG),超声心动图(UCG)以及实验室检查诊断为房颤或健康对照(根据2016年ESC房颤管理指南)。纳入标准:年龄40~80 岁。患者均完善ECG,DCG,UCG 以及实验室检查。排除标准:无肿瘤性疾病以及其他累及免疫系统的疾病,无任何严重威胁生命的重大疾病。
1.2 采集血样 这项研究得到了南京江宁医院伦理服务委员会的批准[伦审批第(20180135)号]。所有患者的静脉血样均于上午空腹获得,以1 000 r/min 离心10 min 后分离血清,低温(-80 ℃)冷冻保存。
1.3 RNA 提取和鉴定 随机选取房颤组和对照组各3 例,进行circRNAs 定量分析。利用Nano-Drop ND-1000从每个样本中提取总RNA。基于Arraystar标准协议进行样品制备和微阵列杂交。简单地说,总RNA 被RaseR 酶(Epicentre,Inc.)消化以去除线性RNA 并保留circRNAs。然后,利用随机启动方法(Arraystar Super RNAKit;Arraystar)将富集的circ RNAs扩增并转录成荧光circ RNAs。将标记的circRNAs杂交到Arraystar人circRNAs阵列V2(8x15K,Arraystar)上。随后,阵列被Agilent扫描仪G2505C 扫描。使用Agilent特征提取软件(版本11.0.1.1)分析获得的阵列图像。使用R 软件limma包进行量化归一化和后续数据处理。两组间差异表达的circ RNAs通过火山图过滤鉴定是否具有统计学意义。
1.4 房颤组与对照组circRNAs的差异表达 房颤组与对照组circ RNAs差异表达共发现6 165 个circRNAs上调,6 744 个circ RNAs下调。利用R软件lima包进一步过滤分析,用t 检验估计两组间差异表达的统计学意义。在t 检验中P 值小于0.05,差异倍数大于2 的被认为是差异表达显著的circRNAs。
1.5 逆转录-定量PCR(RT-qPCR)验证circRNAs
为了进一步验证在入选患者中circRNAs的差异表达,在22例房颤患者和24例对照者中,筛选出2个上调的circRNAs和2个下调的circRNAs。两组血样离心,取血清进行RNA 提取。用Nano-Drop ND-1000对每个样品的总RNA 进行定量。利用基因AMPP CR 系统9700 合成了cDNA。利用Taq Man miRNA 逆转录-定量聚合酶链反应(RTqPCR)测定候选circRNAs。对46个样品进行了4个候选circ RNAs的检测。2个circ RNAs定量与我们之前的差异表达分析结果一致。
1.6 GO 和KEGG 富集分析 目标基因的差异表达用GO 富集分析模块(GO:Term Finder,http://search.cpan.org/dist/GO-Term Finder/)评 估 和 确定circRNAs靶基因的差异。用R 功能进行不同circRNAs靶基因的KEGG 富集分析。显著富集的GO 分析基因和KEGG 通路符合校正后P <0.05的标准。
1.7 Circ RNAs和微小RNA(miRNA)相互作用的分析 TargetScan(http://www.targetscan.org/)和miRanda(http://www.microRNA.org/)可以预测circRNAs与微小RNA(miRNA)的相互作用。通过RT-qPCR 鉴定circRNAs的差异表达,利用上述两个分析软件,构建ceRNA 网络,对circ RNAs与miRNA 的相互作用进行了详细分析。
1.8 心肌纤维化相关生物因子的检测 利用ELISA 方法测量组织生长因子-β[TGF-β,生工生物工程(上海)股份有限公司,C505060]的水平以及这两组患者血样中的Ⅰ型前胶原氨基端前肽(PINP,上海康朗生物科技有限公司,货号:DL-PICP-Hu)、Ⅲ型前胶原氨基端前肽(PⅢNP,上海康朗生物科技有限公司,货号:DL-PⅢCP-Hu)和N 端前脑利钠肽(NT-proBNP)水平。
1.9 统计分析 计量资料用平均数±标准差或中位数表示。采用未配对t 检验或Mann-Whitney U检验对两组连续变量进行比较。多次比较通过方差分析进行评估。用Spearman分析确定不同组间的线性相关性。P<0.05有统计学意义。统计分析采用SPSS19.0软件包(SPSSInc,IBM,美国)进行。
2 结果
2.1 两组的基线特征 两组人群基线特征如表1。两组患者NT-proBNP含量和左房直径比较差异有显著性(P 均<0.05)。
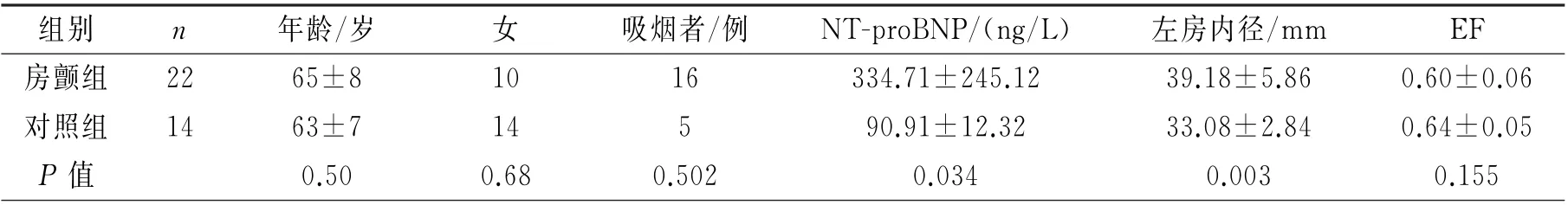
表1 两组患者的基线特征
2.2 两组circ RNAs差异表达的确定 经过circ RNAs提取和分析(图1),根据差异分析中的P<0.05以及两组样本量中差异circ RNAs表达量(Fold Change,FC)比值大于2,选取P值小且差异表达量比值大的hsa_circRNAs_101216,hsa_circ RNAs_102485作为房颤与对照组之间的上调表达差异circ RNAs,而hsa_circ RNAs_102158和hsa_circ RNAs_001678作为下调表达差异circRNAs被挑选进行RT-qPCR 的定量验证。由于引物设计困难,2个circRNAs未成功验证(表2)。hsa_circRNAs_101216(hsa_circ_0006903)和hsa_circ RNAs_001678(hsa_circ_0000517)通过两组患者中的定量分析,证实为在房颤组中表达上调和表达下调。
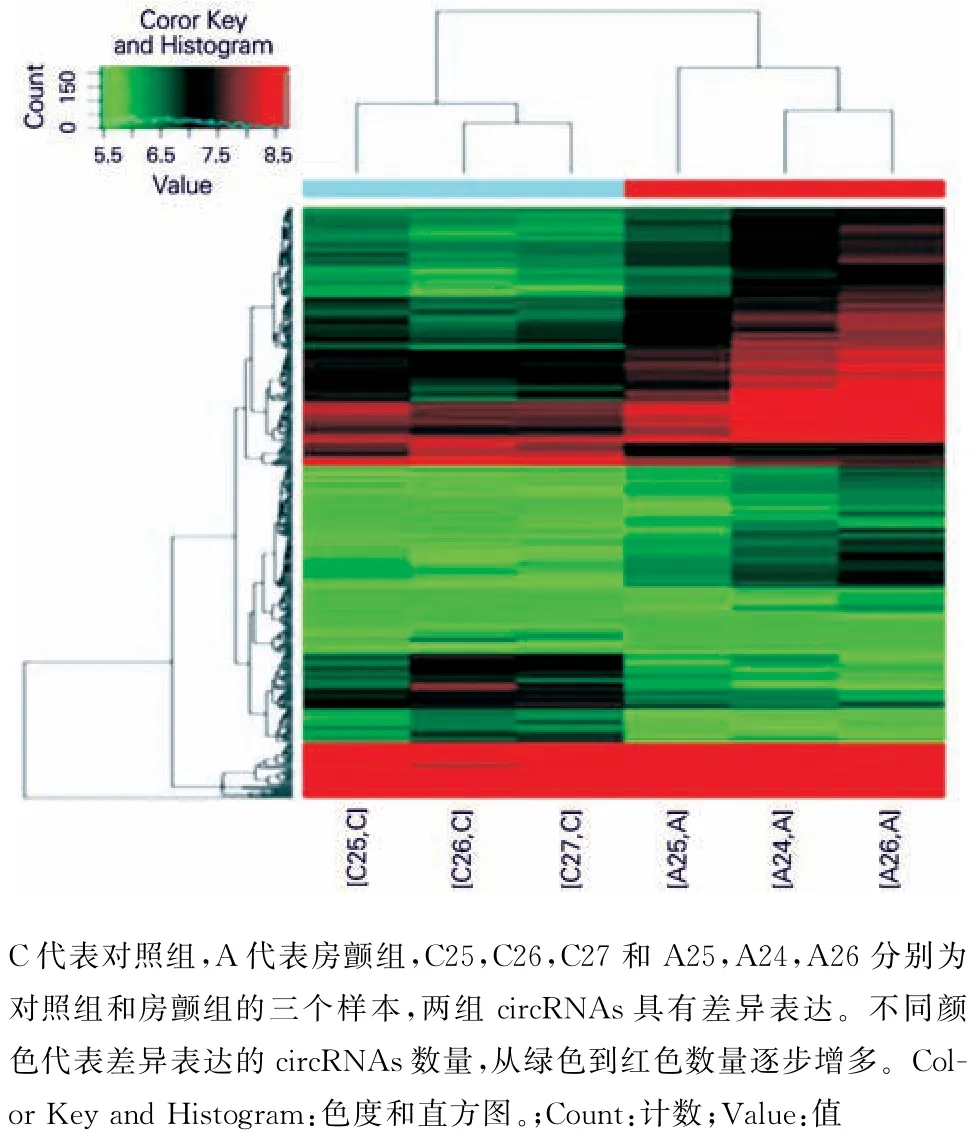
图1 不同表达的circRNAs在两组中的层次聚类
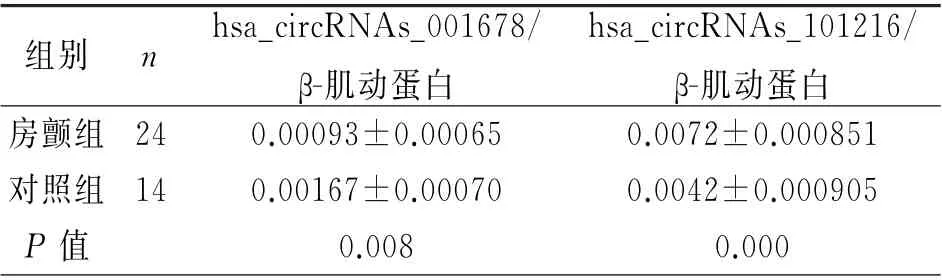
表2 两组间差异表达的circRNAs
2.3 circRNAs GO 和KEGG 分析的差异表达GO 分析(http://www.geneontology.org)中的基因和基因产物属性包括三个领域:生物过程、细胞成分和分子功能。
生物过程的富集评分表明,能量代谢、浸润反应、坏死和凋亡是失调的circ RNAs调节基因的主要生物学功能(图2)。同时,笔者发现下调的circRNAs(hsa_circ_0000517)调节基因ME3和TNFRS F19更多地参与能量代谢过程。相反地,上调的circRNAs(hsa_circ_0006903)调节基因PIH1D1、LDHC和TNFRS F19参与调节了细胞成分的发生和发展。
2.4 Circ RNAs和miRNA 的相互作用 通过ceRNA 的分析发现hsa_circ_0000517与hsa-miRNA-671-5p、hsa-miRNA-596,hsa_circ_0006903 与hsamiR-326、hsa-miR-330-5p之间的相互作用(图3)。
2.5 CircRNAs与心肌纤维化生物标志物的相关性分析 房颤组的TGF-β、PINP、PⅢNP 和NTProBNP水平均高于对照组;Hsa_circ_0006903 与TGF-β、PⅢNP和PINP呈定量正相关,见表3。

图2 两组间差异表达circRNAs的GO 分析生物功能的富集评分
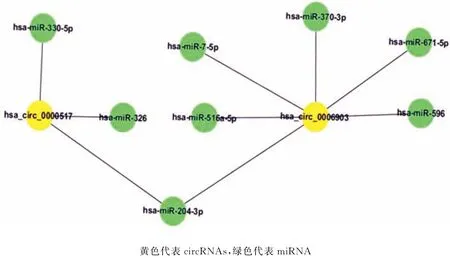
图3 circRNAs和mRNA 的相互作用关系网络图

表3 差异表达的circRNAs和心肌纤维化的生物标志物的相关分析
2.6 circRNAs与患者临床特征间的相关性分析综合分析hsa_circ_0000517,hsa_circ_0006903与患者临床特征,进行相关分析发现hsa_circ_0000517与左房直径成负相关,hsa_circ_0006903与左房直径成正相关(表4)。
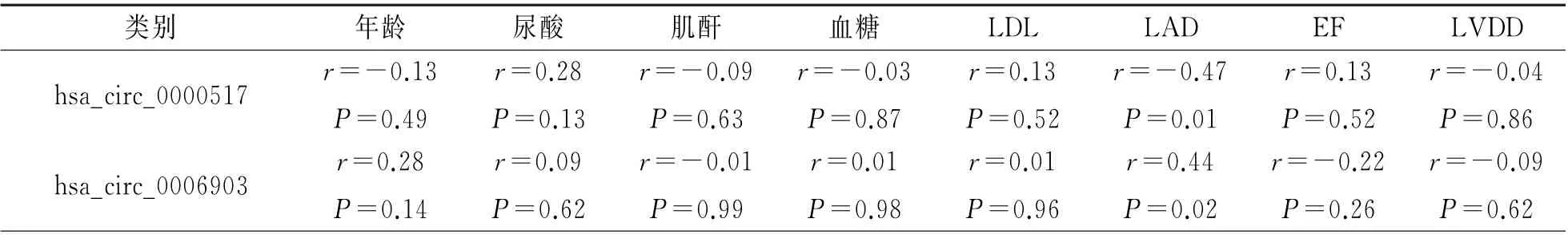
表4 差异表达的circRNAs和患者临床特征的相关分析
3 讨论
目前,由于高密度微阵列的推广应用,许多非编码RNA 被认为是基因表达的新的和有效的调节因子。非编码RNA 包括miRNAs、INc RNAs、circ RNAs、核糖体RNAs 和核内小核糖核蛋白颗粒(snRNPs)以及各种调节RNA[3]。其中,circRNAs由于其稳定性和保守结构,是理想的诊断和治疗候选RNA。
本研究试图确定一种房颤相关新的生物标志物并研究circ RNAs在非瓣膜性房颤中的发病机制。房颤组与对照组共鉴定出12909个差异circ RNAs(FC>2;P<0.05),包括6165个上调的circRNAs和6744个下调的circRNAs。通过RT-qPCR 的方法,笔者验证了circRNAs定量数据,选择了差异表达的环形RNA(hsa_circ_0000517,hsa_circ_0006903)进行分析。RT-qPCR 结果表明,RNA 测序鉴定的circRNAs可靠,值得进一步研究。参与房颤患者发病调节的circRNAs可能在房颤进展机制中起关键作用。
目前许多科学家认为,circ RNAs能够降低心脏成纤维细胞中纤维化相关基因的表达水平[2,4]。还有一些学者认为,circRNAs可能涉及离子通道功能的过度修饰,包括钾通道和细胞内钙离子的处理和释放[5-6],这些机制可能导致房颤。Raman等[7]发现适应性基因表达变化可以满足心脏的代谢需求。他们指出能量代谢在房颤进展中的重要性。代谢分析表明,糖酵解途径的中间代谢产物,包括2-磷酸甘油酸(2P G)、1,3双磷酸甘油酸(1,3P G)和丙酮酸的含量较大[8]。在房颤的代谢重塑过程中,葡萄糖代谢增加,脂质代谢降低,以维持三羧酸循环性动脉粥样硬化[9]。这些结果与本研究一致。生物过程富集评分表明,调节丙酮酸代谢过程是circRNAs调节基因的重要功能之一。下调表达的circ RNAs(hsa_circ_0000517)调节基因ME3和TNFRS F19参与了能量代谢浸润反应的过程,可能导致心肌缺乏能量供应,导致心肌细胞凋亡和坏死,相反地,上调表达的Circ RNA(hsa_circ_0006903)调节基因PIH1D1和LDHC参与细胞生物发生的降解,可能导致心肌纤维化发生的加速,这一结果部分说明了circRNAs在房颤发病过程中的作用。
在本研究中,笔者发现hsa_circ_0000517 和hsa_circ_0006903调节丙酮酸代谢过程,这表明房颤患者存在能量代谢不足,这可能是hsa_circ_0000517和-0006903之间调节平衡的结果,在这一过程中两种circRNAs发挥相反作用,可能加速或减少肌球蛋白细胞的纤维化进展。
心房结构重塑参与房颤的发展和维持,如左房直径的形态变化。NT-proBNP 被证明与阵发性房颤到持续性房颤的进展相关,而它也是房颤事件的显著预测因子[10]。本研究表明,房颤组的NT-proBNP水平高于对照组。说明房颤组患者存在心房结构重塑,这可能是心房心肌的纤维化过程。本研究表明,hsa_circ_0006903与TGF-β、PⅢNP和PINP呈正相关。而TGF-β、PINP、PⅢNP是心肌纤维化的生物标志物,说明与调控失调的circ RNAs水平有一定的关系。
研究显示,与心血管疾病相关的促炎细胞因子和激素,包括肿瘤坏死因子(TNF)-α,IL-6和血管紧张素-Ⅱ[11]。这些细胞因子的释放也加速了房颤的发生[12]。对环状RNA 的GO 分析表明,环状RNA可以介导这些促炎细胞因子的释放或形成,特别是TNF-α,这也表明circ RNAs参与了房颤的发展。
每个miRNA 调控几十到数百个不同的靶基因,从而从根本上影响细胞生物学。许多研究已经证明了miRNA 参与房颤的发生和发展[13-14]。然而,本研究并未发现这些已知的miRNA 与房颤的关系。笔者发现,既往研究显示micro-RNA204-3p调节葡萄糖代谢[15],结合GO 分析,hsa_circ_0006903 和hsa_circ_0000517 可能通过micro-RNA204-3p调节丙酮酸的形成。这一结果还需要进一步的研究。
但是,本研究仍然有一些局限性,未进行详细的追踪随访,来观察这部分患者的各项指标变化。另外,由于不能获取患者心肌活检标本,无法对心肌的纤维化程度进行定量的评估。

