MDMPR:小鼠发育代谢表型库
朱文静,刘志玮
资源与平台
MDMPR:小鼠发育代谢表型库
朱文静,刘志玮
苏州大学,剑桥–苏大基因组资源中心,苏州 215123
小鼠发育代谢表型库(Mouse Developmental and Metabolic Phenotype Repository, MDMPR)是一个致力于小鼠资源和表型数据实时共享的开放性平台,它依托于科技部重点研发计划“发育编程及其代谢调节”专项项目“建立小鼠发育代谢表型库”。该项目预计在5年内完成500个发育代谢相关小鼠敲除模型的建立,并对其表型数据进行标准化的解析、建立表型数据库。MDMPR作为一个资源及数据集成的库,由多个子系统作为支撑,包括ES细胞数据库、项目管理系统、繁育管理系统、精子库管理系统、表型分析系统,信息化管理深入到项目中每个环节,从基因突变ES细胞制备、基因突变小鼠制备、小鼠繁育,精子冻存到最终的表型分析、数据处理及展示,保证了MDMPR产生数据的真实性及实时性。MDMPR除了不断地推进项目进行,增加自身产生的数据外,也在积极的整合其他的资源及数据,如人特异性基因敲除ES细胞库、蛋白相互作用数据库(STRING)、核心转录调节环路(dbCoRc)和Enhancer-Indel数据库,今后还将进一步整合,帮助发育代谢及其他领域的研究人员能够一站式的获取所需资源和数据、加快研究进程,最终服务于全人类的医疗事业。
小鼠发育代谢表型库;资源库;开放共享平台
作为哺乳动物,小鼠()和人()的基因组同源性高达90%以上,在组织结构和功能、生理学稳态、生殖、行为和疾病发生机制等方面也高度相似。更重要的是,临床疾病大多有其遗传基础,基因突变小鼠模型可以真实模拟人类基因组变异导致的疾病,是研究病理机制和发现潜在靶标的最佳模式动物。人类和小鼠基因组计划完成后,美国、欧盟、加拿大、日本等批准了一系列重大工程项目,系统化建立基因突变小鼠模型,这些项目包括美国国立卫生院的小鼠基因敲除计划(Knockout Mouse Project, KOMP)、欧盟第七框架的欧洲小鼠条件性敲除计划(The European Conditional Mouse Mutagenesis Program,EUCOMM),和加拿大的北美小鼠条件性敲除计划(The North American Conditional Mouse Mutagenesis Project, NorCOMM)等,中国也有支撑计划(如The Chinese Conditional Mouse Mutagenesis, ChiCOMM)[1~6]。随着小鼠敲除模型的不断积累,国际上成立了敲除小鼠联盟(The International Knockout Mouse Consortium, IKMC)[7])。在2011年更是将单纯制备基因修饰小鼠模型的IKMC联盟,发展为系统性无偏见功能性解析小鼠表型的国际小鼠表型分析联盟(International Mouse Phenotyping Consortium, IMPC)[8~11])。迄今为止,全球14个国家的25个研究机构参与了这个国际大科学计划,杂志将该计划和国际核聚变计划、国际加速器计划并列为三大国际大科学计划,并指出系统分析基因功能对疾病解析及新药开发将起到核心推动作用[12]。该计划也是G7国家认可的生命科学领域最大的合作项目,受到领域内研究机构的空前重视。为实施该计划,美国国立卫生院、欧盟、英国医学研究理事会、加拿大卫生部、日本理化研究院及Wellcome Trust基金会都投入了大量经费,目前由现任NIH院长Francis Collins博士担任IMPC领导者。
在我国,疾病模型研究和资源平台建设在过去几年也取得了长足的发展,依托国家遗传工程小鼠资源库等小鼠基因组改造技术平台,科技部于2006年启动了国家科技支撑计划重点项目“人类重大疾病小鼠模型的建立与应用”,使得我国与世界同步开展了基因敲除计划(ChiCOMM),主要承担方为南京大学模式动物研究所(Model Animal Research Center, Nanjing University, MARC),也借此发展成为国内最大的遗传突变小鼠资源库。ChiCOMM形成了与国际对接的小鼠表型分析技术体系及相关技术标准,显著推动了国内敲除小鼠模型的共享及开发。随后在2014年,由苏州大学和英国剑桥大学Sanger研究所共同成立了剑桥–苏大基因组资源中心(Cambridge- Suda Genomic Resource Center, CAM-SU GRC),一个隶属于苏州大学的校级研究机构。剑桥-苏大基因组资源中心依托Sanger研究所提供的资源优势,建立亚太地区的干细胞资源中心,同时建立小鼠标准化基因组功能解析平台,从此成为一个提供小鼠突变胚胎干细胞、疾病模型以及基因功能解析技术的生物技术平台。CAM-SU GRC同时成为继英国、德国、美国后第4个小鼠突变干细胞资源中心,也是亚洲唯一的代表,负责亚太地区48个国家的资源分发工作。在多年的运行期间,剑桥–苏大基因组资源中心已经向国内外上百个课题组提供了细胞及小鼠资源,极大地促进了研究的进展。在国际上,剑桥–苏大基因组资源中心也与南京大学模式动物研究所一起在国际小鼠表型分析联盟指导委员会成员中占得两个席位,这不仅是我国对人类科学事业应尽的责任和义务,同时也是我国社会经济发展的客观需要。
紧跟国际生命科学研究的发展,科技部于2018年启动了国家重点研发计划“发育编程及其代谢调节”重点专项,提出需建立小鼠突变品系的发育与代谢特征的系统分析,对现有和新创建的小鼠突变品系的发育与代谢进行系统性分析,发现重要基因的新功能;建立和实时更新突变品系的表型数据,形成有效的数据共享网络系统。在此背景下,由苏州大学剑桥–苏大基因组资源中心牵头,南京大学模式动物研究所作为主要参与方,联合复旦大学、中国科学院遗传与发育生物学研究所、东华大学、军事医学科学院等单位申报并获批了“建立小鼠发育代谢表型库”项目,本项目的核心就是联合并利用国内优势资源和技术,在5年内建立不少于500个具有自主知识产权的小鼠品系及其发育、代谢表型的数据库,同时重点开展小鼠表型数据与临床特征之间的关联研究,促进人类疾病遗传基础研究领域的变革型发展。小鼠发育代谢表型库(Mouse Developmental and Metabolic Phenotype Repository, MDMPR)作为项目最为主要的成果展示,也于2020年底正式上线(小鼠发育代谢表型库访问地址:www.cam-su.org),受到相关领域内人员极大的关注,随着项目的进行,资源、表型数据的不断丰富,必将极大的推进相关领域研究的进展。
1 国内外同类资源库现状
国外的同类数据库起步较早,目前数据量较大,应用比较广泛的数据库有Mouse Genome Informatics (MGI, http://www.informatics.jax.org/)及International Mouse Phenotyping Consortium (IMPC, https://www.mousephenotype.org/),各自有不同的侧重点。MGI更关注于基因信息的整合,集成基因表达、功能注释、蛋白信息、相关小鼠品系资源信息等,可以使用户较全面的获取基因相关信息,开展研究。IMPC更专注于发布自身项目的数据,包括细胞、小鼠及表型信息,是目前世界上表型信息最为全面的数据库,并且基于其标准化的分析流程,能够进行大数据层面的平行比较,将基因按照表型分类进行归类,给从表型入手的研究者提供了一个非常好的入手点。
国内的此类数据库起步较晚,而且大部分尚处在资源展示的阶段,如国家遗传工程小鼠资源库(http://www.nrcmm.cn/)。另有很多的品系资源保存在商业化的公司内,如上海南方模式生物科技股份有限公司(https://www.modelorg.com/)、江苏集萃药康生物科技股份有限公司(http://www.gempharmatech. com/cn/)和赛业生物(https://www.cyagen.com/cn/zh- cn/),其商业化的运作模式对于资源开放共享方面存在着诸多的限制,不利于研究领域内的资源共享。此外,对于小鼠模型进行系统性的表型解析,并将数据全部开放,在国内尚处于空白阶段。在科技部支持下建立的MDMPR,秉持资源开放共享的理念,对国内的研究领域是一个非常好的补充,也是紧跟国际发展趋势的。
2 MDMPR项目基因选择
小鼠中大约有20,000个基因,而在有限的项目经费支持下,我们只能完成约500个基因的小鼠制备以及功能解析,这样对于目标基因的挑选提出了很大的挑战性,怎么样能从有限的数量中找到尽可能多的有改变的基因。因此,在项目启动初期,我们也做了很多工作,首先利用国际公认权威的人类基因组元件、远端转录调控数据库进行比对整合获得人类4D调控组数据库,进一步与人类疾病相关基因组突变数据库(Motif Break、Mutation Targeted Pair)及对应转录组数据进行关联分析,得到存在潜在逻辑调控关系的人类疾病关联基因数据库,而后再根据GWAS对基因进行深层筛选和分类得到1000多个人类疾病相关的发育代谢相关基因。通过与小鼠基因组进行比对,结合国际表型分析联盟的研究进展并与小鼠基因组进行比对从中深层筛选得到514个小鼠同源基因。另外也从国内的发育、代谢领域专家中征集了部分基因,共同构成MDMPR候选基因。项目进展至今,早期获得的一些基因突变小鼠的初步表型数据也证实了我们筛选基因的方式是有效的,已经发现了数个控制小鼠存活的关键基因,并且是国际上尚未有相关报道的,相信随着项目的深入进行,更多有意义的表型改变会出现。
3 MDMPR技术路线
3.1 标准化基因遗传工程小鼠敲除策略
MDMPR同时采用了两种不同的技术手段去建立基因遗传工程小鼠,其中剑桥-苏大基因组资源中心主要基于现有的突变小鼠胚胎干细胞资源(图1),南京大学模式动物研究所基于CRISPR/Cas9敲除技术(图2),分别建立基因敲除小鼠模型。
小鼠的遗传背景和微生物级别是影响实验结果的重要因素:不同遗传背景的小鼠对同一个基因的改变可能呈现不同量甚至是不同质的反应;同样,小鼠的微生物级别反映小鼠的生理健康状态,其差异也会造成实验结果的波动。对小鼠遗传背景和微生物进行质量控制可以保证使用最少的实验资源完成既定实验目标,最大限度实现实验效益。这部分工作主要包括:(1)小鼠模型制备过程中遗传质控;(2)小鼠繁育过程中的遗传质控;(3)小鼠繁育过程中的微生物质控。
在项目进行期间,依据现有实验动物遗传质量控制和微生物学等级的相应国标[13,14],各单位根据实际情况制定规范,定期对小鼠的遗传背景(C57BL/6)和微生物级别(无特定病原体动物)进行质控,保障了实验动物的可靠性与实验数据的可重复性。截止2020年11月,检测结果见表1,CAM-SU GRC及MARC的微生物质控标准参见附表1及附表2。
3.2 标准化的发育、代谢表型分析流程
以“国际小鼠表型分析联盟”的生理病理表型分析技术流程为基础,本项目发展了重点针对代谢和发育功能的专有分析流程(图3)。在交配获得进入流程的实验小鼠过程中,利用纯和的雌性或雄性与杂合鼠进行交配,对其生殖能力进行评估;另外存活检测作为一个重要的检查点,决定该基因敲除小鼠品系是否会进入胚胎分析流程以及是用杂合还是纯和敲除小鼠进行成体流程分析。
为了保证小鼠在表型分析流程中产生数据的一致性以及可重复性,所有的表型分析操作基于SOP (Standard Operating Procedure)来进行,采集的具体参数,包括实验设置相关参数(metadata)以及具体实验参数(parameter)的参数种类、数据类型、数值选项也通过SOP进行规范,从制度上保障了产生数据的可靠性(图4)。目前已经建立了111个SOP、492个参数,随着项目进行,SOP及参数数量还将进一步增加,MDMPR即是建立在这些基础参数之上。

图1 基于突变小鼠胚胎干细胞的敲除策略
“Knockout First” allele (tm1a)是在目的基因内含子中插入一个SA-IRES-reporter片段(含有报告基因lacZ和一个启动子驱动的Neo),lacZ的转录在SA的作用下受目的基因启动子控制,并在polyA处终止,从而达到终止目的基因转录、破坏基因表达的目的。Flp的表达可以删除SA-IRES-reporter片段,将tm1a转变成条件敲除基因(tm1c),从而恢复基因功能。在tm1a的基础上,Cre的表达可以删除Neo和critical region从而变成lacZ标记的allele (tm1b)同时敲除基因;在tm1c的基础上,Cre的表达可以删除tm1c allele上的critical region而变成移码突变的tm1d,从而使该基因不表达。

图2 基于CRISPR/Cas9的敲除策略
基于CRISPR/Cas9的敲除策略,针对目标外显子两侧设计一对sgRNA,通过Cas9介导的同源重组方式将供体DNA(目标外显子外侧包含一对loxp序列及外侧的同源臂)形成Flox基因,在Cre作用下能够切除目标外显子,造成基因敲除。该策略可能存在两种副产物,其一是同源重组未能发生,造成目标外显子缺失;另可能造成目标外显子部分缺失,也可能引起frame-shift而造成基因敲除。

表1 遗传背景和微生物级别质控情况汇总
CAM-SU GRC:剑桥–苏大基因组资源中心;MARC:南京大学模式动物研究所。
4 MDMPR系统建设
4.1 MDMPR系统架构
建立的细胞、小鼠资源及表型数据通过数据库进行存储,进而通过网页前端将信息通过合理的方式开放给公众进行查询及使用。尽管数据库的目的在于存储细胞、小鼠及表型的信息,但构建时并不仅仅局限于此,还包含了小鼠制备、繁育、品系保种等过程中相关的实验记录及数据。小鼠发育代谢表型库是一个非常庞大的信息化系统,主要包括ES细胞数据库、项目管理系统、繁育管理系统、精子库管理系统及表型分析系统,各系统间信息互相交互,共同形成MDMPR中的对外展示信息。同时,为了更加方便用户使用,我们在进行多个数据库的整合工作,目前已经将蛋白相互作用数据库(STRING)、核心专利调节环路(a database of Core transcriptional Regulatory Circuitries, dbCoRC),Enhancer-Indel数据库进行了整合,使用户能够获取更多的关注基因的咨询,将MDMPR发展成为一站式的小鼠信息资源数据库(图5)。

图3 标准化的发育、代谢表型分析流程

图4 实验参数结构
4.2 MDMPR数据流
数据起始源头来自于ES细胞数据库,项目启动,开始复苏后,克隆信息进入项目管理系统,对复苏状态,QC结果进行记录,相关克隆信息同时返回ES细胞数据库;QC通过的克隆会进行注射,经项目管理系统记录注射数据及嵌合鼠结果,产生的雄性嵌合鼠自动在繁育管理系统中生成,进入实际饲养管理中;饲养管理系统基于移动端开发,通过二维码形成的地址访问相对应的笼位、小鼠信息,常规的繁育操作如配繁、生仔报告、剪尾、分笼、处死均通过“动物管家”APP进行,确保了数据库内存储的活体信息是实时更新的;表型分析项目从繁育管理系统中实际存在的小鼠开始建立,通过建立标准化的分析流程,标准化的数据录入格式,自动化的数据处理方式,保障了数据的真实性,排除人工带入的误差;品系经精子冻存后,冻存样本信息导入精子库管理系统,同时返回繁育管理系统,这样共同构成品系的真实存储状况;MDMPR前端展示数据从各个系统中进行调用,共同构成展示部分(图5)。
4.3 MDMPR前端展示界面设计
数据合理的展示方式实际是用户最为关注的点,我们以使用者的角度,对MDMPR中来源于不同系统的数据有机组合,构成前端展示的界面,使用户能够在一个界面获取到所需数据的概览,如在检索结果的主页即能查看到细胞、小鼠、表型、相关数据库的情况,第一时间掌握资源及数据的储存情况;同时大量使用直观的图示,使用户能够在最短的时间内获取到最为重要的信息,极大的提升了使用的体验度;此外创造性的加入了关注功能,基于全部项目进度的信息化,能够在关注基因状态改变的同时自动化的向用户推送信息,掌握第一手资讯(图6)。

图5 MDMPR系统架构
5 MDMPR应用实例
MDMPR除了对于单个基因的表型数据进行分析外,基于其数据量、产生数据方式带来的平行可比性,可以对数据进行大规模的表型与基因关联分析,筛选出关键基因。我们曾经成功的利用IMPC联盟ICS、TCP、RBRC、WTSI四个中心的小鼠饮食和活动的数据作为模型训练的数据初步开发了深度卷积神经网络方法并对少数指标进行了表型筛选,筛选了10种饮食或者活动异常的基因型小鼠,并针对基因敲除小鼠进行了生物节律的深入表型验证和分析,验证了其在外界光照牵引小鼠昼夜节律中的作用,进一步的功能分析暗示其可能通过调控生物钟核心SCN (Suprachiasmatic Nucleus)神经元之间的同步影响外界光照对昼夜节律的牵引,成果已发表在[15]。IMPC也已有多方面的尝试,在大数据比较的基础上找到了一些新的发育相关基因[9,16~18]。以此为例,在MDMPR数据广度和深度的不断充实下,可以通过类似的大规模表型与基因关联分析,发掘出一些新的基因功能。

图6 MDMPR前端页面设计
6 结语与展望
MDMPR最主要的核心在于其资源与数据的开放共享,这在国内属于创新性的尝试,可以将国家的研究经费转化出最大的价值,被更多的相关研究领域的人所利用。标准化的表型分析流程,信息化的流程及数据管理,保障了产生数据的真实性及及时性,能够为研究者在项目起始阶段提供初期实验数据,对立项及研究方向提供指引,是研究者开展研究的一个利器。虽然MDMPR尚处在起步阶段,资源及表型的数据尚在积累中,从数量上来说还不足,但随着项目的不断进展,更多的资源和数据将会加入,不断充实表型库,让更多用户能够获得有用信息,促进研究的进展。在系统架构上,我们也在不断的进行优化,提升用户在使用上的体验,通过整合其他的数据库,力争将MDMPR建设成一站式的资源数据平台。尽管项目投入有限,我们也将充分利用现有条件,通过技术手段的提升,压缩成本,尽可能在完成项目目标的基础上,增加更多的资源与数据。同时,我们也积极在国内外寻求合作的机会,招募有意愿的研究单位共同加入到此项目中,扩充研究的宽度,使资源的使用效率最大化。目前已有数家合作单位参与到项目中,承担如痛觉、生殖、应激行为等方面的分析,进一步拓展了表型库的覆盖范围,并且已经获得了一些有意义的数据,数据库也在同步准备中,将在不久后开放展示。相信在不久的将来,MDMPR将建设成为国内最大、最全、开放程度最高、实用性最强的资源库,极大地推进代谢、发育甚至其他领域的研究进展。
致谢
感谢科技部重点研发计划“发育编程及其代谢调节”重点专项“建立小鼠发育代谢表型库”项目参与单位苏州大学、南京大学、复旦大学、中国科学院遗传与发育生物学研究所、东华大学、军事医学科学院的项目组成员对于此项目所付出的努力,感谢南京百迈斯信息科技有限公司在MDMPR数据库建设中的全力支持。
附录:
附加材料详见文章电子版www.chinagene.cn。
[1] Freudenthal B, Logan J, Pipelines SIM, Croucher PI, Williams GR, Bassett JHD. Rapid phenotyping of knockout mice to identify genetic determinants of bone strength., 2016, 231(1): R31–46.
[2] Mishra A, Bubela T. Legal agreements and the governance of research commons: lessons from materials sharing in mouse genomics., 2014, 18(4): 254–273.
[3] Adams D, Baldock R, Bhattacharya S, Copp AJ, Dickinson M, Greene NDE, Henkelman M, Justice M, Mohun T, Murray SA, Pauws E, Raess M, Rossant J, Weaver T, West D. Bloomsbury report on mouse embryo phenotyping: recommendations from the IMPC workshop on embryonic lethal screening., 2013, 6(3): 571–579.
[4] Sung YH, Baek IJ, Seong JK, Kim JS, Lee HW. Mouse genetics: catalogue and scissors., 2012, 45(12): 686–692.
[5] Mallon AM, Iyer V, Melvin D, Morgan H, Parkinson H, Brown SDM, Flicek P, Skarnes WC. Accessing data from the International Mouse Phenotyping Consortium: state of the art and future plans., 2012, 23(9–10): 641–652.
[6] Auwerx J, Avner P, Baldock R, Ballabio A, Balling R, Barbacid M, Berns A, Bradley A, Brown S, Carmeliet P, Chambon P, Cox R, Davidson D, Davies K, Duboule D, Forejt J, Granucci F, Hastie N, de Angelis MH, Jackson I, Kioussis D, Kollias G, Lathrop M, Lendahl U, Malumbres M, von Melchner H, Müller W, Partanen J, Ricciardi-Castagnoli P, Rigby P, Rosen B, Rosenthal N, Skarnes B, Stewart AF, Thornton J, Tocchini-Valentini G, Wagner E, Wahli W, Wurst W. The European dimension for the mouse genome mutagenesis program., 2004, 36(9): 925–927.
[7] Austin CP, Battey JF, Bradley A, Bucan M, Capecchi M, Collins FS, Dove WF, Duyk G, Dymecki S, Eppig JT, Grieder FB, Heintz N, Hicks G, Insel TR, Joyner A, Koller BH, Lloyd KCK, Magnuson T, Moore MW, Nagy A, Pollock JD, Roses AD, Sands AT, Seed B, Skarnes WC, Snoddy J, Soriano P, Stewart DJ, Stewart F, Stillman B, Varmus H, Varticovski L, Verma IM, Vogt TF, von Melchner H, Witkowski J, Woychik RP, Wurst W, Yancopoulos GD, Young SG, Zambrowicz B. The knockout mouse project., 2004, 36(9): 921–924.
[8] Rozman J, Rathkolb B, Oestereicher MA, Schütt C, Ravindranath AC, Leuchtenberger S, Sharma S, Kistler M, Willershäuser M, Brommage R, Meehan TF, Mason J, Haselimashhadi H, Consortium I, Hough T, Mallon AM, Wells S, Santos L, Lelliott CJ, White JK, Sorg T, Champy MF, Bower LR, Reynolds CL, Flenniken AM, Murray SA, Nutter LMJ, Svenson KL, West D, Tocchini-Valentini GP, Beaudet AL, Bosch F, Braun RB, Dobbie MS, Gao X, Herault Y, Moshiri A, Moore BA, Kent Lloyd KC, McKerlie C, Masuya H, Tanaka N, Flicek P, Parkinson HE, Sedlacek R, Seong JK, Wang CL, Moore M, Brown SD, Tschöp MH, Wurst W, Klingenspor M, Wolf E, Beckers J, Machicao F, Peter A, Staiger H, Haring HU, Grallert H, Campillos M, Maier H, Fuchs H, Gailus-Durner V, Werner T, de Angelis MH. Identification of genetic elements in metabolism by high-throughput mouse phenotyping., 2018, 9(1): 288.
[9] Dickinson ME, Flenniken AM, Ji X, Teboul L, Wong MD, White JK, Meehan TF, Weninger WJ, Westerberg H, Adissu H, Baker CN, Bower L, Brown JM, Caddle LB, Chiani F, Clary D, Cleak J, Daly MJ, Denegre JM, Doe B, Dolan ME, Edie SM, Fuchs H, Gailus-Durner V, Galli A, Gambadoro A, Gallegos J, Guo SY, Horner NR, Hsu CW, Johnson SJ, Kalaga S, Keith LC, Lanoue L, Lawson TN, Lek M, Mark M, Marschall S, Mason J, McElwee ML, Newbigging S, Nutter LMJ, Peterson KA, Ramirez-Solis R, Rowland DJ, Ryder E, Samocha KE, Seavitt JR, Selloum M, Szoke-Kovacs Z, Tamura M, Trainor AG, Tudose I, Wakana S, Warren J, Wendling O, West DB, Wong L, Yoshiki A, International Mouse Phenotyping Consortium, Jackson Laboratory , Infrastructure Nationale Phenomin, Institut Clinique de la Souris (ICS), Charles River Laboratories, Harwell MRC, Toronto Centre for Phenogenomics, Wellcome Trust Sanger Institute, RIKEN BioResource Center, MacArthur DG, Tocchini-Valentini GP, Gao X, Flicek P, Bradley A, Skarnes WC, Justice MJ, Parkinson HE, Moore M, Wells S, Braun RE, Svenson KL, de Angelis MH, Herault Y, Mohun T, Mallon AM, Henkelman RM, Brown SDM, Adams DJ, Lloyd KCK, McKerlie C, Beaudet AL, Bućan M, Murray SA. High-throughput discovery of novel developmental phenotypes., 2016, 537(7621): 508–514.
[10] Brown SDM, Chambon P, de Angelis MH, Eumorphia Consortium. EMPReSS: standardized phenotype screens for functional annotation of the mouse genome., 2005, 37(11): 1155.
[11] Skarnes WC, Rosen B, West AP, Koutsourakis M, Bushell W, Iyer V, Mujica AO, Thomas M, Harrow J, Cox T, Jackson D, Severin J, Biggs P, Fu J, Nefedov M, de Jong PJ, Stewart AF, Bradley A. A conditional knockout resource for the genome-wide study of mouse gene function., 2011, 474(7351): 337–342.
[12] Mouse megascience., 2010, 465(7298): 526.
[13] 魏强, 贺争鸣, 田克恭, 李红, 黄韧, 范薇, 屈霞琴. GB 14922.2-2011 实验动物微生物学等级及监测. 2011.
[14] 岳秉飞, 高翔, 鲍世民, 张连峰, 邢瑞昌. GB 14923-2010 实验动物哺乳类实验动物的遗传质量控制. 2010.
[15] Zhang T, Xie PC, Dong YY, Liu ZW, Zhou F, Pan DJ, Huang ZY, Zhai QC, Gu Y, Wu QY, Tanaka N, Obata Y, Bradley A, Lelliott CJ, Sanger Institute Mouse Genetics Project, Nutter LMJ, McKerlie C, Flenniken AM, Champy MF, Sorg T, Herault Y, De Angelis MH, Durner VG, Mallon AM, Brown SDM, Meehan T, Parkinson HE, Smedley D, Lloyd KCK, Yan J, Gao X, Seong JK, Wang CKL, Sedlacek R, Liu Y, Rozman J, Yang L, Xu Y. High-throughput discovery of genetic determinants of circadian misalignment., 2020, 16(1): e1008577.
[16] Bowl MR, Simon MM, Ingham NJ, Greenaway S, Santos L, Cater H, Taylor S, Mason J, Kurbatova N, Pearson S, Bower LR, Clary DA, Meziane H, Reilly P, Minowa O, Kelsey L, International Mouse Phenotyping Consortium, Tocchini-Valentini GP, Gao X, Bradley A, Skarnes WC, Moore M, Beaudet AL, Justice MJ, Seavitt J, Dickinson ME, Wurst W, de Angelis MH, Herault Y, Wakana S, Nutter LMJ, Flenniken AM, McKerlie C, Murray SA, Svenson KL, Braun RE, West DB, Lloyd KCK, Adams DJ, White J, Karp N, Flicek P, Smedley D, Meehan TF, Parkinson HE, Teboul LM, Wells S, Steel KP, Mallon AM, Brown SDM. A large scale hearing loss screen reveals an extensive unexplored genetic landscape for auditory dysfunction., 2017, 8 (1): 886.
[17] Moore BA, Leonard BC, Sebbag L, Edwards SG, Cooper A, Imai DM, Straiton E, Santos L, Reilly C, Griffey SM, Bower L, Clary D, Mason J, Roux MJ, Meziane H, Herault Y, International Mouse Phenotyping Consortium, McKerlie C, Flenniken AM, Nutter LMJ, Berberovic Z, Owen C, Newbigging S, Adissu H, Eskandarian M, Hsu CW, Kalaga S, Udensi U, Asomugha C, Bohat R, Gallegos JJ, Seavitt JR, Heaney JD, Beaudet AL, Dickinson ME, Justice MJ, Philip V, Kumar V, Svenson KL, Braun RE, Wells S, Cater H, Stewart M, Clementson-Mobbs S, Joynson R, Gao X, Suzuki T, Wakana S, Smedley D, Seong JK, Tocchini-Valentini G, Moore M, Fletcher C, Karp N, Ramirez-Solis R, White JK, de Angelis MH, Wurst W, Thomasy SM, Flicek P, Parkinson H, Brown SDM, Meehan TF, Nishina PM, Murray SA, Krebs MP, Mallon AM, Lloyd KCK, Murphy CJ, Moshiri A. Identification of genes required for eye development by high-throughput screening of mouse knockouts., 2018, 1: 236.
[18] Moore BA, Flenniken AM, Clary D, Moshiri AS, Nutter LMJ, Berberovic Z, Owen C, Newbigging S, Adissu H, Eskandarian M, McKerlie C, International Mouse Phenotyping Consortium, Thomasy SM, Lloyd KCK, Murphy CJ, Moshiri A. Genome-wide screening of mouse knockouts reveals novel genes required for normal integumentary and oculocutaneous structure and function., 2019, 9(1): 11211.
附表1 CAM-SU GRC微生物质控标准
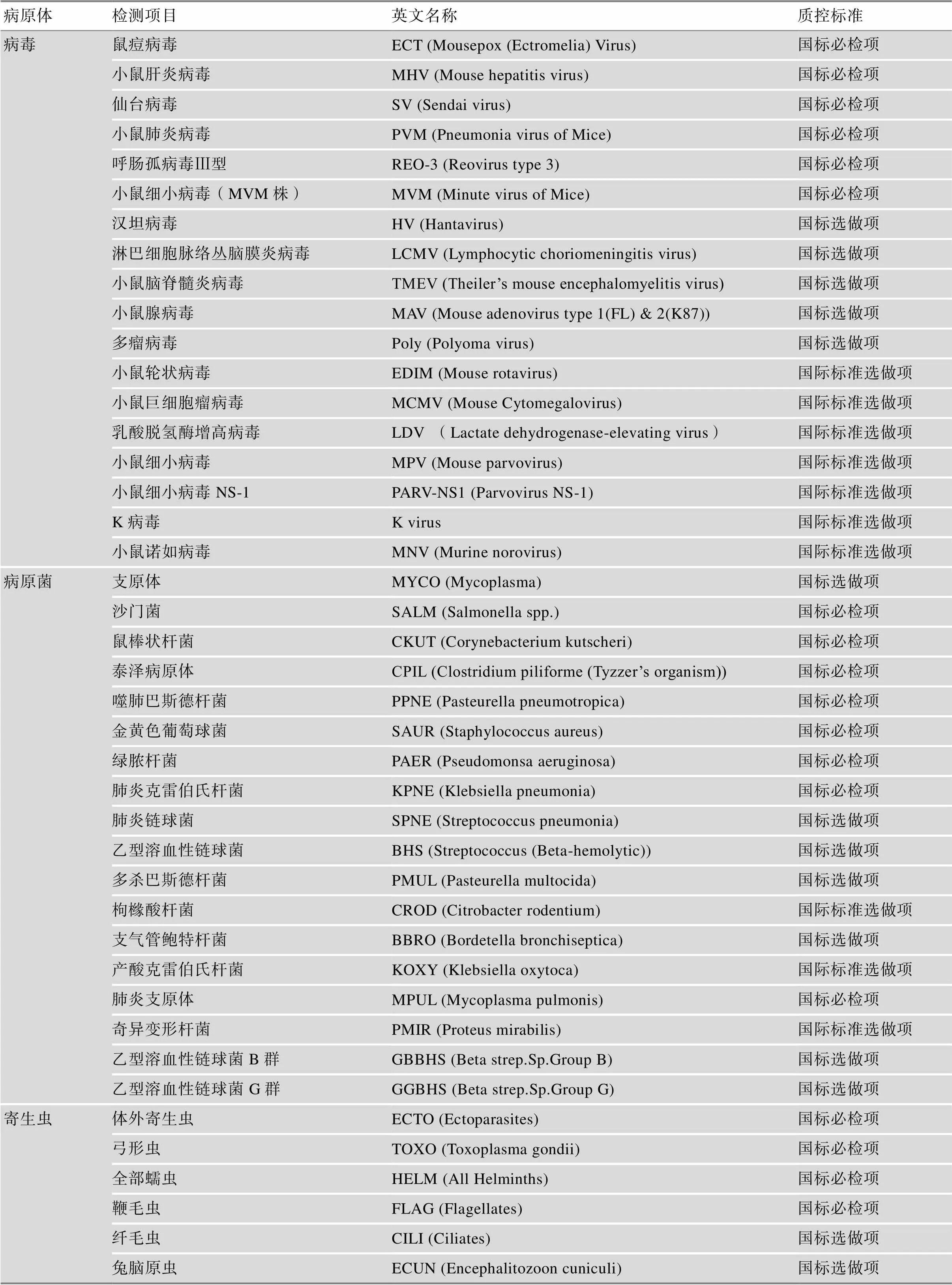
Supplementary Table 1 Microbiological quality control standards of CAM-SU GRC
附表2 MARC微生物质控标准
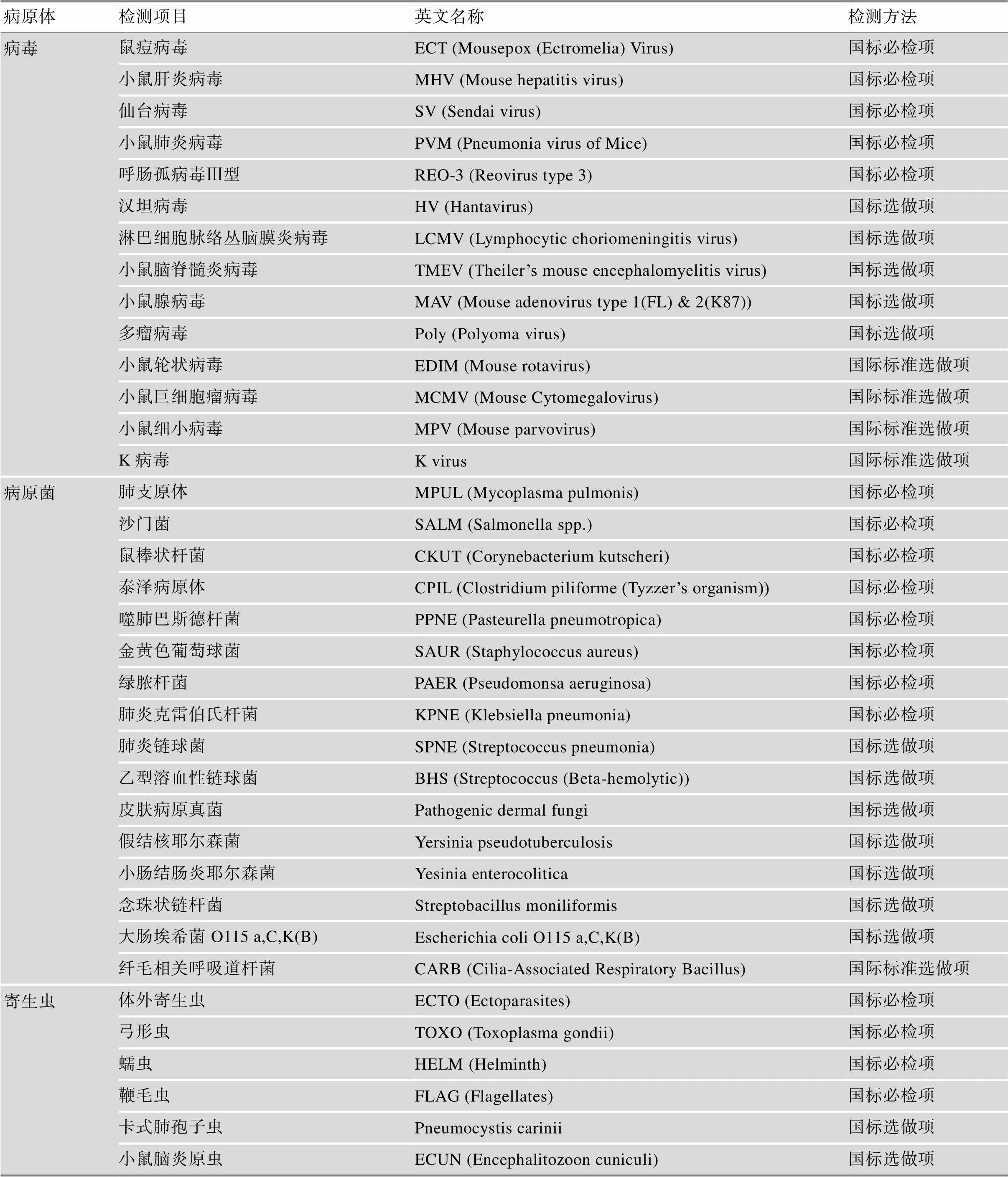
Supplementary Table 2 Microbiological quality control standards of MARC
MDMPR: Mouse Developmental and Metabolic Phenotype Repository
Wenjing Zhu, Zhiwei Liu
Mouse Developmental and Metabolic Phenotype Repository (MDMPR) is an open access, real-time database which dedicates to share mouse resources and phenotype data. MDMPR is supported by the National Key Research and Development Project “Establishment of Mouse Developmental and Metabolic Phenotype Repository” within the Key Project of “Developmental Programming and Its Metabolic Regulation” from the Ministry of Science and Technology of the People’s Republic of China’s program. In the next 5 years, MDMPR will create 500 mutant mouse models related to development and metabolism, perform standard phenotyping analysis, and establish a phenotype database. MDMPR is a combination of resources and data repository, has several sub-systems, including the ES cell database, the project management system, the breeding management system, the sperm bank management system and the phenotyping database. These systems digitalize all data and ensure their authenticity in real-time. Besides the gradual increase of data during the project, MDMPR will also integrate other resources, such as human KO ES cell database, STRING database, database of Core Transcriptional Regulatory Circuitries and Enhancer-Indel database. MDMPR is anticipated to contribute to various areas of developmental and metabolic research to investigators through more convenient accesses to the resources and data in one-stop manner, thereby accelerating the research processes and ultimately serving the medical causes of human health.
Mouse Development and Metabolic Phenotype Repository; resource repository; open-access sharing platform
2021-01-05;
2021-03-04
科技部重点研发计划项目(编号:2018YFA0801100)资助[Supported by the National Key R&D Program of the Ministry of Science and Technology of China (No. 2018YFA0801100)]
朱文静,硕士,实验技术员,研究方向:生物化学与分子生物学。E-mail: zhuwenjing@cam-su.org
刘志玮,博士,研究方向:生物化学与分子生物学。E-mail: liuzhiwei@cam-su.org
10.16288/j.yczz.21-005
2021/3/16 11:31:37
URI: https://kns.cnki.net/kcms/detail/11.1913.R.20210315.0958.005.html
(责任编委: 陈帅)

