植物PHD结构域蛋白的结构与功能特性
王天一,王应祥,尤辰江
综 述
植物PHD结构域蛋白的结构与功能特性
王天一,王应祥,尤辰江
复旦大学生命科学学院,植物科学研究所,上海 200433
植物同源结构域(plant homeodomain, PHD)是锌指结构域家族的一类转录调控因子,其最主要的功能是可以识别各种组蛋白修饰密码,包括组蛋白甲基化和乙酰化等;此外PHD结构域还可以与DNA结合。含有PHD结构域的蛋白,或者本身具有组蛋白修饰酶活性,或者可以与各类组蛋白修饰酶相互作用,还有部分与DNA甲基化相关,具有E3泛素连接酶活性,或者还可以作为染色质重塑因子,以各种不同的作用方式,在植物的生长发育过程中发挥了重要的作用。本文主要综述了结合各种类型组蛋白(包括H3K4me3/0、H3K9me3、H3R2和H3K14ac)以及DNA的PHD结构域的结构特点及其结合特异性、PHD结构域在植物中的进化保守性以及植物中已经发现的含有PHD结构域蛋白的功能及作用机制,为进一步了解该类蛋白在植物生长发育过程中如何发挥作用提供了参考。
PHD结构域;组蛋白密码;组蛋白修饰识别;转录调控;表观遗传
植物同源结构域(plant homeodomain, PHD)是真核生物进化过程中一种保守的锌指结构域。PHD结构域发挥功能最主要的方式是识别各类组蛋白修饰,它们对于各类组蛋白修饰的识别也具有一定的特异性,除此之外还可以识别一些DNA序列。含有PHD结构域的蛋白作为转录调控因子,参与生物体的各项生命过程。如在植物中,含有PHD结构域的蛋白参与了包括胚胎分生组织萌发、根系发育、发芽、开花、减数分裂,以及减数分裂后的花粉发育等重要过程,对植物的生长发育具有十分重要的作用[1]。
关于植物中含有PHD结构域的蛋白,Mouriz等[1]按照其功能分类进行了综述,本文则是从PHD结构域与各类组蛋白翻译后修饰的结合特异性、PHD结构域在植物中的进化保守性,以及各类PHD结构域蛋白的作用机制方面进行论述,主要针对拟南芥()中PHD蛋白的作用机制进行解析,为进一步了解其如何发挥调控功能提供参考。
1 PHD的结合特异性
组蛋白翻译后修饰主要包括甲基化、乙酰化等多种形式,这些翻译后修饰通过招募与组蛋白结合的效应蛋白来调控下游基因的表达。其中,识别组蛋白甲基化修饰的结构域主要为PHD结构域和Royal家族的Chromo、Tudor、PWWP (Pro-Trp-Trp- Pro)和MBT (malignant brain tumour)结构域等;而识别组蛋白乙酰化的主要为 Bromo、DPF (double PHD finger)和YEATS结构域[2]。除此之外,对于DNA的识别主要有螺旋–转角–螺旋(Helix-Turn-Helix, HTH)、锌指结构、亮氨酸拉链(basic Leucine zipper, bZIP)和碱性–螺旋–环–螺旋(basic Helix- Loop-Helix, bHLH)结构域等,而PHD结构域就属于锌指结构域家族。
PHD结构域是一类比较小的蛋白结构域,由50~80个氨基酸残基组成,其不同家族成员序列表现出较低的氨基酸相似性,但该结构域可以折叠成高度保守的球状结构[3]。保守的PHD折叠,包含了一对反向平行的双链β-sheet和一个C端的α-helix (不是所有的PHD都有),2个锌原子被固定在Cys4-His-Cys3基序上,形成一个“cross-brace”的拓扑结构[4]。
PHD结构域最主要功能是与各种组蛋白修饰特异结合,作为组蛋白密码识别器来调控下游基因的表达。研究发现,PHD结构域可以识别的组蛋白密码包括H3K4me3/2/0[5,6]、H3K9me3[7~9]、H3K36[10]、H3R2me2/0[11,12]、H3K14ac以及H4乙酰化等[13,14]。研究发现,PHD结构域通常和其他识别模块(reader modules)成对出现,例如与Bromo、Chromo、Tudor、PWWP、MBD、SRA或者PHD finger本身,一起作为一个组合来发挥作用[15]。模块配对可以显著地提升PHD的识别能力,提高与组蛋白之间结合的亲和力和特异性,从而提供新的调控能力。例如BPTF通过PHD-Bromo结构域识别“H3K4me3+H4K16ac”[16]。
本文对已经研究出晶体结构以及结合特性的PHD蛋白进行了总结。
1.1 识别H3K4me3
PHD与组蛋白H3K4me3的结合通常依靠一个包含2~4个芳香族残基的笼子(aromatic cage)。在这个笼子中,K4me3由范德华力和π键稳定[5](图1A)。不同的PHD中aromatic cage氨基酸组成不一样,但是位于序列的相似位置,其中一个色氨酸(Trp)残基,在位点和残基类型上都完全保守,是识别结合甲基化赖氨酸的PHD中最特异的残基[17]。
在结合过程中,组蛋白H3的肽段形成第三条反向平行的β链,与PHD现存的双链β-sheet配对[5]。PHD蛋白的β1链和H3K4me3的R2-T6残基之间形成分子间氢键,稳定了结合复合体的特征骨架[18]。
1.2 识别未修饰的H3
研究表明,BHC80[19]的PHD结构域可以与未修饰的组蛋白H3的前8个残基结合(图1B),并且当K4位点发生甲基化时,这个结合会被阻止,但是K9和K14位点的甲基化不会对其造成影响[19]。类似的,AIRE[20]的PHD结构域也可以与未修饰的组蛋白H3氮端的前8个残基结合。
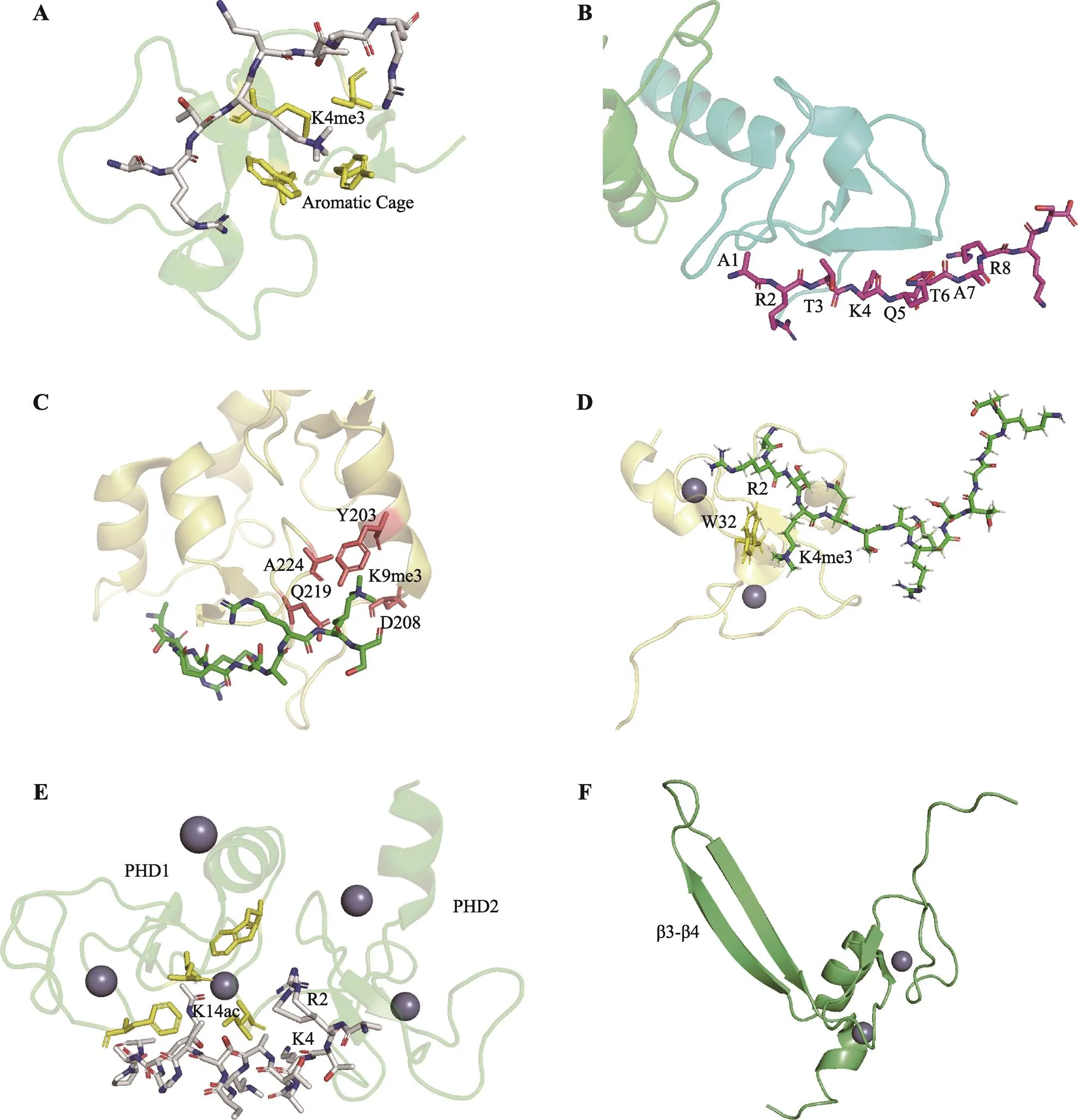
图1 PHD结构域的晶体结构图
A:ING2的PHD结构域结合H3k4me3 (2g6q[23]);B:BHC80的PHD结构域结合未修饰的H3 (2puy[19]);C:ATRX的PHD结构域识别H3K9me3 (3ql9[24]);D:BPTF的PHD结构域结合H3K4和H3R2 (2fuu[25]);E:DPF3b的PHD1识别乙酰化修饰(5i3l[26],黄色代表结合K14ac的疏水性口袋);F:BRPF2的PHD2结构域(2lq6[27])。图中所用模型来自PDB数据库(Protein Data Bank),晶体结构展示软件为PyMOL (version 2.4)。
与H3K4me3-PHD类似,未修饰的组蛋白H3尾巴也形成一个β链,与PHD的β1链配对。但是,这一类的PHD很明显缺失了芳香族的残基,由一大片的酸性残基作为替代,并且PHD的第1个与锌离子结合的半胱氨酸残基的N端会存在一个或者一对保守的酸性残基[19,20]。
1.3 识别H3K9me3
ATRX通过其ADD结构域识别H3K9me3。ADD (ATRX-DNMT3-DNMT3L)结构域富含半胱氨酸残基,是4个不同的蛋白ATRX、DNMT3A、DNMT3B和DNMT3L共同拥有的结构。ATRX的ADD结构域由1个N端类GATA锌指(GATA-like finger)、1个PHD结构域、1个长的C端螺旋(C-terminal helix),通过广泛的疏水性相互作用组装在一起,形成一个球状的结构域[21]。
在ATRX的ADD结构域识别K9me3的过程中,涉及到一个非典型的极性口袋(polar pocket),该口袋只含有一个芳香族的酪氨酸(Tyr),并且富含极性的氨基酸(polar residues)[8]。除了分子表面的范德华力以外,K9me3的识别还涉及到了芳香族Tyr203与甲基化赖氨酸之间的阳离子π作用,以及Tyr203、Asp207、Gln219和Ala224与甲基化的赖氨酸之间的非传统的C-H…O氢键的相互作用[21](图1C)。
类似地,在TRIM33[7]和CHD4[9]中也存在PHD的一个芳香族残基与K9之间形成π键,以及非传统的氢键作用。对于TRIM33,它具有一个PHD-Bromo盒子(cassette),其Bromo结构域与H3的K14、K18、K23乙酰化修饰结合,PHD结构域与H3K9me3结合。在结合K9me3的过程中,K9me3与芳香族Trp889之间形成π键,与PHD的羰基氧之间形成非传统的C-H…O氢键[7];而在CHD4的PHD2中,K9me3与芳香族Phe451之间形成π键,与其他残基之间形成氢键,这样的模式稳定了PHD-H3K9me3的结构[9]。
1.4 识别H3R2
在许多蛋白(例如BPTF[5]、ING4[22]和UHRF[11]等)中,H3K4和H3R2的识别发生在同一个PHD的不同位点。在这个过程中,H3R2的侧链和H3K4me3分别进入到2个相邻的识别口袋,这两个口袋通过一个保守的Trp残基分隔开(图1D)。其中K4me3的结合口袋是芳香族笼子,通过π键,疏水相互作用和范德华力,稳定PHD与三甲基集团之间的结合。而R2口袋通常是酸性的,通过Asp、Glu和Gln等残基与R2之间形成盐桥或者氢键来进行配对[5]。UHRF1的PHD同样依赖于Asp347和Asp350与H3R2之间形成氢键进行结合[11]。
1.5 识别乙酰化
识别乙酰化组蛋白的PHD通常是串联的两个,例如DPF3b[14]、MOZ[13]和MORF[28]的DPF模块可以与H3K14以及其他乙酰化的组蛋白尾巴相互作用。
研究发现,DPF3b的2个串联的PHD可以形成一个组合的球状结构域。其中PHD1可以结合H3K14ac,PHD2可以结合H3K4me0和H3R2me0(图1E)。PHD2结合H3K4me0的口袋,与前述结合H3K4me0的PHD类似,拥有酸性和疏水性的残基。而PHD1结合H3K14ac的口袋是由Phe264、Leu296、Trp311和Ile314组成的疏水性口袋。PHD1在β2表面产生了一个窄的沟槽,当复合体形成,H3的T11和K14ac的侧链插入到这个沟槽中,由一系列氢键和范德华力稳定[14]。类似的,MOZ通过其Phe211、Leu242、Trp257和Ile260组成相应的疏水性口袋来识别K14ac[13]。
1.6 识别DNA
BRPF1/2[27]中的PHD可以结合DNA,它们都拥有2个PHD结构域,中间由一个锌原子连接。BRPF1/2的PHD1对于未修饰的H3有高度的特异性。PHD2呈鞍状结构,可以与DNA相互作用,与一般的PHD结构域不同,PHD2具有2个与锌离子配位的组氨酸残基,其序列特征为C4HC2H,并且还含有一个额外的β3-β4发夹结构(图1F)。PHD2对于DNA的结合能力,可能是由于PHD结构域中具有静电势(electrostatic potential)的表面的作用[27]。
2 植物中PHD蛋白功能及作用机制
染色质对基因的转录调控主要依赖于两种作用:一种是激活因子,主要依赖于组蛋白的乙酰化修饰,使DNA结构变得松散,有利于RNA聚合酶等转录起始因子与DNA的结合;另一种是抑制因子,它们可以通过对组蛋白进行抑制型的甲基化修饰,以招募去乙酰化酶,去掉激活类修饰,进而增强染色质的屏障作用。含有PHD结构域的蛋白,以两种形式参与转录调控作用,或者本身即拥有组蛋白修饰酶活性,或者可以与各种组蛋白修饰酶相互作用。还有部分PHD结构域蛋白与DNA甲基化相关,具有E3泛素连接酶活性,或者可以作为染色质重塑因子,对各种植物的生长发育以及应对各种逆境胁迫过程着重要作用。
PHD结构域蛋白在植物中广泛存在。在植物基因组网站Phytozome12中搜索可以发现,已注释的拟南芥PHD结构域基因有82个,水稻(L.)有84个,无油樟()有67个。以拟南芥MMD1的同源蛋白构建系统发育树,发现该PHD结构域蛋白在植物中广泛存在,在十字花科和豆科中的同源蛋白数量最多,在蔷薇科、芸香科、杨柳科、番木瓜科等科目中也有存在,说明该蛋白在植物进化过程中具有保守性,并且可能在多种植物的生长发育过程中发挥了重要的作用(图2)。

图2 植物中部分PHD结构域蛋白MMD1的系统发育树
图中展示了部分拟南芥MMD1的同源蛋白的进化关系(序列来源于植物基因组网站Phytozome12)。使用IQ-TREE (version 1.6.12),以最大似然法(Maximum Likelihood, ML)构建系统发育树,选用的构树模型为JTT+R6,以葫芦藓科、泥炭藓科和卷柏科的MMD1同源蛋白定根。图中数字为分支的自展值,星号“*”代表自展值为100。拟南芥的MMD1分支标为红色,睡莲的MMD1同源蛋白分支标为蓝色,无油樟的MMD1同源蛋白分支标为绿色。图中标注了几个主要的科目,分别为豆科(粉色)、蔷薇科(绿色)、杨柳科(紫色)、锦葵科(黄色)、十字花科(蓝色)和番木瓜科(橙色)。
对拟南芥中已研究的PHD结构域蛋白进行序列比对,发现它们都具有保守的C4HC3序列特征(图3)。根据拟南芥中含有PHD结构域的蛋白发挥功能的调控机制不同,可以对其进行具体的分类(表1):主要包括本身具有组蛋白修饰酶活性(包括本身具有组蛋白甲基转移酶活性的ATX1/2/3/4/5和ATXR,本身具有组蛋白乙酰基转移酶活性的IDM1/ROS4);可以与组蛋白修饰酶相互作用(包括可以与组蛋白去乙酰化酶相互作用的EBS/SHL,与组蛋白甲基转移酶相互作用的MMD1、AL、VIN3),与DNA甲基化相关的MBD9和ORTH,具有E3泛素连接酶活性的SIZ1,以及可以作为染色质重塑因子的CHR4和PKL等。
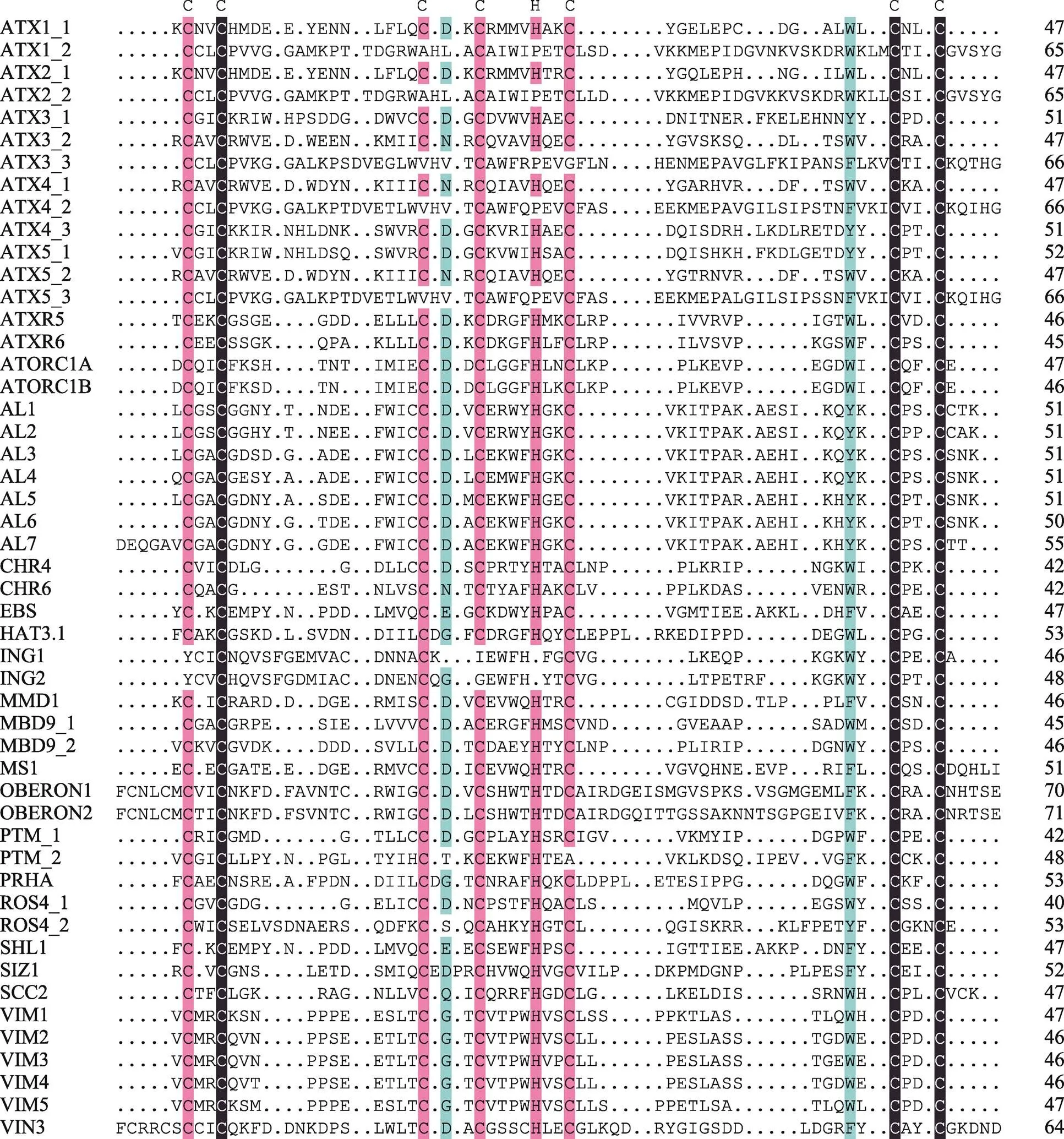
图3 拟南芥中PHD结构域的序列比对
黑色表示同源性为100%,粉色表示同源性为75%,蓝色表示同源性为50%。可以看出PHD结构域保守的C4HC3氨基酸残基序列特征。图中各蛋白对应的TAIR基因号分别为:ATX1 (At2g31650)、TX2 (At1g05830)、ATX3 (At3g61740)、ATX4 (At4g27910)、ATX5 (At5g53430)、ATXR5 (At5g09790)、ATXR6 (At5g24330)、ATORC1A (At4g14700)、ATORC1B (At4g12620)、AL1 (At5g05610)、AL2 (At3g11200)、AL3 (At3g42790)、AL4 (At5g26210)、AL5 (At5g20510)、AL6 (At2g02470)、AL7 (At1g14510)、CHR4 (At5g44800)、CHR6 (At2g25170)、EBS (At4g22140)、HAT3.1 (At3g19510)、ING1 (At3g24010)、ING2 (At1g54390)、MMD1 (At1g66170)、MBD9 (At3g01460)、MS1 (At5g22260)、OBERON1 (At3g07780)、OBERON2 (At5g48160)、PTM (At5g35210)、PRHA (At4g29940)、ROS4 (At3g14980)、SHL1 (At4g39100)、SIZ1 (At5g60410)、SCC2 (At5g15540)、VIM1 (At1g57820)、VIM2 (At1g66050)、VIM3 (At5g39550)、VIM4 (At1g66040)、VIM5 (At1g57800)、VIN3 (At5g57380)。

表1 拟南芥中PHD蛋白的特征与功能
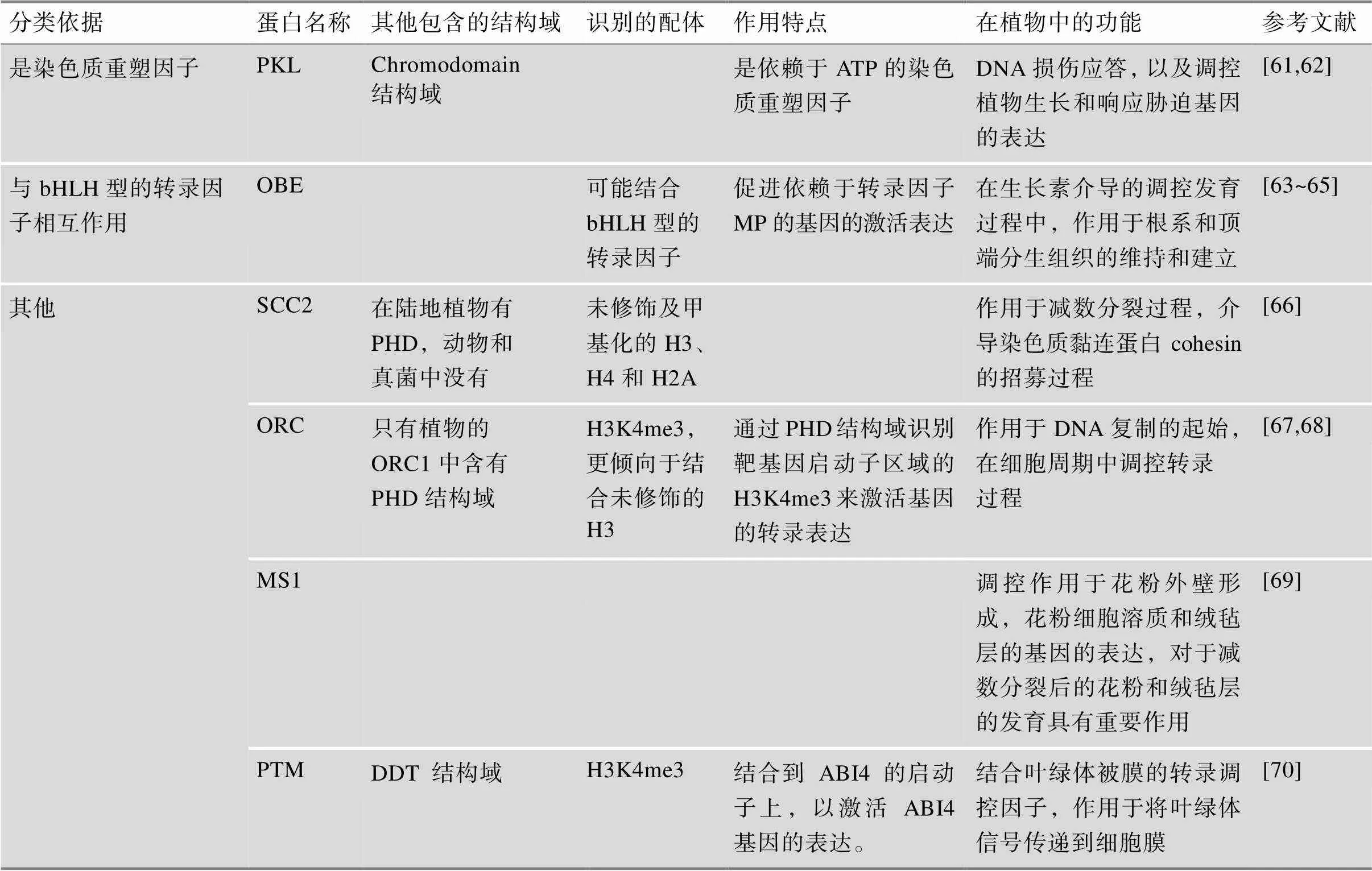
续表
2.1 本身具有组蛋白修饰酶活性
2.1.1 本身具有组蛋白甲基转移酶活性
拟南芥()基因分为两个亚家族,分别为/亚家族和//亚家族。ATX1和ATX2蛋白包含ePHD结构域(extended plant homeodomain)和SET结构域。ePHD结构域包含1个N端pre-PHD (C2HC Zinc finger)、1个长的连接区域和1个PHD (C4HC3Zinc finger),ePHD结构域可以结合双链DNA但是不能结合组蛋白。而SET结构域使ATX1/2具有甲基转移酶活性。ATX1作用于H3K4me3,而ATX2作用于H3K4me2,这两个组蛋白修饰都是活跃转录的标志。
ATX1参与根系、叶片和花器官的发育以及一些逆境胁迫基因的转录调控[29]。研究表明,ATX1在调控目标基因表达时具有两种不同的作用:一种是作为共同激活子参与前起始复合物PIC的形成,招募RNA聚合酶II和TATA结合蛋白;另一种是被磷酸化的RNA聚合酶II招募到转录起始位点下游行使H3K4三甲基化作用[31]。ATX1催化的H3K4me3不作用于转录起始,而是作用于活跃转录的延伸[30]。与其他的PHD结构域结合H3K4me3的功能不同,ATX1的ePHD结构域通过结合PI5P (phosphatidylinositol 5-phosphate),影响ATX1的亚细胞定位,从而调控一些特异依赖于ATX1的基因的表达[71]。
ATX2与ATX1拥有相似的序列,但是它们在调控基因转录方面具有非冗余的功能,在作用于同一个基因的调控时,有不同的靶向特异性,并且使用了不同的分子生物学机制[32]。例如,ATX1和ATX2都使的转录水平上调。在突变体中,的H3K4me2的标记消失伴随着基因转录水平的降低,并且H3K4me3不是转录所需要的(在活跃转录的野生型中也没有H3K4me3)。说明ATX2导致的H3K4me2累积对转录是必要的,而ATX1则并不对的核小体直接进行修饰,发挥了间接的调控作用[32]。
ATX3、ATX4、ATX5同样包含PHD结构域,并且可能编码了H3K4甲基转移酶,参与H3K4的二甲基化和三甲基化。ATX3/4/5具有冗余的功能,可以调控一系列作用于营养生长和生殖生长的基因。它们是迄今为止在拟南芥基因组中发现的唯一具有H3K4me2甲基转移酶活性的蛋白[34]。
ATXR5/6 (ARABIDOPSIS TRITHORAX-RELATED PROTEIN 5/6)是拟南芥中另一对含有PHD结构域的蛋白,具有H3K27me1甲基转移酶活性。ATXR5/6参与植物中染色质结构、基因沉默和异染色质的DNA复制过程[72]。和突变体表现出异染色质成分(heterochromatic elements)、转座子和重复序列的转录激活。ATXR5/6的PHD结构域可以识别未甲基化的H3K4,参与SET结构域结合辅因子以及促进H3K27me1的过程[35]。
除此之外,在酵母和人类的PHD蛋白中也有类似的例子。酿酒酵母()的Spp1是一个含有PHD结构域的蛋白,同时也是组蛋白H3K4甲基转移酶Set1(COMPASS)复合体的成员。Spp1的PHD结构域结合组蛋白H3K4,并且可以调控甲基转移酶COMPASS的活性[73]。在人类中的一些蛋白,包括PHF2、PHF8和KIAA1718等,除了包含PHD结构域以外,还包含1个具有组蛋白去甲基化酶活性的JmjC (Jumonji-C)结构域。这些蛋白的PHD结构域,在活跃转录基因的启动子区域,与含有H3K4me3的核小体结合。而JmjC结构域则可以移除与转录抑制相关的组蛋白H3K9、H3K27的单、双甲基化以及H4K20的单甲基化。这样的双重作用,确保了基因在被转录激活之后,组蛋白转录抑制标记能够恰当地移除[74]。
2.1.2 本身具有组蛋白乙酰基转移酶活性
IDM1 (INCREASED DNA METHYLATION1)是一个组蛋白H3乙酰基转移酶,具有1个MBD结构域(methyl-CpG-binding domain)、1个PHD结构域和1个组蛋白乙酰基转移酶结构域[37]。MBD结构域参与识别甲基化的DNA;PHD结构域可以识别未甲基化的H3K4。IDM1对于DNA甲基化具有负面调控,阻止高度同源的多拷贝基因和其他重复序列的DNA高度甲基化,其抑制作用表现在两个方面:第一,IDM1的H3乙酰化活性可以抑制DNA高度甲基化,乙酰化的H3(H3K18Ac、H3K23Ac)创造了一个允许5-甲基胞嘧啶DNA糖苷酶(参与DNA脱甲基化)作用的染色质环境;第二,PHD结构域抑制DNA高度甲基化。一方面,PHD结构域的突变,会引起IDM1乙酰转移酶活性的丧失,这也说明了PHD结构域可以不依赖于其结合组蛋白H3的功能,去影响IDM1乙酰转移酶的活性;另一方面,PHD结构域只能识别未甲基化的H3K4,其与H3K4me0的结合会被H3K4me2/3抑制。因此,由于PHD结构域的存在,IDM1在H3K4me2/3水平高的位点与甲基化的DNA结合是被抑制的[75]。
在酿酒酵母中,Yng1蛋白包含PHD finger结构域,并且该蛋白是NuA3 HAT复合体(组蛋白乙酰基转移酶)的组成部分。Yng1通过PHD结构域识别H3K4me3,增强了NuA3 HAT复合体对组蛋白H3底物作用的活性,使H3K14乙酰化,从而激活基因的表达[76]。
2.2 与组蛋白修饰酶相互作用
2.2.1 与组蛋白去乙酰化酶相互作用
()和()是拟南芥中的两个旁系同源基因,都包含PHD结构域和BAH (bromo-adjacent homology)结构域,参与染色质介导的开花抑制和种子休眠。研究发现,的过量表达还会引起植物育性的降低[77]。SHL和EBS在调控开花时间上发挥重要作用,主要通过其PHD结构域识别H3K4me2和H3K4me3,分别结合到和(和都是开花调节基因[78])的调控区域。这两种PHD蛋白通过结合组蛋白去乙酰化酶HDA6 (HISTONE DEACETYLASE 6),阻止高水平的H3乙酰化,维持了和的不活跃的染色质构象[79]。EBS与HDA6的相互作用,说明组蛋白甲基化和乙酰化对于开花时间的精确控制是一个关键的因素。另外,在和参与的种子休眠调控的过程中,组蛋白甲基化和乙酰化修饰共同协作对于种子休眠的精确调控也十分重要[39]。和在植物中高度保守,但是在其他真核生物中却没有,说明这些基因介导的调控方式是植物中的一种特有的机制。
2.2.2 与组蛋白甲基转移酶相互作用
拟南芥的MMD1 (MALE MEIOCYTE DEATH 1)对于植物减数分裂过程十分重要[40]。其PHD结构可以识别H3K4me2/3,通过结合目标基因启动子区域的组蛋白标记,招募其他的调控因子到相同的位点,从而调控基因的表达[41]。最近研究发现,MMD1拥有另一个保守的MMD结构域,其与JMJ16 (组蛋白去甲基化酶,具有H3K4me3去甲基酶活性[80])的FYR-C结构域相互作用,从而拓展了JMJ16的H3K9me3去甲基酶活性,使目标基因启动子区域的抑制基因表达的组蛋白修饰减少,从而促进了基因表达,包括编码浓缩蛋白CAP-D3的基因表达。在这个过程中,PHD结构域通过结合H3K4me3,使JMJ16能够准确定位到基因的启动子区域[42]。
拟南芥中AL (ALFIN-LIKE)蛋白是植物中特有的一类转录因子,对于植物的生长发育,以及应对低温、干旱、高盐等非生物胁迫具有重要的调控作用[44,46]。AL蛋白家族包括AL1、AL2、AL3、AL4、AL5、AL6和AL7,除AL3以外都能通过PHD结构域与H3K4me3和H3K4me2结合来调控目标基因的表达[45]。在种子萌发时,AL蛋白通过与PRC1蛋白相互作用,招募PRC2 (一个保守的H3K27甲基转移酶[81])从而促进H3K27me3的积累,将种子发育相关基因的活跃转录的染色质状态转变为不活跃状态,对于种子萌发和早期的幼苗生长十分重要[43]。
植物的春化作用中所需的表观遗传学基因沉默过程,需要PRC2和2个PHD finger蛋白—VRN5和VIN3的参与。VIN3蛋白的PHD结构域与PRC2的相互作用被认为是PcG家族调控基因表达的保守机制[82],PHD结构域蛋白在增强PRC2活性上发挥了重要作用。高水平的H3K27me3能够稳定基因的沉默状态,PHD-PRC2复合体使基因的H3K27me3水平升高,从而使其保持沉默[82]。
类似的调控过程在果蝇()和哺乳动物中也存在。哺乳动物的PHF1蛋白拥有1个N端Tudor结构域和2个PHD结构域,是PRC2的组成部分。PHF1包含两个PHD finger结构域的区域,可以直接与PRC2的催化亚基EZH2 (甲基转移酶活性)相互作用[83]。在果蝇中,(的同源基因)和()(的同源基因)也可以互相结合[84],说明PHF1的PHD finger功能的保守性[85]。此外,研究发现在没有H3K36me3的情况下,PHF1可以增强PRC2的甲基转移酶活性,这可能是由于PHD finger结构域与其他组蛋白翻译后修饰(例如未修饰的H3或H3K4me3,与H3K27me3没有空间冲突)相互作用[86]。
2.3 与DNA甲基化相关
AtMBD9 (METHYL-CPG BINDING DOMAIN 9)蛋白拥有5个与染色质结构修饰调控基因表达相关的结构域,分别为1个MBD结构域、2个PHD结构域、1个Bromo结构域、1个FYRN结构域(N-terminal phenylalanine/tyrosine-rich domain)和1个FYR结构域(C-terminal phenylalanine/tyrosine-rich domain)[53]。AtMBD9是一个转录调控因子,通过DNA甲基化和组蛋白乙酰基化这两种表观遗传途径,分别间接和直接调控基因的表达,从而影响拟南芥的生长发育[52]。研究表明,AtMBD9突变体出现DNA高度甲基化,并且AtMBD9通过调控H4乙酰化来影响开花调节基因的表达,从而调节开花时间,其突变体表现出早开花和根系分支增多的表型[53]。MBD结构域可以结合甲基化的DNA,Bromo结构域可能发挥了催化组蛋白乙酰化反应的作用,因为该结构域在其他组蛋白乙酰基转移酶中经常存在[87],而PHD结构域的功能还没有具体的研究。
拟南芥/(/)基因家族有6个成员,是哺乳动物(,,)的直系同源基因群[55]。-编码的蛋白拥有1个PHD结构域、2个RING结构域和1个SRA(SET RING associated)结构域。研究表明,ORTH蛋白在体外拥有E3泛素连接酶活性,并且可以介导DNA甲基化。而第6个成员/(/)只有1个RING结构域和1个SRA结构域,其SRA结构域作用于结合甲基化的DNA[88]。而在人的UHRF1中,PHD和SRA结构域共同作用,使其结合到甲基化的H3K9上[89]。
正确的甲基化对于基因调控十分重要。MET1 (DNA METHYLTRANSFERASE 1)作用于DNA的CG甲基化。研究发现,ORTH/VIM可以通过识别MET1建立的CG甲基化在相应的位点积累,成为MET1介导的DNA甲基化途径中关键的组成部分[54]。
此外,ORTH2/VIM1的PHD结构域可以与NtSET (SU(VAR)3-9蛋白,烟草()中的一种甲基转移酶)相互作用[90]。因此,该PHD结构域是一个蛋白互作结构域,可能通过招募H3K9甲基转移酶,在DNA甲基化和H3K9组蛋白修饰之间建立联系。
在VIM突变体的靶基因上,活跃的染色质标记如H3K4me3和H3K9/K14ac明显增加,而抑制的染色质标记如H3K9me2和H3K27me3减少。此外,VIM的不足会引起异染色质染色中心(chromocenters) H3K9me2的明显减少[54]。因此,VIM蛋白通过调控激活和抑制的组蛋白修饰,使靶基因沉默,在协调组蛋白修饰和DNA甲基化状态的转变方面发挥了重要作用。
2.4 具有E3泛素连接酶活性
类泛素化(SUMOylation)在调控真核生物生长发育的多个方面发挥了重要的作用。Siz/PISA家族是一类具有SP-RING特征结构域的SUMO E3泛素连接酶,具有保守的特征结构域,包括SAP、PINIT和PHD finger结构域(只有植物成员具有PHD结构域,动物和酵母中没有)[91]。
拟南芥通过调控基因的表达,参与植物的生长发育以及应对干旱、低盐的胁迫的过程[57]。AtSIZ1包含了5个结构域,分别是SAP结构域、Siz/PIAS-RING结构域(作用于发挥SUMO E3连接酶活性)、PINIT结构域、SXS结构域(促进与SUMO的结合)以及PHD结构域。研究人员推测其PHD结构域与染色质重塑复合体有关,或者可能具有E3泛素连接酶活性[58]。
在水稻中OsSIZ1的SP-RING结构域主要发挥了类泛素化作用。OsSIZ1的PHD结构域可以识别H3R2me2和H3K4me3,研究指出PHD结构域可能和DNA结合结构域SAP一起协同作用,通过使一些效应因子(包括转录因子、组蛋白、HDAC组蛋白去乙酰化酶等)发生类泛素化,从而调控基因的表达[56]。
人类的KAP1也是一个含有PHD结构域且具有E3泛素连接酶活性的蛋白。KAP1的PHD结构域作为E3连接酶,介导了分子内的Bromo结构域的类泛素化,参与了基因沉默[92]。
2.5 是染色质重塑因子
依赖于ATP的染色质重塑因子主要包括:SWI/ SNF (SWITCH/SUCROSE NON-FERMENTING)、ISWI (IMITATION SWITCH)、INO80 (INOSITOL 80)和CHD (CHROMODOMAIN-HELICASE-DNA BINDING)。其中CHD是依赖于ATP的染色质重塑因子,在调控基因表达方面具有重要作用。CHD蛋白可以分为3个亚家族,分别为Chd1亚家族(也称为CHD1蛋白)、Mi-2亚家族(也称为CHD3蛋白)和CHD7亚家族[61]。
拟南芥中Mi-2亚家族有3个成员(PKL、CHR4和CHR7),水稻中也有3个(CHR207、CHR729和CHR703)。这些蛋白除了CHR7以外,都拥有1个PHD结构域和2个chromo结构域。拟南芥PKL和CHR4作用于DNA损伤应答[59],与水稻CHR729一样在调控植物生长和响应胁迫基因的表达中发挥重要的作用[60]。
PKL对一些基因的表达具有抑制作用,这些基因位点上有H3K27me3富集,推测该抑制作用与H3K27me3有关。PKL还可以激活一些基因的表达,它出现在例如ACT7 (ACTIN7)和UBQ10 (POLYUBIQUITIN 10)这类不需要依赖PKL表达的基因的启动子区域,并且促进普遍的染色质重塑过程[62]。CHR7没有PHD结构域,但是与PKL在激活基因表达上拥有重叠的功能,这说明PHD结构域可能对激活转录表达是不必要的。
2.6 与bHLH型的转录因子相互作用
(TDR interacting protein 3)是水稻中的一种雄性不育基因,包含PHD结构域,主要于花药发育期间在绒毡层和小孢子中表达[93]。TDR是一个bHLH型的转录因子,通过直接激活其靶基因的表达来调控绒毡层的发育和退化、脂质的代谢以及花粉的形成等过程[93]。研究发现,TIP3可以作为转录激活因子与TDR相互作用,从而影响与绒毡层程序性死亡和花粉壁发育相关基因的表达;并且酵母双杂交实验表明,TIP3的PHD结构域可以在在酵母中与3个bHLH型的转录因子TDR、EAT1和TIP2相互作用[93]。
因此,含有PHD结构域的蛋白还可能是通过不依赖于组蛋白修饰的方式,直接与转录因子结合,将转录因子招募到基因的启动子区域,从而发挥基因表达调控的功能。
3 结语与展望
在植物中,含有PHD结构域的蛋白作为转录调控因子参与了对植物生长发育具有重要作用的基因的转录调控。正如拟南芥中的PHD蛋白,参与种子萌发、根系发育、发芽、开花、减数分裂,以及减数分裂后的花粉发育等过程(图4)。
基因的转录调控与各种染色质修饰以及DNA甲基化密切相关。一般认为DNA甲基化与转录抑制相关,组蛋白乙酰化促进转录激活,而组蛋白甲基化则通过招募各种下游效应蛋白来发挥激活(H3K4me)或抑制(H3K9me)转录的修饰效应[94]。PHD结构域由于不同的氨基酸序列组成和结构特异性,对于不同的组蛋白乙酰化和甲基化修饰的结合也具有特定的偏好性。因此,对于PHD结构域结合特异性的精准研究,将会对其如何发挥转录调控功能具有十分重要的指示作用。目前已有研究在PHD结构域的结合特异性的基础上,开发相应的化学探针(chemical probes)以干扰其与组蛋白H3之间的相互作用[95],以及通过一些小分子抑制子(small molecule inhibitors)来竞争结合PHD结构域[96]。此外,开发具有E3泛素连接酶活性的PHD结构域配体以降解PHD蛋白也可能成为未来的一种研究思路[95]。
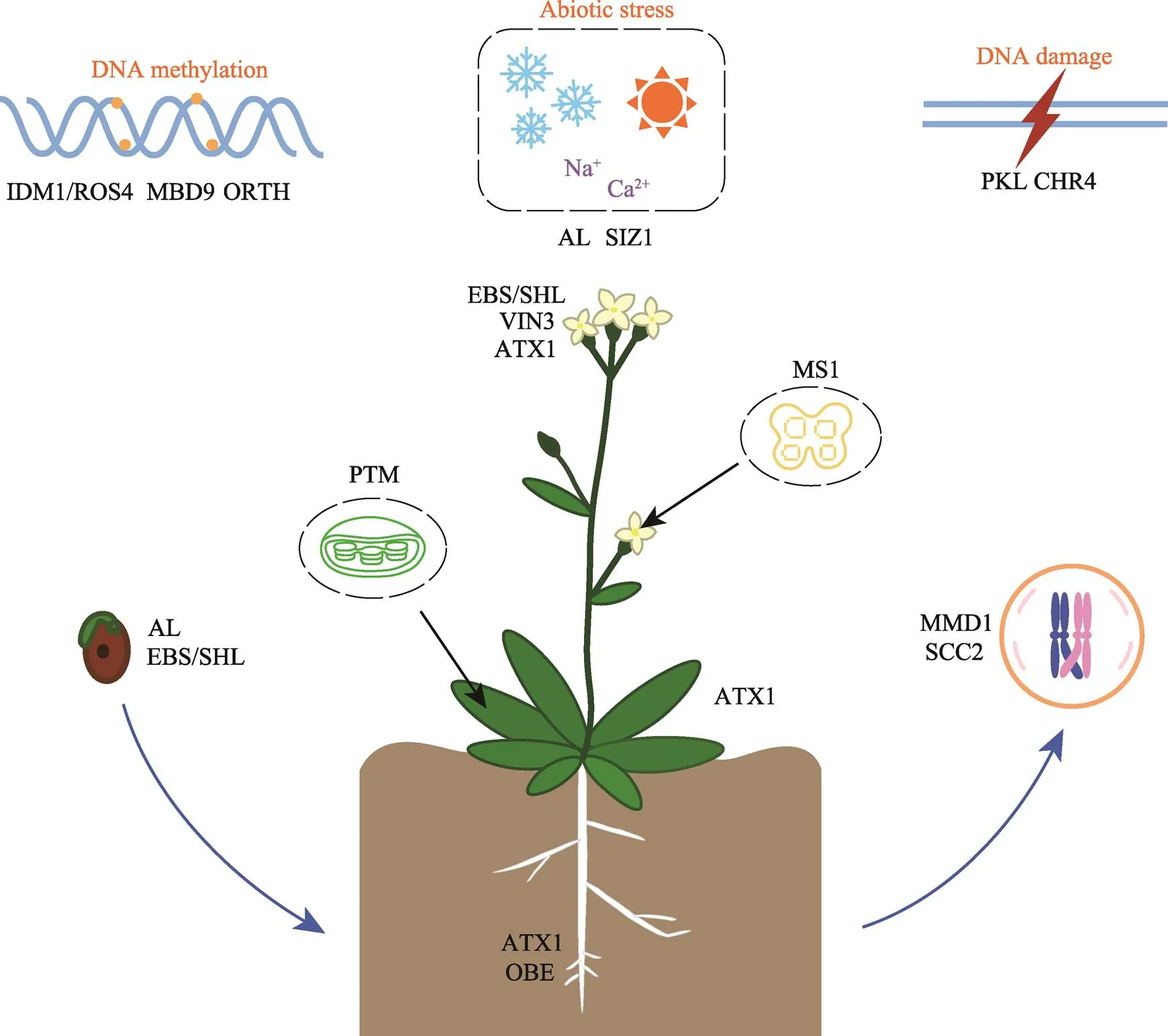
图4 拟南芥中PHD结构域蛋白的功能示意图
图中均为拟南芥的PHD结构域蛋白。可以看出,这些蛋白参与的生长发育相关的转录调控过程主要包括:种子萌发、根系发育、发芽、开花、减数分裂,以及减数分裂后的花粉发育等过程,并且还有蛋白与DNA甲基化相关。同时,对于植物的逆境胁迫和DNA损伤修复过程也发挥了重要的作用。
此外,含有PHD结构域的蛋白不仅在植物中保守存在,在人类基因组中也有约180多个含有PHD结构域的蛋白,这些蛋白同样具有转录调控、细胞周期和凋亡等方面的重要作用。对于植物PHD结构域蛋白的研究,可以更好地了解PHD结构域蛋白的作用机制,以运用于人类的肿瘤和疾病治疗。例如,在基因靶向治疗中,可以将外源性的PHD结构域蛋白导入人体,依靠PHD结构域对组蛋白密码的识别,从而靶向特定基因的转录激活和抑制,以达到抑制肿瘤基因表达等目的。
综上所述,在基因的转录调控过程中,含有PHD结构域的蛋白通过与不同的表观遗传效应蛋白(例如组蛋白甲基转移酶、组蛋白去甲基化酶、组蛋白乙酰基转移酶等)相互作用,以协调恰当的染色质修饰状态或者DNA甲基化状态,共同调控基因的转录表达。因此,研究含有PHD结构域的蛋白,对于植物生长发育和逆境胁迫等调控过程的解析具有十分重要的意义。
[1] Mouriz A, Lopez-Gonzalez L, Jarillo JA, Piñeiro M. PHDs govern plant development, 2015, 10(7): e993253.
[2] Zhao S, Zhang BC, Yang M, Zhu JS, Li HT. Systematic profiling of histone readers in, 2018, 22(4): 1090–1102.
[3] Pascual J, Martinez-Yamout M, Dyson HJ, Wright PE. Structure of the PHD zinc finger from human Williams- Beuren syndrome transcription factor, 2000, 304(5): 723–729.
[4] Kwan AHY, Gell DA, Verger A, Crossley M, Matthews JM, Mackay JP. Engineering a protein scaffold from a PHD finger, 2003, 11(7): 803–813.
[5] Li HT, Ilin S, Wang W, Duncan E M, Wysocka J, Allis CD, Patel DJ. Molecular basis for site-specific read-out of histone H3K4me3 by the BPTF PHD finger of NURF, 2006, 442(7098): 91–95.
[6] Champagne KS, Kutateladze TG. Structural insight into histone recognition by the ING PHD fingers, 2009, 10(5): 432–441.
[7] Xi QR, Wang ZX, Zaromytidou AI, Zhang XH, Chow- Tsang LF, Liu JX, Kim H, Barlas A, Manova-Todorova K, Kaartinen V, Studer L, Mark W, Patel DJ, Massague J. A poised chromatin platform for TGF-β access to master regulators, 2011, 147(7): 1511–1524.
[8] Iwase S, Xiang B, Ghosh S, Ren T, Lewis PW, Cochrane JC, Allis CD, Picketts DJ, Patel DJ, Li H, Shi Y. ATRX ADD domain links an atypical histone methylation recognition mechanism to human mental-retardation syndrome, 2011, 18(7): 769–776.
[9] Mansfield RE, Musselman CA, Kwan AH, Oliver SS, Garske AL, Davrazou F, Denu JM, Kutateladze TG, Mackay JP. Plant homeodomain (PHD) fingers of CHD4 are histone h3-binding modules with preference for unmodified H3K4 and methylated H3K9, 2011, 286(13): 11779–11791.
[10] Shi XB, Kachirskaia I, Walter KL, Kuo JA, Lake A, Davrazou F, Chan SM, Martin DGE, Fingerman IM, Briggs SD, Howe L, Utz PJ, Kutateladze TG, Lugovskoy AA, Bedford MT, Gozani O. Proteome-wide analysis inidentifies several PHD fingers as novel direct and selective binding modules of histone H3 methylated at either lysine 4 or lysine 36, 2007, 282(4): 2450–2455.
[11] Hu L, Li Z, Wang P, Lin Y, Xu Y. Crystal structure of PHD domain of UHRF1 and insights into recognition of unmodified histone H3 arginine residue 2, 2011, 21(9): 1374–1378.
[12] Wang CK, Shen J, Yang ZZ, Chen P, Zhao B, Hu W, Lan WX, Tong XT, Wu HM, Li GH, Cao CY. Structural basis for site-specific reading of unmodified R2 of histone H3 tail by UHRF1 PHD finger, 2011, 21(9): 1379–1382.
[13] Qiu Y, Liu L, Zhao C, Han CC, Li FD, Zhang JH, Wang Y, Li GH, Mei YD, Wu M, Wu JH, Shi YY. Combinatorial readout of unmodified H3R2 and acetylated H3K14 by the tandem PHD finger of MOZ reveals a regulatory mechanism for HOXA9 transcription, 2012, 26(12): 1376–1391.
[14] Zeng L, Zhang Q, Li Sd, Plotnikov AN, Walsh MJ, Zhou M. Mechanism and regulation of acetylated histone binding by the tandem PHD finger of DPF3b, 2010, 466(7303): 258–262.
[15] Wang ZX, Patel DJ. Combinatorial readout of dual histone modifications by paired chromatin-associated modules, 2011, 286(21): 18363–18368.
[16] Ruthenburg AJ, Li HT, Milne TA, Dewell S, Mcginty RK, Yuen M, Ueberheide B, Dou YL, Muir TW, Patel DJ, Allis CD. Recognition of a mononucleosomal histone modification pattern by BPTFmultivalent interactions, 2011, 145(5): 692–706.
[17] Sanchez R, Zhou MM. The PHD finger: A versatile epigenome reader, 2011, 36(7): 364–372.
[18] Gatchalian J, Kutateladze TG. PHD fingers as histone readers. Histone Recognition, Zhou M, Cham:Springer International Publishing, 2015, 27–47.
[19] Lan F, Collins RE, De Cegli R, Alpatov R, Horton JR, Shi XB, Gozani O, Cheng XD, Shi Y. Recognition of unmethylated histone H3 lysine 4 links BHC80 to LSD1-mediated gene repression, 2007, 448(7154): 718–722.
[20] Chakravarty S, Zeng L, Zhou M. Structure and Site-Specific recognition of histone h3 by the PHD finger of human autoimmune regulator, 2009, 17(5): 670–679.
[21] Argentaro A, Yang JC, Chapman L, Kowalczyk MS, Gibbons RJ, Higgs DR, Neuhaus D, Rhodes D. Structural consequences of disease-causing mutations in the ATRX-DNMT3-DNMT3L (ADD) domain of the chromatin-associated protein ATRX, 2007, 104(29): 11939–11944.
[22] Hung T, Binda O, Champagne KS, Kuo AJ, Johnson K, Chang HY, Simon MD, Kutateladze TG, Gozani O. ING4 mediates crosstalk between histone h3 k4 trimethylation and h3 acetylation to attenuate cellular transformation, 2009, 33(2): 248–256.
[23] Peña PV, Davrazou F, Shi XB, Walter KL, Verkhusha VV, Gozani O, Zhao R, Kutateladze TG. Molecular mechanism of histone H3K4me3 recognition by plant homeodomain of ING2, 2006, 442(7098): 100–103.
[24] Iwase S, Xiang B, Ghosh S, Ren T, Lewis PW, Cochrane JC, Allis CD, Picketts DJ, Patel DJ, Li H, Shi Y. ATRX ADD domain links an atypical histone methylation recognition mechanism to human mental-retardation syndrome, 2011, 18(7): 769–776.
[25] Li HT, Ilin S, Wang W, Duncan EM, Wysocka J, Allis CD, Patel DJ. Molecular basis for site-specific read-out of histone H3K4me3 by the BPTF PHD finger of NURF, 2006, 442(7098): 91–95.
[26] Li W, Zhao A, Tempel W, Loppnau P, Liu Y. Crystal structure of DPF3b in complex with an acetylated histone peptide, 2016, 195(3): 365–372.
[27] Liu L, Qin S, Zhang JH, Ji P, Shi YY, Wu JH. Solution structure of an atypical PHD finger in BRPF2 and its interaction with DNA, 2012, 180(1): 165–173.
[28] Qin S, Jin L, Zhang JH, Liu L, Ji P, Wu M, Wu JH, Shi YY. Recognition of unmodified histone h3 by the first PHD finger of Bromodomain-PHD finger protein 2 provides insights into the regulation of histone acetyltransferases monocytic leukemic zinc-finger protein (MOZ) and MOZ-related factor (MORF), 2011, 286(42): 36944–36955.
[29] Napsucialy-Mendivil S, Alvarez-Venegas R, Shishkova S, Dubrovsky JG.(ATX1) is required for cell production, patterning, and morphogenesis in root development, 2014, 65(22): 6373–6384.
[30] Ding Y, Ndamukong I, Xu ZS, Lapko H, Fromm M, Avramova Z. ATX1-generated H3K4me3 is required for efficient elongation of transcription, not initiation, at ATX1-regulated genes, 2012, 8(12): e1003111.
[31] Ding Y, Avramova Z, Fromm M. Two distinct roles of ARABIDOPSIS HOMOLOG of TRITHORAX1 (ATX1) at promoters and within transcribed regions of ATX1-Regulated genes, 2012, 23(1): 350–363.
[32] Saleh A, Alvarez-Venegas R, Yilmaz M, Le O, Hou GC, Sadder M, Al-Abdallat A, Xia YN, Lu GQ, Ladunga I, Avramova Z. The highly similarhomologs of trithorax ATX1 and ATX2 encode proteins with divergent biochemical functions, 2008, 20(3): 568–579.
[33] Liu Y, Zhang A, Yin H, Meng Q, Yu X, Huang S, Wang J, Ahmad R, Liu B, Xu ZY. Trithorax‐group proteins ARABIDOPSIS TRITHORAX4 (ATX4) and ATX5 function in abscisic acid and dehydration stress responses, 2018, 217(4): 1582–1597.
[34] Chen LQ, Luo JH, Cui ZH, Xue M, Wang L, Zhang XY, Pawlowski WP, He Y. ATX3, ATX4, and ATX5 encode putative H3K4 methyltransferases and are critical for plant development, 2017, 174(3): 1795–1806.
[35] Jacob Y, Feng S, Leblanc CA, Bernatavichute YV, Stroud H, Cokus S, Johnson LM, Pellegrini M, Jacobsen SE, Michaels SD. ATXR5 and ATXR6 are H3K27 monomethyltransferases required for chromatin structure and gene silencing, 2009, 16(7): 763–768.
[36] Li Q, Wang XK, Sun H, Zeng J, Cao ZD, Li Y, Qian WQ. Regulation of active DNA demethylation by a Methyl- CpG-Binding domain protein in arabidopsis thaliana, 2015, 11(5): e1005210.
[37] Qian WQ, Miki D, Lei MG, Zhu XH, Zhang HM, Liu YH, Li Y, Lang ZB, Wang J, Tang K, Liu RY, Zhu JK. Regulation of active DNA demethylation by an alpha- crystallin domain protein in, 2014, 55(3): 361–371.
[38] Qian S, Lv X, Scheid RN, Lu L, Yang Z, Chen W, Liu R, Boersma MD, Denu JM, Zhong X, Du J. Dual recognition of H3K4me3 and H3K27me3 by a plant histone reader SHL, 2018, 9(1): 2425.
[39] Narro-Diego L, López-González L, Jarillo JA, Piñeiro M. The PHD-containing protein EARLY BOLTING in SHORT DAYS regulates seed dormancy in, 2017, 40(10): 2393–2405.
[40] Yang XH, Makaroff CA, Ma H. The arabidopsis MALE MEIOCYTE DEATH1 gene encodes a PHD-Finger protein that is required for male meiosis, 2003, 15(6): 1281–1295.
[41] Wang J, Niu B, Huang J, Wang H, Yang X, Dong A, Makaroff C, Ma H, Wang Y. The PHD finger protein MMD1/DUET ensures the progression of male meiotic chromosome condensation and directly regulates the expression of the condensin gene CAP-D3, 2016, 28(8): 1894–1909.
[42] Wang J, Yu CY, Zhang SB, Ye JY, Dai H, Wang HK, Huang JY, Cao XF, Ma JB, Ma H, Wang YX. Cell-type- dependent histone demethylase specificity promotes meiotic chromosome condensation in, 2020, 6(7): 823–837.
[43] Molitor AM, Bu ZY, Yu Y, Shen WH. Arabidopsis AL PHD-PRC1 complexes promote seed germination through H3K4me3-to-H3K27me3 chromatin state switch in repression of seed developmental genes, 2014, 10(1): e1004091.
[44] Nulu NPC, Sundaravelpandian K, Yu SM, Schmidt W. ALFIN-LIKE 6 is involved in root hair elongation during phosphate deficiency in, 2013, 198(3): 709–720.
[45] Lee WY, Lee D, Chung W, Kwon CS. Arabidopsis ING and Alfin1-like protein families localize to the nucleus and bind to H3K4me3/2plant homeodomain fingers, 2009, 58(3): 511–524.
[46] Wei W, Zhang YQ, Tao JJ, Chen HW, Li QT, Zhang WK, Ma B, Lin Q, Zhang JS, Chen SY. The Alfin-like homeodomain finger protein AL5 suppresses multiple negative factors to confer abiotic stress tolerance in, 2015, 81(6): 871–883.
[47] Kim DH, Sung S. Coordination of the vernalization response through a VIN3 and FLC gene family regulatory network in arabidopsis, 2013, 25(2): 454–469.
[48] Kim DH, Sung S. The binding specificity of the PHD-Finger domain of VIN3 moderates vernalization response, 2017, 173(2): 1258–1268.
[49] Sung S, Amasino RM. Vernalization inis mediated by the PHD finger protein VIN3, 2004, 427(6970): 159–164.
[50] Bond DM, Wilson IW, Dennis ES, Pogson BJ, Jean Finnegan E. VERNALIZATION INSENSITIVE 3 (VIN3) is required for the response ofseedlings exposed to low oxygen conditions, 2009, 59(4): 576–587.
[51] Kim DH, Zografos BR, Sung S. Vernalization-Mediated VIN3 induction overcomes the LIKE-HETEROCHROMATIN PROTEIN1/POLYCOMB REPRESSION COMPLEX2- Mediated epigenetic repression, 2010, 154(2): 949–957.
[52] Yaish MWF, Peng M, Rothstein SJ. AtMBD9 modulatesdevelopment through the dual epigenetic pathways of DNA methylation and histone acetylation, 2009, 59(1): 123–135.
[53] Peng M, Cui Y, Bi YM, Rothstein SJ. AtMBD9: A protein with a methyl-CpG-binding domain regulates flowering time and shoot branching in, 2006, 46(2): 282–296.
[54] Kim J, Kim JH, Richards EJ, Chung KM, Woo HR. Arabidopsis VIM proteins regulate epigenetic silencing by modulating DNA methylation and histone modification in cooperation with MET1, 2014, 7(9): 1470– 1485.
[55] Kraft E, Bostick M, Jacobsen S E, Callis J. ORTH/VIM proteins that regulate DNA methylation are functional ubiquitin E3 ligases, 2008, 56(5): 704–715.
[56] Shindo H, Suzuki R, Tsuchiya W, Taichi M, Nishiuchi Y, Yamazaki T. PHD finger of the SUMO ligase Siz/PIAS family in rice reveals specific binding for methylated histone H3 at lysine 4 and arginine 2, 2012, 586(13): 1783–1789.
[57] Catala R, Ouyang J, Abreu IA, Hu YX, Seo H, Zhang XR, Chua NH. The arabidopsis e3 SUMO ligase SIZ1 regulates plant growth and drought responses, 2007, 19(9): 2952–2966.
[58] Miura K, Rus A, Sharkhuu A, Yokoi S, Karthikeyan AS, Raghothama KG, Baek D, Koo YD, Jin JB, Bressan RA, Yun DJ, Hasegawa PM. TheSUMO E3 ligase SIZ1 controls phosphate deficiency responses, 2005, 102(21): 7760–7765.
[59] Shaked H, Avivi-Ragolsky N, Levy AA. Involvement of the arabidopsis SWI2/SNF2 chromatin remodeling gene family in DNA damage response and recombination, 2006, 173(2): 985–994.
[60] Hu Y, Lai Y, Zhu D. Transcription regulation by CHD proteins to control plant development, 2014, 5: 223.
[61] Murawska M, Brehm A. CHD chromatin remodelers and the transcription cycle, 2011, 2(6): 244–253.
[62] Zhang H, Bishop B, Ringenberg W, Muir WM, Ogas J. The CHD3 remodeler PICKLE associates with genes enriched for trimethylation of histone h3 lysine 27, 2012, 159(1): 418–432.
[63] Thomas CL, Schmidt D, Bayer EM, Dreos R, Maule AJ. Arabidopsis plant homeodomain finger proteins operate downstream of auxin accumulation in specifying the vasculature and primary root meristem, 2009, 59(3): 426–436.
[64] Saiga S, Möller B, Watanabe-Taneda A, Abe M, Weijers D, Komeda Y. Control of embryonic meristem initiation inby PHD-finger protein complexes, 2012, 139(8): 1391–1398.
[65] Saiga S, Furumizu C, Yokoyama R, Kurata T, Sato S, Kato T, Tabata S, Suzuki M, Komeda Y. The Arabidopsis OBERON1 and OBERON2 genes encode plant homeodomain finger proteins and are required for apical meristem maintenance, 2008, 135(10): 1751–1759.
[66] Wang HK, Xu WY, Sun YJ, Lian QC, Wang C, Yu CY, He CP, Wang J, Ma H, Copenhaver G P, Wang Y. The cohesin loader SCC2 contains a PHD finger that is required for meiosis in land plants, 2020, 16(6): e1008849.
[67] de la Paz Sanchez M, Gutierrez C, Dean C. Arabidopsis ORC1 is a PHD-Containing H3K4me3 effector that regulates transcription, 2009, 106(6): 2065–2070.
[68] Li SS, Yang ZL, Du X, Liu R, Wilkinson AW, Gozani O, Jacobsen SE, Patel DJ, Du J. Structural basis for the unique multivalent readout of unmodified h3 tail by arabidopsis ORC1b BAH-PHD cassette, 2016, 24(3): 486–494.
[69] Ito T, Nagata N, Yoshiba Y, Ohme-Takagi M, Ma H, Shinozaki K. Arabidopsis MALE STERILITY1 encodes a PHD-Type transcription factor and regulates pollen and tapetum development, 2007, 19(11): 3549– 3562.
[70] Sun X, Feng P, Xu X, Guo H, Ma J, Chi W, Lin R, Lu C, Zhang L. A chloroplast envelope-bound PHD transcription factor mediates chloroplast signals to the nucleus, 2011, 2: 477.
[71] Alvarez-Venegas R, Sadder M, Hlavacka A, Baluska F, Xia YN, Lu GQ, Firsov A, Sarath G, Moriyama H, Dubrovsky JG, Avramova Z. The arabidopsis homolog of trithorax, ATX1, binds phosphatidylinositol 5-Phosphate, and the two regulate a common set of target genes, 2006, 103(15): 6049–6054.
[72] Jacob Y, Stroud H, Leblanc C, Feng SH, Zhuo LT, Caro E, Hassel C, Gutierrez C, Michaels SD, Jacobsen SE. Regulation of heterochromatic DNA replication by histone H3 lysine 27 methyltransferases, 2010, 466(7309): 987–991.
[73] He C, Liu N, Xie DY, Liu YH, Xiao YZ, Li FD. Structural basis for histone H3K4me3 recognition by the N-terminal domain of the PHD finger protein Spp1, 2019, 476(13): 1957–1973.
[74] Fortschegger K, Shiekhattar R. Plant homeodomain fingers form a helping hand for transcription, 2011, 6(1): 4–8.
[75] Qian WQ, Miki D, Zhang H, Liu YH, Zhang X, Tang K, Kan YC, La HG, Li XJ, Li SF, Zhu XH, Shi XB, Zhang KL, Pontes O, Chen XM, Liu RY, Gong ZZ, Zhu JK. A histone acetyltransferase regulates active DNA demethylation in, 2012, 336(6087): 1445–1448.
[76] Taverna SD, Ilin S, Rogers RS, Tanny JC, Lavender H, Li HT, Baker L, Boyle J, Blair LP, Chait BT, Patel DJ, Aitchison JD, Tackett AJ, Allis CD. Yng1 PHD finger binding to h3 trimethylated at k4 promotes NuA3 HAT activity at k14 of h3 and transcription at a subset of targeted ORFs, 2006, 24(5): 785–796.
[77] Müssig C, Kauschmann A, Clouse SD, Altmann T. ThePHD-finger protein SHL is required for proper development and fertility, 2000, 264(4): 363–370.
[78] Fornara F, de Montaigu A, Coupland G. SnapShot: Control of flowering in arabidopsis, 2010, 141(3): 550.
[79] López-González L, Mouriz A, Narro-Diego L, Bustos R, Martínez-Zapater JM, Jarillo JA, Piñeiro M. Chromatin- Dependent repression of thefloral integrator genes involves plant specific PHD-Containing proteins, 2014, 26(10): 3922–3938.
[80] Liu P, Zhang SB, Zhou B, Luo X, Zhou XF, Cai B, Jin YH, Niu D, Lin JX, Cao XF, Jin JB. The histone H3K4 demethylase JMJ16 represses leaf senescence in, 2019, 31(2): 430–443.
[81] Laugesen A, Højfeldt JW, Helin K. Molecular mechanisms directing PRC2 recruitment and H3K27 methylation, 2019, 74(1): 8–18.
[82] Lucia FD, Crevillen P, Alexandra MEJ, Greb T, Dean C. A PHD-Polycomb Repressive Complex 2 triggers the epigenetic silencing of FLC during vernalization, 2008, 105(44): 16831–16836.
[83] Sarma K, Margueron R, Ivanov A, Pirrotta V, Reinberg D. Ezh2 requires PHF1 to efficiently catalyze h3 lysine 27 trimethylation, 2008, 28(8): 2718–2731.
[84] Nekrasov M, Klymenko T, Fraterman S, Papp B, Oktaba K, Kocher T, Cohen A, Stunnenberg HG, Wilm M, Muller J. Pcl-PRC2 is needed to generate high levels of H3-K27 trimethylation at Polycomb target genes, 2007, 26(18): 4078–4088.
[85] O'Connell S, Wang L, Robert S, Jones CA, Saint R, Jones RS. Polycomblike PHD fingers mediate conserved interaction with enhancer of zeste protein, 2001, 276(46): 43065–43073.
[86] Musselman CA, Avvakumov N, Watanabe R, Abraham CG, Lalonde ME, Hong Z, Allen C, Roy S, Nuñez JK, Nickoloff J, Kulesza CA, Yasui A, Côté J, Kutateladze TG. Molecular basis for H3K36me3 recognition by the Tudor domain of PHF1, 2012, 19(12): 1266–1272.
[87] Hassan AH, Awad S, Prochasson P. The Swi2/Snf2 bromodomain is required for the displacement of SAGA and the octamer transfer of SAGA-acetylated nucleosomes, 2006, 281(26): 18126–18134.
[88] Johnson LM, Bostick M, Zhang XY, Kraft E, Henderson I, Callis J, Jacobsen SE. The SRA Methyl-Cytosine-Binding domain links DNA and histone methylation, 2007, 17(4): 379–384.
[89] Patnaik D, Esteve PO, Pradhan S. Targeting the SET and RING-associated (SRA) domain of ubiquitin-like, PHD and ring finger-containing 1 (UHRF1) for anti-cancer drug development, 2018, 9(40): 26243–26258.
[90] Liu SM, Yu Y, Ruan Y, Meyer D, Wolff M, Xu L, Wang N, Steinmetz A, Shen WH. Plant SET- and RING- associated domain proteins in heterochromatinization, 2007, 52(5): 914–926.
[91] Ouyang J, Gill G. SUMO engages multiple corepressors to regulate chromatin structure and transcription, 2009, 4(7): 440–444.
[92] Zeng L, Yap KL, Ivanov AV, Wang XQ, Mujtaba S, Plotnikova O, Rauscher Iii FJ, Zhou MM. Structural insights into human KAP1 PHD finger–bromodomain and its role in gene silencing, 2008, 15(6): 626–633.
[93] Yang ZF, Sun LP, Zhang PP, Zhang YX, Yu P, Liu L, Abbas A, Xiang XJ, Wu WX, Zhan XD, Cao LY, Cheng SH. TDR INTERACTING PROTEIN 3, encoding a PHD-finger transcription factor, regulates Ubisch bodies and pollen wall formation in rice, 2019, 99(5): 844–861.
[94] Martin C, Zhang Y. The diverse functions of histone lysine methylation, 2005, 6(11): 838–849.
[95] Amato A, Lucas X, Bortoluzzi A, Wright D, Ciulli A. Targeting ligandable pockets on plant homeodomain (PHD) zinc finger domains by a Fragment-Based approach, 2018, 13(4): 915–921.
[96] Wagner EK, Nath N, Flemming R, Feltenberger JB, Denu JM. Identification and characterization of small molecule inhibitors of a plant homeodomain finger, 2012, 51(41): 8293–8306.
Structural and functional characteristics of plant PHD domain-containing proteins
Tianyi Wang, Yingxiang Wang, Chenjiang You
,,,,
Plant homeodomain (PHD) is a class of transcription factor in the Zinc finger domain family. The most important function of which is to recognize various histone modifications, including histone methylation and acetylation, etc. They can also bind to DNA. Proteins with PHD domains, some of which possess histone modification enzyme activity, or can interact with histone modification enzymes, and some are associated with DNA methylation, with E3 ubiquitin ligase activity, or even can be chromatin remodeling factors. As transcriptional regulators, they play an important role in plant growth and development. In this review, we summarize the structural features and substrate binding specificity of PHD domains (including H3K4me3/0, H3K9me3, H3R2, H3K14ac) and DNA, the conservation of plant PHD domain in evolution, the molecular mechanism of known PHD domain-containing proteins in plants, providing a reference for further understanding of the involvement of these proteins during plant growth and development.
plant homeodomain (PHD); histone code; histone modification reader; transcriptional regulation; epigenetics
2020-12-01;
2021-02-09
国家自然科学基金项目(编号:31925005)资助[Supported by the National Natural Science Foundation of China (No. 31925005)]
王天一,在读博士研究生,研究方向:植物减数分裂。E-mail: 20110700106@fudan.edu.cn
尤辰江,博士,青年研究员,研究方向:植物表观遗传。E-mail: cjyou@fudan.edu.cn
10.16288/j.yczz.20-412
2021/3/11 17:17:54
URI: https://kns.cnki.net/kcms/detail/11.1913.R.20210310.1027.002.html
(责任编委: 宋任涛)

