基于开源平台的医学数据集成应用与研究
李越飞,李少云,冯虎翼,周凯欣,周乐明
(1.重庆市第五人民医院大数据实验室,重庆 400062;2.中国科学院大学生命科学学院,北京 100049;3.重庆市卫生健康统计信息中心大数据应用发展部,重庆 401120)
随着医院信息系统在医院信息化建设和现代化管理中的普及,海量增长的医疗数据已经成为了宝贵的科研资源。科学而有效的利用这些数据,对于医学研究和药物研发等都是极其重要的方法和手段。然而,绝大多数医院信息系统仅服务于医院的诊疗流程,存在系统数据存储和标准的不统一,质量控制不完善等问题。这使得医院所有的业务系统数据集几乎都无法达到“科研数据集”的标准[1]。基于医院临床数据构建医疗大数据集成平台,形成健康医疗大数据的生态体系,进一步发挥数据的资源优势,已成为越来越多大型研究型医院以及临床专家的共识。然而,健康高效的医疗大数据科研生态体系的形成,需要大量的资金投入与长时间持续的数据治理,这对于大部分中小型医院不太现实。与此同时,医疗科研需求越来越多。如果能快速有效的通过开源数据集成工具,从面向医疗流程设计的数据库中抽取数据并转换成科研数据集[1],将会使大部分中小型医院获益。数据集成的三个基本环节:抽取(extract)、转换(transform)、加载(load)简称ETL[2]。抽取是将数据从已有的数据源中提取出来,转换是对原始数据进行处理,加载是将数据写入目标数据库。开源技术已经成为整个互联网时代的支撑技术,其透明性、可控性、安全性及稳定性深受业界青睐。采用开源平台及技术来实现数据ETL,能够有效提升科研效率,节省科研经费,具有可观的应用价值。本文结合重庆市第五人民医院的医疗信息系统及中国科学院科技服务网络计划(STS)项目的数据需求为实例,总结运用开源平台及技术实现从医疗数据集到科研数据集的转换及清理,现报道如下。
1 系统设计及框架
1.1 系统现状及需求分析 以“适配特定人群院内医疗真实世界数据[3]”为例,需要提供以患者为导向的信息如下:①患者基本信息;②历次发药记录、药品目录;③门诊、住院医嘱、诊断情况;④病历、手术记录;⑤检查、检验项目结果。上述数据并非存储在单一的系统里,而是分别存在医院的患者体检系统(PEIS)、电子病历(EMR)和医院信息系统(HIS)中。这些系统由不同的服务商提供,其数据存储在不同的数据库中,如PEIS 的数据存储在SQL Server,EMR 的数据存储在Oracle,HIS 的数据存储在SybaseASE。从这些异构数据库提取所需数据的难点在于:①三个系统是医院的主要业务系统,数据量大,增量快,存储在异构数据库中;②数据关系分散,单一数据库查询后再与其它数据合并需要繁琐的关联操作;③异构数据库如果没有有效的数据集成处理,无法进行统计和数据分析;④缺乏患者主索引(EMPI)[3],PEIS 的体检数据与其它系统的患者信息没有共有的唯一标识符做关联。集成这三个异构数据库中的数据,并保证数据的持续增量,为科研数据需求提供优质数据,是本次的研究目标。
1.2 数据集成框架设计 鉴于以上难点和实例需求,本文医学数据集成作业框架,见图1,将实例需求拆分为具体的任务:①异构数据源同步;②任务执行与调度;③数据清洗与整理;④数据视图解释;⑤数据探索及结果计算,以上具体作业抽支撑了对数据的存储、归纳和分析[4]。

图1 数据集成的作业流程
2 实现方法
2.1 异构数据源同步 由于数据存储在异构数据库,数据的集成必须先以全量数据同步的方式进行异构数据整合。为满足这一需求,采用DataX 来实现数据的同步。DataX 是阿里巴巴开源的离线数据同步工具/平台,实现各种异构数据源之间高效的数据同步。与被广泛使用的数据集成工具PDI/Kettle 相比,DataX 设计的数据传输通道充当了缓冲层,不光有多种流量控制模式可以选择,还在限流的同时以各种策略支持任务的切分和并发。所以DataX 运行时对源头数据库产生的压力比较小,同时全量读取速度优于PDI/Kettle,并且能根据数据量进行智能的性能调优,更加适合做数据同步工作。而后者则是擅长做数据的清理和转化等复杂任务。因而本研究将DataX 作为数据同步的首选方案。
本次数据同步流程图见图2。DataX 作为框架中的中枢模块支撑,提供了数据的读/写功能及同步工作所要求的所有配置规范,如基于JDBC 驱动读写的数据源、指定字段等,都可以在JSON 格式的配置文件上定义。当一个同步工作开始时,以带参数运行的方式调用DataX 的Python 程序入口,读入事先规定的JSON 文件配置片段,按照预先制定的流程来执行同步任务,便可将异构数据源数据全量高效地同步到MySQL 数据库。以HIS 中的发药主表为例,当每条记录体积平均为633.8 kb 时,平均同步速度达每秒11596 条,20 min 可完成13683325 条数据的同步。与之相比,相同条件下,PDI/Kettle 的同步任务速度最快仅能达到每秒2540 条,还会对源头数据库造成压力。

图2 数据同步流程
2.2 任务执行与工作流调度 为了很好地组织起这样的复杂执行计划,使数据能稳定地进行同步到目标数据库并能自动进行全局增量,需要一个工作流调度系统来调度执行。目前开源的主流数据调度平台 有 Azkaban、Oozie、DolphinScheduler、Quartz、airflow、Zookeeper、XXL-Job 等,这些平台都有各自不可替代的特性。在本研究应用场景中,由LinkedIn开源的Azkaban 批量工作流任务调度器既有便捷的部署优势,又能在所构造各种工作流内以规定流程执行任务,这些特性能够快速、清晰地组织起一系列DataX 数据同步任务,达到数据增量更新的效果。本研究中的Azkaban 调度逻辑见图3,Azkaban 平台的两个执行器节点分别部署在不同位置的服务器上,调度平台使用一组加权择优算法,根据节点当前执行任务数、CPU、内存使用情况的综合分析,判断和选择资源最优的执行器,保证执行器的高度可用性。

图3 Azkaban 工作流调度逻辑
2.3 数据整理与清洗 医学数据挖掘工作中,事先常需要大量的观察研究以便对数据进行有效的整合和清洗。在本研究实例中,利用开源可视化数据集成工具PDI/Kettle 的独特优势,简化了海量数据的管理,增加了处理数据的种类和速度。通过PDI/Kettle 的可视化编程来进行字符串清理、字段清理、数据校验、排重等工作。如去除文本字段前后的空格、数字或标点符号;统一时间、日期、数字的格式;在字符串替换时引入正则表达式,结合字符切分等操作步骤,从各类诊疗文本中提取有用信息存入目标数据库的新字段里。
虽然PDI/Kettle 的字符串操作、字段选择、过滤行等可视化编程控件提供了行之有效的工具,但是并不能完成所有清理工作。对于更加复杂的情况,基于效率和可操作性的考虑,本次并没有使用PDI/Kettle 复杂的自定义模块,而是使用易用性更高的Python 的数据分析包Pandas 来完成分类、智能数据对齐和局部去重等综合整理。
本次对维度数据和度量数据两类数据进行清洗[5,6]。维度数据清洗包括患者个案信息在数据匹配过程中的必要清洗,以及对个体的行为在时间序列中的逻辑合理性判断中所发现的异常数据进行清洗。在此过程中,筛查和丢弃可信度低的数据。此外,清洗度量数据时,对可以进行数学计算的变量,如检查检验值、年龄等分布情况,进行了一定的数据分析并剔除离群值。
2.4 数据视图解释 数据清理和集成后,临床医疗服务数据,包含实验室测试结果、处方、临床资料、体检记录等组织在一起,形成以患者为中心的医学科研数据。这些数据可实现自动同步,也可以根据需求单独对指定的数据表调整同步频率和时间,获取当前最新数据。
本研究中,查询获得的科研数据集见图4,在目标数据源中集成了原本分散的医疗数据,成功匹配个案35,971 个,可以根据医学研究需求自由组织“特定人群院内真实世界医疗数据”,在一定程度上满足了医学科研对数据的需求及医院现阶段下临床科研队列的数据分析需要。
2.5 数据分析挖掘 对于“特定人群院内真实世界医疗数据”中的“特定人群”,可以指定为研究组、对照组人员名单,也可以指定为使用某些药物或者接受某种治疗的患者。如本研究中的特定人群被规定为“使用胰岛素、二甲双胍、格列吡嗪、利格列汀等药物的36 岁以下的患者”,组织他们的用药频次和血糖检验数据。具体步骤如下:①查询每种药发药记录,编写Python 脚本,调用collections 模块中Counter类的most_common()方法分别算出每个患者id 出现次数,得到给药频次,回写进数据库临时表;②以第一次发药时间减去出生日期的方法判断患者第一次取药时的年龄;③桥接基本信息和血糖检验值。这样可从患者数据视图出发进行探索,实现了取出复杂条件下的科研个案队列。
上述过程涉及到数据表中发药明细表体积最大(1.7G),含发药记录27,804,621 条,通过依靠患者基本信息进行数据集的初步筛选,可以有效缩短多表关联时的查询耗时。针对不同特征的查询结果,用Pandas_profiling 探索分析或者按需寻找疾病、用药、年龄等不同因素之间的关系,探索各变量间的相关性,检查是否存在冗余。这种检查的意义在于医学数据科学中常会研究各种因素的相互作用,如药品、治疗方法和疾病之间的作用,如果变量之间相关性强,统计建模时需剔除冗余的变量[5]。
3 关键技术问题及解决方案
3.1 SybaseASE 的采集接口适配 DataX 没有专用于SybaseASE 数据库的插件,使用通用的关系型数据源读取插件RDBMS_Reader 可实现DataX 从SybaseASE 数据库的读取。需要在其对应配置中注册SybaseASE 的JDBC 驱动支持,并且为读取插件使用驱动时根据数据源正确指定正确字符集,如CP936。

图4 实例数据串联图
3.2 开源工具的组件依赖冲突 相比商用软件,开源工具在文档说明和技术服务等多个方面都存在不足之处。以本项目使用的开源工具为例,Azkaban 官方并未提供安装包,只能通过源码编译。这需要依赖如node.js、ant 等诸多环境以及项目自动化构建工具Gradle 来编译Azkaban。使用Azkaban 工作流调度DataXJob 是实现数据自动增量更新的关键所在,但Azkaban 平台本身并不能直接运行DataX 的Python 任务。所以通过利用在LinuxShell 读写时间戳并运行datax.py,根据目标数据源情况选择不同的DataX 配置判断作全量或增量更新。在这个过程中,Azkaban 需要Python3 环境,而DataX 默认了Python2,因此将Azkaban、DataX 放置在不同的Docker 容器中,利用容器化部署解决Python 版本冲突问题。
3.3 确保数据匹配的精准度 PEIS 数据库的体检记录和其它两个数据库的患者记录之间没有共有的ID 关联,这给数据的关联查询带来了一定的难度。为保证查询结果的正确性,本研究按照EMPI 身份数据标准采用匹配专用字段,并选取多个用户特征字段所建立的全面的关联规则,来保证数据的匹配精准度。匹配策略见图5,身份证号、姓名、性别、和电话被选取为匹配策略的核心数据元素,而出生日期、家庭住址、工作单位等信息被选为辅助验证元素,根据匹配情况按需引入。这种策略的优势在于既不会丢失可能有用的个案数据,同时提高了数据匹配的效率。具体策略如下:①核心元素全部一致即可判定的个案为同一患者;②如果仅有一个核心元素不一致的情况,辅助验证元素将被引入,用来进一步判断个案是否匹配。当所有的辅助验证元素均能匹配时,该个案被认为是匹配的,反之则不匹配。单个核心元素的不一致有可能是在询问和录入时由于输入错误所造成的。③当不一致的核心元素超过一个,遵循严格匹配的原则,该个案不能达到匹配条件。
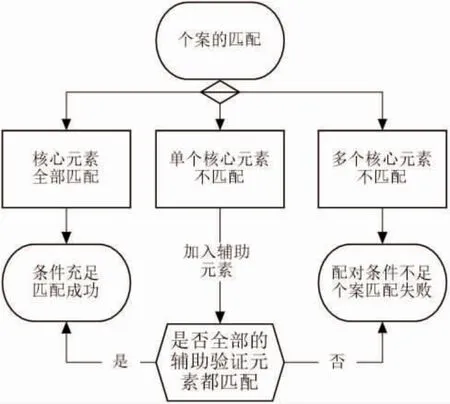
图5 患者个案匹配策略
4 应用现状
4.1 亟待解决的问题 本研究使用开源工具(平台)完成ETL,虽然开源技术更加灵活自由,能够实现更多的个性化定制需求,也省略了许多商业活动步骤,加快了知识转化的速度,但并不能完全代替商业软件,如可视化编程的PDI/Kettle 开发过程比商业可视化编程工具的Informatica 开发过程困难。除此之外,许多开源工具不提供可视化操作界面,活用开源工具通常需要一定的学习成本。数据清理过程中,异构数据的某些维度因难以共享一致性而无法重新设计,这正是由于缺乏能跨多个系统、设施的患者标识造成的。
4.2 改进对策和思路 对于开源工具/平台功能限制的问题,可以通过灵活运用多种开源工具,相互补充以提升数据挖掘的效率,通过大数据工程师的设计、开发、运维能力来解决各种开源框架在实际工作中相互配合时产生的报错和兼容性问题。当开源软件提供的功能不能满足需要时,可以通过二次开发来拓展开源软件的功能,定制适用自身工作场景的插件。而对于相对简单的场景和需求,可以在不改变开源软件的情况下,通过手动编写脚本的方式来解决。从而减少由于不必要的二次开发带来的工作量。根据需求和现有资源,购入适当规模的商业软件,和开源软件协同工作,会有助于提高开发和系统运行的效率。对于数据一致性问题,采取优先确保在数据转换和处理过程中医生诊断、检查、检验、日期保持一致性的策略,以提高数据匹配和清理的准确性和有效性。
5 总结
利用开源的ETL 工具可实现医疗业务数据到科研数据集的转化,在数据集成中进行数据的匹配和清理,为科研课题的提供进一步分析和研究提供了有效的查询平台。

